乳腺癌相关长链非编码RNA的生物信息学分析*
2018-12-03张莉张开炯吴立春杭永伦
张莉,张开炯,吴立春,杭永伦
610041成都,四川省肿瘤医院·研究所,四川省癌症防治中心,电子科技大学医学院 检验科(张莉、吴立春);646000四川 泸州,西南医科大学附属医院 检验科(张开炯、杭永伦)
乳腺癌是女性最常见的恶性程度较高的肿瘤[1]。根据2015年中国癌症统计资料显示,乳腺癌的发病率位居我国女性恶性肿瘤的首位,病死率位居第4,并呈现逐年上升的趋势[2]。随着手术、放化疗等治疗水平的提高,乳腺癌患者的总体生存率有了较大的改善,但乳腺癌起病隐匿且恶性程度高,当患者出现了典型症状时往往己处于肿瘤中晚期,此时治疗效果不甚理想,严重影响患者的预后[3-4]。因此,寻找便捷且高效能的乳腺癌诊断标志物迫在眉睫[5]。
近年来,研究者们对肿瘤发生发展机制的认识越来越深入,已经从蛋白编码基因逐渐拓展到非编码基因,如非编码RNA(non-coding RNA)[6-7]。长链非编码RNA(long non-coding RNA, lncRNA)是一类长度大于 200 个核苷酸的非编码 RNA,广泛地参与了包括细胞周期调控、细胞凋亡和分化及表观遗传等生物学进程[8]。LncRNA的异常表达与肿瘤的发生发展密切相关。研究发现,在多数肿瘤组织和细胞中存在若干特异性表达的lncRNAs,这些lncRNAs不仅能将肿瘤组织和正常组织区分开,而且其表达水平还与肿瘤分子亚型、分化程度、侵袭转移及预后程度密切相关[9-10]。近年来已有若干乳腺癌相关的lncRNA相继被报道[10-11],而如何寻找一种特异性更高灵敏度更强的lncRNA用作乳腺癌的特异性诊断,值得我们进一步探索和深入研究。
目前,如何找到方便有效的肿瘤筛查和预防分子标记一直是肿瘤研究的重点。基因芯片作为一种高效、大规模的生物信息学技术,可以检测和分析肿瘤组织与正常组织之间差异表达的基因,为筛选新的肿瘤标志物提供机会。为了从分子水平了解乳腺癌的发病机制,为乳腺癌的筛查和防治提供新的靶点,我们采用R语言Limma函数包,对美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)平台公共基因芯片数据平台(gene expression omnibus,GEO)数据库中的乳腺癌基因芯片表达数据集进行分析,鉴定乳腺癌中具有显著差异的lncRNA并重新注释,分析其表达以筛选乳腺癌相关的lncRNA,并采用定量PCR的方法进一步验证其表达情况,以评价基因芯片筛选结果的可靠性,从而为乳腺癌生物标志物的筛选提供策略和依据。
1 材料与方法
1.1 标本的收集
选取四川省肿瘤医院2015年5月~2016年9月初诊的乳腺癌女性患者50例,采集患者肿瘤组织标本及其对应癌旁组织标本(距肿瘤大于5cm)。 患者年龄27~69岁,中位年龄48岁。研究获得患者知情同意及医院伦理委员会批准。全部病例通过病理确诊,术前未进行放、化疗等任何治疗,病例资料齐全。
1.2 主要试剂与设备
1.2.1 主要试剂组织 RNA提取试剂盒(大连宝生物),PrimeScriptTM RT reagent Kit(大连宝生物),SYBR®Premix Ex TaqTM II扩增试剂盒(大连宝生物),PCR引物(上海生工)。
1.2.2 主要设备 C1000TM Thermal Cycle PCR仪(Bio-RAD,美国),ABI7500Fast荧光定量PCR仪器(ABI,美国),NanoDrop ND-2000紫外分光光度计(Thermo,美国)。
1.3 实验方法
1.3.1 乳腺癌中差异表达lncRNA分析 从NCBI的GEO公共基因芯片数据平台下载乳腺癌基因芯片数据GSE33447,包含8对乳腺癌组织及正常组织,采用Limma函数包对乳腺癌差异表达的lncRNA进行分析,以FC≥2,adj.P<0.05为筛选标准。为了提高数据的可靠性,采用DNA元件百科全书(ENCODE)对筛选出的差异表达的lncRNA进一步注释,只保留ENCODE中包含的lncRNA。
1.3.2 标本制备 乳腺癌患者新鲜组织标本及相邻的对照组织切下后迅速液氮冷冻,并储存于-80℃冰箱备用。
1.3.3 总RNA的提取及表达水平检测 按照TRIZOL 法提取组织中的总RNA(按照试剂说明书进行操作),用紫外分光光度法测定总RNA浓度和纯度,剔除不合格标本不参与后续实验,并将其逆转录为cDNA储存以备用。选取组织中稳定表达的β-actin作为内参。引物序列由NCBI引物在线工具进行设计,经BLAST比对后送上海生工合成。采用宝生物试剂盒(PrimeScriptTM RT reagent Kit)进行逆转录反应,并进一步采用SYBR®Premix Ex TaqTM II试剂盒,ABI7500实时定量PCR扩增仪扩增cDNA。引物序列见表1。
表1实时荧光定量PCR引物序列
Table 1. Primer sequences of real-time PCR
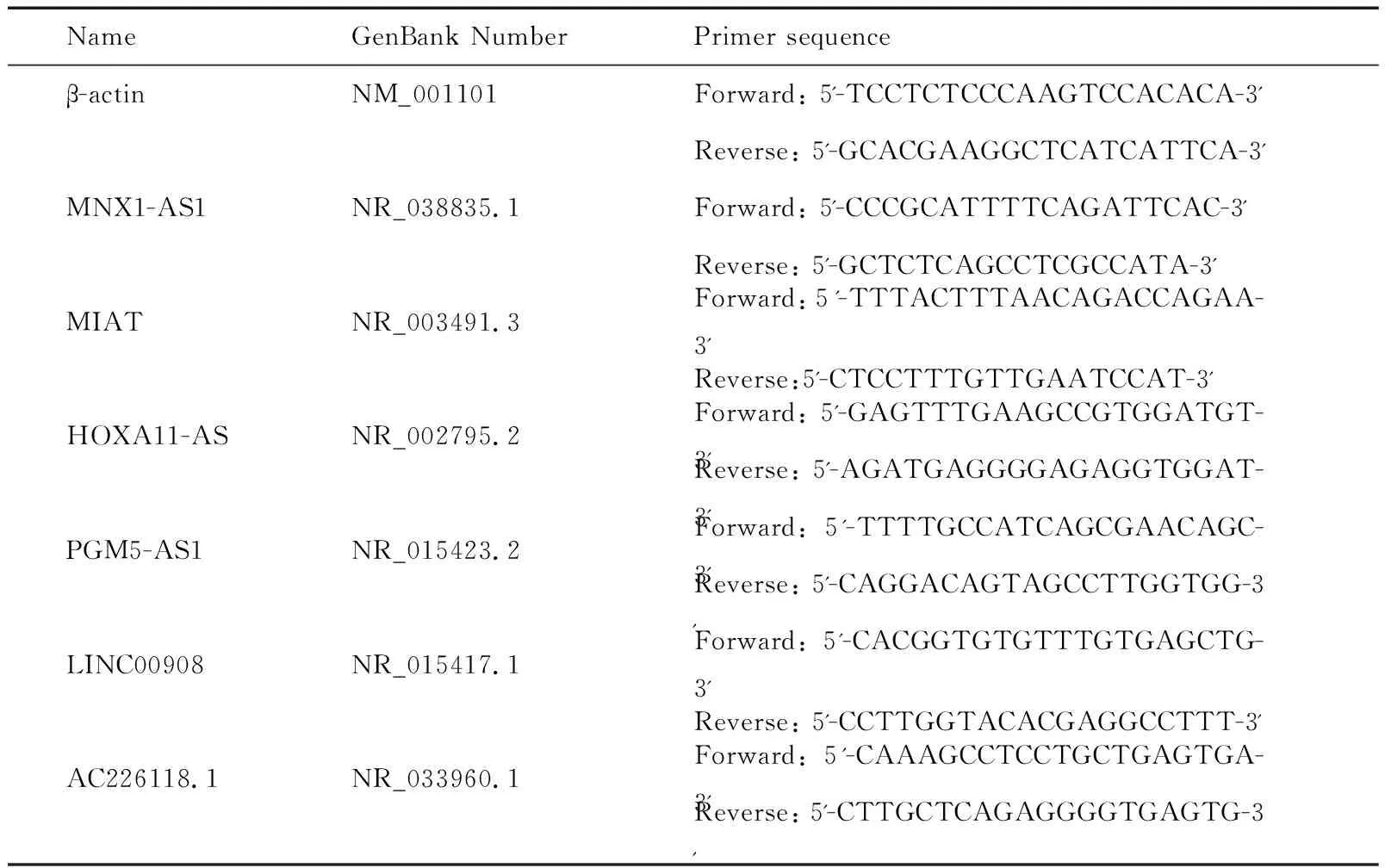
NameGenBank NumberPrimer sequenceβ-actinNM_001101Forward: 5'-TCCTCTCCCAAGTCCACACA-3'Reverse: 5'-GCACGAAGGCTCATCATTCA-3'MNX1-AS1NR_038835.1Forward: 5'-CCCGCATTTTCAGATTCAC-3'Reverse: 5'-GCTCTCAGCCTCGCCATA-3'MIATNR_003491.3Forward:5'-TTTACTTTAACAGACCAGAA-3'Reverse:5'-CTCCTTTGTTGAATCCAT-3'HOXA11-ASNR_002795.2Forward: 5'-GAGTTTGAAGCCGTGGATGT-3'Reverse: 5'-AGATGAGGGGAGAGGTGGAT-3'PGM5-AS1NR_015423.2Forward: 5'-TTTTGCCATCAGCGAACAGC-3'Reverse: 5'-CAGGACAGTAGCCTTGGTGG-3'LINC00908NR_015417.1Forward: 5'-CACGGTGTGTTTGTGAGCTG-3'Reverse: 5'-CCTTGGTACACGAGGCCTTT-3'AC226118.1NR_033960.1Forward: 5'-CAAAGCCTCCTGCTGAGTGA-3'Reverse: 5'-CTTGCTCAGAGGGGTGAGTG-3'
1.3.4 生物信息学分析 采用starbase 2.0(http://starbase.sysu.edu.cn/)进行lncRNAs靶基因预测;采用DAVID(https://david.ncifcrf.gov/)数据库对lncRNAs靶基因进行基因本体论(Gene Ontology,GO)和KEGG信号通路富集分析。
2 结 果
2.1 乳腺癌差异表达的lncRNA
采用Limma函数包对乳腺癌基因芯片数据集GSE33447进行差异分析,发现227个差异表达的lncRNA,其中135个lncRNA表达上调,92个lncRNA表达下调(FC≥2,adj.P<0.05)。差异表达的lncRNA如图1。由于芯片所检测出的lncRNA大部分信息和功能未知,我们进一步采用NONCODE对差异表达的lncRNA进行注释,为确保所筛选出的lncRNA结果可靠,剔除没有被ENCODE所收录的45个lncRNA,只保留ENCODE中包含的47个差异表达的lncRNA,在47个差异表达的lncRNA中,17个表达上调,30个表达下调,结果见表2。
表2 ENCODE中47个乳腺癌差异表达的lncRNAs
Table 2. Forty-seven differentially expressed lncRNAs of breast cancer included in GENCODE
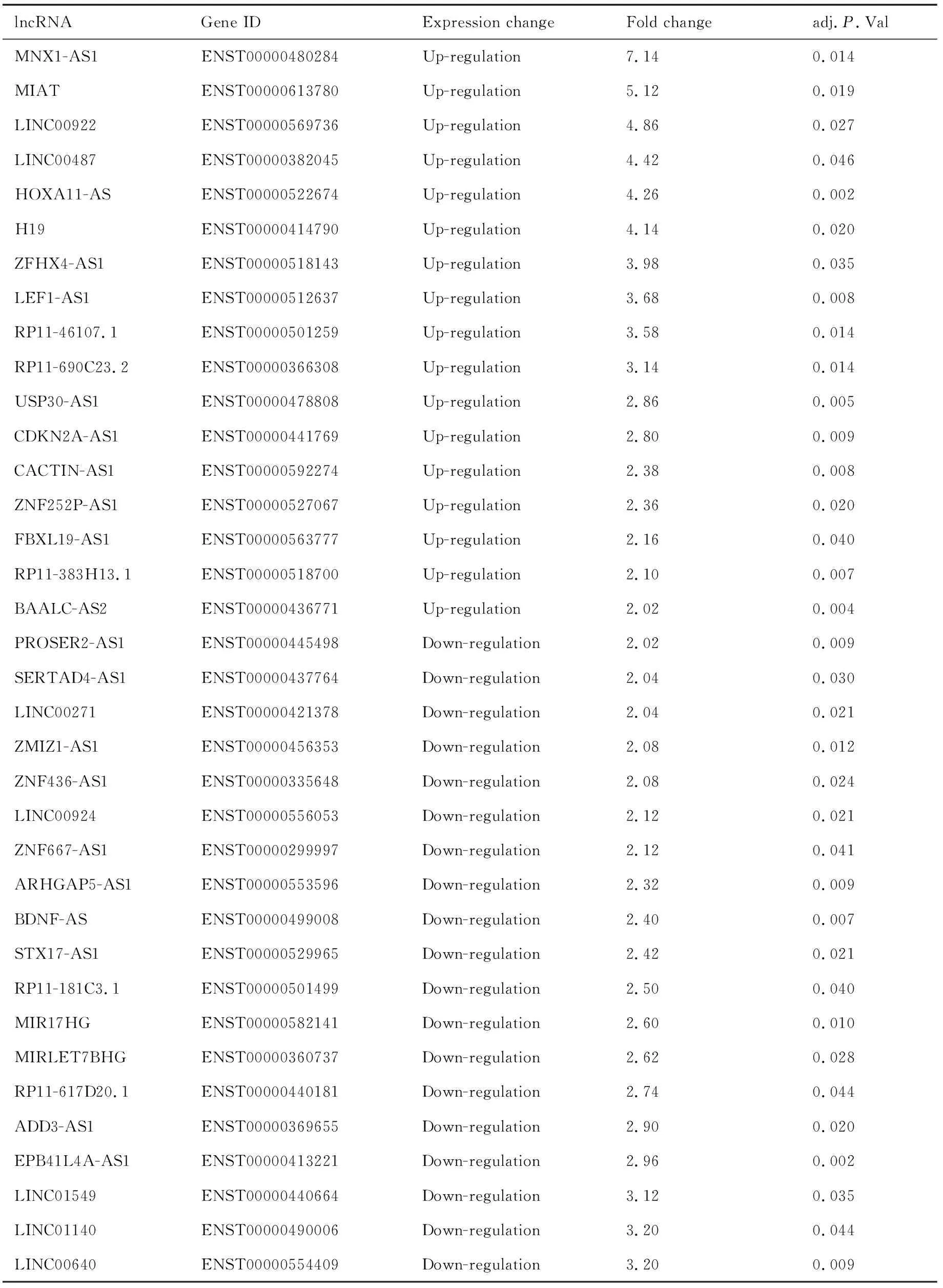
lncRNAGene IDExpression changeFold changeadj.P.ValMNX1-AS1ENST00000480284Up-regulation7.140.014MIATENST00000613780Up-regulation5.120.019LINC00922ENST00000569736Up-regulation4.860.027LINC00487ENST00000382045Up-regulation4.420.046HOXA11-ASENST00000522674Up-regulation4.260.002H19ENST00000414790Up-regulation4.140.020ZFHX4-AS1ENST00000518143Up-regulation3.980.035LEF1-AS1ENST00000512637Up-regulation3.680.008RP11-46107.1ENST00000501259Up-regulation3.580.014RP11-690C23.2ENST00000366308Up-regulation3.140.014USP30-AS1ENST00000478808Up-regulation2.860.005CDKN2A-AS1ENST00000441769Up-regulation2.800.009CACTIN-AS1ENST00000592274Up-regulation2.380.008ZNF252P-AS1ENST00000527067Up-regulation2.360.020FBXL19-AS1ENST00000563777Up-regulation2.160.040RP11-383H13.1ENST00000518700Up-regulation2.100.007BAALC-AS2ENST00000436771Up-regulation2.020.004PROSER2-AS1ENST00000445498Down-regulation2.020.009SERTAD4-AS1ENST00000437764Down-regulation2.040.030LINC00271ENST00000421378Down-regulation2.040.021ZMIZ1-AS1ENST00000456353Down-regulation2.080.012ZNF436-AS1ENST00000335648Down-regulation2.080.024LINC00924ENST00000556053Down-regulation2.120.021ZNF667-AS1ENST00000299997Down-regulation2.120.041ARHGAP5-AS1ENST00000553596Down-regulation2.320.009BDNF-ASENST00000499008Down-regulation2.400.007STX17-AS1ENST00000529965Down-regulation2.420.021RP11-181C3.1ENST00000501499Down-regulation2.500.040MIR17HGENST00000582141Down-regulation2.600.010MIRLET7BHGENST00000360737Down-regulation2.620.028RP11-617D20.1ENST00000440181Down-regulation2.740.044ADD3-AS1ENST00000369655Down-regulation2.900.020EPB41L4A-AS1ENST00000413221Down-regulation2.960.002LINC01549ENST00000440664Down-regulation3.120.035LINC01140ENST00000490006Down-regulation3.200.044LINC00640ENST00000554409Down-regulation3.200.009
(Table 2 continues on next page) (continued from previous page)
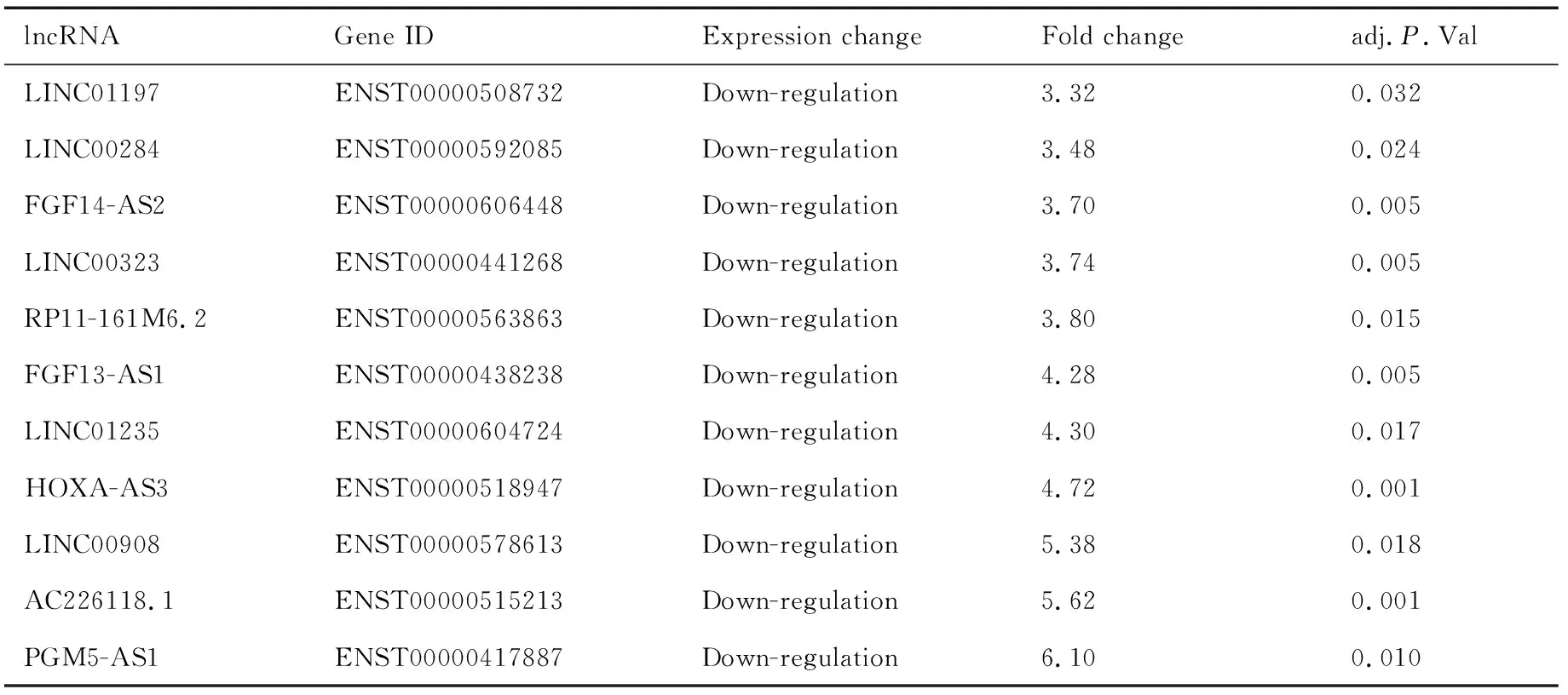
lncRNAGene IDExpression changeFold changeadj.P.ValLINC01197ENST00000508732Down-regulation3.320.032LINC00284ENST00000592085Down-regulation3.480.024FGF14-AS2ENST00000606448Down-regulation3.700.005LINC00323ENST00000441268Down-regulation3.740.005RP11-161M6.2ENST00000563863Down-regulation3.800.015FGF13-AS1ENST00000438238Down-regulation4.280.005LINC01235ENST00000604724Down-regulation4.300.017HOXA-AS3ENST00000518947Down-regulation4.720.001LINC00908ENST00000578613Down-regulation5.380.018AC226118.1ENST00000515213Down-regulation5.620.001PGM5-AS1ENST00000417887Down-regulation6.100.010
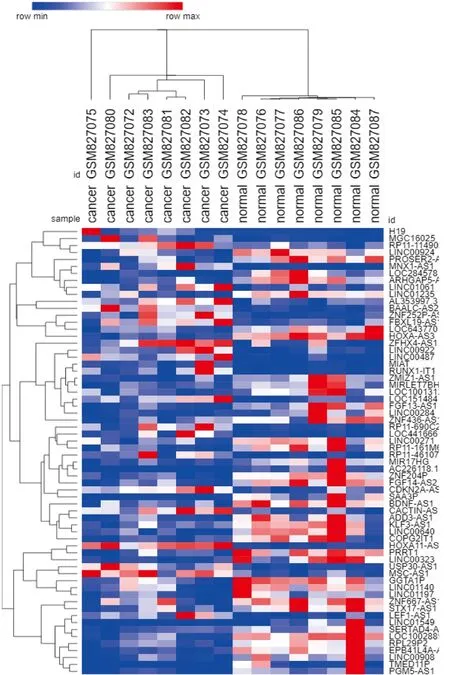
图1 8对乳腺肿瘤组织及其癌旁对照组织中差异表达lncRNA聚类分析图
Figure 1. A comparison between differentially expressed lnsRNA profiles in 8 breast cancer tissue and those in 8 normal breast tissue:Hierarchical clustering
2.2 lncRNAs参与的生物信息学功能分析
采用starbase 2.0对47个差异表达的lncRNAs靶基因进行预测,并采用DAVID数据库对差异表达lncRNAs靶基因进行GO和KEGG信号通路富集分析,以了解差异基因所具有的生物学意义以及所参与的重要生物学途径。结果如图2、图3所示。通过GO富集分析发现,lncRNAs广泛地参与了基因的转录及转录后调控、基因沉默及细胞代谢等生物学进程;通过KEGG信号通路富集分析发现,lncRNAs参与了乳腺癌、膀胱癌以及前列腺癌进程,并参与了PI3K-Akt、Ras、TNF以及p53等信号通路。

图2 GO富集分析乳腺癌中异常表达的lncRNAs
Figure 2. GO enrichment analysis of differentially expressed lncRNAs in breast cancer
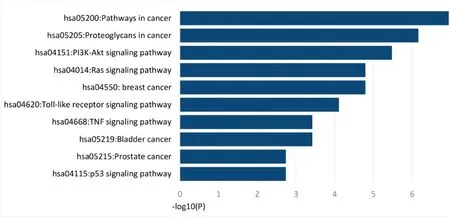
图3 KEGG信号通路分析乳腺癌中差异表达的lncRNAs
Figure 3. KEGG signaling pathway analysis of differentially expressed lncRNAs in breast cancer
2.3 采用qRT-PCR方法验证lncRNAs在乳腺癌组织中的表达水平
为了明确生物信息学筛选的可靠性,分别选取乳腺癌中3个高表达(MNX1-AS1,MIAT,HOXA11-AS)和3个低表达(PGM5-AS1,LINC00908,AC226118.1)的lncRNA,采用qRT-PCR的方法检测其在50例乳腺癌病理组织和50例癌旁组织中的表达水平,采用t检验对差异表达的lncRNA进行分析,发现MNX1-AS1、MIAT、HOXA11-AS在乳腺癌病理组中的表达水平明显高于其癌旁非肿瘤对照组,而PGM5-AS1、LINC00908和AC226118.1在乳腺癌病理组中的表达水平低于其癌旁非肿瘤对照组,差异有统计学意义(P均<0.05),结果如图4。
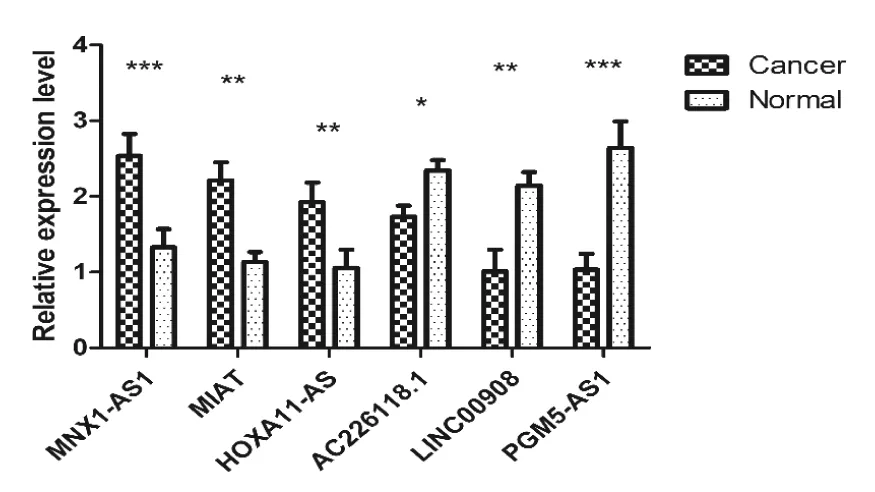
图4 LncRNAs在乳腺癌组织中相对于癌旁组织的表达水平
Figure 4. Comparision between relative expression levels of lncRNAs in breast cancer tissue and those in control tissue
*P<0.05,**P<0.01,***P<0.001
2.4 评价lncRNA潜在诊断价值
为了评价lncRNA作为诊断标志物的潜在价值,我们选取乳腺癌中高表达的MNX1-AS1作为研究对象,利用SigmaPlot 12.5进行ROC曲线绘制,发现MNX1-AS1曲线下面积(area under the curve,AUC)为0.921,当cut-off为1.425时,灵敏度和特异性分别为0.933和0.800。结果如图5。
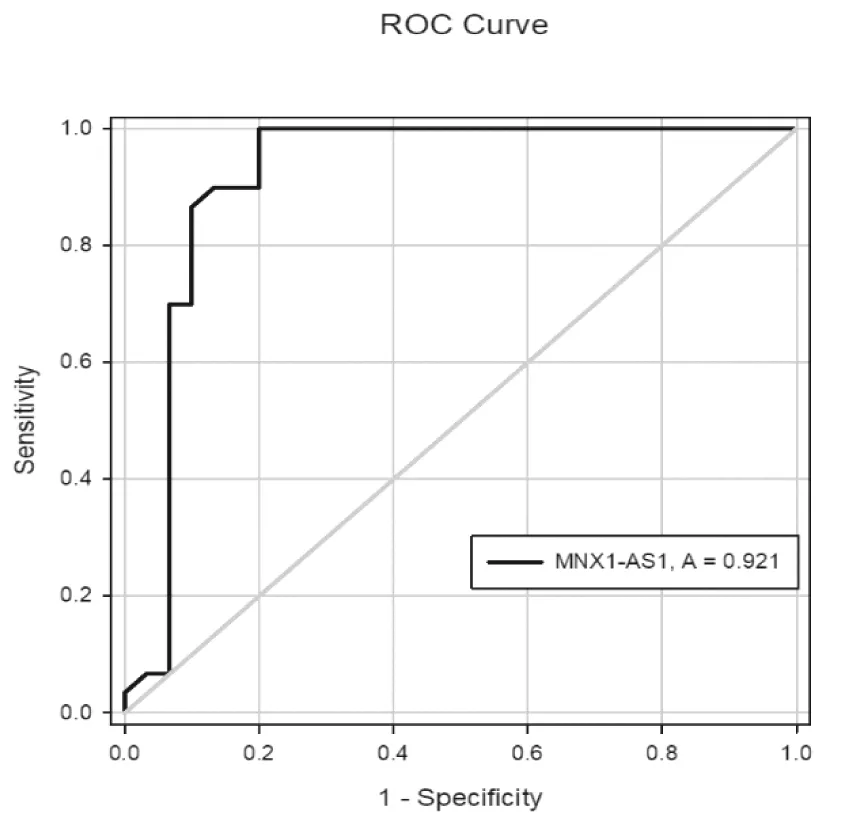
图5 MNX1-AS1对乳腺癌的潜在诊断价值
Figure 5. Potential diagnostic value of MNX1-AS1 for breast cancer
3 讨 论
LncRNA广泛地被报道参与人类许多疾病的发生,如肿瘤[12]、自身免疫性疾病[13]、炎症和感染[14]等。目前,lncRNA的检测方法主要为lncRNA特异芯片[15]、tiling芯片[16]以及RNA-seq测序[17]等,然而这些方法价格不菲,且需要特殊仪器检测。近年来,有研究发现通过对现有基因芯片进行再分析,能够挖掘到芯片原使用者不曾注意到的信息,从而发挥更大的效用[18-19]。通过筛选GEO数据库中含有lncRNA探针的芯片并重新注释,可以得到其匹配的lncRNA的名称,以分析lncRNA的表达情况。这种方法可以充分利用现有丰富的基因芯片数据资源,同时也节省了研究成本。
本研究选取乳腺癌相关基因芯片数据集GSE33447,利用基于R语言的Limma函数包对基因芯片原始数据进行分析,获得每个探针的倍数变化值。根据该值,筛选差异表达的探针以获得相应转录物的表达值。最后,通过实时荧光定量PCR进一步验证差异表达的lncRNA。通过分析基因芯片数据,共筛选出227个差异表达的lncRNAs,其中135个lncRNAs上调,92个lncRNAs下调。虽然这些lncRNA具有明确的名称,但大部分基因功能并不十分清楚。为了确保这些lncRNAs的可靠性,采用ENCODE对筛选出的差异表达的lncRNA进一步注释,只保留ENCODE中包含的47个差异表达的lncRNA,其中17个表达上调,30个表达下调。
近年来,已有少量本文中所筛选出的差异表达lncRNA在乳腺癌中的报道。例如MIAT、H19、HOXA11-AS等。Luan等[20]研究发现,MIAT在乳腺癌细胞系和乳腺癌组织中表达增高,且MIAT 表达水平与TNM分期和淋巴结转移相关。敲除MIAT可体外抑制乳腺癌细胞增殖、上皮-间质转化(epithelial-mesenchymal transition,EMT)以及侵袭转移进程,并促进乳腺癌细胞凋亡,且在体内抑制肿瘤生长。随后,Alipoor等[21]进一步研究发现,高表达的MIAT可作为乳腺癌诊断和治疗的新型肿瘤标志物。Vennin等[22]发现,H19在乳腺癌中表达增高,高表达的H19可通过H19/miR-675轴调控肿瘤细胞的增殖、凋亡、迁移以及肿瘤的远端转移。Zhou等[23]研究证实,H19可通过差异海绵吸附的方式竞争结合miR-200b/c和let-7b,从而调控乳腺癌细胞EMT和间质-上皮转化(MET)及侵袭转移进程,抑制H19将降低乳腺癌的远端转移。目前,已有较多HOXA11-AS在多种肿瘤中异常高表达的报道。例如非小细胞肺癌[24]、结直肠癌[25]、胃癌[26]及宫颈癌[27]等,其表达水平与肿瘤细胞的侵袭和转移显著相关。最近,Li等[28]采用qRT-PCR的方法对50例乳腺癌组织及其癌旁组织中的HOXA11-AS进行检测,发现乳腺癌组织中的HOXA11-AS明显高于癌旁对照组织。敲除HOXA11-AS可在体内体外抑制EMT相关分子标志物(E-cadherin, N-cadherin, Vimentin),从而抑制EMT进程,进而影响肿瘤细胞的侵袭转移[28]。通过上述文献报道,初步证实了采用生物信息学方法筛选差异表达目的基因的可靠性。
为了进一步确认生物信息学筛选基因芯片结果的可靠性,我们选取乳腺癌中暂无报道的MNX1-AS1、PGM5-AS1,LINC00908和AC226118.1,以及已经在乳腺癌中报道的MIAT,HOXA11-AS,采用qRT-PCR的方法验证这些lncRNA在乳腺癌中的表达水平。结果显示,乳腺癌组织中MNX1-AS1、MIAT、HOXA11-AS表达增加;PGM5-AS1,LINC00908和AC226118.1表达降低,差异具有统计学意义。为了评价lncRNA作为诊断标志物的潜在价值,我们对乳腺癌中高表达的MNX1-AS1绘制ROC曲线,发现AUC为0.921,当cut-off为1.425时,灵敏度和特异性分别为0.933和0.800。由此可见,通过生物信息学方法筛选肿瘤相关lncRNA,在解决研究的盲目性以及节约时间和成本上起到一定的作用,也可为lncRNA与乳腺癌之间进一步研究提供理论依据。
综上所述,通过生物信息学方法可为筛选肿瘤相关lncRNA提供一个方便、快捷的途径,为寻找肿瘤新型标志物提供了新的思路,为后续实验奠定了基础。在后续研究中,我们将进一步扩大样本,分析lncRNA与乳腺癌病理类型、TNM分期、淋巴结转移、雌激素以及孕激素水平等相关性,并进行细胞功能实验明确lncRNA所具有的生物学功能和参与的信号通路,为lncRNA作为乳腺癌新型生物标志物以及临床转化提供依据。
作者声明:本文第一作者对于研究和撰写的论文出现的不端行为承担相应责任;
利益冲突:本文全部作者均认同文章无相关利益冲突;
学术不端:本文在初审、返修及出版前均通过中国知网(CNKI)科技期刊学术不端文献检测系统学术不端检测;
同行评议:经同行专家双盲外审,达到刊发要求。
