栽培绿豆V1128抗豆象基因定位
2018-11-29刘长友苏秋竹范保杰曹志敏张志肖程须珍
刘长友 苏秋竹 范保杰 曹志敏 张志肖 武 晶 程须珍 田 静,*
栽培绿豆V1128抗豆象基因定位
刘长友1苏秋竹1范保杰1曹志敏1张志肖1武 晶2程须珍2田 静1,*
1河北省农林科学院粮油作物研究所/ 河北省作物遗传育种实验室, 河北石家庄 050031;2中国农业科学院作物科学研究所, 北京 100081
对抗豆象资源中蕴藏的抗豆象基因进行定位, 是对其充分利用的前提和基础。本研究通过对抗豆象栽培绿豆V1128和感豆象栽培绿豆冀绿7号杂交形成的F2分离群体进行抗豆象鉴定, 分析V1128抗豆象遗传规律; 并利用混合群体分离分析法(BSA法)筛选抗感池间的多态性标记, 进而利用QTL IciMapping 4.0对V1128抗豆象基因进行染色体定位分析。结果表明, V1128对绿豆象的抗性由具有主效作用的显性单基因控制, 暂命名其为“”。在将抗豆象性状作为质量性状的条件下, 按照显性单基因的定位方法, 将抗豆象基因定位在绿豆染色体5上, 位于标记DMB158和VRBR-SSR033 (标记VRID5、VRBR-SSR032与VRBR-SSR033的连锁群位置相同)之间, 两侧遗传距离分别为4.4 cM和5.8 cM, 所在物理区间约288 kb。将抗豆象性状作为数量性状, 采用完备区间作图法(ICIM)对种子被害率进行QTL定位, 同样在标记DMB158和VRBR-SSR033之间检测到1个主效QTL, 其LOD值为38.04, 可以解释表型变异(PVE)的71.64%, 来自父本V1128的等位基因具有明显减少种子被害率的效应。该研究结果可以为绿豆抗豆象分子标记辅助育种及抗豆象基因的精细定位和克隆提供有用信息。
绿豆; 抗豆象; V1128;; QTL
绿豆()作为一种医食同源的作物, 在健康饮食和膳食结构调整中发挥着越来越重要的作用[1]。近年来, 我国绿豆品种的产量和品质都有大幅提高[2]。然而, 绿豆储藏过程中的豆象危害却一直是困扰农民和商家的主要难题。
豆象是危害绿豆等食用豆类作物的严重仓储害虫。危害绿豆的豆象种类主要有绿豆象()、四纹豆象()、鹰嘴豆象()、巴西豆象()和灰豆象()等, 其中在我国存在的主要是绿豆象和四纹豆象, 且绿豆象对生产的危害最严重[3-4], 若处置不及时, 可在3~6个月内造成整仓绿豆全部受损[5-6]。生产上防治豆象危害一般采用磷化铝熏蒸法, 这种处理方法存在农药残留超标和造成环境污染的隐患, 不符合安全生产的目标。培育抗豆象绿豆品种, 利用作物本身的抗性是一种最经济有效的防治豆象危害的手段。然而, 以杂交选育为主要手段的常规育种途径存在选择周期长、抗豆象鉴定工作繁琐等诸多问题。对抗豆象基因进行定位, 开拓抗豆象育种方法, 利用分子手段辅助抗豆象育种, 成为当前十分紧迫的研究课题。
到目前为止, 筛选鉴定出的抗豆象绿豆种质资源主要有TC1966、ACC41、V2802、V2709、V1128和V2817 6份[7-10]。其中TC1966和ACC41是2份野生绿豆资源。研究表明, TC1966的抗豆象基因和ACC41的抗豆象基因均为单显性主基因遗传[8,11-12], 但它们的豆象抗性还可能存在其他微效基因的修饰作用[13]。Talekar等[9]和Somta等[14]先后发现栽培绿豆V2802和V2709高抗绿豆象和四纹豆象。Somta等[10]发现另外2份栽培绿豆V1128和V2817对绿豆象和四纹豆象具有完全抗性。利用不同类型的分子标记和作图群体, TC1966的基因和ACC41的基因已经被定位在不同的连锁标记之间[12,15-18]。孙蕾等[19]以V2709为研究材料, 将其抗豆象基因定位在1个RAPD和1个STS标记之间。Chotechung等[20]和Kaewwongwal等[21]分别将V2802和V2709的抗豆象基因定位在绿豆第5染色体上, 并预测可能是V2802的抗豆象候选基因, 而和可能是V2709的抗豆象候选基因。
在抗豆象育种中, 应用抗豆象野生种容易引入一些不利性状, 如蔓生、炸荚等。加强对栽培抗豆象绿豆资源的研究利用可以有效避免这些问题。V1128是对绿豆象和四纹豆象均具有完全抗性的栽培绿豆资源, 在人工种子试验中表现出比V2817更强的抗性[10]。因此, V1128在绿豆抗豆象育种中具有更大的潜力。然而, 到目前为止, V1128抗豆象基因的遗传定位研究尚无人开展。对其进行抗豆象基因定位有利于补充对目前尚不明晰的绿豆抗豆象基因的认识, 并加强其在抗豆象育种中的应用。因此, 本研究利用感豆象亲本冀绿7号和抗豆象亲本V1128杂交组配获得的F2群体对V1128抗豆象基因进行遗传定位分析, 为V1128及其他绿豆抗豆象资源抗性基因来源及基因克隆提供参考。
1 材料与方法
1.1 试验材料
抗豆象材料V1128由泰国农业大学Peerasak Srinnives教授惠赠, 该材料籽粒无光泽; 感豆象材料为河北省农林科学院粮油作物研究所育成品种冀绿7号, 该材料籽粒有光泽。以冀绿7号作母本, V1128作父本, 杂交获得F1。籽粒无光泽相对于籽粒有光泽为显性, 因此可以从F1植株所收获籽粒判断是否为真杂交种。选择来源于同一F1植株的种子繁殖F2分离群体, 共获得158个单株, 用于抗豆象遗传分析和构建遗传连锁图谱。
1.2 DNA提取
从每个F2单株取幼嫩的三出复叶, 立即投入液氮, –80°C保存, 以备提取DNA。利用植物基因组提取试剂盒(TIANGEN, 北京)提取DNA。使用1%琼脂糖凝胶检测DNA质量。利用超微量核酸蛋白检测仪(Nanodrop ND-2000, 美国)测定DNA浓度, 并稀释到10 ng μL–1。
1.3 抗豆象鉴定及遗传分析
从每个鉴定单株随机选取90粒健康种子, 分成3个重复, 每个重复30粒, 分别放入直径5 cm、高1.8 cm的圆形小塑料盒中。同时, 分别放入抗豆象亲本V1128和感豆象亲本冀绿7号作为对照。将小塑料盒并排放入大塑料箱内(66 cm×40 cm×15 cm), 每个大塑料箱中放入刚羽化1~3 d的成虫作为虫源(平均每份被鉴定材料3~8对), 至每粒种子着卵量3~5粒时, 除去成虫; 养虫室温度(29±2)℃, 湿度60%~70%。待感虫亲本完全被蛀食后(约40~60 d), 调查每份材料的受害粒数。3个重复取平均值用于后续数据分析。
参照Young等[15]和Kaewwongwal等[21]的豆象抗性分级方法, 种子被害率0~80%视为抗, 其中0~20%视为纯合抗性, 21%~80%视为杂合抗性; 种子被害率81%~100%视为纯合感豆象。利用Microsoft Excel 2010进行卡方测验, 检测F2群体的抗感分离比是否符合3 (抗)∶1 (感)的遗传分离规律, 利用SPSS 16.0统计分析软件计算F2群体种子被害率的频率分布。
1.4 DNA标记分析
分别选择10个完全抗豆象和10个完全感豆象F2单株的DNA混合组成抗感池。利用两亲本和抗感池进行多态性分子标记筛选, 选择在两亲本间表现多态且抗池带型与V1128相同, 感池带型与冀绿7号相同的标记用于后续F2群体的基因型分析。
PCR扩增及电泳分析参照Liu等[22]的方法。在亲本和抗感池间共计筛选了3767个不同类型的分子标记, 包括SSR、EST-SSR、STS和Indel标记。最终获得在两亲本间表现多态且抗池带型与V1128相同, 感池带型与冀绿7号相同的分子标记31个, 其中VRID1和VRID5为InDel标记, 其余均为SSR标记, 多态性标记信息见表1。
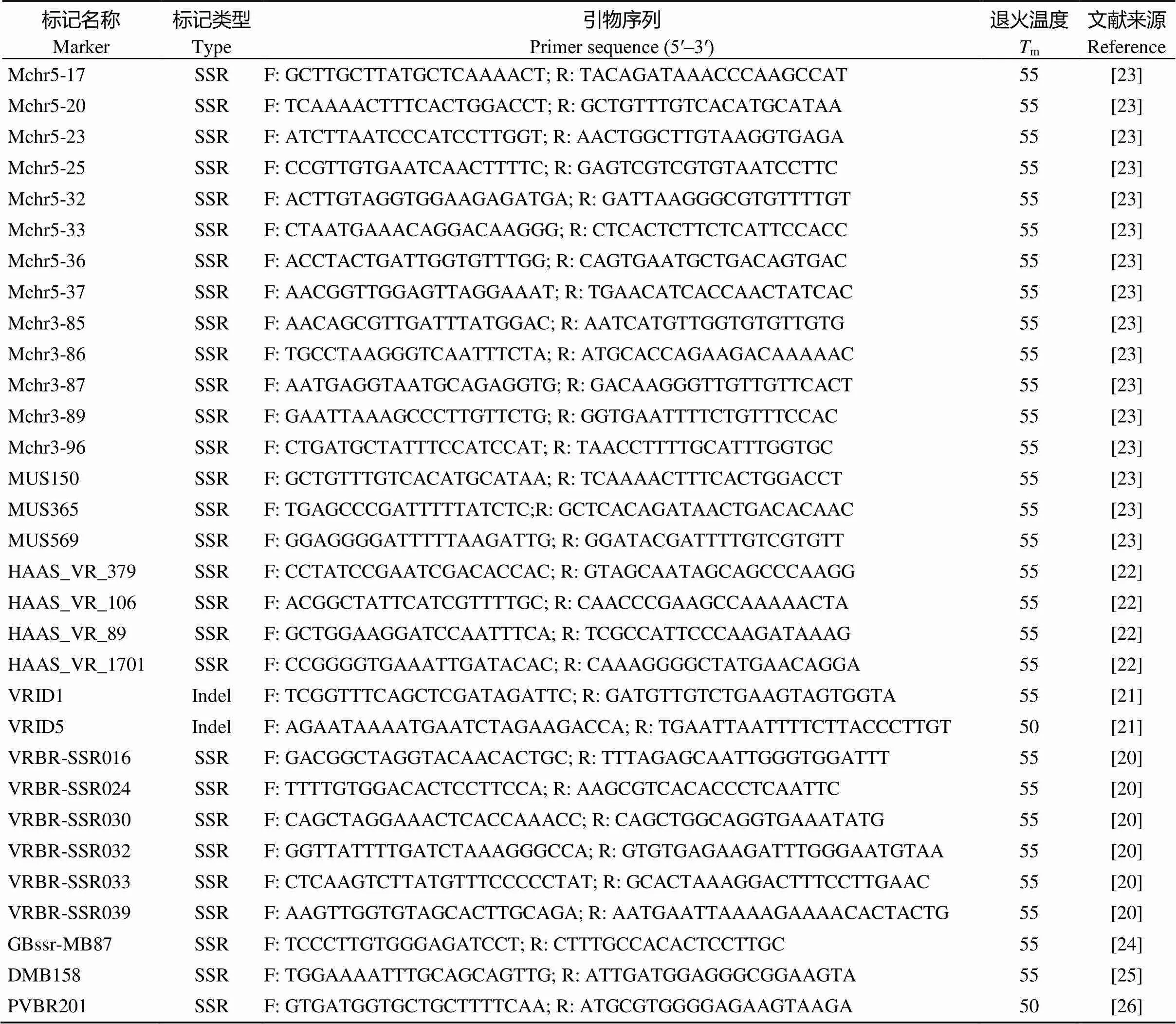
表1 多态性标记信息
1.5 抗豆象基因定位
利用QTL IciMapping 4.0构建连锁图谱[27]。在LOD=5.0时进行标记分组, 利用Kosambi作图方法计算相邻标记间的遗传距离[28]。采用两种方法定位抗豆象基因, 第一种, 将抗豆象性状作为质量性状, 根据上述分级方法分类, 纯合抗豆象记作“A”, 纯合感豆象记作“B”, 杂合抗豆象记作“H”, 由此, 将抗豆象基因“”作为一个表型标记构建到连锁图中, 对其进行连锁定位; 第二种, 将抗豆象性状作为数量性状, 以F2群体的种子被害率定位QTL。QTL作图方法为完备区间作图法(ICIM)[29], QTL的显著性LOD阈值在=0.01时作10 000次置换试验(Permutation test)确定, 其他参数设置采用QTL IciMapping 4.0软件的默认参数。
2 结果与分析
2.1 亲本及F2群体的豆象抗性鉴定与遗传分析
经抗豆象鉴定, 亲本V1128表现为完全抗豆象(种子被害率=0), 而亲本冀绿7号表现为完全感豆象(种子被害率=100%)。F2群体的抗性频率分布趋向于二项分布(图1), 抗感分离比为117 (抗)∶41 (感), 卡方测验符合3 (抗)∶1 (感)的分离规律, χ2=0.034 (χ20.05,=1= 3.84)。将37份种子被害率0~20%的材料视为纯合抗性个体, 80份种子被害率21%~80%的材料视为杂合抗性个体, 41份种子被害率81%~100%视为纯合感豆象个体, 卡方测验符合1∶2∶1的分离规律, χ2= 0.228 (χ20.05,=2= 5.99)。说明抗豆象亲本V1128对绿豆象的抗性由具有主效作用的显性单基因控制。延续前人的命名规则, 将该基因暂命名为“”。
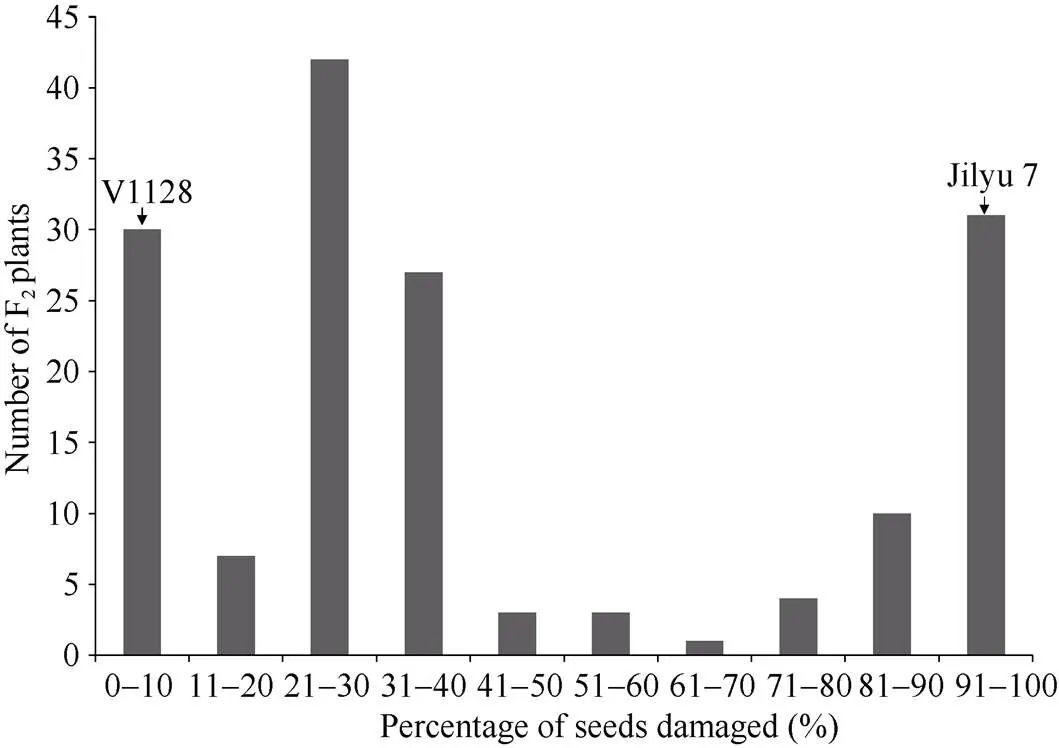
图1 F2群体种子被害率分布图
2.2 基因定位
利用31个多态性分子标记对158个F2单株进行基因型分析, 获得每个个体的基因型数据。在将抗豆象性状作为质量性状的条件下, 按照显性单基因的定位方法, 抗豆象基因作为一个表型标记与31个多态性分子标记共同进行标记间连锁分析。在LOD=5.0时, 所有31个分子标记和表型标记均位于同一分组内, 形成长度为73.9 cM的连锁群。在该连锁群中, 抗豆象基因被定位于标记DMB158和VRBR-SSR033 (标记VRID5、VRBR-SSR032与VRBR-SSR033的连锁群位置相同)之间, 两侧遗传距离分别为4.4 cM和5.8 cM (图2)。
利用F2群体的分子标记基因分型数据单独进行连锁图谱构建, 在LOD=5.0时, 形成1个长度为65.7 cM的连锁群。将抗豆象性状作为数量性状, 采用完备区间作图法(ICIM)对种子被害率进行QTL定位, 在该连锁群上检测到1个主效QTL, 位于标记DMB158和VRID5 (标记VRBR-SSR032、VRBR-SSR033与VRID5的连锁群位置相同)之间, LOD值为38.04, 可以解释表型变异(PVE)的71.64%, 其加性效应和显性效应值分别为57.78和22.92 (图3)。来自父本V1128的等位基因具有明显减少种子被害率的效应。
为了将与其他研究的抗豆象QTL定位结果进行比较, 利用7个共有标记将V1128抗豆象基因的QTL分析结果与V2709的QTL进行共线性分析, 所有7个共有标记均表现出极好的共线性。本研究中定位的V1128的抗豆象QTL与V2709的抗豆象位点QTL位于临近的标记区间内(图4), 2个QTL间存在部分重叠。

图2 抗豆象基因Br3连锁定位分析
利用QTL IciMapping 4.0软件进行标记连锁分析, LOD=5; 连锁群左右两侧分别标示图距(单位为cM)和标记名称。
Linkage map constructed by using QTL IciMapping 4.0 software, LOD=5; map distances (cM) and marker names are shown on the left and right sides of the linkage group, respectively.
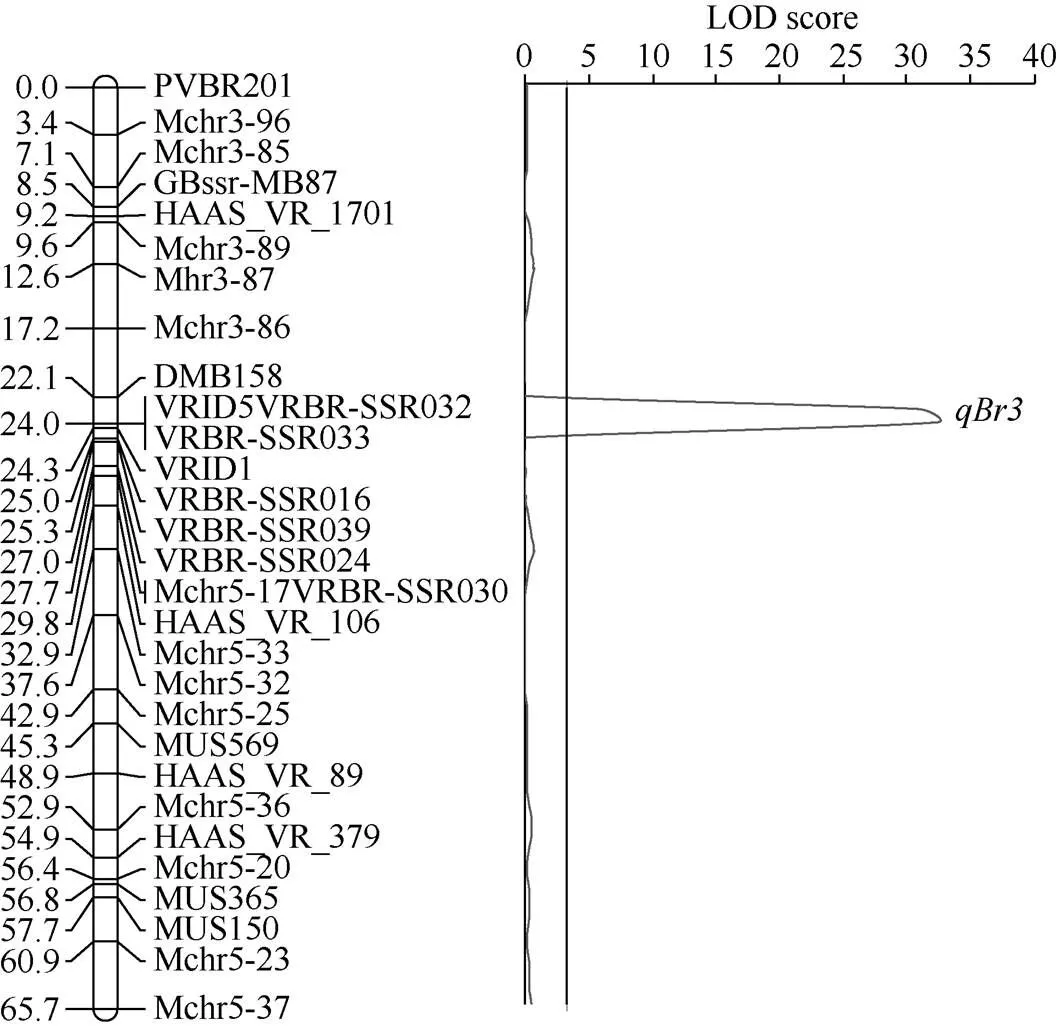
图3 抗豆象QTLqBr3的定位图谱
利用QTL IciMapping 4.0软件的完备区间作图法(ICIM)进行QTL 定位分析; 连锁群左右两侧分别标示图距(单位为cM)和标记名称; 竖线标示LOD阈值(3.18)。
QTL mapping based on inclusive composite interval mapping method (ICIM) using QTL IciMapping 4.0 software; map distances (cM) and marker names are shown on the left and right sides of the linkage group, respectively; the vertical line shows the LOD threshold (3.18).
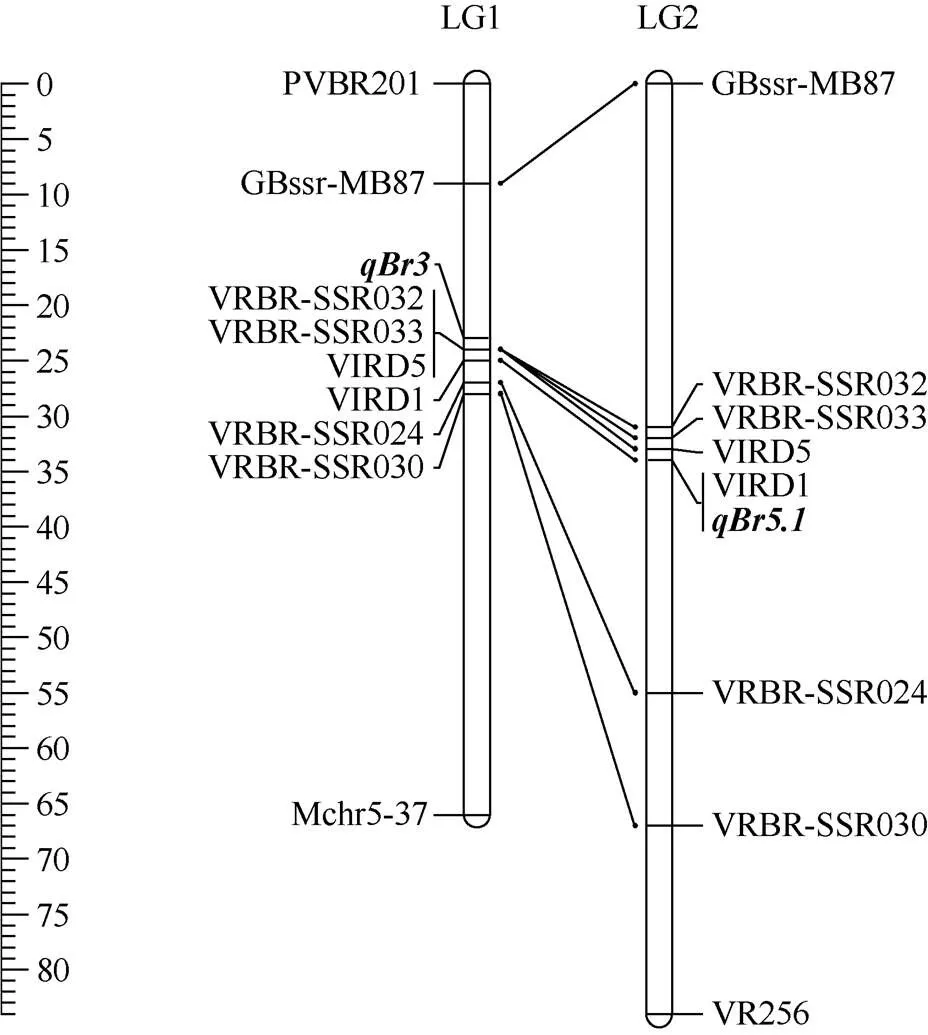
图4 QTL定位结果共线性比较分析
基于7个共有标记, 利用MapChart 2.2软件[30]作本研究与Kaewwongwal等[21]构建的连锁图谱的共线性图谱; 左侧标尺标示连锁群长度, 单位为cM; “LG1”为本研究构建的连锁图谱, “LG2”为Kaewwongwal等构建的连锁图谱; QTL名字用斜体字标示。
A synteny map between the genetic linkage map from this study and the linkage map constructed by Kaewwongwal et al.[21], this map was drown using MapChart 2.2 software[30]based on seven common markers; the scale bars on the left of the figure indicate the length of each linkage group in cM; “LG1” is the linkage group constructed in this study, “LG2” was constructed by Kaewwongwal et al.; the names of QTLs are shown in italics.
为了确定基因的染色体位置, 利用其两侧标记的引物序列与已经公布的绿豆基因组数据库[31]进行BLAST分析。DMB158位于绿豆第5染色体(Chr5: 5 597 658… 5 597 891), VRID5位于绿豆第5染色体(Chr5: 5 410 272… 5 410 493), VRBR-SSR032位于绿豆第5染色体(Chr5: 5 310 107…5 310 281), VRBR-SSR033位于绿豆第5染色体(Chr5: 5 380 081…5 380 180)。若取4个标记间的最远距离(VRBR-SSR032和DMB158之间), 可以将定位在绿豆第5染色体约288 kb的物理区间内(5 310 107… 5 597 891)。在此区间内共有11个注释基因(表2), 其中有5个基因编码RD22类蛋白, 2个基因编码多聚半乳糖醛酸酶抑制剂类蛋白, 3个基因编码未知蛋白;则为假基因。
3 讨论
从第一份抗豆象野生绿豆资源TC1966被发现以来, 研究者就没有停止过对不同抗豆象资源的遗传规律、基因定位、抗豆象机制等的研究。大量的研究表明, 不同抗豆象绿豆资源中均存在主效的显性抗豆象基因[8,11-12]。同TC1966、ACC41、V2802、V2709的抗豆象遗传规律相似, V1128的抗豆象特性也由主效的显性单基因控制。但从不同抗豆象单株的种子被害率存在差异、抗豆象性状表现出一定程度的数量性状遗传特征来看, 很可能也存在其他微效基因起修饰作用。这与Chen等[13]对TC1966抗豆象遗传规律的分析结论相同。
基于抗豆象绿豆资源中存在主效抗性单显性基因, 可以将抗豆象性状作为质量性状定位, 如Young等[15]、Kaga等[12]、Wang等[18]、孙蕾等[19]在对TC1966、ACC41、V2709的抗豆象基因定位研究中均采用了此策略。同时, 抗豆象性状表现出一定程度的数量性状遗传特征, 因此可以将其按照数量性状QTL定位的方法定位。如Chotechung等[20]、Kaewwongwal等[21]、Mei等[17]、Hong等[32]、Chen等[13]均采用数量性状QTL定位的方法, 根据种子被害率对V2802、V2709、ACC41、TC1966的抗豆象性状进行QTL定位。本研究同时采用上述2种方法均将定位在相同的标记区间内。2种方法的结果可以相互验证, 从而增加定位的准确度。

表2 V1128抗豆象基因位点范围内基因注释
V1128抗豆象基因所在的物理区间(5 310 107… 5 597 891)包含在Kaga等[12]对TC1966抗豆象基因的定位标记区间内(Bng143~Bng110, 物理区间为Chr.5: 5 052 031…6 779 027)。同时, 该区间包含Chotechung等[20]对V2802抗豆象基因定位的标记区间(VrSSR013~ DMB158, 物理区间为Chr.5: 5 561 387…5 597 891), 而与Kaewwongwal等[21]对V2709抗豆象基因定位的标记区间(VRID5~VRBR-SSR037, 物理区间为Chr.5: 5 410 272… 5 647 621)存在大部分重叠。表明这些不同的抗豆象种质可能具有相同或紧密连锁的抗豆象基因。.
Chotechung等[20]在V2802的抗豆象分离后代群体中发现标记DMB158与抗豆象性状共分离, 该标记位于()基因内部, 认为基因很可能是V2802的抗豆象候选基因。另外一个与紧密连锁的基因()与抗豆象性状不存在共分离, 认为不是抗豆象基因。然而Liu等[33]在NM92ÍTC1966的重组自交系群体中发现DMB158与抗豆象性状不完全连锁, 表明不是TC1966的抗豆象基因。根据绿豆基因组注释结果, Kaewwongwal等[21]确定V2709的定位区间内共有8个注释基因, 其中包括和。VrPGIP1和VrPGIP2蛋白同属于多聚半乳糖醛酸酶抑制剂类蛋白, 研究表明该类蛋白在植物抵御微生物病原体侵染及昆虫危害中起作用。如菜豆的基因在抗菌核病中发挥作用[34]; 菜豆PvPGIP3/PvPGIP4蛋白可以抑制盲蝽象的多聚半乳糖醛酸酶[35]。因此, 预测和可能是V2709的抗豆象候选基因。本研究构建的连锁图谱与Kaewwongwal等[21]构建的连锁图谱具有很好的共线性。但在本研究中DMB158与抗豆象性状之间并不存在共分离。因此,可能不是V1128的抗豆象基因。在定位区间内的11个注释基因中, 5个为RD22类蛋白编码基因。研究表明RD22类蛋白可能与植物抵御生物或非生物胁迫有关。如拟南芥的RD22蛋白参与耐脱水胁迫过程[36]。然而, 不能排除这类基因在抗豆象过程中发挥作用的可能性。同样, 在11个注释基因中还有3个未知蛋白的编码基因, 它们是否在抗豆象过程中起作用还无法判断。因此, 目前还不能仅仅依靠定位区间内基因功能分析的结果来判断V1128抗豆象基因的归属。有待进一步利用较大的近等基因系群体, 从中寻找交换单株, 对抗豆象基因精细定位。
致谢:对泰国农业大学Peerasak Srinnives教授提供抗豆象栽培绿豆种质V1128表示感谢。
[1] Yao Y, Cheng X, Ren G. A 90-day study of three bruchid-resistant mungbean cultivars in Sprague-Dawley rats., 2015, 76: 80–85
[2] 王丽侠, 程须珍, 王素华. 绿豆种质资源、育种及遗传研究进展. 中国农业科学, 2009, 42: 1519–1527 Wang L X, Cheng X Z, Wang S H. Advances in research on genetic resources, breeding and genetics of mungbean (L.),, 2009, 42: 1519–1527 (in Chinese with English abstract)
[3] 程须珍, 王素华, 金达生, 杨又迪, 吴绍宇, 周吉红. 绿豆抗豆象遗传的初步研究. 植物遗传资源学报, 2001, 2(4): 12–15 Cheng X Z, Wang S H, Jin D S, Yang Y D, Wu S Y, Zhou J H. Preliminary study on heredity of mungbean resistance to bruchid., 2001, 2(4): 12–15 (in Chinese with English abstract)
[4] 刘长友, 田静, 范保杰, 曹志敏, 苏秋竹, 张志肖, 王素华. 豇豆属3种主要食用豆类的抗豆象育种研究进展. 中国农业科学, 2010, 43: 2410–2417 Liu C Y, Tian J, Fan B J, Cao Z M, Su Q Z, Zhang Z X, Wang S H. Advances in breeding research on bruchid-resistant cultivars of three mainfood legumes., 2010, 43: 2410–2417 (in Chinese with English abstract)
[5] Tomooka N, Kashiwaba K, Vaughan D A, Ishimoto M, Egawa Y. The effectiveness of evaluating wild species: searching for sources of resistance to bruchid beetles in the genussubgenus., 2000, 115: 27–41
[6] 金文林, 谭瑞娟, 王进忠, 张志勇, 刘长安, 濮绍京, 赵波. 田间小豆绿豆象卵空间分布型初探. 植物保护, 2004, 30(6): 34–36 Jin W L, Tan R J, Wang J Z, Zhang Z Y, Liu C A, Pu S J, Zhao B. Preliminary study on spatial distribution pattern ofegg in adzuki bean field., 2004, 30(6): 34–36 (in Chinese with English abstract)
[7] Fujii K, Miyazaki S. Infestation resistance of wild legumes () to adzuki bean weevil,(L.) (Coleoptera: Bruchidae) and its relationship with cytogenetic classification., 1987, 22: 229–230
[8] Lambrides C J, Imrie B C. Susceptibility of mungbean varieties to the bruchid species(F.),(Gyll.),(L.), and(Say.) (Coleoptera: Chrysomelidae)., 2000, 51: 85–90
[9] Talekar N S, Lin C P. Characterization of(Coleoptera: Bruchidae) resistance in mungbean., 1992, 85: 1150–1153
[10] Somta C, Somta P, Tomooka N, Ooi A C, Vaughan D A, Srinives P. Characterization of new sources of mungbean ((L.) Wilczek) resistance to bruchids,spp. (Coleoptera: Bruchidae)., 2008, 44: 316–321
[11] Kitamura K, Ishimoto M, Sawa M. Inheritance of resistance to infestation with adzuki bean weevil inand successful incorporation to., 2008, 38: 459–464
[12] Kaga A, Ishimoto M. Genetic localization of a bruchid resistance gene and its relationship to insecticidal cyclopeptide alkaloids, the vignatic acids, in mungbean (L. Wilczek)., 1998, 258: 378–384
[13] Chen H M, Ku H M, Schafleitner R, Bains T S, Kuo G C, Liu C A, Nair R M. The major quantitative trait locus for mungbean yellow mosaic Indian virus resistance is tightly linked in repulsion phase to the major bruchid resistance locus in a cross between mungbean [(L.) Wilczek] and its wild relativessp.., 2013, 192: 205–216
[14] Somta P, Ammaranan C, Ooi P, Srinives P. Inheritance of seed resistance to bruchids in cultivated mungbean (, L. Wilczek)., 2007, 155: 47–55
[15] Young N D, Kumar L, Menancio-Hautea D, Danesh D. RFLP mapping of a major bruchid resistance gene in mungbean (, L. Wilczek)., 1992, 84: 839–844
[16] Miyagi M, Humphry M, Ma Z Y, Lambrides C J, Bateson M, Liu C J. Construction of bacterial artificial chromosome libraries and their application in developing PCR-based markers closely linked to a major locus conditioning bruchid resistance in mungbean (L. Wilczek)., 2004, 110: 151–156
[17] Mei L, Cheng X Z, Wang S H, Wang L X, Liu C Y, Sun L, Xu N, Humphry M E, Lambrides C J, Li H B, Liu C J. Relationship between bruchid resistance and seed mass in mungbean based on QTL analysis., 2009, 52: 589–596
[18] Wang L, Wu C, Zhong M, Zhao D, Mei L, Chen H, Wang S, Liu C, Cheng X. Construction of an integrated map and location of a bruchid resistance gene in mungbean., 2016, 4: 360–366
[19] 孙蕾, 程须珍, 王素华, 王丽侠, 刘长友, 梅丽, 徐宁. 栽培绿豆V2709抗豆象特性遗传及基因初步定位. 中国农业科学, 2008, 41: 1291–1296 Sun L, Cheng X Z, Wang S H, Wang L X, Liu C Y, Mei L, Xu N. Heredity analysis and gene mapping of bruchid resistance of a mungbean cultivar V2709., 2008, 41: 1291–1296 (in Chinese with English abstract)
[20] Chotechung S, Somta P, Chen J, Yimram T, Chen X, Srinives P. A gene encoding a polygalacturonase-inhibiting protein (PGIP) is a candidate gene for bruchid (Coleoptera: bruchidae) resistance in mungbean ()., 2016, 129: 1673–1683
[21] Kaewwongwal A, Chen J, Somta P, Kongjaimun A, Yimram T, Chen X, Srinives P. Novel Alleles of two tightly linked genes encoding polygalacturonase-inhibiting proteins (VrPGIP1 and VrPGIP2) associated with the Br Locus that confer bruchid (spp.) resistance to mungbean () accession V2709., 2017, 8: 1692
[22] Liu C, Fan B, Cao Z, Su Q, Wang Y, Zhang Z, Wu J, Tian J. A deep sequencing analysis of transcriptomes and the development of EST-SSR markers in mungbean ()., 2016, 95: 527–535
[23] Liu C, Wu J, Wang L, Fan B, Cao Z, Su Q, Zhang Z, Wang Y, Tian J, Wang S. Quantitative trait locus mapping under irrigated and drought treatments based on a novel genetic linkage map in mungbean (L.)., 2017, 130: 2375–2393
[24] Gwag J G, Chung J W, Chung H K, Lee J H, Kyung-Ho M A, Dixit A, Park Y J, Cho E G, Kim T S, Lee S H. Characterization of new microsatellite markers in mung bean,(L.)., 2007, 6: 1132–1134
[25] Somta P, Seehalak W, Srinives P. Development, characterization and cross-species amplification of mungbean () genic microsatellite markers., 2009, 10: 1939–1943
[26] Grisi M C, Blair M W, Gepts P, Brondani C, Pereira P A, Brondani R P. Genetic mapping of a new set of microsatellite markers in a reference common bean () population BAT93ÍJalo EEP558., 2007, 6: 691–706
[27] Meng L, Li H, Zhang L, Wang J. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations., 2015, 3: 269–283
[28] Kosambi D D. The estimation of map distances from recombination values., 1943, 12: 172–175
[29] Li H, Ribaut J M, Li Z, Wang J. Inclusive composite interval mapping (ICIM) for digenic epistasis of quantitative traits in biparental populations., 2008, 116: 243–260
[30] Voorrips R E. MapChart: software for the graphical presentation of linkage maps and QTLs., 2002, 93: 77–78
[31] Kang Y J, Kim S K, Kim M Y, Lestari P, Kim K H, Ha B K, Jun T H, Hwang W J, Lee T, Lee J, Shim S, Yoon M Y, Jang Y E, Han K S, Taeprayoon P, Yoon N, Somta P, Tanya P, Kim K S, Gwag J G, Moon J K, Lee Y H, Park B S, Bombarely A, Doyle J J, Jackson S A, Schafleitner R, Srinives P, Varshney R K, Lee S H. Genome sequence of mungbean and insights into evolution withinspecies., 2014, 5: 5443, doi: 10.1038/ ncomms6443
[32] Hong M G, Kilhyun K, Jahwan K, Jinkyo J, Minjung S, Changhwan P, Yulho K, Hongsik K, Yongkwon K, Sohyeon B. Inheritance and quantitative trait loci analysis of resistance genes to bruchid and bean bug in mungbean (L. Wilczek)., 2015, 3: 39–46
[33] Liu M S, Kuo T C Y, Ko C Y, Wu D C, Li K Y, Lin W J, Lin C P, Wang Y W, Schafleitner R, Lo H F, Chen C Y, Chen L F O. Genomic and transcriptomic comparison of nucleotide variations for insights into bruchid resistance of mungbean ([L.] R. Wilczek)., 2016, 16: 46
[34] Vasconcellos R C, Lima T F, Fernandesbrum C N, Chalfunjunior A, Santos J B. Expression and validation ofgenes for resistance to white mold () in common beans (L.)., 2016, 15: 15038269
[35] D'Ovidio R, Raiola A, Capodicasa C, Devoto A, Pontiggia D, Roberti S, Galletti R, Conti E, O’Sullivan D, Lorenzo G D. Characterization of the complex locus of bean encoding polygalacturonase-inhibiting proteins reveals subfunctionalization for defense against fungi and insects., 2004, 135: 2424–2435
[36] Yamaguchi-Shinozaki K, Shinozaki K. The plant hormone abscisic acid mediates the drought-induced expression but not the seed-specific expression of, a gene responsive to dehydration stress in., 1993, 238: 17–25
Genetic Mapping of Bruchid Resistance Gene in Mungbean V1128
LIU Chang-You1, SU Qiu-Zhu1, FAN Bao-Jie1, CAO Zhi-Min1, ZHANG Zhi-Xiao1, WU Jing2, CHENG Xu-Zhen2, and TIAN Jing1, *
1Institute of Food and Oil Crops, Hebei Academy of Agricultural and Forestry Sciences / Hebei Laboratory of Crop Genetic and Breeding, Shijiazhuang 050031, Hebei, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
It is an urgent research topic to map the bruchid resistance gene and to carry out bruchid resistance breeding using molecular marker-assisted method in mungbean. This study was carried out to identify a F2isolated group formed by the hybrid of a bruchid-resistant cultivar “V1128” and a bruchid-susceptible cultivar “Jilyu 7”, and to analyze the genetic regularity of V1128 in resistance to bruchids. The bulked segregant analysis (BSA) method was used for screening the polymorphic markers. Genetic linkage map construction and quantitative trait locus (QTL) mapping were conducted using software QTL IciMapping 4.0. The results showed that the bruchid resistance of V1128 was controlled by a dominant gene with main effect. According to previous naming rules, the bruchid resistance gene of V1128 was temporarily named as “”. When we treated bruchid-resistance as a quality trait,was used as a marker for linkage map construction and was positioned between the markers DMB158 and VRBR-SSR033 (VRID5, VRBR-SSR032, and VRBR-SSR033 are located at the same map position). The genetic distances ofaway from the two markers were 4.4 cM and 5.8 cM, respectively.was positioned on chromosome 5 in the physical range of about 288 kb. By using the inclusive composite interval mapping (ICIM) to locate the seed damage rate, a main QTL locus with LOD score of 38.04 was identified in the marker intervals from DMB158 to VRBR-SSR033, contributing 71.64% of the observed phenotypic variation. The allele of male parent V1128 had a significant effect on reducing the rate of seed damage. The results can provide useful information for the molecular marker-assisted breeding of mungbean, and the fine localization and cloning of.
mungbean; bruchid resistance; V1128;; QTL
2018-05-08;
2018-08-20;
2018-09-18.
10.3724/SP.J.1006.2018.01875
通信作者(Corresponding author): 田静, E-mail: nkytianjing@163.com, Tel: 0311-87670655
E-mail: Liuchangyou2006@aliyun.com.cn
本研究由国家自然科学基金项目(31601367), 国家现代农业产业技术体系建设专项(CARS-08), 河北省科技计划项目(16227508D)和河北省现代农业科技创新工程项目(F18R494004-01)资助。
This work was supported by the National Natural Science Foundation of China (31601367), the China Agriculture Research System (CARS-08), the Science and Technology Program of Hebei (16227508D), and the Modern Agricultural Science and Technology Innovation Program of Hebei Province (F18R494004-01).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20180915.1120.004.html
