针对高维数据的动态网格子空间聚类算法HDGCLUS
2018-10-26刘晨赫刘小晴
刘晨赫,刘小晴,刘 青,苏 蕉,杨 楠,肖 林
(中国人民大学 信息学院计算机系,北京 100872)
1 引 言
聚类分析是一种重要的无监督学习方法,信息时代大量涌现的基因表达数据、图像、文本数据等都具有很高的维度,为了解决高维数据聚类难题,产生了子空间聚类算法,它是一种在局部子空间而非全维度空间上进行搜索和识别数据中稠密簇的聚类方法.
CLIQUE[1]是较早尝试在子空间中搜索稠密簇的算法,由IBM Almaden研究中心数据挖掘课题组提出和实现.CLIQUE综合了传统聚类算法中基于网格的聚类和基于密度的聚类的特点,从单一维度开始自底向上地搜索存在于子空间中的稠密簇.此后国内外提出了大量子空间聚类算法,以期提高这种聚类方法的质量和效率,德国慕尼黑大学HANS-PETER KRIEGEL教授对高维数据聚类算法做了详细的综述[2].依据不同的搜索策略,可以将子空间聚类分为自底向上和自顶向下的两大类.著名的CLIQUE算法和ENCLUS[3]、MAFIA[4]、GPUMAFIA[5]、并行框架[6]以及SUBCLU[7]等都是自底向上的子空间聚类算法.自顶向下的子空间聚类算法有PROCLUS[8]、ORCLUS[9]、FINDIT[10]、δ-Clusters[11]以及COSA[12]等.
随着硬件平台的提升,近年来通过并行处理[5,6]和云计算[17,18]来提高子空间聚类的计算效率.由于子空间聚类方法在聚类的同时对数据进行降维和特征提取,有文献结合机器学习领域的研究热点如深度网络、图模型等来获得低维的数据表示[13-15],也取得较好效果.
CLIQUE算法虽然存在较多缺点,比如较高的时间复杂度、敏感的参数设置,以及采用固定网格划分、MDL剪枝等技术,容易破坏稠密区域的边缘或者丢失一些有用信息等,但也具有模型简明易扩展、能对任意形状的数据聚类、对数据输入顺序不敏感、聚类结果易于解释以及对硬件配置和平台要求不高的优点,尤其在生物信息学中对疾病聚类的同时发现致病原因的应用效果明显.本文基于CLIQUE算法提出了改进的HDGCLUS(High-Dimensional Genomic data subspace CLUStering) 子空间聚类算法.新算法采用基于稀疏区域的动态网格划分技术,实现了网格的动态划分和稠密区域的动态合并,并加入了边界调整技术,减少了初始候选稠密单元个数,避免了人工输入网格参数和边界数据信息的丢失,提高了聚类质量和算法效率.同时新算法采用静态剪枝和信息增量动态剪枝相结合的技术,进一步降低了算法复杂度,优化了算法性能.
2 HDGCLUS算法
2.1 CLIQUE算法基本概念与符号
假设输入为n维数据矩阵S,矩阵的行表示样本,列表示属性,那么S=A1*A2*A3…*An.其中A1、A2等代表相应属性维度.S的行表示为V={V1,V2,V3,…,Vm},其中每个样本Vi={Vi1,Vi2,…,Vin},即Vi的第j个分量Vij∈Aj.
定义1. 子空间
对于一个 n维的数据集合,其在任意K个维度上组成的空间,被称为 K维子空间.
定义2. 网格参数 ξ
CLIQUE有两个输入参数:网格参数 ξ和密度参数τ.算法初始时基于网格参数将数据集S的每一个维度都分割成均匀的 ξ个区间,每个区间描述为一个类似于 Ui=[li,hi) 的前闭后开网格.
数据集中的一个样本点集 V={V1,V2,…,Vk}当且仅当对于每一个 Ui都有li≤ Vi< hi成立时,才称其在单元 U={U1,U2,…,Uk}中.
定义3. 密度参数 τ
CLIQUE算法根据密度参数来判定单元格是否为稠密单元格.数据单元 U是稠密的,当且仅当selectivity(U)>τ,其中 selectivity(U) 称为单元格 U的选择率,定义为:
selectivity(U)=单元格中样本数/数据集总样本数
定义4. 单元格连通
两个K维空间中的单元格u1,u2称为连通的,仅当这两个单元格有一个公共的面,或者这两个单元格u1,u2都跟另外一个单元格u3连通.
定义5. 公共面

定义6. 区域
区域是指由单元格组成、具有规则形状的,并且每一条边都与坐标轴平行的一个类矩形,可以用区间的交的形式表示.
称R是最大区域,当且仅当 R∩C=R,即没有一个R的超集R’也包含于C,其中C是区域 R包含的一个聚类.
CLIQUE是自底向上的子空间聚类算法,算法步骤如下[1]:
第1步. 根据网格参数ξ的值将原数据的每一维度划分成相等的区间;
第2步. 初始K=1;
第3步. 扫描数据集,找出K维子空间中落在每个候选单元格的数据点数,根据密度阈值τ找出K维子空间中的所有稠密单元;
第4步. MDL修剪;
第5步. 由K维子空间中的稠密单元集,生成K+1维子空间中的侯选稠密单元集,若K+1维子空间中的侯选稠密单元集不为空,跳转第3步;
第6步. 随机选取一稠密单元格,用深度优先策略找出K维空间中的簇;
第7步. 用贪心算法求出覆盖每个簇的最大区域;
第8步. 根据最大区域计算出每个聚类的最小覆盖描述,并以析取范式的形式输出结果;
2.2 HDGCLUS的动态网格划分
由以上对CLIQUE算法的描述和实验可知,该算法对输入的网格参数 ξ和密度参数τ敏感,采用固定等长的网格划分,不仅容易破坏稠密区域的边缘,降低最终聚类结果的准确性,而且由于增加了网格参数,人为增加了聚类结果的不准确性.在HDGCLUS算法中,实现了网格的动态划分和稠密区域合并,并加入了边界调整技术,降低了算法初始候选稠密单元个数,避免了人工输入网格参数和边界数据信息的丢失,提高了聚类质量和算法效率.
2.2.1 确定网格划分数目
当高维数据维度无限大时,其在低维空间的投影以概率1逼近正态分布[16].
因此,为了更好地反映高维数据在低维投影的分布情况,根据信息理论,将高维数据在每一维上的投影按数据的统计特征划分为多个网格区间,N=min{(1+log2S)d,k}其中,N为每一维上网格划分的个数,S为样本数,d为子空间维度数目(一维空间d值为1),k为全局空间维度数目即属性个数.
通过将每一维度划分为N个不同的网格,更真实的反映了高维数据在低维的分布情况,同时也避免了网格参数的敏感性对聚类结果的影响.
2.2.2 密度阈值参数设定

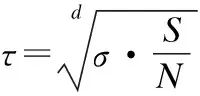
2.2.3 单一维度簇识别
CLIQUE根据用户输入的密度阈值进行稠密网格的识别,同时删除稀疏网格.这样可能破坏稠密区域的边界,导致稠密区域边界上的点无法有效识别.聚类可被看作稠密区域的集合,而稠密区域又是由稀疏区域分隔开的,因此本文将聚类重新定义为由稀疏区域分隔开的稠密区域.关联规则指出,K维上稠密区域,在任一K-1维上的投影亦是稠密的.反过来说,任一K-1维上的稀疏区域,在K维上也是稀疏的.那么单一维度上的稀疏区域,也即是高维空间中的稀疏区域在一维空间上的投影,因此一维空间上的稀疏区域构成了类的边界,本算法也将立足于此进行稠密区域的划分.
依据聚类为稀疏区域分隔开的稠密区域这一思想,按照平均误差最小的原则,本文将每一维度上簇的分界定义为与稠密区域相邻的稀疏区域的中间值.这样调整了稠密区域的边界,避免了稠密区域边界上的点信息的丢失.

图1 单一维度簇识别样例图Fig.1 Clusters in single dimension
图1的平行坐标系展示了具有3个属性的64个样本.第一步的网格划分,将64个样本点划分为7个网格,在V1维度上,可以看到[0,10),[10,20),[30,40) 为稠密单元格,其余为稀疏单元格.传统的稠密单元识别算法,将会丢失[20,22)之间的数据点,造成聚类信息的不完整.
本算法通过相邻稠密单元格的合并,以及在稀疏区域中点的划分,可以有效的识别单一维度上的簇.具体各维度上的簇的表示如下:
V1:[0,25),[25,45)
V2:[0,25),[25,45)
V3:不存在密集区域
HDGCLUS算法中单一维簇识别步骤如下:
1)依次输入每一维度,并进行排序;
2)统计每一维度的最大值max和最小值min;
3)将每一维度[min,max]区间,划分为(1+log2S) 个前闭后开的网格区间;
4) 根据密度阈值τ确定稠密单元格,并将相邻稠密单元格合并;
5) 按照稠密区域相邻的稀疏区域的中间值为簇边界进行网格划分.
2.3 识别子空间
HDGCLUS识别子空间的过程分为静态剪枝和信息增量动态剪枝两部分,目的在于提高计算效率.
2.3.1 静态剪枝
静态剪枝是指在前面单一维度簇识别过程中,对每一维度进行检测,将满足以下两种情况中任意一种的维度删除:
a.该维度上不存在任何稠密区域;
b.该维度上只有唯一的一个稠密区域;
情况a表明高维空间在这个维度上的投影是分散的或者是均匀的,那么该维度对最终类的发现没有任何贡献,因此将其删除.
情况b显示高维空间上的类在此维度上的投影只有一个稠密区域,即任何类在该维度上的投影都在同一个区域,那么这个维度对识别高维空间上的类也没有任何贡献的,因此将其删除.
2.3.2 信息增量动态剪枝
在子空间自底向上迭代搜索的过程中,我们对每一个发现的子空间进行判断,同时定义了一个兴趣度增量参数,如果该子空间的兴趣度增益大于给定的兴趣度增量参数,则该子空间不再增长.这样动态的剪枝策略,不仅有效地避免了子空间盲目地增长,降低了算法复杂度,同时也有助于生成无冗余的聚类描述.
1)熵值
根据信息熵的概念,在子空间聚类过程中,存在簇的子空间往往比不存在簇的子空间拥有更小的熵值.
设X为一个离散随机变量,其概率分布为:
则随机变量X的信息熵定义为:
(1)
本文信息熵的对数的底数取值为2,信息熵的单位为比特(bit).
2)互信息
互信息(Mutual Information)在信息论中是衡量两个事件集合之间相关性大小的度量,设事件X和Y,则Y对X的互信息量定义为X的后验概率与先验概率比值的对数,即:
(2)
根据熵的连锁规则可知I(X,Y)=I(Y,X),
即有,
I(X,Y) =H(X)-H(X|Y)=H(Y)-H(Y|X)
=H(X)+H(Y)-H(X,Y)
其中H(X,Y)是事件X和Y的联合熵(Joint Entropy),定义为:
H(X,Y)=-∑p(x,y)·log2p(x,y)
(3)
3) 子空间信息增量
单一维度两两之间的相关性可以用信息熵中的互信息I(X,Y)来衡量,同理多个维度之间的相关性我们也用熵来衡量,并将其定义为兴趣度In(X)(1维子空间的兴趣度为0)[3]:
(4)
其中:
H({X1,X2,…,Xn})
=-∑x1…∑xnp(x1,…,xn)log2p(x1,…,xn)
(5)
x1,x2,…, xn是 X1,X2,…,Xn的特定值,相应的 p(x1,x2,…,xn) 为 X1,X2,…,Xn这些变量同时出现的概率.在全维空间中,假设存在维度A和维度B,两维之间数据分布相似,那么这两个维度之间具有较强的相关性.A和B共同形成的子空间AB已经能很好的表述A和B形成的类,为了得到最小无冗余的子空间,AB子空间无需继续增长.更一般化的来说,在算法自底向上的搜索过程中,对于任一候选的k维子空间Sk,如果In(Sk)的值大于给定的阈值ε,则该子空间不再增长.
为了更直观的衡量维度之间相关性的增长,本文定义一个新的函数,兴趣度增量In_gain.子空间 {X1,X2,…,Xn}的兴趣度增量定义为:
In_gain({X1,X2,…,Xn})=
In({X1,X2,…,Xn})-max{In({X1,X2,…,Xn}-{Xi})}
(6)
兴趣度增量是子空间增加一个维度的最小兴趣度增量,1维子空间的兴趣度增量为0.在算法自底向上的搜索过程中,对于任一候选的k维子空间Sk,如果In_gain(Sk)的值大于给定的阈值ε′,则该子空间不再增长.
2.4 识别聚类
识别聚类的过程是根据稠密单元格的集合Dk(集合中单元格全部都在同一个k维空间S中),得出集合Dk的一个划分{Dk1,Dk2,…,Dkq},满足处于Dki中所有稠密单元格都是相邻的,并且处在不同的Di、Dj中的单元格是不相邻的.
2.5 生成聚类描述
根据2.4产生集合Dk的每一个划分{Dk1,Dk2,…,Dkq},找到聚类的最大覆盖,通过最大覆盖生成最小描述,并以析取范式的形式表示出来.
2.6 HDGCLUS算法性能分析
与CLIQUE算法相比,HDGCLUS算法同样采用自底向上的搜索过程,但动态网格划分以及静态剪枝和信息增量动态剪枝相结合的剪枝方法大大减少了稠密网格数目以及子空间迭代搜索次数,降低了算法时间复杂度.假定S为样本数,D为数据集维数,K为子空间大小,HDGCLUS算法整体的时间复杂度为O(S*logS *K),同时算法在子空间搜索过程中需要存储每一个稠密区域信息,空间复杂度为O(D*logS).
3 实 验
本文实验均在Windows7操作系统,英特尔酷睿i3 CPU,4GB内存硬件平台上进行,软件开发实现语言为JAVA,数据处理平台为MATLAB R2012a.
3.1 实验数据集
为了检测HDGCLUS算法在不同高维数据集上的良好伸缩性,实验选取了4组真实数据集:高维度的SoyBean[19],较高维度的Leukemia[20]、DLBCL[21]以及超高维Breast Cancer[22],数据集具体信息如表1所示.
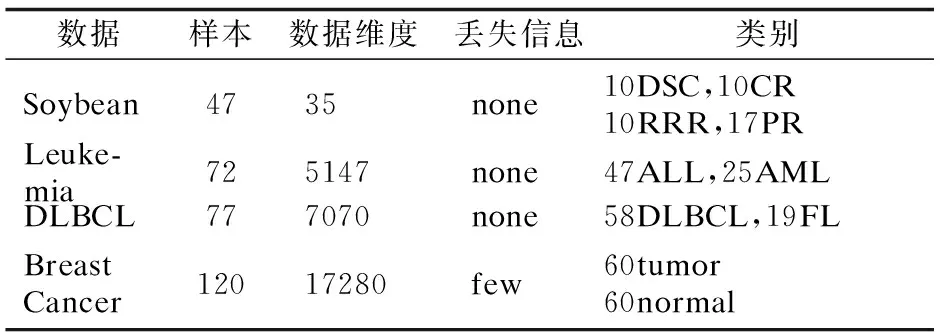
表1 实验数据集Table 1 4 Datasets
3.2 评价指标
由于子空间聚类是在局部样本集以及局部空间中搜索可能存在的聚类,因此部分样本和维度可能并不在聚类结果中,为了更为客观的展示HDGCLUS算法与其他算法的性能,本文选取信息检索中的准确率(Precision)和F1值(F-Measure)两者作为评价指标.
在信息检索结果评价中,准确率和召回率(Recall)是两个基本指标,准确率定义为返回结果中相关文档所占的比例,召回率描述了返回的相关文档占所有相关文档的比例.在聚类结果评价中,准确率可以很好地衡量参与聚类的样本集中正确聚类的样本比例.
本文采用F1值作为衡量聚类结果的另一评价指标.F1值融合了准确率P值和召回率R值,定义为P值和R值的调和平均值:
(7)
由于聚类结果中存在多个簇,因此本文定义整个聚类的P值为所有簇的P值的平均值,同理F1值也为所有簇的F1值的平均值.假设k为聚类结果中簇的个数,则聚类的P值和F1值定义如下:
(8)
(9)
针对基因表达数据,我们还进行了NCBI生物验证,其生物学意义十分显著,此处限于篇幅不作介绍.
3.3 实验结果
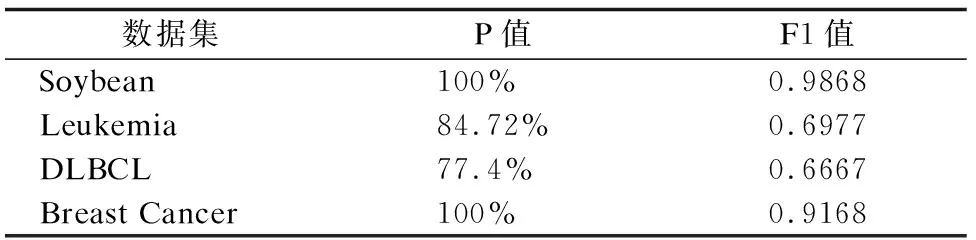
表2 聚类结果Table 2 Clustering results
3.4 同类算法对比实验
本文将HDGCLUS与应用广泛的聚类算法K-Means、CLIQUE、SUBCLU、以及PROCLUS进行对比实验,进一步验证了HDGCLUS算法在聚类结果准确性以及算法时间复杂度上的优越性.考虑到传统算法如K-means对处理超高维数据能力太弱,在此仅展示Soybean数据集结果.5种聚类算法的参数设置见表3,聚类结果的P值和F1值如表4所示.

表3 5种聚类算法的参数设置Table 3 Parameter setting of five algorithms

表4 各算法在SoyBean数据集上的运行评价值Table 4 Evaluation values of clustering on Soybean data
各算法对数据集SoyBean聚类的运行时间如图2所示.

图2 Soybean数据集上各算法运行时间Fig.2 Running time of five algorithms on Soybean data
4 结束语
本文设计实现了针对超高维数据的子空间聚类算法HDGCLUS,分析并实验验证了HDGCLUS在处理超高维数据时的优良性能.通过研究分析可知HDGCLUS依然存在不足,我们将在以下方面做进一步的探索:
1)子空间聚类算法对数据类别分布敏感,因此在今后的工作中需要进一步优化算法,解决新算法在类别样本数分布不均匀的数据集上聚类效果不理想的问题.
2)进一步降低算法的参数敏感度,可在下一步工作中实现自适应选择的兴趣度增益阈值参数ε.
