DNA甲基化测序技术及其在哺乳动物中的应用研究进展
2018-10-15段昕妤陈善元
段昕妤, 肖 蘅, 陈善元
(云南大学 生命科学学院, 昆明 650091)
DNA甲基化是指在DNA甲基转移酶的催化下,以S-腺苷甲硫氨酸(SAM)为甲基供体,将甲基转移到胞嘧啶的5′C上,DNA的碱基结合甲基的过程[1]。在哺乳动物中,正常的DNA甲基化在基因表达等方面具有重要作用。但是,异常的DNA甲基化却会引起人类疾病的发生,如癌症、衰老、老年痴呆等。随着甲基化研究的不断深入,甲基化的检测方法也在不断改进更新,本文将对哺乳动物DNA甲基化测序技术作简要介绍,以期为甲基化测序技术的选择提供参考依据。
1 DNA甲基化简介
DNA甲基化现象是1948年Hotchkiss在小牛胸腺DNA中发现的[2],直到1988年,人们才逐渐认识到DNA甲基化对基因功能的影响,进而展开了该领域的研究。作为表观遗传学的重要修饰方法之一,DNA甲基化在很多生物代谢进程中起着关键性作用,如基因表达、胚胎发育、细胞增生、细胞分化和染色体稳定性等。更重要的是,DNA甲基化可以随着细胞分裂在世代间遗传[3]。除了可遗传性,DNA甲基化还具有其他特性:1)普遍性。DNA甲基化现象普遍存在于原核生物和真核生物中,哺乳动物基因组中约有5%~10%是CpG位点,其中约80%为甲基化CpG位点(mCpG)[4]。2)分布规律性。DNA甲基化主要发生于启动子、转座子、增强子、沉默子和基因本体等部位的CpG二核苷酸上。3)时间特异性和空间特异性。时间特异性指的是在生命不同时期DNA甲基化程度存在巨大差异,而空间特异性即组织特异性,指的是不同的组织细胞即便拥有一样的基因序列却有不一样的DNA甲基化模式,以此调控基因在不同组织内的特异性表达。4)可逆性。去甲基化(demethylation)是与甲基化一样存在于生物中却完全相反的过程,且细胞内只要存在甲基化就必然存在去甲基化,甲基胞嘧啶的水平和模式由DNA甲基化和去甲基化共同决定。除去甲基化与去甲基化的动态调控,甲基化状态还会受到环境、年龄、性别等因素影响。其中,DNA甲基转移酶(DNA methyltransferase, DNMTs)也是影响DNA甲基化的重要因素之一,哺乳动物存在3个甲基化转移酶家族:DNMT1、DNMT2、DNMT3(DNMT3A和DNMT3B)[5]。DNMT1在细胞分裂期DNA进行复制修复及维持正常甲基化的过程中起关键作用;DNMT2是主要的tRNA甲基转移酶[6];DNMT3含有3个亚基,3A和3B催化CpG岛进行从头甲基化,而3L(DNMT3-like)是一个不具有催化活性的调节蛋白,主要负责3A和3B的调控[7]。DNMT1是DNMT酶家族中研究最多的一种酶,很多研究发现DNMT1的上调与DNA异常甲基化有关,会导致抑癌基因沉默、原癌基因激活导致的肿瘤发生[8]。
在对甲基化作用研究有一定了解的基础上,还发现DNA甲基化在克隆技术、家畜育种、人类疾病等方面也扮演着重要角色。从目前克隆动物的繁育情况来看,动物克隆效率低且克隆胚胎流产率极高,分析原因可能是基因启动子区域异常的DNA甲基化引起异常的基因表达从而使得克隆动物表型异常或发育缺陷[9]。此外,DNA甲基化修饰还参与调控家畜重要性状的表达,如Fang等[10]比较分析了日本黑牛(Wagyu)与中国草原红牛背最长肌的甲基化差异与肉质性状的关系。在人类疾病方面,DNA的甲基化状态往往与疾病的发生密切相关,了解复杂疾病的甲基化致病机理,摸清治病基因的甲基化模式,既有助于理解复杂疾病的产生也有利于复杂疾病的治疗。DNA甲基化具有可逆性,对异常甲基化还原药物的研发、复杂疾病的治疗都有较高价值。随着DNA甲基化在医学、畜牧育种等领域逐渐凸显出重要作用,甲基化的检测也成了现阶段的研究热点。以下将对一些常用测序方法进行介绍和综合比较,以期为甲基化测序技术的选择提供思路。
2 DNA甲基化测序技术的发展
在测序技术普及之前,高效液相色谱法(HPLC)是经典的甲基化测定方法,但只能测定总的DNA甲基化水平,对特定序列无法测定。而对特定DNA序列测定的经典方法是Southern杂交分析法,但此方法敏感性较低,样品需求量较大。之后,结合PCR技术与酶切技术出现了新的检测技术,例如甲基化敏感的限制性指纹法(MSRF)、甲基化敏感扩增多态性法(MASP)、限制性标记基因组扫描法(RLGS)和CpG岛扩增结合代表差异分析技术(MCA-RDA)等。随着测序技术的不断发展和哺乳动物基因组测序的完成,对哺乳动物全基因组水平DNA甲基化的检测也逐渐实现。结合新一代测序技术(next-generation sequencing, NGS)的甲基化测定法是目前最新、最具前景的全基因组DNA甲基化分析方法,能对DNA甲基化进行定量测定且产生大量的序列信息。基于NGS进行全基因组DNA甲基化水平的方法主要分为3大类:1)核酸内切酶消化法;2)亲和富集法;3)重亚硫酸盐转化法。以下将对上述3类方法进行详细介绍。
2.1 基于核酸内切酶消化法的测序方法
2.1.1 HELP-seq
连接子介导PCR的HpaⅡ小片段富集测序法(HpaⅡtiny fragement enrichment by ligation-mediated PCR, HELP-seq)指用限制性内切酶Ⅱ(HpaⅡ)与甲基化敏感同裂酶(MspⅠ)对同一基因组序列进行消化,产生不同的代表性序列,然后对此序列进行连接子介导的PCR,之后将此DNA样本在基因组芯片上进行共杂交分析或测序分析。但此方法受限于甲基化敏感性限制性核酸内切酶,对甲基化序列的识别具有局限性,如采用HpaⅡ仅能分析基因组内8%的CpG岛[11]。
2.1.2 MSCC
甲基化敏感酶切计数测序法(methyl-sensitive cut counting, MSCC)是基于全基因组的DNA甲基化测序方法,利用基因组内全部CCGG位点对HpaⅡ酶的敏感性来进行甲基化位点测定,能被HpaⅡ酶识别的位点均能被检测到,并且该方法不局限于使用HpaⅡ一种酶。MSCC有利于从高甲基化事件中辨别中甲基化事件,且测序深度越深,精确度越高[12]。但是MSCC在酶切过程中会计入较多杂位点,杂位点会影响甲基化位点的统计,且杂位点不易被排除。
2.2 亲和富集法
2.2.1 MeDIP-seq
甲基化DNA免疫共沉淀测序法(methylated DNA immunoprecipitation sequencing, MeDIP-seq),是通过使用5-甲基胞嘧啶抗体富集高甲基化的DNA片段,然后对全基因组中DNA高甲基化区域进行高通量测序的方法[13]。此方法成本低、数据处理简单且对高度甲基化区域敏感性高。但在实际应用过程中却存在一些问题,如经免疫共沉淀后得到的MeDIP产物产量较低难以达到测序建库的标准,且反应过后双链DNA会变为单链DNA,而单链DNA并不适合测序文库的构建。采用该方法进行甲基化水平测定时,推荐增添一个成本较低的互补实验即甲基化敏感限制性内切酶测序(methylation-sensitive restriction enzyme sequencing, MRE-seq)构成M&M方法(MeDIP-seq和MRE-seq);相较单一测序,M&M具有更高的准确性和重复性[14]。许多研究者采用MeDIP-seq研究了多种疾病的全基因组DNA甲基化水平,如王汨[15]通过比较伴童年创伤抑郁症患者、不伴童年创伤抑郁症患者和健康人之间的甲基化水平发现,各对照组间NR3C1基因内含子区的DNA甲基化水平均有改变,推测这些区域的甲基化的改变与童年创伤、抑郁症有一定的相关性。
2.2.2 MBD-seq
甲基结合蛋白测序法(methyl-CpG binding domain protein-enriched genome sequencing, MBD-seq)检测全基因组DNA甲基化的原理与MeDIP-seq基本相同,区别在于该方法用MBD2b蛋白富集甲基化DNA片段来取代MeDIP-seq的甲基化胞嘧啶抗体富集,对富集到的DNA片段进行高通量测序从而检测全基因组范围内的甲基化位点,对高甲基化位点更敏感。Frattini等[16]利用MBD-seq研究了山羊在全基因组水平下的下丘脑和卵巢的DNA甲基化状态,并绘制了山羊的甲基化图谱。Neary等[17]利用MeDIP-seq和MBD-seq两种方法对患精神疾病的小鼠进行甲基化测序,发现MeDIP-seq比MBD-seq能识别更多的差异甲基化区域(differentially methylated regions, DMRs),且小鼠基因组的甲基化改变影响其基因的表达。

图 1 3类DNA甲基化测序技术流程图Fig 1 Workflow of three DNA methylation sequencing technologies
2.2.3 MethylCap-seq
甲基化DNA捕捉测序法(methylated DNA capture by affinity purification, MethylCap-seq)是一项基于捕捉带MeCP2基团MBD从而进行甲基化测序的技术[18]。该技术由两部分组成,一个是通过携带mCpG的MBD来捕捉甲基化DNA片段,另一个是对洗脱后的DNA进行测序。梯度洗脱可以将不同甲基化状态的基因分成不同的部分,从而高效率地洗脱出甲基化DNA片段。洗脱后的DNA基因组按mCpG密度分层分布在不同部位,相比全基因组来说降低了复杂性。用该方法可以得到详细的全基因甲基化区域图谱且可以检测不同基因区域的DNA甲基化水平。Meyer等[19]利用MethylCap-seq对70个人类大脑组织进行了研究,发现比较之前的测序方法,MethylCap-seq能识别更多甲基化区域且敏感性较高。但MethylCap-seq也存在不足之处,即有限的测序覆盖度,无法满足高覆盖度测序实验的要求。
2.3 重亚硫酸盐转化法
2.3.1 WGBS
全基因组重亚硫酸盐测序法(whole genome bisulfite sequencing, WGBS)是前期用重亚硫酸盐(bisulfite)处理,将基因组中未发生甲基化的C碱基转换成U碱基,进行PCR扩增后变成T碱基,与原本具有甲基化修饰的C碱基区分开来,再对PCR产物进行高通量测序的方法。WGBS在1992年由Frommer等[20]提出,被公认为一项革命性的创新,已成为绘制单碱基DNA甲基化图谱的首选方法。该方法因其通量高、耗时少、单碱基分辨率、高覆盖率的优势被称为甲基化测序的“黄金标准”,避免了酶切不完全可能导致的假阳性问题,且敏感性较高,仅需1 ng 的DNA量即可进行建库检测。此外,WGBS还具有精准性高等优点,每个胞嘧啶的甲基化程度都能被精密地定量检测出来。但也存在一些缺陷,WGBS测序价格昂贵且数据量庞大,在多样品研究中,WGBS在单碱基分辨率下无法分辨5-羟甲基胞嘧啶(5hmC)和5-甲基胞嘧啶(5mC),对5mC数量统计存在一定影响。测序完成后需要高级计算分析来确定DNA甲基化模式和内在发生的改变,被限制于比较样品间个体的CpG位点。WGBS虽然降低了序列的复杂度,却未能解释产生不完全转化和序列错误的问题。在实际应用中,例如,Zhang等[21]利用WGBS方法对小鼠脑组织进行甲基化测序,寻找到新的甲基化位点可用于抵抗未来威胁人类健康的疾病。此外,Zhang等[22]对多产和低产湖羊的卵巢组织进行WGBS测序,描绘了湖羊卵巢组织的全基因组甲基化图谱。
2.3.2 RRBS
简化基因组重亚硫酸盐测序(reduced representation bisulfite sequencing, RRBS)是结合新一代测序的重亚硫酸盐转化和限制性内切酶消化的方法,可有效检测单碱基分辨率的DNA甲基化水平,与WGBS相比分辨率相当但费用较低。RRBS富集高CpG的基因区域,可以减少样品DNA的需求量,优化了未甲基化胞嘧啶的转化,使重亚硫酸盐处理过程中的DNA损耗降到最低[23],为定量分析DNA甲基化提供了高敏感性的测定方法。RRBS减小了由不完全胞嘧啶转化为尿嘧啶或PCR或测序错误造成的假阳性甲基胞嘧啶出现的可能性,建库时仅需要非常小的片段(500~600 bp),是因为在重亚硫酸盐反应时温度很高且pH值很低,会造成脱嘌呤和双链断裂,小片段更能减少PCR过程中的损伤。由于WGBS价格昂贵且数据量庞大,RRBS是一种可用于替代WGBS的方法。RRBS已被用于重要基因区域的高覆盖度、高灵敏度的测序,如Aniruddha等[24]利用RRBS描绘了斑马鱼大脑的甲基化图谱,此外Korkmaz等[25]也对不同年龄牛的纤维母细胞进行RRBS测序后发现免疫相关的甲基化位点可能与不同的脂多糖应答反应有关。另一方面,因为RRBS结合了重亚硫酸盐转化和限制性内切酶消化,它同时也具有二者的缺点。同理,RRBS无法分辨5hmC与5mC,也无法解释不完全转化与序列错误的问题。
2.3.3 oxBS-seq
WGBS和RRBS在单碱基分辨率下无法分辨5hmC和5mC,5hmC是5mC脱甲基成胞嘧啶过程的中间产物,在某些哺乳动物细胞和组织中丰度较高,而采用Booth等[26]提出的氧化-重亚硫酸盐测序法(oxidative bisulfite sequencing, oxBS-seq),将DNA样品5hmC读为胸腺嘧啶(T),而5mC仍被读为胞嘧啶(C),随后再将经过氧化处理和未处理的样本进行测序比较,即可在单碱基分辨率下分辨5hmC和5mC。换言之,oxBS-seq是改良升级版的WGBS,继承了WGBS的优点的同时还弥补了其缺点,但该方法本身也存在一定的局限性,如因氧化条件会对基因组DNA造成严重的降解和损伤。为了降低DNA损伤和降解对测序的影响,一般要求DNA建库量在100 ng以上。实际应用中,Booth等[26]通过oxBS-seq初次构建了小鼠胚胎干细胞CpG富集区域5hmC和5mC单分辨率的甲基化图谱。Hernandez Mora等[27]发现人类胎盘和大脑的5hmC可能与印记位点转录相关。
2.4 测序技术综合比较
以上测序方法都为广泛应用于高通量表观遗传学研究中的技术手段,上述介绍着重于各技术核心。图1从样本处理、富集建库、软件分析等方面简要总结了3大类测序技术的工作流程。此工作流程图利用不同类型箭头展示不同的DNA甲基化高通量测序步骤,浅蓝色注释框与注释圈表示可使用的生物信息软件。在选择合适的测序技术时,还需要考虑该方法的覆盖度、灵敏度、分辨率、特异性和费用等是否符合实验目的要求,为更简洁明了地展示各方法的优点与缺点,采用5等级分段法对以上技术手段进行综合比较(表1),以期为选择最适测序手段提供参考依据。
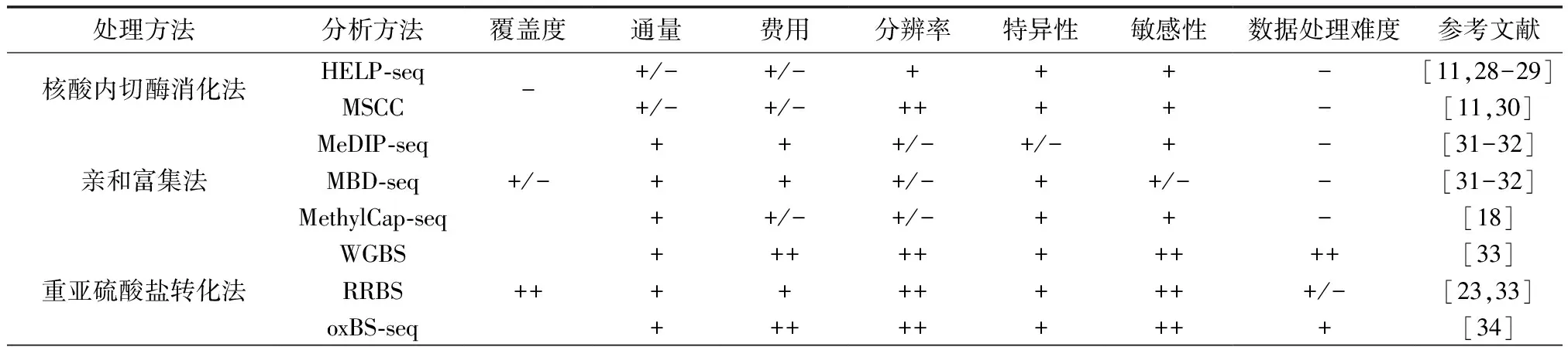
表1 不同测序方法的比较
注:测序技术各指标分为5等级,依次++>+>+/->->--
3 小结及展望
近年来,DNA甲基化已成为各领域研究的热点,尤其在家畜育种和人类疾病临床应用方面具有十分重要的意义。基于NGS检测全基因组DNA甲基化的方法通常分为核酸内切酶消化法、亲和富集法、重亚硫酸盐转化法3类,每种方法各有其优缺点,但从理论、方法、测序平台等方面来看,检测DNA甲基化的测序技术一直在不断地改进和完善。核酸内切酶消化法仅能识别一部分的甲基化位点,且不能识别出甲基化位点在染色体上的具体位置。亲和富集法成本较低,检测物种范围较广,但达不到单碱基分辨率,且只对高、中水平甲基化敏感,难以检测低水平甲基化位点。重亚硫酸盐转化法具有高敏感、高覆盖度、高分辨率的优势,使之成为甲基化位点检测的首选方法,但高昂的价格、庞大的数据量也限制了它的广泛应用。就目前而言,在实际应用过程中,为了获取更加准确可靠的甲基化数据,可联合不同的分析方法,或选择合适的方法后进行技术方面的微调来达到实验的目的。测序只是实验中的一个重要步骤,后续的数据分析也同测序技术一样重要,有必要结合其他分子生物学技术,开发或改进生物信息分析工具,共同分析解读甲基化数据,来挖掘数据背后的信息。相信不久的将来,测序成本会大幅度降低,高通量甲基化测序方法会得到广泛的应用,也会开发出更多新型甲基化检测技术及分析方法,为动物遗传育种、人类疾病治疗等领域提供理论依据与技术支撑。
