癌症 TCGA 数据库中乳腺癌预后数据的挖掘
2018-08-20MianKhizarHayat王铭裕李硕磊
Mian Khizar Hayat, 王铭裕, 李硕磊
(兰州大学 生命科学学院 生物物理所, 兰州 730000)
乳腺癌是危害女性身心健康的最主要的恶性肿瘤,男性乳腺癌患者比较少见,Cancer Statistics 在 2017 年的统计数据显示乳腺癌在女性癌症发病中占据了 30%的比例[1]。近年来, 乳腺癌的发病率逐年上升,并且年轻化趋势明显[2]。根据世界卫生组织国际癌症研究中心(IARC)最新的全球肿瘤流行病统计数据,乳腺癌仍是欠发达国家女性因肿瘤引起死亡的首要原因[3]。乳腺癌是由多基因突变、多蛋白相互作用,并结合遗传因素和环境因素等多方面原因引发的,其分子病理机制尚未完全揭示。Nikzainal等分析了 560 例乳腺癌病人的全基因组数据,发现了携带 93 个编码蛋白的癌基因可能驱动乳腺癌生成,而且其研究对象集中在体细胞突变基因,但也不否定可能存在非高频突变基因驱动癌症的发生发展[4],而且预后是疾病治疗的一个重要指标。因此,我们利用 TCGA 数据库中有关于乳腺癌的数据,运用生物信息学的手段,从乳腺癌预后的相关基因来进行探究。
1 材料与方法
1.1 数据库
为了保证癌症组织信息与正常组织信息是在病人的同一时期获得的,我们在癌症基因图谱(The Cancer Genomes Atlas, TCGA)数据库中选取 113 对同时检测癌区和癌旁正常组织的样品,调取其转录组数据(RVAseqV2,raw count),这样就排除了个体癌组织与正常组织取样时间的差异。其病理诊断与人种等信息见表 1。

表1 113 位病人的性别、确诊年龄、病理诊断和人种等基本信息Table 1 The basic information of gender, age at diagnosis, histological type and race of the selected 113 patients
1.2 筛选差异表达基因
对调取的样本转录组数据通过 R 语言 DESeq 包来进行差异表达分析,通过Padj<0.05并且Abs(log2fold change)>1, 来筛选差异表达基因。
1.3 乳腺癌相关基因的选择
利用 R 语言 clusterProfiler 包对差异基因进行基因本体 GO(包括生物过程 biological process、细胞组分 cellular component 和分子功能 molecular function 3个方面)和代谢通路KEGG 富集分析。同时,利用 DOSE 对差异基因进行疾病本体 DO(Disease Ontology)富集分析(Enrichment Analysis)。其中 GO 富集我们取P<0.01,KEGG 富集分析和 DO 富集分析我们取P<0.05。
1.4 分析乳腺癌相关基因对预后的影响
利用 R 语言 Survival 包,采用数据库中所用癌症的表达数据(共 1097 例)对这些乳腺癌相关基因进行生存分析。
1.5 分析筛选出的 8 个基因与临床乳腺癌标志物 ER、PR 和 HER2 的关系
我们调取了 113 位病人的上述 8 个基因的表达数据,并对照每个病人 ER、PR 和 HER2 的免疫组化结果,依据 ER、PR 和 HER2 免疫组化为阳性、阴性、三阳性和三阴性时,分析每个基因的表达水平与 ER、PR 和 HER2 的相关性。
2 结果
2.1 正常组织与乳腺癌中的差异表达基因
我们检测了 113 对乳腺癌及其对应正常癌旁组织,分析了乳腺癌的分期情况并检测了3种乳腺癌重要的已知突变基因(her2,er 和 pr 基因)。并从其转录组数据库中分析出差异表达基因,共得到 1428 个差异表达基因,结果如图 1 所示。
2.2 从生物过程、细胞组分和分子功能3个方面分析等显出的1428个差异表达基因
利用R语言clusterProfiler包对差异基因进行基因本体GO分析,分别包括生物过程biological process分析、细胞组分 cellular component 分析和分子功能 molecular function 分析(见图2)。
从 GO 的生物过程分析,我们可以看到这些差异表达基因主要集中在细胞过程,单一器官过程和单一器官细胞过程。而从细胞组分和分子功能两方面的分析可以看到,差异表达基因主要集中在蛋白结合方面。
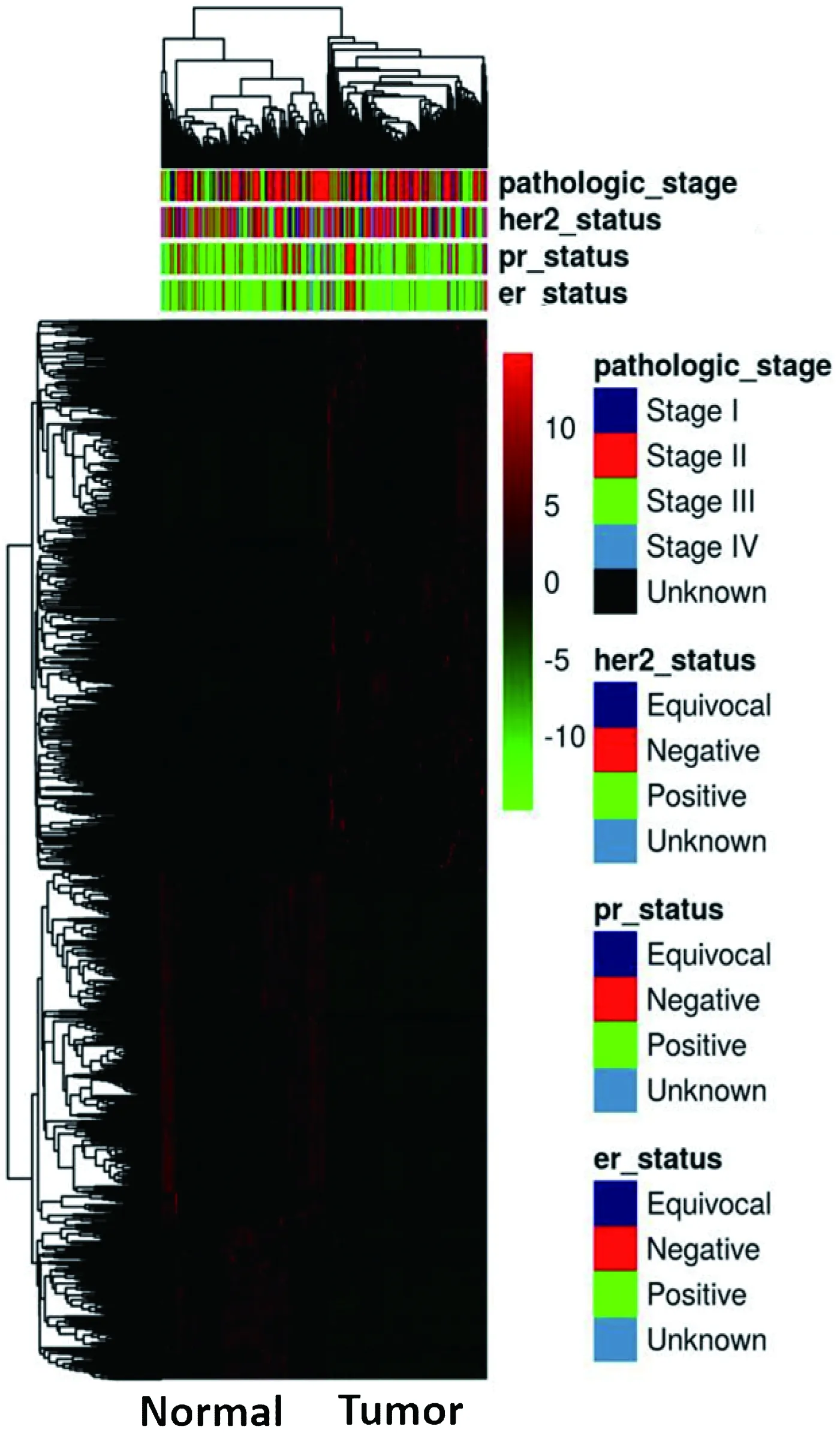
图1 113 对乳腺癌区和癌旁正常组织样品基因差异表达图谱Fig 1 Differential expression genes in 113 pairs breast cancer and its normal tissue adjacent to carcinoma
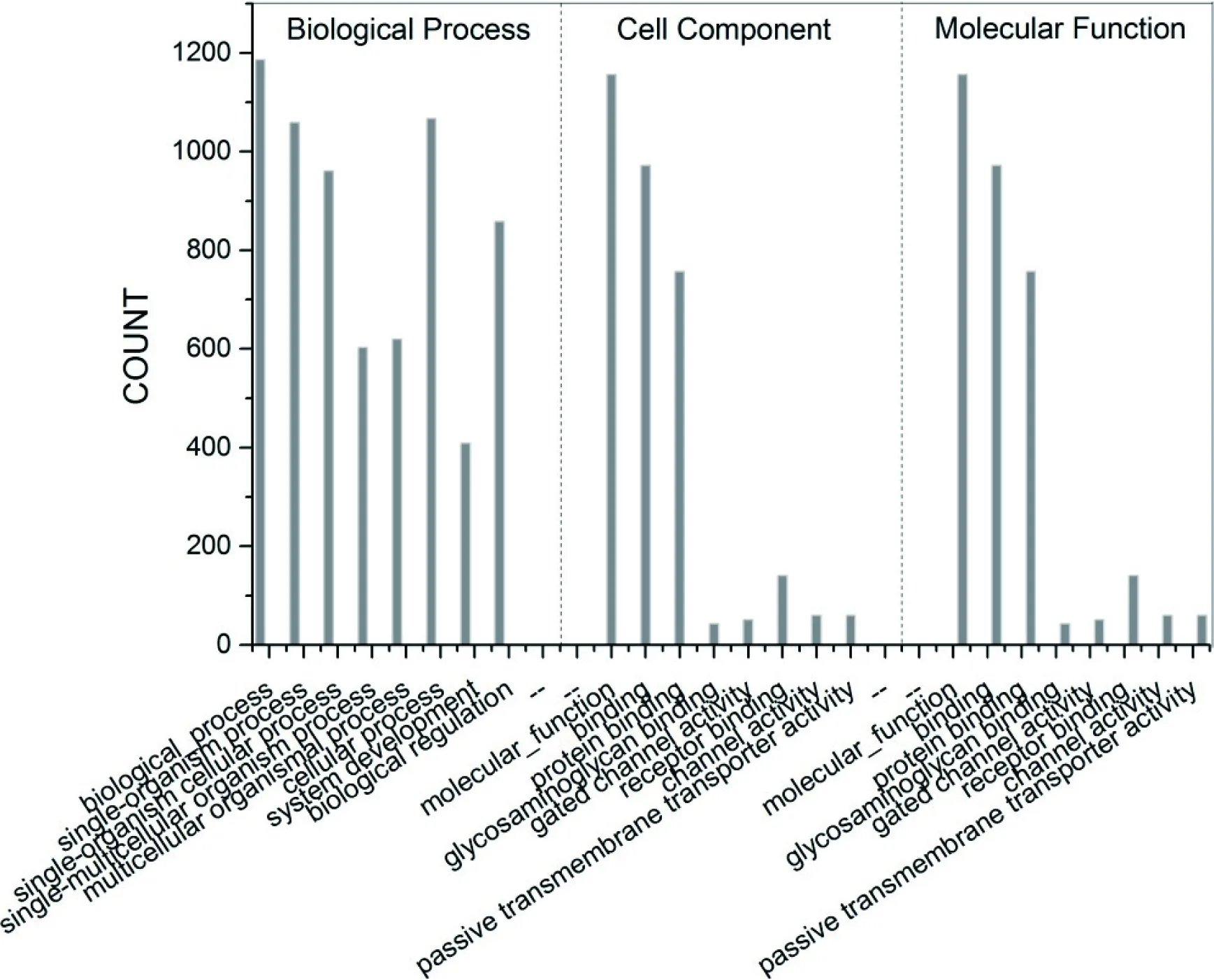
图2 基因本体 GO 分析的细胞组分分析,生物过程分析和分子功能分析Fig 2 Gene Ontology analysis of biological process, cell component and molecular function
2.3 差异表达基因在细胞周期和信号通路中的位置
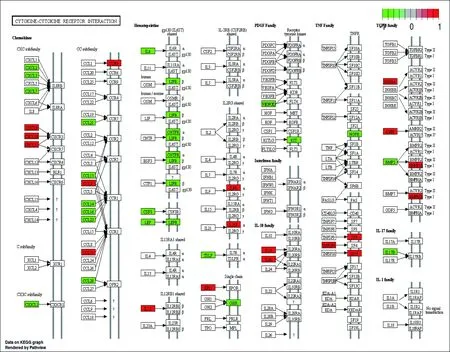
图3 差异基因细胞因子-受体互作图Fig 3 Cytokine-cytokine receptor interaction net
红色代表上调基因;绿色代表下调基因
从上面的结果我们推测这些差异基因可能主要通过细胞信号通路和细胞周期来起作用, 因此我们进行了代谢通路 KEGG 富集分析,以期能够了解差异表达显著的基因在细胞信号通路中所处的位置,并了解这些差异基因与哪些基因或转录因子发生作用有利于更好地揭示乳腺癌的发病机制。细胞信号通路结果如图 3 所示,细胞周期分析结果如图 4 所示。
红色代表上调基因;绿色代表下调基因
从分析结果看来,差异基因是通过CXC亚家族、CC亚家族、PDGF家族、TNF家族、IL-10家族和TGF-β家族等参与信号通路调节进而影响乳腺癌的发生发展。在细胞周期层面上看,差异基因参与了 G1 期、S 期、G2 期和 M 期整个细胞周期过程,提示差异基因可能通过影响细胞周期来影响乳腺癌的发生发展过程。
2.4 乳腺癌中主要差异表达基因的分析
通过上面的KEGG分析,我们找到了关于细胞周期和信号通路方面的差异基因,接下来我们通过疾病本体 DO 分析找到了与乳腺癌相关联的差异基因。我们选取显著性前 15 的类群(包含 68 个差异表达基因)做散点图,并对这 68 个基因做差异表达图谱(Heatmap),结果如图 5 所示。
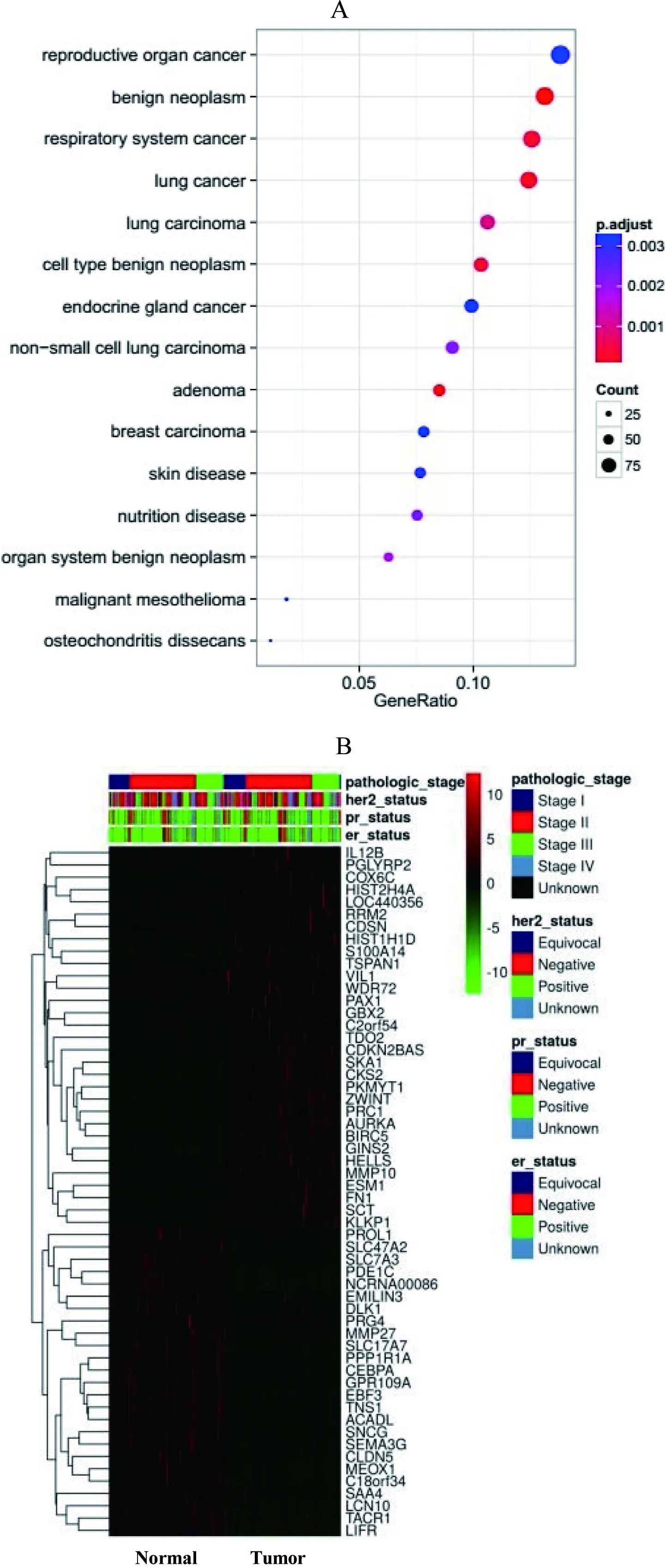
图5 A代表关于 DO 富集显著性前15的类群散点图;B代表经富集分析筛选的68个差异表达显著基因的差异表达图谱Fig 5 A represent disease Ontology of top 15 significance disease; B shows 68 differential expression genes after DO analysis
2.5 生存分析得到 8 个预后关键基因
通过上述DO分析,我们得到了68个差异显著基因,并对这68个基因进行了OS(Overall Survival)生存分析,从而得到了8个显著性的关键基因,PGLYRP2、SEMA3G、PROL1、SLC7A3、SKA1、BIRC5、RRM2和AURKA。如图 6 所示。
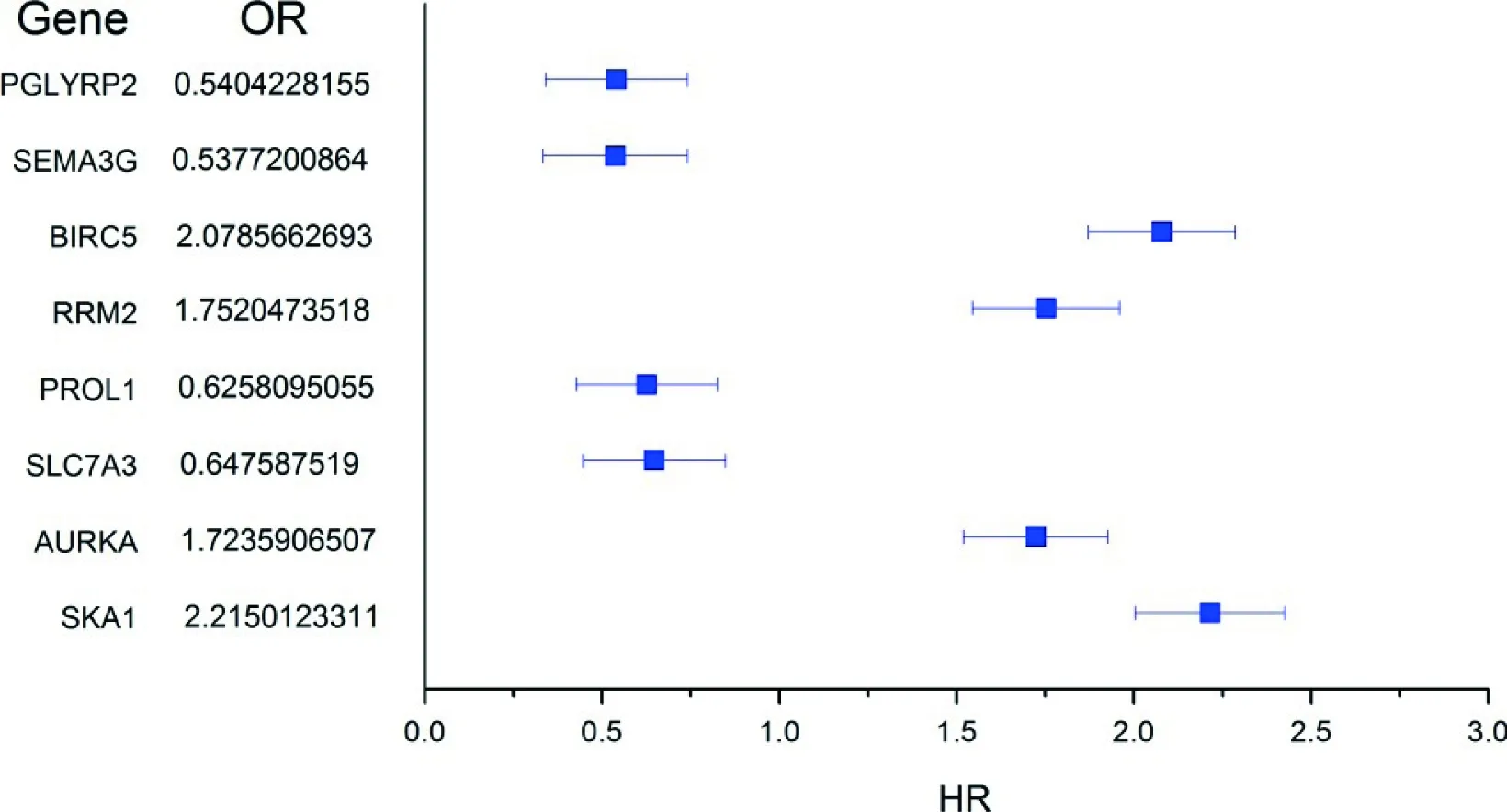
图6 8 个显著差异基因的生存分析结果Fig 6 Survival analysis of 8 significance differential expression genes
从图6中可以看到乳腺癌病人 PGLYRP2、SEMA3G、PROL1及SLC7A3 的高表达能够起到良好预后的作用。而 SKA1、BIRC5、RRM2和AURKA 基因的高表达反而预示着预后不良。

图 7 预后不良相关的 4 个基因在 ER、PR 和 HER2 检测为阳性和阴性时的表达水平Fig 7 The expression levels of 4 poor prognosis-associated genes in ER,PR and HER2 of positive and negative
“N”代表阴性;“P”代表阳性;“Tri-N”代表三阴性;“Tri-P”代表三阳性
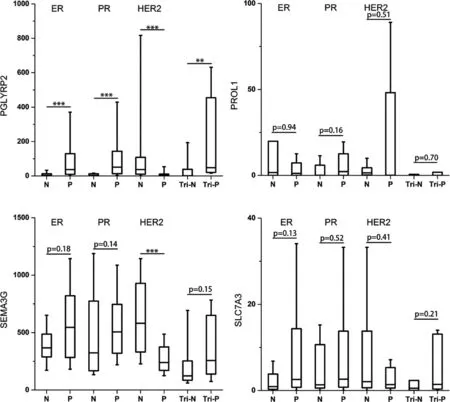
图 8 预后良好相关4基因在 ER、PR 和 HER2 检测为阳性和阴性时的表达水平Fig 8 The expression levels of 4 good prognosis-associated genes in ER,PR and HER2 of positive and negative
“N”代表阴性;“P”代表阳性;“Tri-N”代表三阴性;“Tri-P”代表三阳性
2.6 8个预后相关基因与临床乳腺癌标志物 ER、PR 和 HER2 的关系
我们按照良好预后和不良预后将上述 8 个基因分为两组,以 ER、PR 和 HER2 表达阳性和阴性,三阴性和三阳性为横坐标,分析预后良好相关基因和预后不良相关基因与 ER、PR 和 HER2 的相关性。从分析结果来看,在预后不良组中,AURKA和RRM2 在三阴性和三阳性时,其表达水平并没有明显的相关性,而BIRC5 和SKA1 在三阴性和三阳性时,其表达水平具有明显的相关性,提示三阴性和三阳性条件下,BIRC5 和SKA1 具有更好的预后指导意义(见图7)。在良好预后组中,PROL1、SEMA3G和SLC7A3 在三阳性和三阴性条件下,并没有明显的相关性,而PGLYRP2 在三阳性和三阴性时具有明显的相关性,而且在 ER、PR 和 HER2 单阳性和单阴性时也具有明显的相关性,提示 PGLYRP2 具有更好的预后指导意义(见图8)。
3 讨论
PGLYRP2 是一种 N-乙酰胞壁酸-L-丙氨酸酰胺酶, 它能够将细菌细胞壁肽聚糖中MurNAc 和 L-Ala 之间的共价键水解从而起到抗菌的作用[5]。人 PGLYRP-2 主要在肝脏中表达, 并且可以分泌到血液中,通过血液循环遍布全身来建立先天免疫系统[6]。有研究结果表示,肝脏和血液中的 PGLYRP2 均有基因 pglyrp2 基因编码,而且 PGLYRP2 也能够调控小鼠脑组织发育[7]。目前,对 PGLYP2 的研究主要集中在其作为一种 PGLRYs 蛋白在先天免疫系统中防止病原体侵染宿主细胞的分子机制这一方向,而 PGLYRP2 蛋白与在乳腺癌的发生过程中起着怎样的作用,需要我们进一步深入地研究。SEMA3G属于信号素(Semaphorin)家族,这类分子在神经系统外还起着调节细胞的增殖、黏附和迁移的作用,更重要的是发现其对肿瘤的生长、迁移、免疫反应、血管生成起重要调节作用[8]。SEMA3C 可通过激活整合素蛋白磷酸化和 VEGF120 的分泌来刺激胃癌血管生成并增强体外内皮细胞的黏附性;Sema3E 可以与 PlexinD1 直接结合;SEMA3E/PlexinD1 信号通过启动 R-Ras 失活影响整合素的活化状态和激活 ADP-核糖激化因子6(Arf6)促进细胞内物质运输来发挥促肿瘤血管生成作用。SEMA3A 可以通过增加血管通透性来抗肿瘤血管生成;抑制整合素激活,阻断整合素介导的内皮细胞的迁移和黏附;同时还可以阻断下游 PlexinA4 受体对肿瘤的促进作用。SEMA3B 具有肿瘤细胞的恶性增殖和肿瘤血管生成的抑制作用;SEMA3F 能够抑制 VEGF 和 FGF 介导的 ERK1/2 的激活和下调 NP2 水平以抑制体外内皮细胞的增殖[9]。SEMA3G 对于肿瘤的发生与抑制起着怎样的作用尚未阐明,鉴于 SEMA3G 家族其他成员与肿瘤发生与抑制起着非常重要的作用,以及参考挖掘 TCGA数据库中乳腺癌相关的数据,我们大胆推断SEMA3G一定与乳腺癌有着紧密的联系,相信不久便有进一步的实验验证。PROL1属于我们筛选的乳腺癌差异表达基因,其研究尚不深入;SLC7A3是溶质转运蛋白家族的一员,但是在SLC7A3 敲除小鼠中会阻碍 AMPK-PPAR-alpha 信号通路,并且会在葡萄糖短缺时导致脂质累积[10]。
SKA1 基因编码的蛋白是纺锤体与动粒相关复合体(spindle and kinetochore complex,SKA)的亚基之一,其余 SKA2 和 SKA3 编码的蛋白亚基共同组成纺锤体与动粒相关复合体。SKA 能够促进微管蛋白和动粒的稳定结合,调控微管蛋白的解聚和真核生物有丝分裂过程中染色体向两端的移动[11-12]。已有研究表明,SKA1 的沉默能够抑制神经胶质细胞瘤、肝癌、胃癌、口腔鳞状细胞癌等多种肿瘤细胞的恶性增殖[13],但 SKA1 是否在调节乳腺肿瘤细胞恶性增殖的一个关键因子,需要进一步的验证。RRM2基因编码的蛋白是核糖核苷酸还原酶(Ribonucleotide reductase, RR)的小亚基单位 RRM2。核糖核苷酸还原酶在 DNA 的合成、修复和细胞增殖过程中起着关键的调控作用。已有大量研究表明,RRM2 基因在胰腺癌、胃癌、绒癌、膀胱癌、直肠癌、乳腺癌等多种人体恶性肿瘤细胞中表达水平异常升高,降低RRM2的表达水平有利于降低核糖核苷酸还原酶的活性,加速恶性肿瘤细胞的凋亡[14]。RRM2 被认为是癌症治疗的重要靶点,以 RRM2 作为抗肿瘤药物的靶点有研发出多种药物:3—AP、RRM2 的小干扰 RNA 等[15]。这也就验证了 RRM2 在乳腺癌等其他癌症的发生过程中起着重要的作用,但是,进一步深入研究 RRM2 有利于完整地在揭示乳腺癌的分子病理机制,为乳腺癌的治疗提供一个新的策略。AURKA 基因编码丝氨酸/苏氨酸激酶,属于 Aurora 激酶家族。AURKA 参与中心体的复制、分离和成熟,在真核生物有丝分裂的细胞周期中起着重要作用。AURKA 的表达异常往往导致染色体的异倍性,由此导致的基因组的不稳定增加了基因突变的频率,被认为是恶性肿瘤发生的重要原因之一。同时,异常表达的 AURKA 还能参与到细胞内各种信号通路,直接或间接的促进肿瘤的发生[16]。已有大量研究显示,乳腺癌中AURKA表达水平异常升高。不仅在乳腺癌中,AURKA和SKA3,DSN1 过表达之后也会诱导结直肠癌的发展[17],近期也有研究发现 AURKA 在三阴性乳腺癌模拟血管生成过程也参与了进来[18],而且 AURKA 也参与维持乳腺癌肿瘤干细胞形态[19]。BIRC5(survivin)属于凋亡抑制蛋白(inhibitor of apoptosis, IAP)家族,具有抑制细胞凋亡、促进细胞转化、参与真核细胞有丝分裂、血管生成以及耐药性的产生等生物学功能。抑癌基因Tp53 和 BIRC5 有着密切的关系,Tp53 对 BIRC5 具有负调节作用。当野生型的抑癌基因 Tp53突变时,就会解除在 mRNA 和蛋白质水平对 BIRC5 的抑制作用,导致 BIRC5 异常高表达。高表达的 BIRC5 启动抗细胞凋亡机制抑制细胞凋亡,从而导致细胞恶性增殖形成恶性肿瘤, 并可增强肿瘤细胞的耐药性[20-22]。因此,BIRC5 与乳腺癌的发生也应该有着密切的关系。
通过调研分析发现,RRM2和AURKA基因参与乳腺癌的发生过程;SKA1和BIRC5的异常表达会导致恶性肿瘤的产生;SEMA3G也与肿瘤的生成促进作用或肿瘤生成的抑制作用有关。一方面,验证了TCGA数据库数据挖掘部分结果;另一方面,增加了结果的可靠性。但是这8个基因PGLYRP2、SEMA3G、PROL1、SLC7A3、SKA1、BIRC5、RRM2和AURKA在乳腺癌预后中怎样发挥作用,仍需结合大量临床样本的检测数据和体内外功能性实验来验证和研究,以期在基因调控水平上达到预后良好的结果。
