实时荧光PCR法检测食品中亚利桑那沙门氏菌
2018-07-09王青龙蔡雪凤周艳霞张跃川朱晓龙巩有博耿健强何湘漪
王青龙,蔡雪凤,周艳霞,曹 悦,张跃川,朱晓龙,巩有博,耿健强,何湘漪
(北京市食品安全监控和风险评估中心(北京市食品检验所),北京 100094)
沙门氏菌(Salmonella)是食品检测过程中最常见的食源性致病菌之一,其在世界范围内引起的食物中毒病例也常常排在首位[1-3]。据报道,美国每年沙门氏菌感染的病例就超过40 000例[3-4]。根据世界卫生组织的相关报道,全球每年伤寒感染的人超过1 600万,其中50万~60万人会病死[4]。在我国,每年发生的食源性细菌中毒事件中,由沙门氏菌引起的食物中毒案例经常是排在第一位,约占总比例的40%[5]。
目前,根据有关文献报道已经确认的沙门氏菌的血清型就有2 500多种,在我国已经发现了200多种沙门氏菌的血清型[6-8],其中能够感染人类致病的菌株主要有伤寒沙门氏菌、肠炎沙门氏菌、鼠伤寒沙门氏菌、乙型副伤寒沙门氏菌、丙型副伤寒沙门氏菌、猪霍乱沙门氏菌、婴儿沙门氏菌和鸡沙门氏菌,目前也有文献报道亚利桑那沙门氏菌感染人类致病的案例[9-11]。引起人类感染沙门氏菌主要是由于食用了被污染的食品,感染沙门氏菌后一般会引起痢疾、胃炎、肠炎、伤寒等病症,严重的还会引起败血症[12-14]。目前,食品沙门氏菌的检测方法主要还是使用传统培养和生化鉴定的方法,该方法需进行选择性增菌培养、生化鉴定、血清分型等步骤,一般要5~6 d才能得出检测结果,不利于及时快速的进行诊断、病源的查找、疫情不能够及时得到控制。通过实验证明该方法对于亚利桑那沙门氏菌和沙门氏菌区分比较难,如果对于沙门氏菌的检测经验不足,往往会导致亚利桑那沙门氏菌会被漏检。因此,建立一种能够快速检测食品中沙门氏菌和亚利桑那沙门氏菌的方法是非常必要和急需的,对于以后沙门氏菌的快速检测也有着非常重要的意义。
本研究根据GenBank公布的亚利桑那沙门氏菌和其他沙门氏菌的gudD基因序列,通过序列比对设计引物和Taqman探针。通过种间特异性和不同属间的特异性实验来验证实时荧光聚合酶链式反应(real-time polymerase chain reaction,RT-PCR)扩增体系的特异性、对沙门氏菌检测的敏感性以及与传统检测方法结果的相符合性。以期为亚利桑那沙门氏菌的快速鉴定和检测提供支持。
1 材料与方法
1.1 材料与试剂
1.1.1 实验菌种
不同血清型标准菌株10株,样品菌株88株,其中亚利桑那沙门氏菌(Salmonella arizonae)3株;其他食源性致病菌等非沙门氏菌29株:分别来源于美国典型菌种保藏中心(American type culture collection,ATCC),中国医学微生物菌种保藏管理中心(national center for medical culture collection,CMCC),中国工业微生物菌种保藏管理中心(China center of industrial culture collection,CICC),中国普通微生物菌种保藏管理中心(China general microbiological culture collectioncenter,CGMCC)。分离菌株88株由本实验室分离保存。菌株信息见表1。上述菌株经过生化实验和血清学实验进行验证。
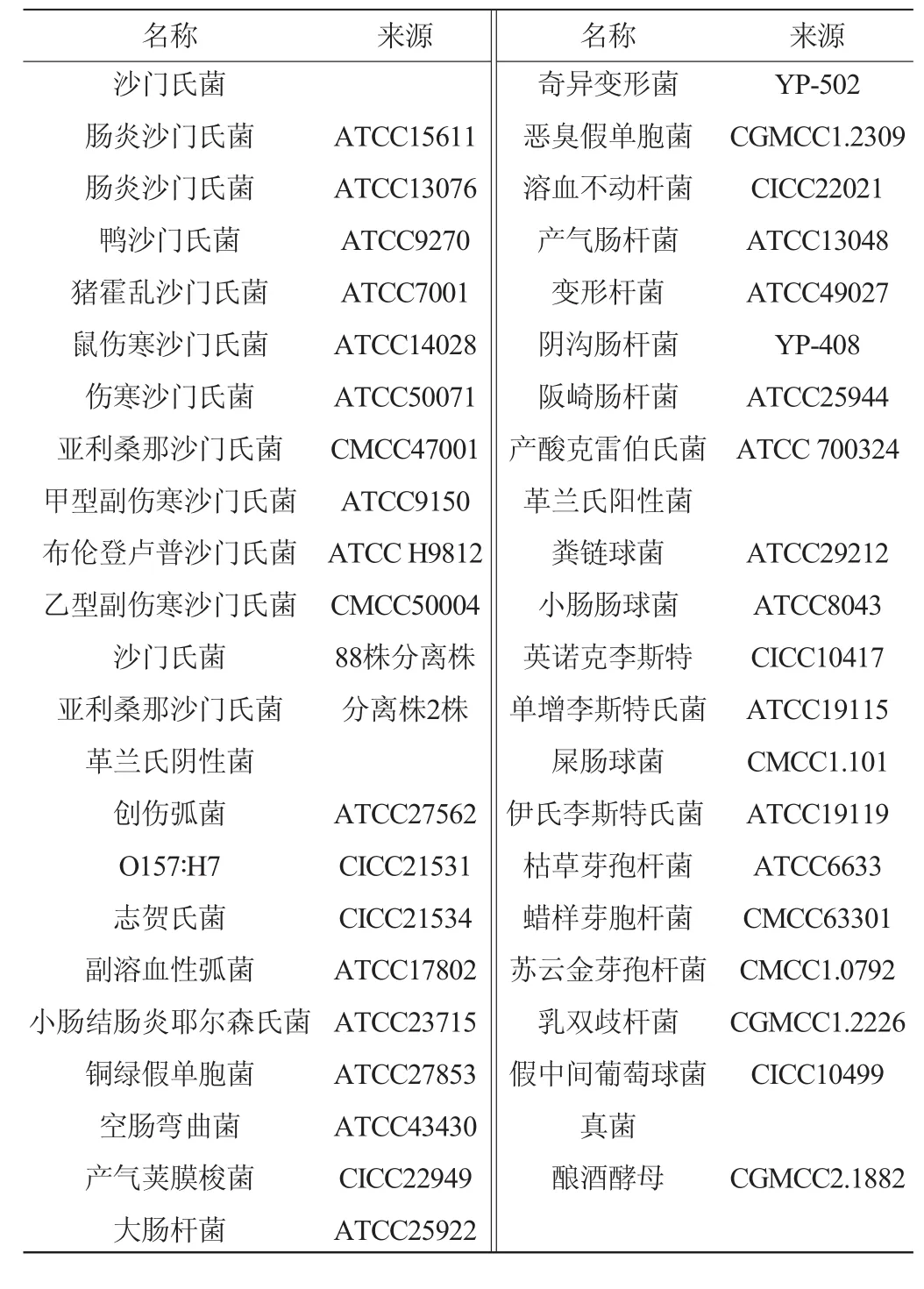
表1 实验用菌株信息Table 1 Information table of experimental strain
1.1.2 试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取液、Taq Man Master Mix:美国ABI公司;亚硒酸盐胱氨酸增菌液(selenitecystinebroth,SC增菌液)、缓冲蛋白胨水(buffered peptone water,BPW):北京陆桥有限公司。
1.2 仪器与设备
ABI QuantStudio 7实时荧光PCR仪:美国ABI公司;VITEKRCompact微生物分析系统:法国生物梅里埃公司。
1.3 方法
1.3.1 引物探针设计
从GenBank上亚利桑那沙门氏菌和其他沙门氏菌的gudD基因已经公布的序列,通过序列比对选择一段目标片段来设计引物和探针,使用该引物和探针实时荧光PCR扩增。引物探针用primer5软件设计,由上海生物工程有限公司合成。引物探针序列见表2。

表2 实时荧光PCR引物和探针序列Table 2 Primers and the probe sequence of real-time PCR
1.3.2 DNA提取和特异性试验
所有沙门氏菌菌株接种于BPW缓冲蛋白胨水,于37℃培养18~24 h,取1 mL接种于SC培养基37℃培养18~24 h。取1 mL于12 000 r/min离心5 min,去上清,用1 mL生理盐水悬浮,12 000 r/min离心5 min,去上清,重复上述步骤两次。最后用200 μL细菌裂解液悬浮,沸水浴5 min,冷却2 min,15 000 r/min离心10 min,取上清备用。其他非沙门氏菌菌株作为阴性对照菌株用相应的培养基过夜培养,取1 mL菌悬液转移至离心管,按照沙门氏菌相同的步骤提取DNA备用,所有菌株的DNA均用于特异性试验。
1.3.3 实时荧光PCR反应体系和参数
实时荧光PCR反应体系为25μL:2×MasterMix12.5μL、引物对(10 μmol/L)各1 μL、探针(10 μmol/L,0.5 μL、模板DNA2μL、去离子水8μL。实时荧光PCR反应参数为:50℃,2min,95℃预变性10min,94℃变性5s,60℃退火延伸40 s;同时收集FAM荧光,进行40个循环,4℃保存反应产物。
实时荧光PCR扩增曲线指数期明显,Ct<35可直接判定为阳性,Ct值为35~40判定为可疑,需要进行重复实验,Ct>40判定为阴性。
1.3.4 模拟样品检测和灵敏度试验
取确定为沙门氏菌阴性的样品(鸡肉、鸭肉),采用GB4789.4—2016《食品安全国家标准食品微生物学检验沙门氏菌检验》进行培养鉴定,确认无沙门氏菌污染。分别称取25g,每份样品中分别添加3个不同稀释浓度(100CFU/mL、101CFU/mL、102CFU/mL)的亚利桑那沙门氏菌和沙门氏菌的菌悬液(添加水作为阴性对照),制成14份模拟样品。每份样品中加入225mLBPW缓冲蛋白胨水,用拍打器拍打2 min,37℃培养18~24 h,取1 mL移入9 mL SC增菌液,37℃培养18~24 h,然后取1 mL增菌液提取DNA用于实时荧光PCR扩增。同时,不同稀释浓度的模拟污染样品也用于实时PCR方法的检测灵敏性试验。所有模拟样品按照GB 4789.4—2016《食品安全国家标准食品微生物学检验沙门氏菌检验》,与实时荧光PCR方法同步进行培养鉴定。
2 结果与分析
2.1 亚利桑那沙门氏菌特异性检测
取表1中所列的3株亚利桑那沙门氏菌、9株沙门氏菌标准菌株的DNA进行实时荧光PCR扩增,结果如图1所示。由图1可知,3株亚利桑那沙门氏菌的DNA样本采用YF/YR引物和YP探针均扩增出阳性扩增曲线,而其他沙门氏菌DNA样本检测结果为阴性。结果表明,本试验设计的亚利桑那沙门氏菌的引物和探针特异性很好。

图1 亚利桑那沙门氏菌实时荧光PCR特异性扩增图谱Fig.1 Specific amplification pattern ofSalmonella arizonaeby real-time PCR
2.2 其他沙门氏菌特异性检测

图2 其他沙门氏菌实时荧光PCR特异性扩增图谱Fig.2 Specific amplification pattern of ordinarySalmonellaby real-time PCR
取表1中所列的9株沙门氏菌标准菌株、3株亚利桑那沙门氏菌的DNA进行实时荧光PCR扩增,结果如图2所示。由图2可知,9株沙门氏菌标准菌株采用SF/SR引物和SP探针均扩增出阳性扩增曲线,而亚利桑那沙门氏菌DNA样本检测结果为阴性。结果表明,本试验设计的沙门氏菌的引物和探针特异性很好,与亚利桑那沙门氏菌能够明显鉴别。
2.3 非沙门氏菌特异性实验结果
分别使用YF/YR引物YP探针和SF/SR引物SP探针对表1中29株非沙门氏菌的DNA进行实时荧光PCR扩增,结果如表3所示。由表3可知,本研究设计的亚利桑那沙门氏菌的YF/YR引物YP探针和其他沙门氏菌的SF/SR引物SP探针对非沙门氏菌均有非常好的特异性。因此本次设计的引物和探针能够用于以后沙门氏菌和亚利桑那沙门氏菌的食品检测。
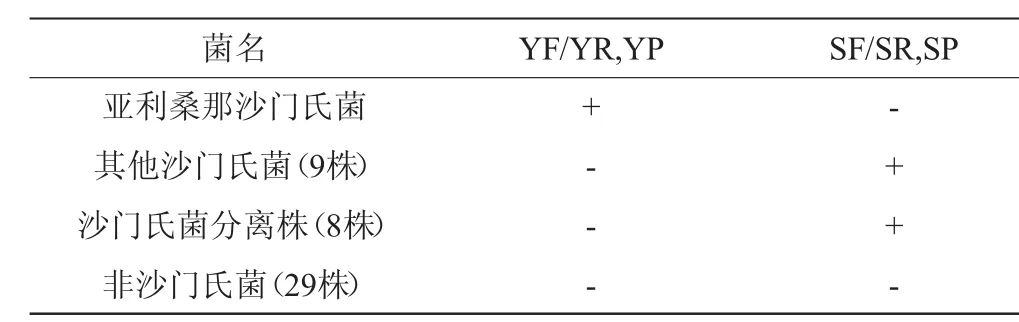
表3 非沙门氏菌特异性实验结果Table 3 Results of specific experiments of non-Salmonella
2.4 模拟样品检测和灵敏度试验结果
14份样品中分别添加3个不同稀释浓度(100CFU/mL、101CFU/mL、102CFU/mL)的亚利桑那沙门氏菌和沙门氏菌,与此同时,按照GB 4789.4—2016《食品安全国家标准食品微生物学检验沙门氏菌检验》检测模拟污染样品,结果见表4。
由表4可知,12份样品的亚利桑那沙门氏菌和沙门氏菌的检测结果均为阳性,不同添加浓度的灵敏度试验结果可见,模拟污染样品的实时PCR检测灵敏度可以达到1~10 CFU/mL的添加浓度。

表4 模拟样品检测和灵敏度试验结果Table 4 Results of simulated sample test and sensitivity test
3 结论
沙门氏菌的传统方法鉴定需要经过病原的选择性分离、生化鉴定、血清学分型等步骤来鉴定,但是在血清型实验过程中不同血清型之间容易交叉反应,从而会干扰血清型实验的准确性,因此在血清型检测的过程中往往会存在一定比例的假阳性结果。随着近些年分子生物学技术快速的发展,目前已经报道的分子方法中主要是针对16SrRNA、invB、invA、SpvC、fimA、SEFl4、agfA、ssaQ、sefA、sdf、fimY等[15-16]为目标基因,采用普通PCR或实时荧光定量PCR检测沙门氏菌,这些方法只是能够检测沙门氏菌的某个或者某些毒力基因,不能具体确定到血清型。
本文建立的TaqMan探针实时PCR检测食品中亚利桑那沙门氏菌和其他沙门氏菌,具有很好的特异性。实时荧光定量PCR扩增灵敏性试验结果表明,检测灵敏度可达到1~10 CFU/mL的添加浓度。经模拟样品验证,所建立的实时荧光定量PCR方法与传统方法的检测结果相一致,具有检测周期短、操作简便的优势,具有很好的应用前景。
[1]TATAVARTHY A,CANNONS A.Real-time PCR detection ofSalmonella species using a novel target:the outer membrane porin F gene(ompF)[J].Lett Appl Microbiol,2010,50(6):645-652.
[2]MANZANO M,COCOLIN L,ASTORI G,et al.Development of a PCR microplate-capture hybridization method for simple,fast and sensitive detection ofSalmonella serovarsin food[J].Mol Cell Probes,1998,12(4):227-234.
[3]YANG B,QU D,ZHANG X,et al.Prevalence and characterization of Salmonella serovars in retail meats of marketplace in Shaanxi,China[J].Int J Food Microbiol,2010,141(1-2):63-72.
[4]SIEMBIEDA J L,MILLER W A,BYRNE B A,et al.Zoonotic pathogens isolated from wild animals and environmental samples at two California wildlife hospitals[J].J Am Vet Med Assoc,2011,238(6):773-783.
[5]YANG B W,QU D,ZHANG X L,et al.Prevalence and characterization ofSalmonella serovarsin retail meats of marketplace in Shaanxi,China[J].Int J Food Microbiol,2010,141(1):63-72.
[6]HOORFAR J,LIND P,BELL M M,et al.Seroreactivity ofSalmonella-infected cattle herds against a fimbrial antigen in comparison with lipopolysaccharide antigens[J].J Vet Med,2010,43(1-10):461-467.
[7]LIU D,HAASE A M,LINDQVIST L,et al.Glycosyl transferases of O-antigen biosynthesis inSalmonella enterica:identification and characterization of transferase genes of groups B,C2,and E1[J].J Bacteriol,1993,175(11):3408.
[8]BARROW P A.ELISAs and the serological analysis ofSalmonellainfections in poultry:a review[J].Epidemiol Infect,1992,109(3):361-369.
[9]彭易根,张正群.亚利桑那沙门氏菌食物中毒72例临床分析[J].现代医药卫生,2011,27(4):571-572.
[10]李 明,利 娟.亚利桑那沙门氏菌引起胆管炎合并胆石症致败血症一例[J].中国临床研究,2004,17(1):6.
[11]刘 杰,黄淑华,陈 磊,等.开封市首次检出2株亚利桑那沙门菌的鉴定与分析[J].河南预防医学杂志,2013,24(4):261-262.
[12]SHI S A,PARK M T,CHOI H,et al.Salmonellabacteriophage and antibacterial composition comprising the same.US8329442[P].2012-11-12.
[13]COX N A,BERRANG M E,CASON J A.Salmonellapenetration of egg shells and proliferation in broiler hatching eggs--a review[J].Poult Sci,2000,79(11):1571-1574.
[14]VRIESEKOOP F,SHAW R.The Australian bush fly(Musca vetustissima)as a potential vector in the transmission of foodborne pathogens at outdoor eateries[J].Foodborne Path Dis,2010,7(3):275.
[15]TURCOTTE C,WOODWARD M J.Cloning,DNA nucleotide sequence and distribution of the gene encoding the SEF14 fimbrial antigen of Salmonella enteritidis[J].J Gen Microbiol,1993,139(7):1477.
[16]GUO X,CHEN J,BEUCHAT L R,et al.PCR detection ofSalmonella entericaserotype Montevideo in and on raw tomatoes using primers derived from hilA[J].Appl Environ Microbiol,2000,66(12):5248-5252.
