miR-29a促进人乳腺癌MCF-7细胞体外迁移和侵袭的机制
2018-07-02汤婷婷朱琴华
吴 悠,汤婷婷,朱琴华,金 亮,潘 怡
(中国药科大学生命科学与技术学院,南京 210009)
乳腺癌是一种发生在乳腺上皮组织的恶性肿瘤,是全世界女性中发病率和死亡率最高的肿瘤。乳腺癌癌细胞转移造成的疾病是绝大多数乳腺癌患者死亡的根本原因[1],因此寻找抑制乳腺癌细胞转移的潜在药物十分必要。
微小核糖核酸(miRNA)是一类由内源基因编码的长度为18~24个核苷酸的非编码单链RNA分子。miRNA与乳腺癌的发生发展、侵袭与转移、血管生成、细胞周期、细胞凋亡等密切相关[2]。不同的miRNA在肿瘤中的表达情况不同,对肿瘤的发生及转移也起到不同的作用[3]。研究表明,miR-29a在乳腺癌组织中有所升高,并促进了乳腺癌细胞的增殖和转移[4-5]。因此,本研究考察miR-29a对乳腺癌转移的生物学作用,研究miR-29a调控乳腺癌转移的新机制,为抗乳腺癌的药物筛选提供了实验依据和新思路。
1 材 料
1.1 试 剂
DMEM高糖培养基、胎牛血清(美国Gibco公司);Lipofectamine 2000、Trizol试剂(美国Invitrogen公司);苯甲基磺酰氟(PMSF)、RIPA裂解液、BCA蛋白定量试剂盒、胰蛋白酶、双荧光素酶报告基因检测试剂盒(上海碧云天生物技术有限公司);兔抗人GAPDH单克隆抗体、辣根过氧化物酶标记的羊抗兔IgG(美国CST公司);兔抗人HBP1多克隆抗体(美国SAB公司);miR-29a模拟物、miR-29a抑制剂、模拟物阴性对照、抑制剂阴性对照、miRNA实时荧光定量RT-PCR定量试剂盒(上海吉玛制药技术公司)。
1.2 仪 器
实时荧光定量PCR仪(美国Roche公司);酶标仪(美国Biotek公司);显微图象采集系统(日本Olympus公司)。
1.3 细 胞
人乳腺癌细胞株MCF-7、人乳腺癌细胞株MDA-MB-231、人胚肾细胞系HEK-293T由中国药科大学生物技术中心保存。
2 方 法
2.1 细胞培养
用含10%胎牛血清+1%青链霉素混合液的DMEM高糖培养基置于37 ℃、5% CO2培养箱中培养,待细胞融合率达90%以上时,用胰蛋白酶消化,按照1∶3的比例传代。将细胞以每孔2×105个的细胞比例种于6孔板,置于细胞培养箱中继续培养用于后续实验。
2.2 RT-qPCR实验
用Trizol法提取总RNA,进行反转录得到cDNA,然后以cDNA为模板进行qPCR反应,检测其中miR-29a和HBP1基因mRNA水平的表达,qPCR反应数据以循环阈值(Ct)记录,实验结果利用ΔΔCt法进行计算,miR-29a以u6为内参,HBP1以GAPDH为内参,相对定量各实验组样品内miR-29a和HBP1基因的表达(miR-29a和u6的引物来自miRNA实时荧光定量 RT-qPCR 定量试剂盒;HBP1和GAPDH引物序列见表1)。
Table1 Primer sequences used for qPCR
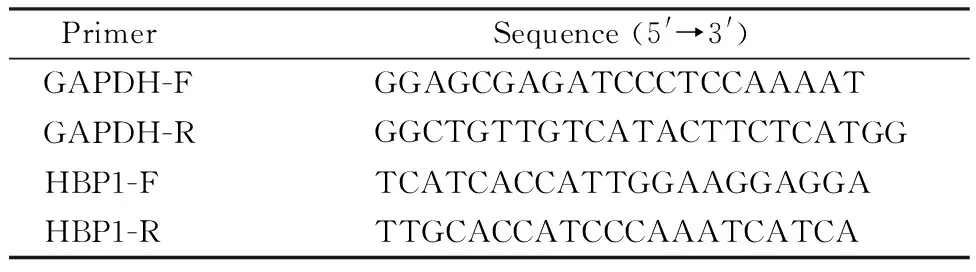
Primer Sequence (5'➝3')GAPDH-FGGAGCGAGATCCCTCCAAAATGAPDH-RGGCTGTTGTCATACTTCTCATGGHBP1-FTCATCACCATTGGAAGGAGGAHBP1-RTTGCACCATCCCAAATCATCA
2.3 细胞转染
将miR-29a模拟物、miR-29a抑制剂、模拟物阴性对照、抑制剂阴性对照(序列见表2)干粉瞬时离心后,用含RNA酶抑制剂的ddH2O溶解,在避光的条件下吹打混匀,配制成终浓度为20 μmol/L的工作液(使用时的终浓度均为100 nmol/L),-20 ℃贮存。取“2.1”项中所述种于6孔板的细胞,按照Lipofectamine 2000试剂说明书进行转染,6 h后更换为新的DMEM培养液,继续培养用于后续实验。
2.4 划痕愈合迁移实验
将种植于6孔板中的细胞按照“2.3”项下方法转染24 h后,利用1 mL枪头在细胞层中形成划痕,于0,48 h在显微镜下观察细胞迁移情况,随机选取3处进行拍照。每组细胞设3个复孔。
Table2 Sequences of miR-29a mimic/inhibitor and their controls
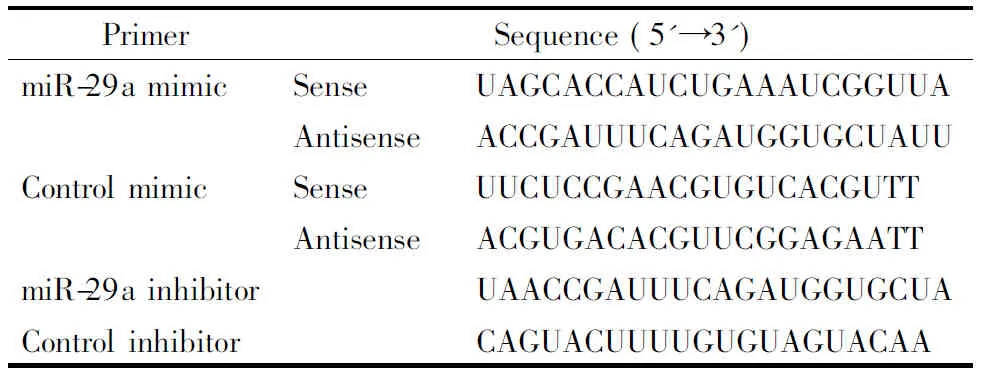
PrimerSequence (5'➝3')miR-29a mimicSenseUAGCACCAUCUGAAAUCG-GUUAAntisenseACCGAUUUCAGAUGGUGC-UAUUControl mimicSenseUUCUCCGAACGUGUCACGUTTAntisenseACGUGACACGUUCG-GAGAATTmiR-29a inhibi-torUAACCGAUUUCAGAUGGUGC-UAControl inhibitorCAGUACUUUUGUGUAGUA-CAA
2.5 Transwell小室侵袭实验
按照“2.3”项下方法转染细胞24 h后,将细胞进行消化,加入无血清DMEM 培养基吹打混匀,调整细胞的浓度为每毫升5×105个,在Transwell小室的下室加入含5% FBS的DMEM培养基800 μL,在上室加入混匀的细胞悬液200 μL(在实验前4~6 h将matrigel胶储液用无血清及抗生素的DMEM培养基按1∶6进行稀释,以每孔50 μL的体积加入到Transwell 侵袭小室中),置于培养箱培养36~48 h后,经4%多聚甲醛固定20 min和结晶紫染色20 min后,用棉签小心擦去膜上面的细胞,用显微图像采集系统进行观察并随机选取视野进行拍照。
2.6 靶基因预测和荧光素酶报告实验
通过生物信息预测数据库Targetscan7.1对miR-29a进行检索,预测其靶基因,然后将预测到的靶基因HBP1作为研究对象。人工合成野生型片段(HBP1-WF/WR),并引入SpeⅠ和HindⅢ 限制性核酸内切酶位点;在野生序列基础上,另外设计合成了种子序列互补序列的突变位点,合成突变型片段(HBP1-MF/MR)(具体序列见表3,粗体表示酶切位点)。包含目标基因的片段经退火后导入pMIR-REPORT质粒。将生长良好的HEK-293T细胞种于24孔板中,于24孔板的每一孔中,分别共转染荧光素酶质粒(或相对应的突变质粒)1 μg、海肾荧光素酶质粒0.3 μg以及相应量的miR-29a模拟物或miR-29a抑制剂或各自对应的阴性对照。转染48 h后按照试剂盒说明书检测荧光水平以及进行数据计算。
Table3 Primer sequences used for luciferase report assay

2.7 Western blot实验
细胞转染48 h后,收集细胞裂解提取蛋白,加入上样缓冲液,100 ℃煮沸10 min,10% SDS-PAGE电泳分离蛋白质,再转印至PVDF膜上,将膜置于含50 g/L脱脂牛奶的TBST室温封闭1 h;再分别加入相应的兔抗人HBP1抗体及兔抗人GAPDH 抗体,4 ℃过夜,次日以TBST洗涤3次,加入HRP标记羊抗兔IgG抗体室温孵育1 h,TBST洗涤3次,ECL显色系统定影显色,观察条带并进行灰度扫描。以GAPDH作为内参对照。
2.8 数据统计
3 结 果
3.1 miR-29a在不同乳腺癌细胞系中的表达
MDA-MB-231属于高转移性恶性乳腺癌细胞系,MCF-7为原位ER阳性乳腺癌细胞系,恶性程度较MDA-MB-231低,本研究比较了不同乳腺癌细胞系中miR-29a的表达水平。RT-qPCR检测结果(图1)显示,miR-29a在MDA-MB-231中的表达明显高于在MCF-7中,miR-29a在恶性程度更高的MDA-MB-231中表达较高提示miR-29a的潜在促肿瘤作用。
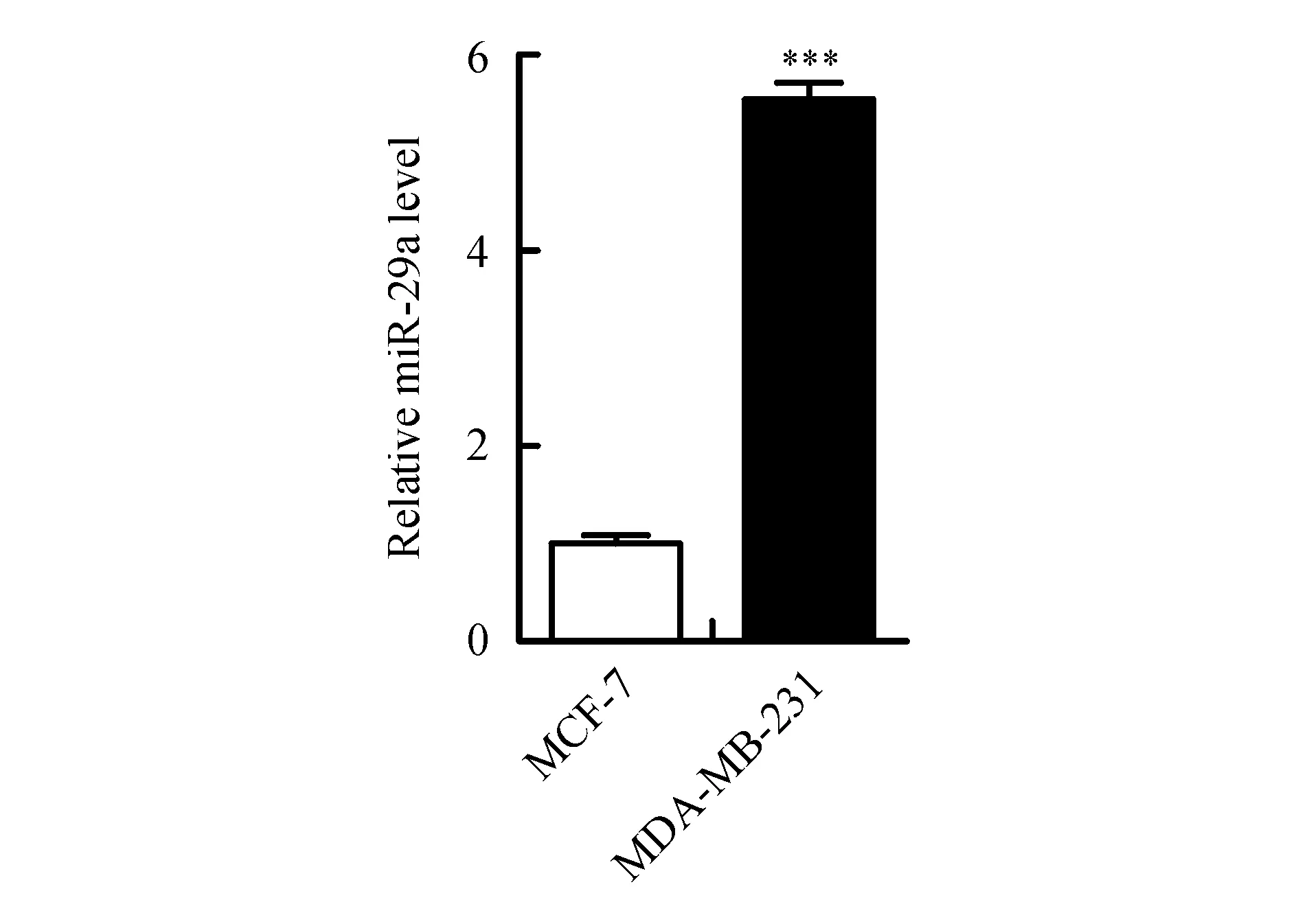
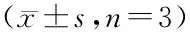
***P<0.001vsMCF-7 group
3.2 过表达和敲除miR-29a的转染效率验证
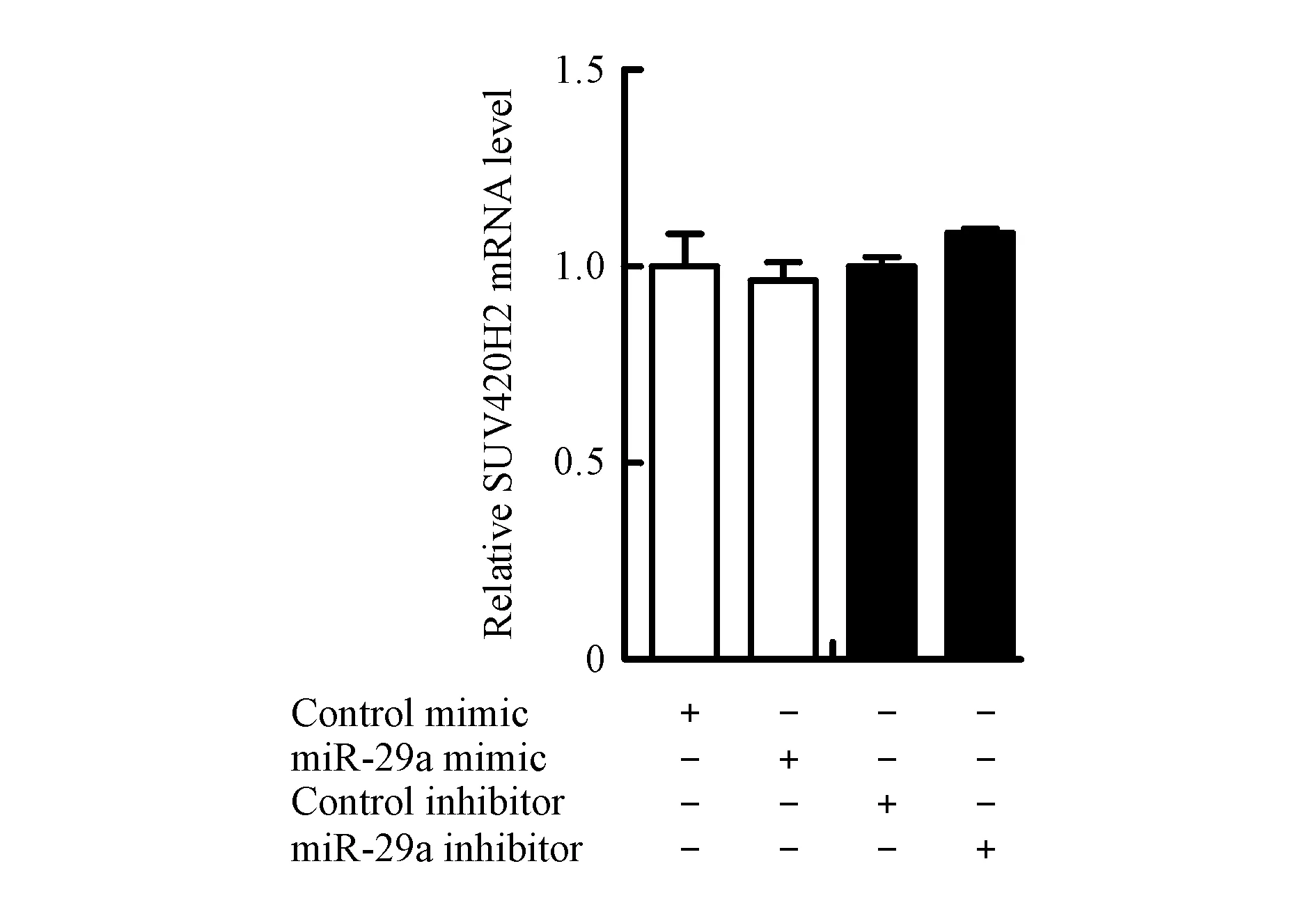
将miR-29a模拟物、miR-29a抑制剂及其阴性对照转染乳腺癌细胞MCF-7,细胞转染48 h后,RT-qPCR检测结果(图2)显示,与模拟物阴性对照组相比,转染miR-29a模拟物的细胞中miR-29a的表达水平上调,差异有统计学意义;与抑制剂阴性对照组相比,转染miR-29a抑制剂的细胞中miR-29a的表达水平下调,差异有统计学意义。总之,以上结果说明有效地实现了miR-29a的过表达和敲除。
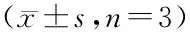
***P<0.001vscontrol mimic group;*P<0.05vscontrol inhibitor group
3.3 miR-29a对人乳腺癌MCF-7细胞的体外侵袭和迁移的促进作用
体外迁移实验结果显示,与模拟物阴性对照组相比,过表达miR-29a的细胞划痕间隙变化显著升高(图3-A);与抑制剂阴性对照组相比,敲除miR-29a的细胞划痕间隙变化显著降低(图3-B)。体外侵袭实验结果显示,过表达miR-29a的细胞迁移和侵袭数增多(图4-A);相应的,敲除miR-29a的细胞侵袭数减少(图4-B),差异有统计学意义。
3.4 靶基因预测及HBP1的荧光素酶验证
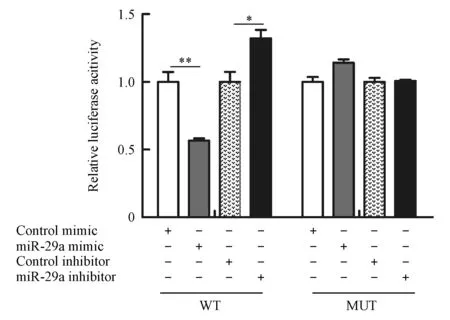
通过预测分析,miR-29a序列与HBP1 3′非翻译区(3′-UTR)存在一个互补结合区,HBP1可能为miR-29a的特异性靶基因。本研究进行了荧光素酶报告实验,实验结果(图5)表明HBP1为miR-29a的直接靶基因,miR-29a与HBP1的3′-UTR区存在直接靶向作用。
3.5 Western blot和RT-qCR检测HBP1蛋白水平和转录水平的表达
Western blot结果(图6)显示,过表达和敲除miR-29a分别可下调和上调HBP1在蛋白水平的表达。然而,RT-qPCR结果(图7)显示,miR-29a的过表达和敲除对HBP1的mRNA水平没有影响。
Figure3 Effects of miR-29a mimic (A) and inhibitor (B) on the migration of MCF-7 cells
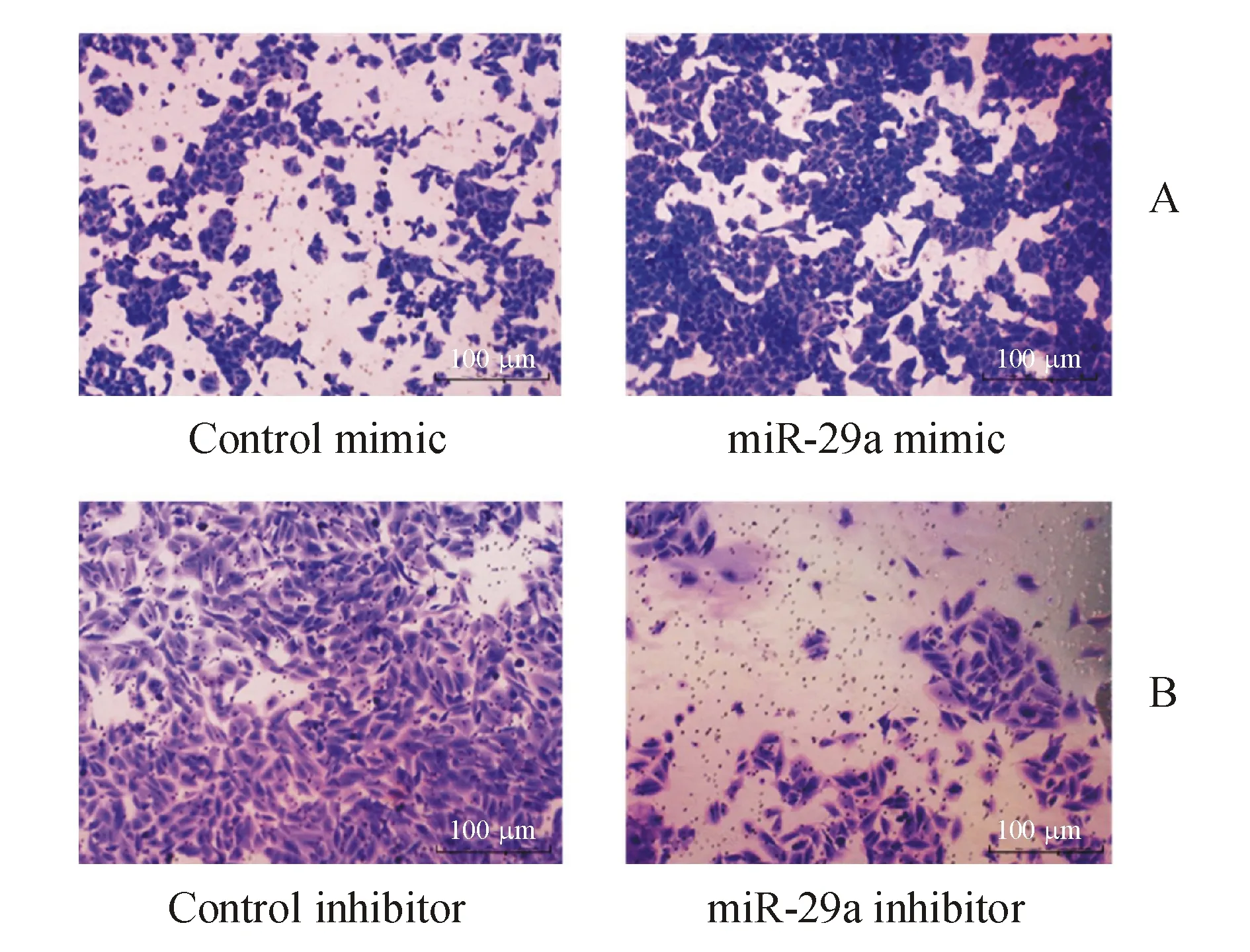
Figure4 Effects of miR-29a mimic (A,36 h) and inhibitor (B,48 h) on the invasion of MCF-7 cells
**P<0.01vscontrol mimic/WT group;*P<0.05vscontrol inhibitor/WT group
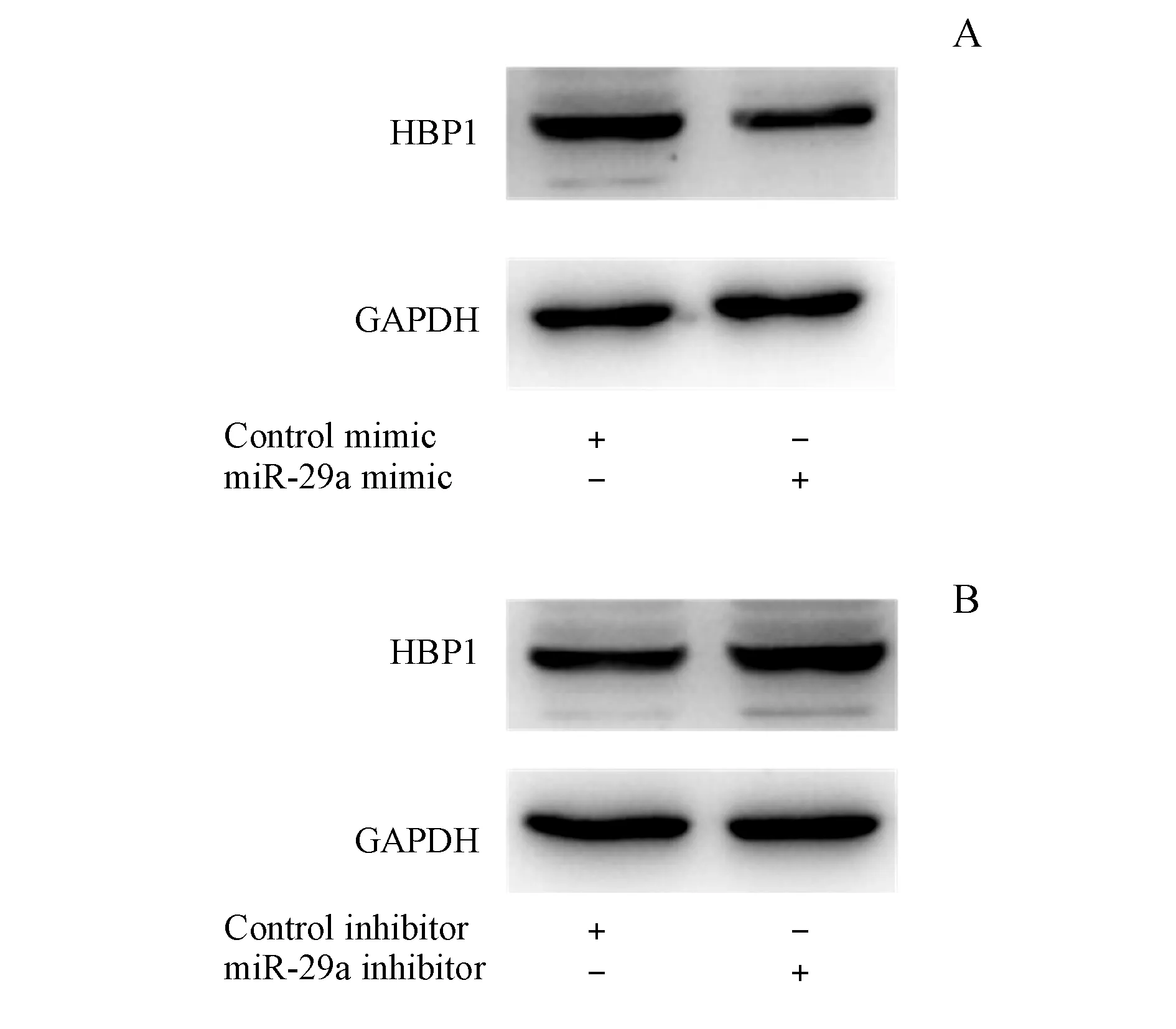
Figure6 Effects of miR-29a mimic (A) and inhibitor (B) on the protein expression of HBP1 in MCF-7 cells
4 讨 论
miRNA参与细胞增殖、迁移、分化和细胞周期等多种生物过程。miRNA主要通过结合靶基因的3′非翻译区(3′-UTR)来控制其靶mRNA的表达,单个基因的UTR区可以具有多个miRNA的结合位点或者某一个miRNA的多个结合位点,这表明由这些miRNA通过复杂的转录后调控影响基因表达[6]。
miR-29a位于染色体7q32.3负链的普通型脆性位点FRA7H内,可能与肿瘤密切相关[7]。miR-29a被报道在多种肿瘤中低表达且是抑癌因子,如胃癌[8]、胰腺癌[9]、宫颈癌[10]、前列腺癌[11]、食管癌[12]等;然而miR-29a也被报道在乳腺癌[4]、结肠癌[13]中表达上调,作为促癌基因促进了肿瘤的发生及发展。本研究首先通过实时荧光定量RT-qPCR检测了乳腺癌细胞系MCF-7和MDA-MB-231中miR-29a的表达水平,发现与MCF-7细胞相比,miR-29a在MDA-MB-231中的表达上调,因为相比MCF-7细胞,MDA-MB-231转移性和恶性程度更高,这提示miR-29a在乳腺癌发生和发展过程中可能起促进的作用。为了进一步明确miR-29a在乳腺癌中的作用,本研究以MCF-7细胞作为研究对象,通过脂质体法将miR-29a mimic及miR-29a inhibitor及其相应的阴性对照转染至细胞中,通过划痕实验和Transwell实验发现,与阴性对照组相比较,转染miR-29a mimic后细胞的迁移和侵袭的能力增加,而转染miR-29a inhibitor后细胞的迁移和侵袭的能力减少,这表明miR-29a对乳腺癌细胞迁移和侵袭能力具有正向调控作用。
此外,为了进一步研究miR-29a调控肿瘤转移的机制,本研究用目前常用的miRNA靶基因预测软件Targetscan 7.1对miR-29a的靶基因进行预测,发现HBP1的3′-UTR区域存在与miR-29a不完全配对的序列,这说明HBP1有可能是miR-29a的靶基因。HBP1(HMG盒转录因子1),是一种核蛋白,人体多种组织中均有表达[14],如大脑、子宫、睾丸、肺以及心脏等。HBP1是一种重要的肿瘤抑制因子,其抑制肿瘤机制有多种[15]:HBP1抑制转录因子c-Myc的活性及表达;通过与p38-MAPK通路作用调控细胞周期的阻滞;参与Ras介导的早衰;HBP1还可阻滞Wnt-β连环蛋白信号通路从而抑制G1进程,进而抑制肿瘤细胞生长。一些临床研究表明,在浸润性乳腺癌中发生HBP1的突变或表达减少,并且HBP1可能调节几种类型的乳腺癌的增殖和侵袭[16-17]。HBP1是通过抑制LEF/TCF转录因子抑制Wnt/b-catenin信号传导[18],而Wnt信号通路是会进一步影响肿瘤细胞的侵袭和转移的[19-20]。综合以上几点推测,HBP1通过Wnt信号通路抑制乳腺癌细胞的迁移和侵袭,因此选择HBP1基因作为下一步研究的对象。
为了验证miR-29a与HBP1之间是否存在调控的关系,本研究先进行了荧光素酶报告基因验证实验,发现miR-29a直接与HBP1基因的3′-UTR区域相结合,然后又检测了miR-29a变化后HBP1的表达情况。Western blot结果显示,与阴性对照组相比较,当细胞转染miR-29a mimic后,HBP1蛋白质的表达下调,而细胞转染miR-29a inhibitor后,HBP1蛋白质的表达上调,说明miR-29a可负向调控HBP1蛋白的表达。然而,RT-qPCR的结果显示,过表达或敲除miR-29a后,HBP1在mRNA水平上的表达没有变化,说明miR-29a调控HBP1的表达是通过结合HBP1的mRNA抑制其蛋白水平表达,而非使其mRNA降解,这符合miRNA调控靶基因的一般规律。
综合以上实验结果,本研究推测,在乳腺癌中高表达的miR-29a通过下调HBP1,从而使乳腺癌细胞获得高的迁移和侵袭能力,促进乳腺癌的转移,其具体机制可能是通过Wnt信号通路,还有待进一步实验研究确认。
参 考 文 献
[1] Redig AJ,McAllister SS.Breast cancer as a systemic disease:a view of metastasis[J].JInternMed,2013,274(2):113-126.
[2] Zhang K,Zhang Y,Liu C,etal.MicroRNAs in the diagnosis and prognosis of breast cancer and their therapeutic potential[J].IntJOncol,2014,45(3):950-958.
[3] Di Leva G,Garofalo M,Croce CM.microRNAs in cancer[J].AnnuRevPathol,2014,9:287-314.
[4] Pei YF,Lei Y,Liu XQ.MiR-29a promotes cell proliferation and EMT in breast cancer by targeting ten eleven translocation 1[J].BiochimBiophysActa,2016,1862(11):2177-2185.
[5] Rostas JW,Pruitt HC,Metge BJ,etal.microRNA-29 negatively regulates EMT regulator N-myc interactor in breast cancer[J].MolCancer,2014,13:200.
[6] Shukla GC,Singh J,Barik S.MicroRNAs:processing,maturation,target recognition and regulatory functions[J].MolCellPharmacol,2011,3(3):83-92.
[7] Debacker K,Kooy RF.Fragile sites and human disease[J].HumMolGenet,2007,16(2):150-158.
[8] Chen L,Xiao H,Wang ZH,etal.miR-29a suppresses growth and invasion of gastric cancer cellsinvitroby targeting VEGF-A[J].BMBRep,2014,47(1):39-44.
[9] Tréhoux S,Lahdaoui F,Delpu Y,etal.Micro-RNAs miR-29a and miR-330-5p function as tumor suppressors by targeting the MUC1 mucin in pancreatic cancer cells[J].BiochimBiophysActa,2015,1853(10 Pt A):2392-2403.
[10] Yamamoto N,Kinoshita T,Nohata N,etal.Tumor-suppressive microRNA-29a inhibits cancer cell migration and invasion via targeting HSP47 in cervical squamous cell carcinoma[J].IntJOncol,2013,43(6):1855-1863.
[11] Pasqualini L,Bu H,Puhr M,etal.miR-22 and miR-29a are members of the androgen receptor cistrome modulating LAMC1 and Mcl-1 in prostate cancer[J].MolEndocrinol,2015,29(7):1037-1054.
[12] Liu C,Duan P,Li B,etal.miR-29a activates Hes1 by targeting Nfia in esophageal carcinoma cell line TE-1[J].OncolLett,2015,9(1):96-102.
[13] Tang W,Zhu Y,Gao J,etal.MicroRNA-29a promotes colorectal cancer metastasis by regulating matrix metalloproteinase 2 and E-cadherin via KLF4[J].BrJCancer,2014,110(2):450-458.
[14] Shih HH,Xiu M,Berasi SP,etal.HMG box transcriptional repressor HBP1 maintains a proliferation barrier in differentiated liver tissue[J].MolCellBiol,2001,21(17):5723-5732.
[15] Huang JS,Xiao R,Pang Y,etal.HBP1—an important tumor inhibitor[J].ChinJBiochemMolBiol(中国生物化学与分子生物学报),2012,28(10):913-918.
[16] Paulson KE,Rieger-Christ K,McDevitt MA,etal.Alterations of the HBP1 transcriptional repressor are associated with invasive breast cancer[J].CancerRes,2007,67(13):6136-6145.
[17] Yee AS,Paulson EK,McDevitt MA,etal.The HBP1 transcriptional repressor and the p38 MAP kinase:unlikely partners in G1 regulation and tumor suppression[J].Gene,2004,336(1):1-13.
[18] Sampson EM,Haque ZK,Ku MC,etal.Negative regulation of the Wnt/b-catenin pathway by the transcriptional repressor HBP1[J].EMBOJ,2001,20(16):4500-4511.
[19] Qin X,Zhang H,Zhou X,etal.Proliferation and migration mediated by Dkk-1/Wnt/b-catenin cascade in a model of hepatocellular carcinoma cells[J].TranslRes,2007,150(5):281-294.
[20] Wang Z,Ma Q.Beta-catenin is a promising key factor in the SDF-1/CXCR4 axis on metastasis of pancreatic cancer[J].MedHypotheses,2007,69(4):816-820.
