实验用狨猴微卫星引物筛选及遗传多样性分析
2018-06-26滕永康刘先菊向志光阮研硕刘云波
滕永康,刘先菊,张 旭,向志光,阮研硕,肖 冲,刘云波
(中国医学科学院医学实验动物研究所,北京 100021)
普通绵耳狨猴(Callithrixjacchus)是一种小型的非人灵长类实验动物,原产于南美巴西的热带雨林,因具有体型较小(成年狨猴体长165~199 mm,体重337~413 g[1])、便于捕捉和固定、饲养成本较低、繁殖效率较高等特点,已广泛用于神经科学、行为学、传染病学、生殖医学、药物开发及药物安全评价等研究领域[2-3]。自上世纪80年代引入我国以来,在病毒学[4-5]、免疫学[6-7]、基因工程[8-9]等研究领域应用较多。清晰的遗传背景是狨猴实验结果的可靠性、准确性、可重复性、以及科学引种、繁殖和生产的基本保障,而尽快建立狨猴的遗传质量监测体系更是十分必要和亟待开展的工作。随着实验动物科学领域的快速发展,微卫星标记检测技术已广泛应用于我国多种实验动物遗传背景监测和种群遗传结构分析,如近交系小鼠及大鼠[10]、封闭群小鼠[11]及大鼠[12]、长爪沙鼠[13]、小型猪[14]等常用实验动物群体已建立了稳定的微卫星标记检测方法,包括非人灵长类动物,如恒河猴[15]及食蟹猴[16],均有相关微卫星标记的遗传学结构分析报道。而狨猴作为新型的实验动物,国外研究者Raveendran等[17]、Katoh等[18]相继开发了狨猴微卫星DNA标记引物,并对狨猴微卫星位点多态性进行分析报道,国内至今尚无报道。鉴于狨猴在生物医学研究领域诸多学科的重要性,以及狨猴目前仍作为“实验用狨猴”,尚无国家、行业或地方标准的推出。本文通过筛选验证得到20对多态性较好的微卫星位点,并就中国医学科学院医学实验动物研究所实验动物北方资源中心引进的实验用狨猴群体遗传结构进行测定分析,为狨猴科学繁育及遗传质量控制提供数据支持。
1 材料和方法
1.1 实验动物
成年狨猴30只,年龄2~4岁,体重330~420 g。由中国医学科学院医学实验动物研究所实验动物北方资源中心[SCXK(京)2014-0011][SYXK(京)2017-0027]提供。温度24℃~29℃,湿度> 40%,并按实验动物使用的3R原则给予人道主义关怀。
1.2 主要试剂及仪器
Easy Pure Blood Genomic DNA Kit(code #EE121-01,全式金);2×EasyTaq PCR Super Mix(AS111-02,全式金);100 bp DNA Ladder(code 3422A,TaKaRa);抗凝剂为乙二胺四乙酸二钾(EDTA-K2);PCR仪(MycyclerTMThermal Cycler型,Bio-Rad);电泳仪(200/2.0 Power Supply型,Bio-Rad);凝胶成像系统(Tanon-1600型,天能);遗传分析仪(3730XL DNA analyzer,ABI)。
1.3 实验方法
1.3.1样本采集和基因组DNA提取
狨猴后肢静脉采血,EDTA-K2抗凝,采血量为每只0.5 mL。用Easy Pure Blood Genomic DNA kit纯化基因组DNA。
1.3.2引物筛选及PCR扩增
通过查阅相关文献,筛选多态性较好的41个狨猴微卫星标记,经NCBI网站(http://www.ncbi.nlm.nih.gov/unists)报道及PCR优化后得到了20个狨猴微卫星标记和20对引物,这些标记基因基本覆盖了狨猴20条染色体上的遗传概貌,具有丰富的多态性,微卫星标记信息详见表1。PCR扩增总体系20 μL,其中2×EasyTaq PCR Super Mix 8 μL,模板DNA量1 μL,上下游引物各0.2 μL(浓度:50 μmol/L),ddH2O 10.6 μL。PCR扩增条件:94℃预变性5 min;94℃变性30 s,退火温度55℃~60℃ 30 s,72℃延伸30 s,共40个循环;72℃继续延伸8 min,扩增产物4℃保存。
1.3.3PCR产物鉴定及电泳结果
用2%琼脂糖凝胶(agrose)电泳,点样量每孔5 μL,以150 V恒压30 min进行PCR产物鉴定;再用6%非变性聚丙烯酰胺凝胶(PAGE)电泳,以80 V恒压1.5 h进行等位基因的分离,上样量5 μL,用EB缓冲液进行染色,Tanon-1600凝胶成像系统进行成像。
1.3.4STR扫描
一次扫描3个微卫星位点,每3个位点的反向引物分别用ROX、FAM、HEX三种荧光标记物进行标记,标记后的引物再进行PCR扩增,其扩增产物按1∶3∶5体积比混合后取1 μL进行毛细管电泳检测和遗传分析仪3730xl DNA analyzer(ABI)扫描分析,通过Genemarker V2.2.0软件统计等位基因片段大小,并对扫描峰图进行数据处理。
1.4 统计学方法
群体内遗传结构分析常采用杂合度和多态信息含量进行分析和评估,将所有样本的微卫星基因型输入Popgene1.32软件,计算不同个体在各个微卫星位点上的观察等位基因数(observed number of alleles, Na)、有效等位基因数(effective numbers of alleles, Ne)、表观杂合度(observed heterozygosity, Ho)、期望杂合度(expected heterozygosity, He)、香隆信息指数(Shannon’s information index, I)及反映哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)的P-val;再用PIC-CALC软件(Version- 0.6)对每个位点进行多态信息含量(polymorphism information content, PIC)计算。
2 结果
2.1 PCR扩增产物电泳结果
图1所示,采用No.3样本基因组DNA扩增的20对狨猴微卫星产物。2%琼脂糖凝胶电泳结果显示,经PCR反复优化,扩增产物丰度较高、特异性较好,初步可以观察到不同微卫星标记扩增片段大小的不同;进一步用6%非变形的聚丙烯酰胺凝胶电泳分离,检测出各个微卫星不同等位基因的片段大小(图2)。
2.2 STR扫描结果
筛选的20个狨猴微卫星标记通过PCR扩增,凝胶电泳鉴定丰度较高,初步用非变性聚丙烯凝胶电泳进行分离,显示出各标记位点不同片段大小的等位基因;进一步STR扫描后,30个样本在20个微卫星标记均检测到扫描波峰,不同样本在各位点标明有效等位基因大小及数量(图3)。STR扫描可以精确到等位基因片段大小1 bp的差异,图3是样本1、5、7、12在微卫星位点Ham26的扫描波峰,图3A只检测到1个等位基因波峰,表示样本为纯合子;图3B~D各检测到2个等位基因波峰,表明所检样本均为杂合子。
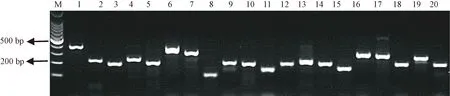
注:(M)DL1000 bp marker。图1 20个狨猴微卫星标记对No.3样本DNA的PCR扩增结果Note.(M)DL1000 bp marker.Fig.1 The PCR amplification results of 20 marmoset microsatellite markers of the sample No.3
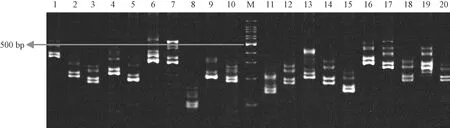
注:(M)DL1000 bp marker。图2 20个微卫星DNA等位基因片段大小的非变性聚丙烯凝胶电泳分离结果Note.(M)DL1000 bp marker.Fig.2 The allele size and number results of 20 microsatellite DNA separated by the non-denatured 6% PAGE
2.3 种群遗传多态性分析
2.3.1多态性分析
30只狨猴在20个微卫星位点共检测147个观察等位基因,平均每个位点有5~10个,其中CAJA17、Ham65、Ham61均有10个等位基因,CAJA1位点有9个等位基因,表现出较高的多态性,20个座位的平均等位基因数为7.35个,有效等位基因数在2.250~6.383个之间,平均等位基因数4.040个。(见表1)
2.3.2杂合度和多态信息含量
20个微卫星位点的观察杂合度、期望杂合度、香隆信息指数以及多态信息含量分析结果详见表2。从表2中可以看出,观察杂合度最大为0.4667,平均为1.533;期望杂合度在0.1424~0.435之间,平均期望杂合度为0.251;香隆信息指数在1.2242~2.0324之间,平均为1.5949,PIC在0.5365~0.8253之间,平均PIC为0.7053。
2.4 Hardy-Weinberg遗传平衡检验
用Popgene1.32软件计算实验用狨猴20个微卫星位点的Hardy-Weinberg遗传平衡检验的P值,结果P值均大于0.05,即30只狨猴在20个微卫星位点处于Hardy-Weinberg平衡,详见表2。
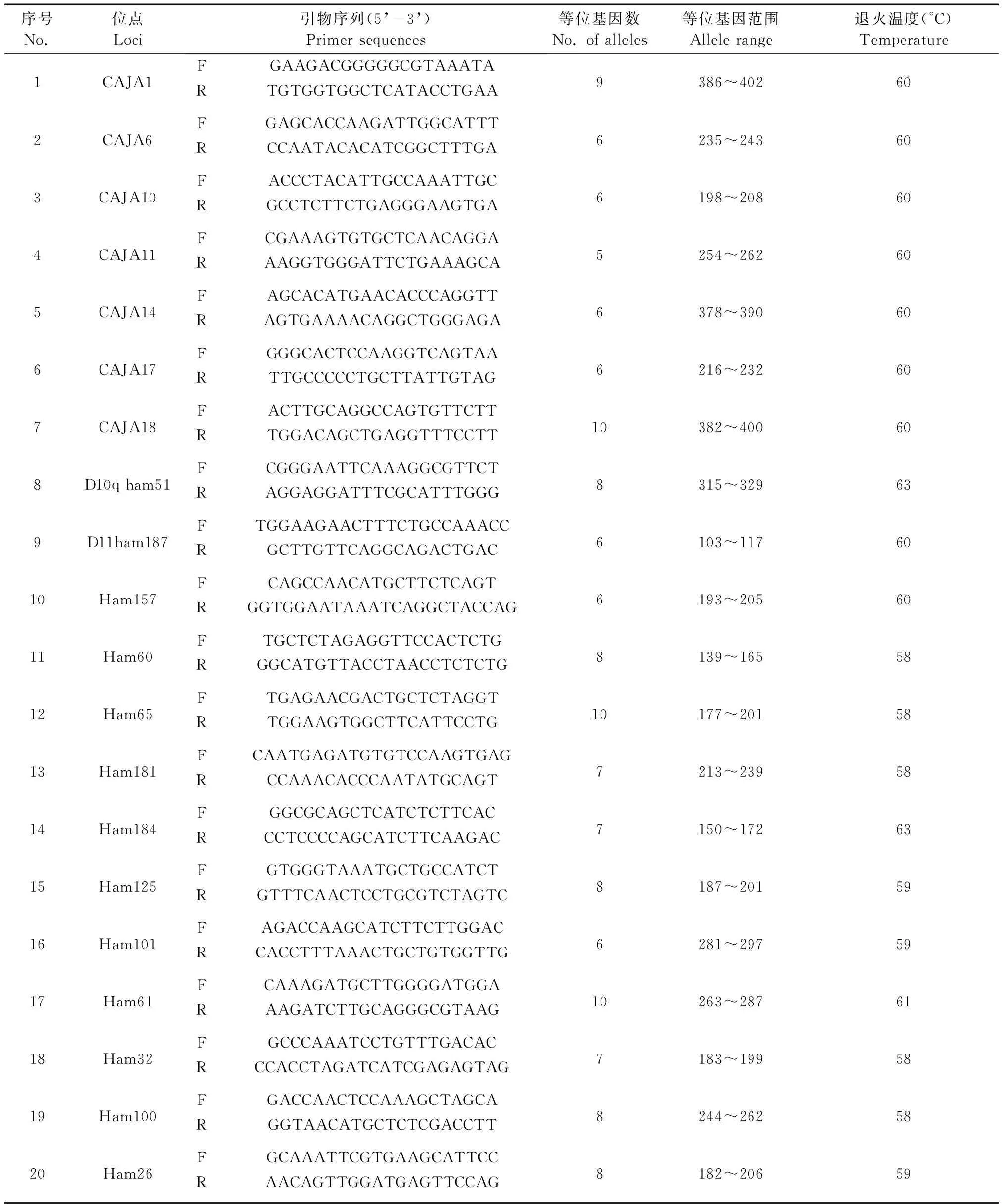
表1 狨猴微卫星DNA标记信息

图3 Ham26对4个样本的STR扫描结果Fig.3 Results of Ham26 scanning of 4 samples from the marmosets
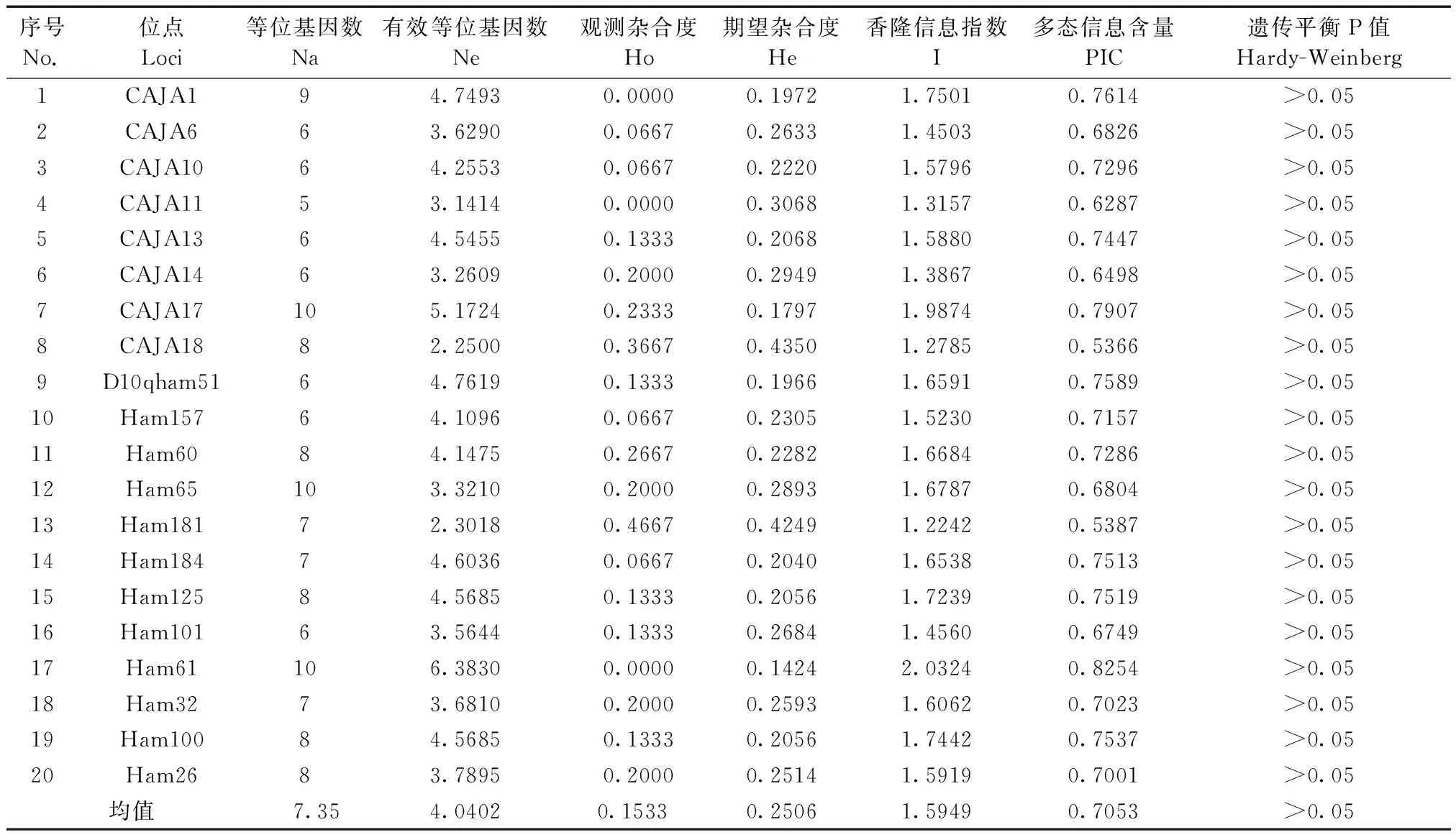
序号No.位点Loci等位基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度He香隆信息指数I多态信息含量PIC遗传平衡P值Hardy-Weinberg1CAJA194.74930.00000.19721.75010.7614>0.052CAJA663.62900.06670.26331.45030.6826>0.053CAJA1064.25530.06670.22201.57960.7296>0.054CAJA1153.14140.00000.30681.31570.6287>0.055CAJA1364.54550.13330.20681.58800.7447>0.056CAJA1463.26090.20000.29491.38670.6498>0.057CAJA17105.17240.23330.17971.98740.7907>0.058CAJA1882.25000.36670.43501.27850.5366>0.059D10qham5164.76190.13330.19661.65910.7589>0.0510Ham15764.10960.06670.23051.52300.7157>0.0511Ham6084.14750.26670.22821.66840.7286>0.0512Ham65103.32100.20000.28931.67870.6804>0.0513Ham18172.30180.46670.42491.22420.5387>0.0514Ham18474.60360.06670.20401.65380.7513>0.0515Ham12584.56850.13330.20561.72390.7519>0.0516Ham10163.56440.13330.26841.45600.6749>0.0517Ham61106.38300.00000.14242.03240.8254>0.0518Ham3273.68100.20000.25931.60620.7023>0.0519Ham10084.56850.13330.20561.74420.7537>0.0520Ham2683.78950.20000.25141.59190.7001>0.05均值7.354.04020.15330.25061.59490.7053>0.05
3 讨论
实验动物是实验动物科学的重要组成,是生命科学的基础支撑、活的“精密仪器”,而实验动物的标准化和动物实验的规范化研究势必会成为实验动物科学的重要工作。在我国,狨猴至今仍被定义为“实验用动物”,即狨猴作为诸多学科研究用的非人灵长类实验动物,目前尚无国家、行业或地方标准的推出。众所周知,健康可用于科学研究的实验动物,除了要求对其携带的病原微生物、寄生虫进行实施质量控制和监测外,还要求实验动物具有清晰和明确的遗传背景。
狨猴是公认的、重要的非人灵长类实验动物模型,美国国家人类基因组研究机构已大力度投资验证了狨猴的基因组序列,这对狨猴的基因和基因型分析具有重要意义[3]。微卫星标记存在于大多数原核及真核生物基因组DNA序列,由2~6个核苷酸串联重复序列构成,物种间和物种个体间的差异及核心序列重复数量的不同,便产生微卫星DNA标记及微卫星DNA的多态性。由于微卫星DNA相对其它分子标记分布广、信息含量高、多态性丰富、特异性好、操作简单、且符合孟德尔的共显性遗传定律,近年来已广泛用于实验动物质量监测及动物群体遗传结构分析[19-20]。本文通过查阅文献[17-18]筛选优化了20个狨猴微卫星DNA标记,并对30只样本进行微卫星DNA扩增和扫描,初步对实验用狨猴群体进行了遗传多样性分析。等位基因频率作为评估遗传变异的前提和基础,是指某一等位基因特定基因座位在所有等位基因中所占的比例,在某一个位点上, 如果它的等位基因频率最大值< 0.95,通常定义为多态性位点,本文检测的所有等位基因频率最高为0.65,表明筛选的20个微卫星位点的遗传多样性较好,有效等位基因数是反映群体遗传变异程度的一个重要指标,如果有效等位基因数与绝对等位基因数越接近,表明该等位基因在群体内均匀分布度就越好,文中涉及的20个微卫星位点,其中D10ham51的有效等位基因数和观察等位基因数较接近,为1.2381;而Ham65位点的有效等位基因数与绝对等位基因数相差最大,为6.679,表明该位点的等位基因的均匀分布度较差,有可能是繁育过程中人工选择因素所导致。
群体杂合度表示在被检测的位点上群体中的杂合子频率,它是反映群体杂合程度的度量单位,群体平均杂合度的高低反映了群体遗传的一致性程度,群体杂合度越高,表明该群体的遗传变异越大,群体遗传多态性越高良好的种群平均杂合度在0.5~0.7。本文用Popgene1.32软件计算平均观测杂合度和平均期望杂合度基本接近,相差范围仅为-0.05~0.3,表明狨猴群体遗传变异较小、遗传一致性较差。而PIC是用以描述微卫星基因座多态性大小的量度单位,Botstein等[21]认为当基因座位PIC > 0.5为高度多态位点,低度多态位点时,PIC < 0.25。本文的20个微卫星基因座位中,PIC范围在0.5365~0.8253,平均为0.7053,所有的位点均大于0.500,表明20个位点均表现为高度多态性,说明所筛选的微卫星基因座用于狨猴群体遗传多样分析较可靠,表明所监测的狨猴群体具有较丰富遗传多样性,选择潜力较大。此外,香隆指数是反映种群均匀性分布的一个重要指标,一般情况下其香隆指数变化在1.5~3之间,本文的20个微卫星基因座位香隆指数平均值为1.594,表明本地狨猴群体分布及群体交配较随机。
Hardy-Weinberg平衡定律是指在一个群体无限大、个体间进行随机交配、没有突变、没有遗传漂变的情况下,群体内一个位点上的基因型频率和基因频率将世代保持不变,即处于遗传平衡状态。Hardy-Weinberg检验P值(HWE)> 0.05时,表示该位点在群体中处于Hardy-Weinberg平衡;当0.01 综上所述,本文从大量的狨猴微卫星DNA座位筛选出分布在不同染色体上20个标记,并随机抽取30只人工饲养繁殖的实验用狨猴样本进行PCR扩增,扩增产物的电泳结果均显示清晰可见的目的条带及DNA多态性,表明采用的微卫星标记能够较好地反映狨猴种群个体间的遗传多样性。STR扫描结果经软件分析显示,除等位基因均匀度分布度较差、群体杂合度的遗传变异较小、遗传一致性较差外,其PIC仍处于高度多态性范围,即具有较丰富遗传多样性,同时香隆指数的平均值进一步表明本地狨猴群体分布及群体繁殖具有随机性。此外,Hardy-Weinberg检验P值也证实了狨猴种群得遗传平衡状态。这些结果均反映了狨猴种群遗传结构的现状,为狨猴科学繁育及遗传检测方法提供了基础资料。
