SELEX法筛选宫颈癌前病变核酸适配子
2018-06-11李文慧石大林贾茹涵杨静张艳艳陈卫韩跃武
李文慧,石大林,贾茹涵,杨静,张艳艳,陈卫,韩跃武
兰州大学 生物化学与分子生物学实验室,甘肃 兰州 730000
世界卫生组织 (WHO) 公布,2012年全世界有超过 50万的宫颈癌新发病例,其中超过 85%的病例来自发展中国家。据全国肿瘤登记中心统计,2011年我国宫颈癌新发病例87 000人,死亡病例23 000人,近30年来宫颈癌发病率呈上升趋势,且发病高峰趋于年轻化。宫颈癌早期没有明显症状和体征,及早发现宫颈癌前病变,并通过治疗阻断癌前病变的发展,可最大程度降低宫颈癌的发生率,因此寻找宫颈癌前病变诊断的生物标志物具有重要意义。1990年 Tuerk等[1]和Ellington等[2]分别报道了应用指数富集配体系统进化技术 (Sysrematie evolution of ligands by exponential enriehmen,SELEX) 筛选得到能与噬菌体 T4 DNA聚合酶和有机染料特异性结合的RNA 片段,并命名为“适配子”或“配体”[3]。该技术原理是利用大容量的随机单链寡核苷酸文库进行体外多轮筛选、扩增,并最终获得与非核酸靶分子高亲和力、高特异性结合的寡核苷酸序列[4]。大容量的随机单链寡核苷酸文库进行体外多轮筛选适配体是折叠成特异性3D结构的具有高亲和力的单链DNA或RNA,能够特异性结合多肽、蛋白质[5]、细胞[6]乃至整个器官的靶分子[7-9]。另外,适配子在一些生理条件下,可通过反义寡核苷酸杂交使其失活,因此可以用于设计解毒剂[10]。近年来,随着研究的不断深入,越来越多实验结果表明相比于抗体,适配子具有更强的亲和力、热稳定性、靶标多样性、高特异性、可化学合成等优点[11-15],可以代替抗体用于疾病的诊断、医药学和工业等领域[16]。本研究采用SELEX技术筛选宫颈癌前病变宫颈脱落细胞特异性适配子库[17-18],并通过特异性、亲和力分析和细胞免疫荧光确立高特异性适配子,可作为宫颈癌前病变诊断标志物,为宫颈癌前病变分子诊断奠定理论基础,提供新思路。
1 材料与方法
1.1 材料
1.1.1 样本
实验所用样本均取自液基薄层细胞检测结果为:正常、炎性、上皮内低级别病变 (CIN1) 上皮内高级别病变 (CIN2、CIN3) 和鳞状细胞癌宫颈脱落细胞,所有样本由甘肃迪安同享医学检验中心提供。
1.1.2 随机ssDNA文库及引物
由Prime Premier 5.0软件设计随机ssDNA文库及引物 (表1)[19],随机ssDNA文库5′端和3′端均为18个碱基固定序列,中间N(54)为54个碱基随机序列。上游引物 P1与 ssDNA文库 5′端固定序列相同,下游引物P2与ssDNA文库3′端固定序列互补,同时合成5′端FITC荧光标记的上游引物 P3。上述均由生工生物工程 (上海)股份有限公司合成。

表1 随机ssDNA文库及引物序列Table 1 Sequences of random ssDNA library and primer
1.2 方法
1.2.1 PCR扩增反应条件优化
经2%琼脂糖凝胶电泳鉴定随机ssDNA文库及引物是否构建成功并确定其条带位置,同时确定dsDNA条带的位置,作为后续实验PCR产物目的条带的参考标准。
以 50 μL 反应体系 (模板、引物各 2 μL,2×PCR Master Mix 25 μL,ddH2O 19 μL) 进行PCR扩增 (95 ℃变性30 s,退火1 min,72 ℃延伸30 s),经2%琼脂糖凝胶电泳鉴定,ImageJ作灰度分析,确定对称PCR最佳退火温度及循环次数[20-22]、间接不对称PCR扩增反应最佳循环数及最佳上下游引物浓度比[23]。
1.2.2 适配子库筛选
参照文献采用SELEX技术进行适配子筛选[24]。筛选过程如下:
①细胞预处理:脱落细胞经磷酸盐缓冲液洗涤数次后用 120目钢滤过滤,细胞计数板计数,冻存于–80 ℃超低温冰箱。每次实验取出细胞冻存管放置37 ℃水浴锅1 min,离心收集细胞。②ssDNA 文库预处理:取 ssDNA文库加入500 μL结合缓冲液,沸水浴5 min,冰浴10 min。③筛选:向预处理的ssDNA文库中加入15 μL 3%BSA以降低非特异性吸附,将其与阴性细胞 (正常、炎性) 37 ℃孵育结合,离心取上清,收集与反筛细胞不结合的核酸。再将收集的核酸与阳性细胞 (上皮内低级别病变 (CIN1)、上皮内高级别病变 (CIN2、CIN3) 和鳞状细胞癌) 37 ℃孵育结合,离心取沉淀,加入洗涤缓冲液洗涤以弃去未与细胞结合的核酸,再加入200 μL洗脱缓冲液,离心取上清,收集得到与正筛细胞结合的核酸。④次级文库获得:收集到的核酸经苯酚∶氯仿∶异戊醇法纯化后,经间接不对称PCR扩增、回收单链作为下一轮筛选的次级文库。⑤测定结合率:以每轮筛选获得的次级文库为模板进行间接不对称PCR扩增,扩增得到荧光素标记的ssDNA。取200 pmol荧光修饰的ssDNA和1 000 μL结合缓冲液,沸水浴5 min,冰浴10 min;分别与2×106个/mL阴性细胞、阳性细胞37 ℃孵育结合30 min,荧光分光光度计测定总荧光强度;回收细胞悬液离心后用洗涤缓冲液洗涤3次,重悬于1 000 μL结合缓冲液中,荧光分光光度计测定荧光强度值,测3次取平均值;不加荧光修饰的ssDNA为空白对照。根据ssDNA文库与细胞结合率=荧光强度平均值/总荧光值×100%,分别计算各轮筛选获得的次级文库与阳性细胞及阴性细胞的结合率判断筛选进度。⑥整个筛选过程通过不断增加筛选压力,如减少加入的ssDNA量、减少孵育时间、增加洗涤次数,以获得高亲和力、高特异性适配子库。经过10–12轮筛选,次级文库与靶细胞结合强度不再增加,达到平台期则筛选终止。筛选条件见表2。⑦将最终获得的 ssDNA核酸适配子库送生工生物工程 (上海) 股份有限公司克隆测序,运用DNAMAN软件进行一级结构同源性分析。
1.2.3 高特异性适配子确立
测序后,选择长度正确且没有双峰的适配子,将其转化菌经培养、质粒抽提,制备荧光素标记的ssDNA。
1) 特异性测定:取200 pmol荧光素标记的适配子加入1 000 μL结合缓冲液,沸水浴5 min,冰浴10 min;分别与2×106个/mL阴性细胞、阳性细胞37 ℃孵育结合30 min,离心去上清,洗涤缓冲液洗涤 3次,重悬于 1 000 μL结合缓冲液中,测定荧光强度值,测3次取平均值。
2) 亲和力分析:制备不同浓度荧光素标记的适配子,取等体积适配子加入1 000 μL结合缓冲液,沸水浴 5 min,冰浴 10 min;分别与 2×106个/mL阳性细胞37 ℃孵育结合30 min,离心去上清,洗涤缓冲液洗涤3次,重悬于1 000 μL结合缓冲液中,测定荧光强度值,测3次取平均值。根据公式Y=Bmax×X/ (Kd+X),运用 SigmaPlot 12.1软件,在非线性分析Regression Wizard公式类型中选择双曲线 (Hyperbola) 的类型,对数据进行分析,结合SPSS 16.0软件得出各适配子与宫颈癌前病变宫颈脱落细胞的平衡解离常数Kd值。
3) 细胞免疫荧光:根据特异性及亲和力分析结果确立高特异性适配子,进一步明确该适配子能够与子宫颈癌前病变宫颈脱落细胞特异性结合。分别取 1×106个/mL阴性细胞、阳性细胞爬片后经4%多聚甲醛固定30 min,0.01 mol/L PBS洗涤3次,每次5 min;抗原修复液沸水浴10 min,0.01 mol/L PBS洗涤 3次,每次 5 min;1%Triton-X100通透10 min,倾去后加入10%正常山羊血清37 ℃恒温封闭30 min;倾去封闭液后加入荧光素标记的适配子 (由生工生物工程 (上海) 股份有限公司合成),4 ℃避光过夜;0.01 mol/L PBS洗涤 3次,每次 5 min;抗荧光衰减封片剂 (含DAPI) 封片后观察。
2 结果与分析
2.1 PCR扩增反应条件优化结果
以文库为模板,经对称PCR后得到90 bp的dsDNA目的条带,即 ssDNA文库及引物构建成功。根据2%琼脂糖凝胶电泳鉴定 (图1),确定对称PCR扩增反应最佳循环数为15个循环;最佳退火温度为49.5 ℃;间接不对称PCR扩增反应最佳循环数为35个循环;最佳上下游引物浓度比为80∶1。
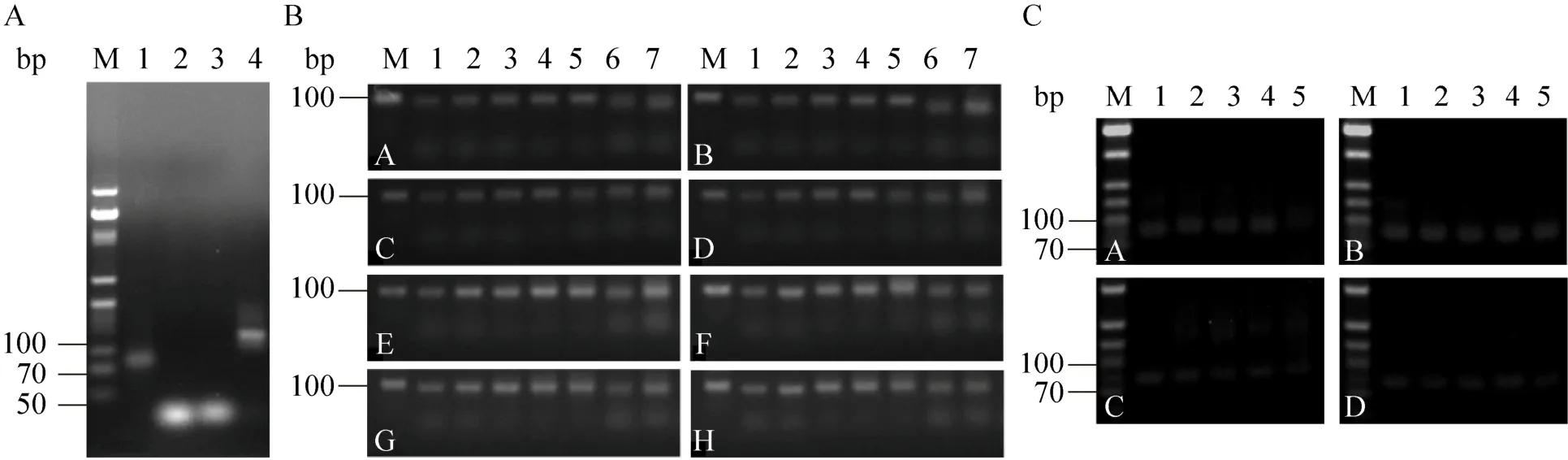
图1 PCR条件优化Fig. 1 Optimization of PCR conditions. (A) The verification result of random ssDNA library, primer and ssDNA PCR products. 1: random ssDNA library; 2−3: primer; 4: ssDNA PCR products. (B) Optimization of symmetry PCR conditions. A−H annealing temperature: 55, 54.5, 53.5, 51.7, 49.5, 47.8, 46.6, 46 ℃; 1−7 cycle times: 5, 8, 10, 12, 15,20, 25. (C) Optimization of indirect asymm PCR conditions. A−D cycle times: 30, 35, 40, 45; 1−5 primer concentration ratio: 10:1, 30:1, 50:1, 80:1, 100:1.
2.2 宫颈上皮内癌变宫颈脱落细胞SELEX筛选
2.2.1 筛选条件
筛选过程中,一方面加入BSA以封闭非特异性结合位点,去除非特异性结合从而提高筛选效率;另一方面,刚开始进行筛选时,如果ssDNA文库加入量过少会导致筛选失败,因此要以较多量的ssDNA和细胞进行前几轮筛选,随着筛选的进行要不断增加筛选压力 (表 2),使特异性的ssDNA能竞争结合在细胞膜表面的特异性物质上而特异性较弱的ssDNA被淘汰。
2.2.2 次级文库鉴定
每轮筛选获得的次级文库经不对称PCR扩增ssDNA,产物经2%琼脂糖凝胶电泳鉴定 (图2),可见90 nt目的片段ssDNA,满足筛选要求,可作为下轮筛选的文库。
2.2.3 结合率测定
在12轮筛选过程中,次级文库与阳性细胞的结合率从17%增加至75%;与阴性细胞的结合率从83%减少至19% (图3)。随着筛选的进行,在第9轮后结合率基本稳定达到平台期,说明特异性高的适配子得到富集,即筛选结束。
2.2.4 核酸适配子库克隆测序及一结构同源性分析
随机挑选40个克隆子进行测序,结果发现大量重序列及2个错误序列,最终确定适配子库共有9条适配子序列 (表3)。DNAMAN 7.0软件进行一级结构同源性分析表明 (图 4) 所有序列无共同的保守序列,但CIN-Ap2、CIN-Ap3、CIN-Ap4和CIN-Ap9含有保守序列GGGAT,剩下的适配子无同源性,且无共有保守序列。
2.3 高特异性适配子确立
2.3.1 适配子特异性及亲和力分析
9条适配子 (除CIN-Ap1) 与阳性细胞和阴性细胞结合力均具有极显著性差异 (P<0.01),其中适配子CIN-Ap3、CIN-Ap4、CIN-Ap5、CIN-Ap6的特异性较高。拟合各适配子与阳性细胞的结合曲线,计算得到平衡解离常数Kd值。根据解离常数Kd值越小,亲和力越大,确定CIN-Ap4、CIN-Ap5具有较高的亲和力 (图 5)。综合上述结果,最终确立 CIN-Ap4作为子宫颈癌前病变宫颈脱落细胞高特异性适配子。
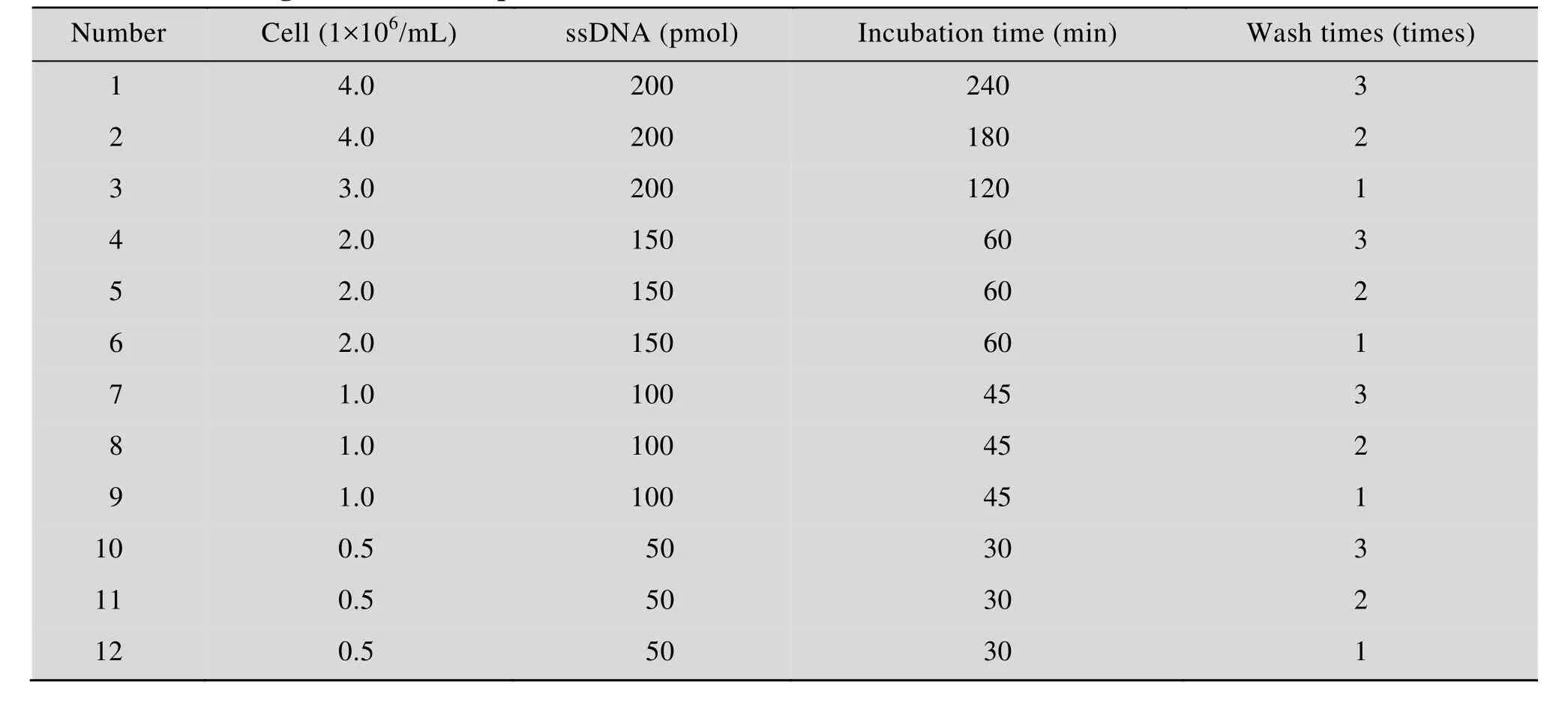
表2 适配子各轮筛选条件Table 2 Screening conditions of aptamer
2.3.2 适配子CIN-Ap4细胞免疫荧光
采用细胞免疫荧光法进一步验证适配子CIN-Ap4与阳性细胞和阴性细胞结合力 (图6),阳性细胞组荧光强度远高于阴性细胞组,说明所确立的适配子CIN-Ap4确实能够与子宫颈癌前病变宫颈脱落细胞高特异性结合,可作为子宫颈癌前病变诊断标志物,为宫颈癌前病变宫颈分子诊断提供理论基础和新思路。

图2 1–12轮筛选获得的ssDNA次级文库产物电泳图Fig. 2 2% agarose gel electrophoresis of ssDNA secondary library. M: marker; 1–12: ssDNA secondary library for each round.
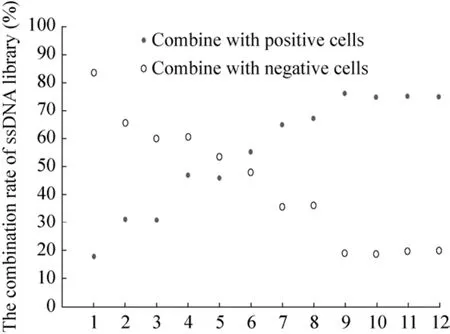
图3 1–12轮ssDNA次级文库与宫颈脱落细胞荧光结合率Fig. 3 The combination rate of ssDNA secondary library and cervical exfoliation cells for 12 rounds.

表3 适配子序列Table 3 Sequences of aptamers

图4 适配子同源性比对结果Fig. 4 Homology comparison.

图5 适配子特异性及亲和力分析结果Fig. 5 Specificity and affinity analysis of aptamers. (A) Specific analysis. (B) Affinity analysis.
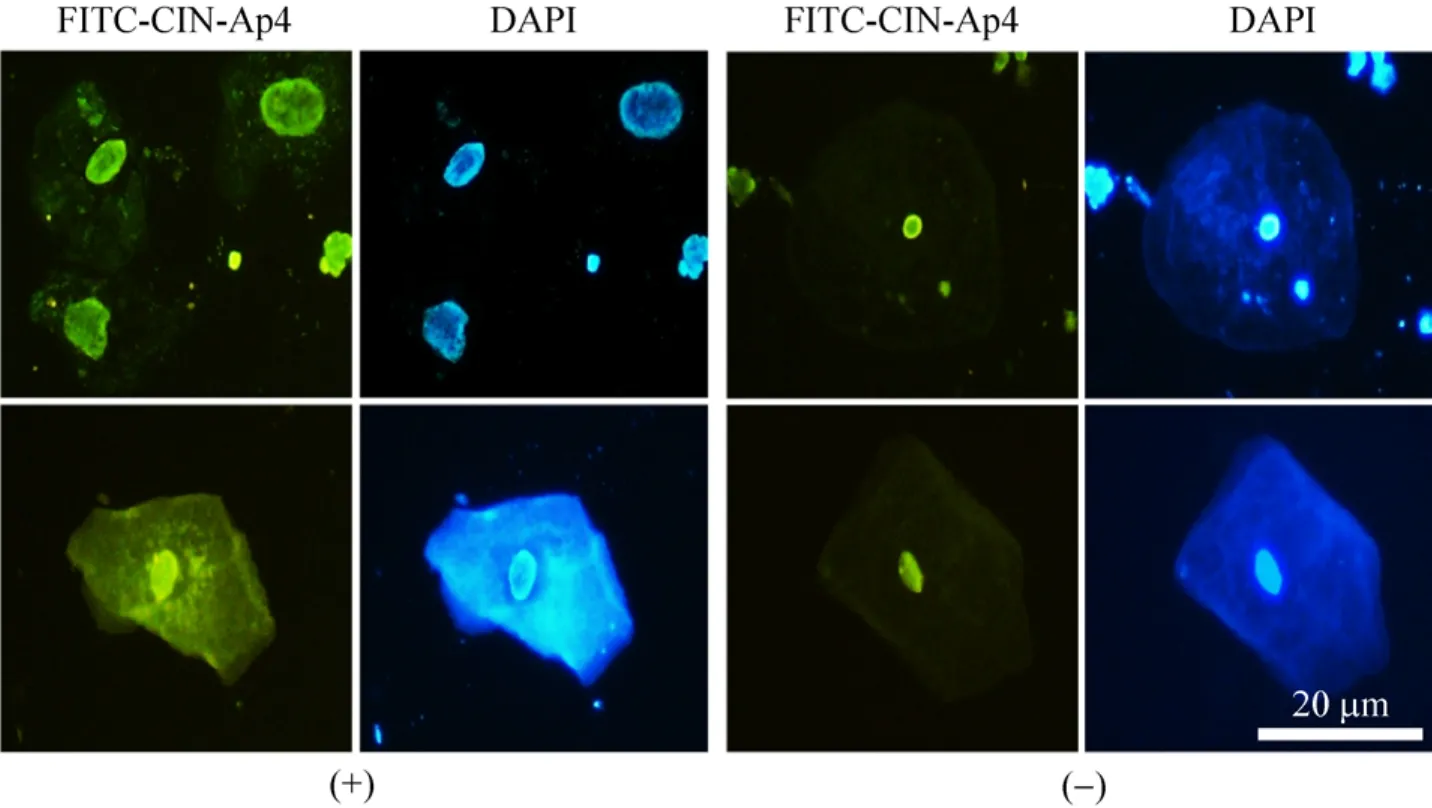
图6 适配子CIN-Ap4细胞免疫荧光结果Fig. 6 Cell immunofluorescence of CIN-Ap4.
3 讨论
对称PCR和不对称PCR是实验过程的关键,对 PCR反应条件进行优化,尤其对不对称 PCR条件进行优化,是获得特异性和亲和力均较强的ssDNA适配子的核心。适配子的筛选过程就是不断重复PCR的过程,所以PCR是筛选适配子的关键。优化PCR条件可以降低非特异性扩增,得到高特异性、高纯度的PCR产物,为今后筛选目标物的适配子奠定一定的基础。但值得一提的是,我们发现在开始的几轮筛选过程中,对称PCR及不对称PCR均能获得较高特异的目的产物。但在后面的几轮筛选过程中,即使在优化的条件下,PCR非特异性产物增多。分析原因可能是筛选之初模板纯度高,所以PCR产物特异性高,但经过多轮筛选后模板纯度降低,故PCR产物特异性降低,尤其是不对称PCR要经过35个如此多的循环,非特异性产物会更多[25-26],因此在筛选过程中可采用多次胶回收的方法以提高每一轮ssDNA纯度。
[1]Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science, 1990, 249(4968):505–510.
[2]Ellington AD, Szostak JW.In vitroselection of RNA molecules that bind specific ligands. Nature, 1990,346(6287): 818–822.
[3]Kou ZW, Wang JP, Zhang XM. Nucleic acid aptamers and its applications in diagnosis and therapy of Alzheimer’s diseases. J Mol Diagn Ther, 2015, 7(4):266–270 (in Chinese).寇哲文, 王家鹏, 张兴梅. 核酸适配子及其在阿尔茨海默病研究中的应用. 分子诊断与治疗杂志, 2015,7(4): 266–270.
[4]Radom F, Jurek PM, Mazurek MP, et al. Aptamers:molecules of great potential. Biotechnol Adv, 2013,31(8): 1260–1274.
[5]Bruno JG. A review of therapeutic aptamer conjugates with emphasis on new approaches. Pharmaceuticals,2013, 6(3): 340–357.
[6]Hernandez LI, Flenker KS, Hernandez FJ, et al. Methods for evaluating cell-specific, cell-internalizing RNA aptamers. Pharmaceuticals, 2013, 6(3): 295–319.
[7]Lee SJ, Kwon YS, Lee JE, et al. Detection of VR-2332 strain of porcine reproductive and respiratory syndrome virus type Ⅱ using an aptamer- based sandwich-type assay. Analytical Chemi, 2013, 85(1): 66–74.
[8]Dong WN, Liao WJ, Lin YP. Aptamers and its review. J Trop Med, 2009, 9(11): 1353–1354 (in Chinese).董婉妮, 廖伟娇, 林勇平. 核酸适配子及其筛选进展.热带医学杂志, 2009, 9(11): 1353–1354.
[9]Bai H, Wang RH, Hargis B, et al. A SPR aptasensor for detection of avian influenza virus H5N1. Sensors, 2012,12(9): 12506–12518.
[10]Zhao ZL, Li Y, Yuan C. Research progress of Cell-SELEX. Biotechnol Bull, 2014 (5): 52–56 (in Chinese).赵仲麟, 李燕, 袁超. Cell-SELEX 技术研究进展. 生物技术通报, 2014 (5): 52–56.
[11]Zhou YC, Cao LT, Chen L, et al. Optimization of asymmetric PCR reaction system and purification of Random single-srtand DNA library. Sichuan Anim Husb Vet Med, 2015, 42(11): 33–35, 37 (in Chinese).周晏丞, 曹立亭, 陈露, 等. 随机 ssDNA文库不对称PCR扩增条件的优化及回收效率比较. 四川畜牧兽医,2015, 42(11): 33–35, 37.
[12]Aptekar S, Arora M, Lawrence CL, et al. Selective targeting to glioma with nucleic acid aptamers. PLoS ONE, 2015, 10(8): e0134957.
[13]Wan J, Ye L, Yang X, et al. Cell-SELEX based selection and optimization of DNA aptamers for specific recognition of human cholangiocarcinoma QBC-939 cells. Analyst, 2015, 140(17): 5992–5997.
[14]Sun H, Zu Y. A highlight of recent advanced in aptamer technology and its application. Molecules, 2015, 20(7):11959–11980.
[15]Colucciello M. Current intravitreal pharmacologic therapies for diabetic macular edema. Postgrad Med,2015, 127(6): 640–653.
[16]Song KM, Lee S, Ban C. Aptamers and their biological applications. Sensors, 2012, 12(1): 612–631.
[17]Tran DT, Janssen KPF, Pollet J, et al. Selection and characterization of DNA aptamers for egg white lysozyme. Molecules, 2010, 15(3): 1127–1140.
[18]Musheev MU, Krylov SN. Selection of aptamers by systematic evolution of ligands by exponential enrichment: addressing the polymerase chain reaction issue. Anal Chim Acta, 2006, 564(1): 91–96.
[19]Tian RY, Lu SY, Li L, et al. Construction of oligonucleotide library in selection of aptamer and optimization of PCR conditions. China Anim Husb Vet Med, 2014, 41(1): 61–65 (in Chinese).田瑞云, 卢士英, 李琳, 等. 适配子筛选中随机寡核苷酸文库的构建和 PCR条件的优化. 中国畜牧兽医,2014, 41(1): 61–65.
[20]Ma ZW, Yuan Y, Chen Q, et al. Genes of HBV C region amplifying and optimization of PCR conditions. J South-Cent Univ Nat: Nat Sci Ed, 2007, 26(1): 30–32(in Chinese).马兆武, 袁媛, 陈强, 等. HBV C区基因扩增与 PCR条件的优化. 中南民族大学学报: 自然科学版, 2007,26(1): 30–32.
[21]Cao LT, Xu LL, Wan X, et al. Optimization of PCR reaction system for random single-strand DNA pool in SELEX technology. J Anhui Agri Sci, 2012, 40(6):3241–3242, 3297 (in Chinese).曹立亭, 许李丽, 万向, 等. SELEX技术中ssDNA文库PCR扩增条件的优化. 安徽农业科学, 2012, 40(6):3241–3242, 3297.
[22]Shao BY, Chen B, Tang MY, et al. Optimization of DNA extraction fromSalmonellaand PCR reaction condition.Food Sci, 2007, 28(7): 331–334 (in Chinese).邵碧英, 陈彬, 汤敏英, 等. 沙门氏菌 DNA 提取及PCR反应条件的优化. 食品科学, 2007, 28(7):331–334.
[23]Wu HB. Selection and characterization of ssDNA aptamers to Ras proteinin vitro[D]. Wuhan: Wuhan University, 2011 (in Chinese).吴红兵. Ras蛋白ssDNA适配子的筛选与鉴定[D]. 武汉: 武汉大学, 2011.
[24]Xu P, Shao MY, Jia RH, et al. Peroxiredoxin-4 as a potential biomarker of early gastric cancer screened by cell-SELEX. Transl Cancer Res, 2017, 6(2): 293–303.
[25]Tolle F, Wilke J, Wengel J, et al. By-product formation in repetitive PCR amplification of DNA libraries during SELEX. PLoS ONE, 2014, 9(12): e114693.
[26]Kalle E, Gulevich A, Rensing C. External and semi-internal controls for PCR amplification of homologous sequences in mixed templates. Microbiol Methods, 2013, 9(95): 285–294.
