干酪乳杆菌细菌素的抗菌机制分析
2018-05-23马佳歌李佳君周诗昊
马佳歌,于 微*,李佳君,周诗昊
(东北农业大学食品学院,黑龙江 哈尔滨 150030)
在食品加工及贮运过程中,腐败菌和致病微生物的生长繁殖会严重威胁食品安全。食品安全与人类健康息息相关,开发可靠、有效的天然防腐剂是目前亟待解决的问题。细菌素是细菌在代谢过程中通过核糖体产生的具有抗菌活性的多肽或蛋白质类化合物,产生细菌素的细菌存在免疫特性,不会伤害菌株本身[1-3]。干酪乳杆菌(Lactobacillus casei)被美国食品与药品监督管理局公认为安全的一种益生菌[4-6],其所产的细菌素具有安全无毒等特性[7-8]。目前商业化的乳酸菌细菌素Nisin由于热不稳定、抑菌谱窄、pH值范围窄、产量低等原因,在食品工业中的应用受到限制[9-10],因此,开发一种应用范围广阔的新型生物防腐剂备受关注,干酪乳杆菌所产的细菌素作为一种新型乳酸菌细菌素受到广泛重视[11-13]。
课题组前期从发酵乳制品中分离出具有抑菌活性的干酪乳杆菌KLDS 1.0338,采用聚合酶链式反应(polymerase chain reaction,PCR)扩增出class IIb细菌素编码基因lacA和lacB,利用重叠延伸PCR技术扩增两端带有酶切位点的重组细菌素基因,从而构建克隆质粒pGEX-4T-1-lacAB。本研究运用生物信息学方法分析干酪乳杆菌细菌素基因及其编码蛋白的结构及特征功能,采用氨基酸定点突变技术构建重组细菌素突变载体,分析细菌素突变体抗菌活性,推测干酪乳杆菌细菌素发挥抑菌活性的关键氨基酸位点,为进一步探索干酪乳杆菌细菌素抗菌机制提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
干酪乳杆菌KLDS1.0338由东北农业大学乳品科学教育部重点实验室工业微生物菌种保藏中心提供;重组质粒pGEX-4T-1-lacAB由东北农业大学乳品科学教育部重点实验室构建;大肠杆菌(Escherichia coli BL21)感受态细胞购自宝生物(大连)有限公司。
1.1.2 试剂
溶菌酶、凝血酶、乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA)、Tris-base上海阳光生物科技有限公司;Triton-X 上海索宝生物科技有限公司;溴酚蓝 北京百奥莱博科技有限公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG) 上海宝曼生物技术有限公司;蛋白Marker宝生物(大连)有限公司;GST融合蛋白纯化试剂盒美国Axygen公司;QuikChange Site-Directed Mutagenesis试剂盒 美国Stratagene公司。
1.2 仪器与设备
SW-CJ-IFD超净工作台 苏州安泰空气技术有限公司;ULT1386-3-V30超低温冰箱 日本Sanyo公司;DH4000A电热恒温培养箱 天津市泰斯特仪器有限公司;GDS-8000凝胶成像系统 美国UVP公司;T-Gradient 96 Thermoblock Modul PCR仪 德国Biometra公司;WH-851旋涡混合器 上海科达测试仪器厂;CHB-100恒温金属浴 杭州博日公司;Z300-K冷冻离心机德国Hermle公司。
1.3 方法
1.3.1 生物信息学分析
根据获得的编码干酪乳杆菌细菌素class IIb类型的相关基因序列,应用DNAMAN软件分别将细菌素lacA和lacB的核酸序列翻译成氨基酸序列,获得蛋白质的基本信息。利用瑞士生物信息学研究所的蛋白组学分析平台(ExPASy)ProtParam tool在线程序(http://web.expasy.org/protparam/)进行蛋白质理化性质分析[14];使用ProtScale 软件(http://web.expasy.org/cgibin/protscale/protscale.pl)预测蛋白质的疏水性和亲水性[15];利用网址http://www.cbs.dtu.dk/services/NetPhos/进行蛋白质磷酸化分析;利用TMHMM Server v.2.0软件(http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测蛋白质的跨膜结构域;利用SOPMA程序(http://npsapbil.ibcp.fr/cgibin/npsa_automat.pl?page=npsa_gor4.html)预测蛋白质的二级结构;采用CBS中的SignalP 4.1 Server软件(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽[16];蛋白质三级结构预测采用http://swissmodel.expasy.org/分析。
1.3.2 细菌素突变表达载体构建
依据细菌素LacA和LacB的生物信息学分析结果和细菌素第2类型(class IIb)抑菌结构GxxxG,选择LacA蛋白中40位V和57位I为突变位点进行定点突变,并设计突变引物,分别是P1/P2和P3/P4。将实验室保存的重组质粒pGEX-4T-1-lacAB作为氨基酸定点突变的DNA模板,氨基酸定点突变PCR扩增体系(50.0 μL):ddH2O 35.5 μL,10×Taq DNA polymerase buffer 5.0 μL,2.5 mmol/L dNTP 4.0 μL,DNA模板1.0 μL,10 μmol/L上游引物2.0 μL,10 μmol/L下游引物2.0 μL,2.5 U/μL Taq DNA polymerase 0.5 μL。PCR条件:95 ℃预变性30 s;95 ℃变性30 s,55 ℃退火1 min,68 ℃延伸2 min/kb,30 个循环。按照QuikChange Site-Directed Mutagenesis试剂盒的实验操作方法,稍作改动[17]。
酶切鉴定:PCR产物冰上孵育5 min,使其温度降至37 ℃以下,用1 µL Dpn I酶单酶切,37 ℃过夜反应,酶切产物用1%琼脂凝胶电泳检测。
引物如下(下划线位点为碱基突变处):
P1:5-CGATCGGGGGAGCGGCAGCAGGCGCAGCTGG-3
P2:5-CCAGCTGCGCCTGCTGCCGCTCCCCCGATCG-3
P3:5-GTTGCAGGCGCTGCAGCAGGATTATCCTTGTG-3
P4:5-CACAAGGATAATCCTGCTGCAGCGCCTGCAAC-3
1.3.3 细菌素突变体的表达及纯化
经鉴定构建正确的突变载体涂布于LB固体平板上,37 ℃培养过夜,按1∶50的比例(200 μL)接种于10 mL LB培养基中扩培,使菌液OD600nm达0.8(使大肠杆菌处于最适合表达外源蛋白的生长状态)。加入IPTG后,37 ℃诱导表达3 h。5 000 r/min、4 ℃离心10 min收集菌体,充分洗涤数次并以8 000 r/min离心2 min。向收集到的菌体中加入细菌裂解液,于4 ℃裂解10 min。
将重组蛋白与GSH-Sepharose 4B悬液混合,于室温作用10 min离心,将悬液全部加入离心柱上,经漂洗和重复洗脱,得到纯化的重组蛋白。采用凝血酶切割重组融合蛋白后,用GST纯化试剂盒纯化去除标签,从而获得单一的目的蛋白LacAB。通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)检测纯化效果。
1.3.4 细菌素突变体抑菌活性检测
牛津杯琼脂扩散法[18-19]:选择金黄色葡萄球菌ATCC25923和大肠杆菌ATCC25922为指示菌种,将过夜培养的指示菌悬液均匀涂于NB平板上,无菌条件下干燥30 min后,将内径为6 mm的牛津杯均匀放置在NB平板上,牛津杯孔内滴入200 μL上清液,37 ℃培养24 h,以抑菌圈直径大小表示抑菌活性强弱[20]。
血球计数板计数法:将指示菌37 ℃培养至菌落数达到106CFU/mL,取1 mL指示菌悬液,加入适量含重组细菌素溶液,至细菌素终质量浓度为1 mg/mL,37 ℃共同培养6 h后,用生理盐水适当稀释菌液,并用血球计数板计算活菌数,实验均重复3 次。
2 结果与分析
2.1 细菌素生物信息学分析
2.1.1 一级结构分析
利用Primer 5.0软件推测lacA开放阅读框编码的蛋白如下:MISKEVGITLKQHDLVLIQRGAKRRNKPSGCIVS TIGGAVAGAAGLNPFTTVAGAAIGLSLCLSTNYIHPA。由蛋白质一级序列分析可知:氨基酸残基数为71,其中包含有8 个碱性氨基酸R 、K、H,1个酸性氨基酸D,26 个亲水性氨基酸 D、E、H、K、Q、R、S、T、N,34 个疏水性氨基酸A、F、I、L、M、P、V、W、Y。
利用Primer 5.0软件推测lacB开放阅读框编码的蛋白如下:MSYNYRQGLDDFQLSGVSGGKKKFDCAATFY TGITDGIGSGTITGLAGGPFGIIGGAVVGGNLGAVGSA IKCLGDGMQ。由蛋白质一级序列分析可知:氨基酸残基数为77,其中包含有5 个碱性氨基酸R、K、H,5 个酸性氨基酸D,23 个亲水性氨基酸D、E、H、K、Q、R、S、T、N,31 个疏水性氨基酸A、F、I、L、M、P、V、W、Y。
2.1.2 理化性质分析
通过ProtParam分析干酪乳杆菌细菌素(class IIb)中LacA和LacB两段氨基酸序列(表1)。依据稳定指数小于40,亲水性平均系数为正值且脂肪指数较高,推测LacA和LacB属于稳定、耐热、疏水性蛋白[21]。
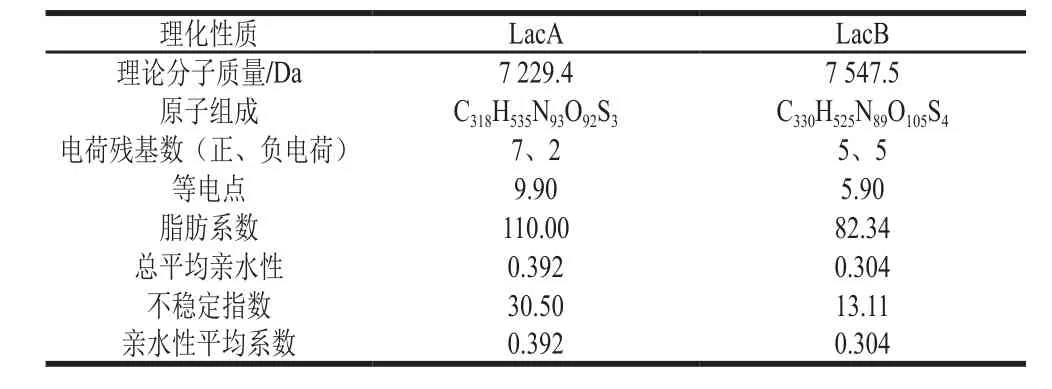
表1 干酪乳杆菌细菌素氨基酸序列分析结果Table 1 Amino acid sequence analysis of bacteriocin from Lactobacillus casei
2.1.3 跨膜结构分析
应用TMHMM2.0预测干酪乳杆菌细菌素的跨膜结构,结果如图1所示,LacA、LacB蛋白具有跨膜结构特点,符合细菌素跨过细胞膜分泌蛋白的作用,同时这也与细菌素与靶细胞膜上的特殊位点相互作用相一致,这种作用可以提高细菌素的有效活性。
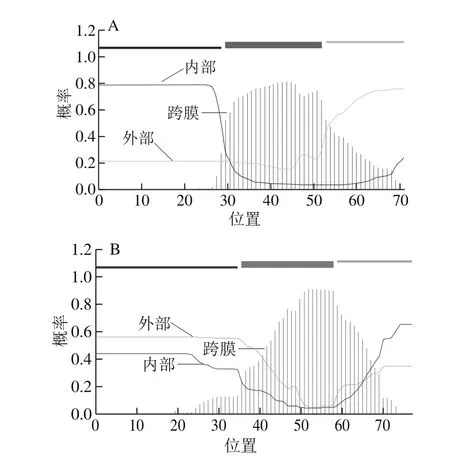
图1 LacA(A)和LacB(B)的跨膜区分析Fig. 1 Analysis of transmembrane area
2.1.4 磷酸化位点分析
应用NetPhos2.0 Server预测细菌素蛋白序列中Ser、Thr和Tyr三种氨基酸残基可能成为的磷酸化位点,如图2所示,分析结果表明LacA中有1 个Thr氨基酸残基磷酸化位点,LacB中无磷酸化位点。由此说明,LacA翻译后进行修饰,推测出该蛋白序列的修饰使结构发生改变有利于提高抗菌肽活性。
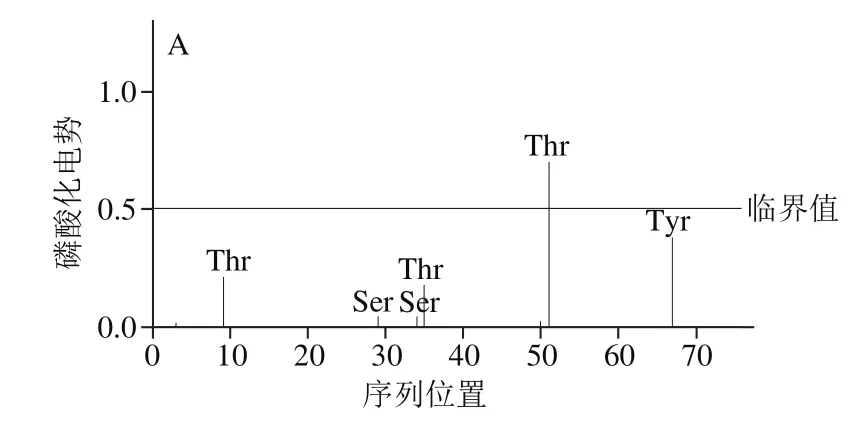
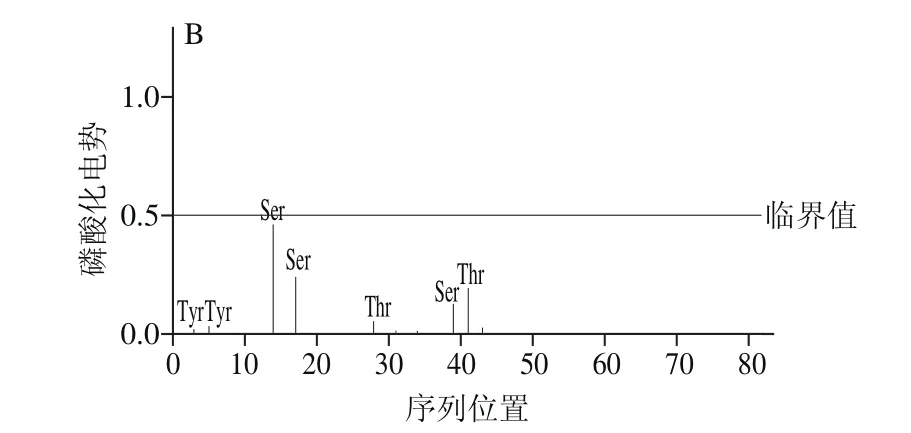
图2 LacA(A)和LacB(B)的磷酸化位点分析Fig. 2 Analysis of phosphorylation sites
2.1.5 信号肽预测结果
信号肽是引导新合成的蛋白质向分泌通路转移的短肽链,其长度约5~30 个氨基酸,这类肽段通常不能存在于成熟蛋白中。如图3所示,LacA的C值、Y值和S值分别在52、33、30位点,分别为0.362、0.508、0.911,预测含有信号肽,切割位点位于28~29氨基酸残基之间。LacB的C值、Y值和S值分别在29、29、19位点,分别为0.415、0.610、0.933,预测含有信号肽,切割位点位于32~33氨基酸残基之间。

图3 LacA(A)和LacB(B)的信号肽预测Fig. 3 Prediction of signal peptide
2.1.6 蛋白质结构预测结果
二级结构中LacA以无规则卷曲为主,α-螺旋、β-折叠、无规则卷曲分别占23.94%、19.72%、56.34%;LacB以无规则卷曲为主,β-折叠和无规则卷曲的比例分别为37.66%、62.34%。结果显示LacA和LacB具备抗菌肽二级结构的特点[22]。
利用Espasy工具中的SWISS-MODLE软件对干酪乳杆菌细菌素LacA和LacB蛋白的三维立体结构进行在线预测,如图4所示。LacA主要由两端松散肽链和中间的α-螺旋构成(图4A),α-螺旋结构中存在GxxxG结构域[23-24],LacB有两个反向的β-折叠和一个α-螺旋,并且两个β-折叠由一段松散肽链组成的铰链区连接(图4B)。
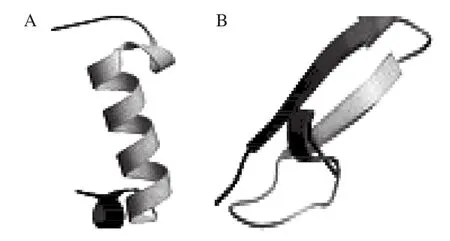
图4 LacA(A) 和LacB(B)的三维结构预测Fig. 4 Predicted tertiary structure of bacteriocin
2.2 氨基酸定点突变
2.2.1 突变载体的PCR扩增
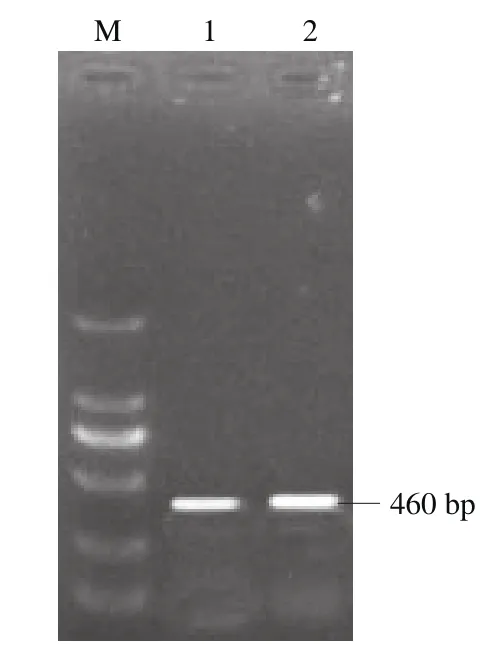
图5 细菌素突变载体PCR扩增结果Fig. 5 Results of PCR amplification of mutant bacteriocin

图6 细菌素突变载体1(A)和突变载体2(B)测序结果Fig. 6 Sequence of mutant plasmid vectors
分别以P1/P2、P3/P4为引物,用实验已构建好的细菌素表达载体为模板,进行细菌素突变载体的PCR扩增,如图5所示。将突变载体转入大肠杆菌BL21中,挑选阳性克隆进行测序、比对,如图6所示,结果表明目的蛋白突变位点发生改变,其余各点均未发生改变,说明突变载体构建成功。
2.2.2 细菌素突变体的诱导表达

图7 细菌素突变体1诱导表达Fig. 7 SDS-PAGE analysis of induced expression of mutant bacteriocin 1
以突变体1为代表,如图7所示,表明已成功诱导表达出突变体细菌素蛋白,氨基酸定点突变没有改变其表达条件与特性。经SDS-PAGE鉴定,诱导的重组蛋白增加一条47 ku的蛋白条带,正好是空载体和目的蛋白之和,说明重组蛋白能在大肠杆菌BL21中成功表达,氨基酸定点突变没有改变其表达条件与特性。
2.2.3 细菌素突变体的纯化
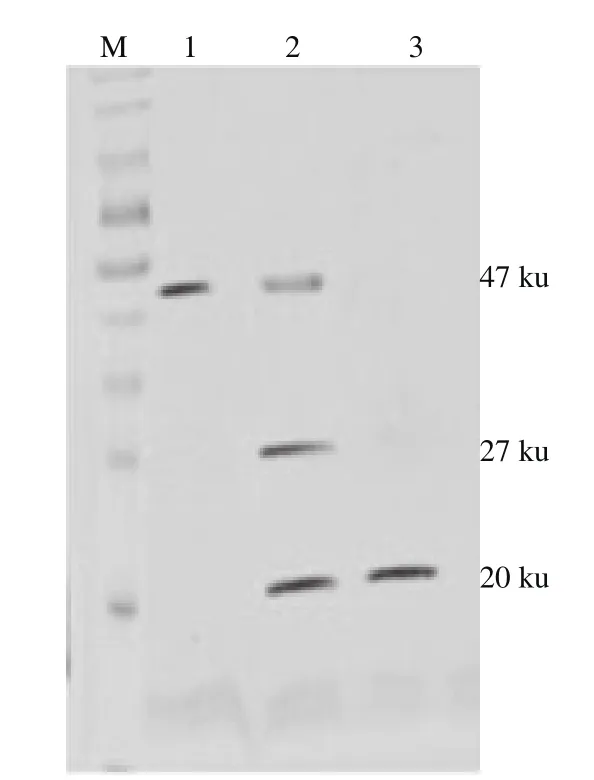
图8 突变体1细菌素纯化SDS-PAGEFig. 8 SDS-PAGE analysis of purified recombinant mutant bacteriocin 1
将诱导表达出的突变体细菌素按1.3.3节方法进行纯化,加入凝血酶切除GST标签,通过洗脱液将目的蛋白从纯化柱上洗脱下来,如图8所示。泳道2与未突变的重组细菌素基本一致,凝血酶切掉了大部分GST标签,但仍有部分带标签的突变体细菌素没有被完全切开。经牛血清白蛋白浓度测定,突变体1和2的质量浓度分别为1.78 mg/mL和1.82 mg/mL,与未突变重组细菌素质量浓度很接近,说明氨基酸定点突变未影响突变体细菌素的表达。
2.2.4 细菌素突变体的抑菌活性
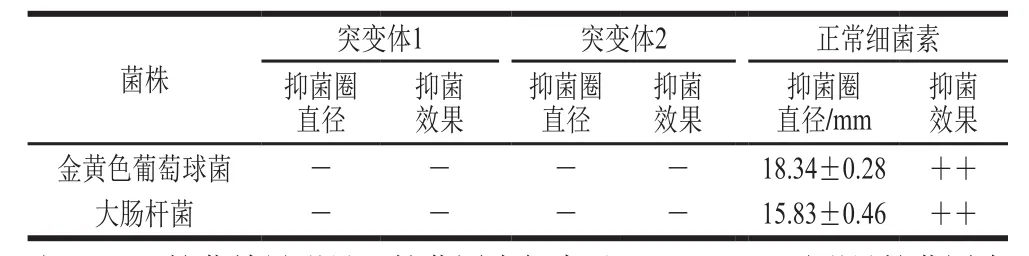
表2 牛津杯琼脂扩散法检测突变体细菌素抑菌活性Table 2 Antimicrobial spectrum and activity of mutant bacteriocins
采用牛津杯琼脂扩散法检测突变体细菌素抑菌活性,结果如表2所示。正常未突变的细菌素对金黄色葡萄球菌和大肠杆菌均有明显的抑菌活性,而突变体1和2对金黄色葡萄球菌和大肠杆菌均无明显的抑菌圈。结果表明,两个突变体均因为氨基酸的定点突变而失去了抑菌活性。

表3 血球计数板计数法检测突变体细菌素抑菌活性Table 3 Antibacterial activity of mutant bacteriocins
采用血球计数板计数法检测突变体细菌素对抑菌活性的影响,以不加细菌素作为阴性对照,如表3所示。结果表明,突变体1和2这两组的活菌数显著大于正常细菌素组,与阴性对照组活菌数无显著性差异,这说明本实验所突变的两个氨基酸均对重组细菌素的抗菌活性有关键性的作用,且这两个氨基酸突变任意一个均会导致重组细菌素失活。
3 结 论
利用生物信息学方法对基因和蛋白序列进行预测、分析,建立理论模型,寻找新型细菌素成为可能[25-27]。分析获得干酪乳杆菌细菌素LacA蛋白具有松散-螺旋-松散的结构,LacB蛋白具有折叠-铰链区-折叠的结构,以及明显的跨膜区域,符合class II细菌素的结构特征,稳定、耐热和疏水的理化性质及结构特征是细菌素发挥抑菌作用所必需的。
通过干酪乳杆菌细菌素结构分析,细菌素存在GxxxG结构域,这种结构代表跨膜螺旋[28-29],在LacA中α-螺旋涉及GxxxG结构,推测LacA中第38~42位氨基酸的GAVAG序列和第54~58位氨基酸的GAAIG序列均对细菌素的抑菌活性有关键作用。
体外定点突变技术是研究蛋白质结构与功能的相互作用的主要方法[30],同时也是蛋白质组学研究的基础。采用氨基酸定点突变技术,选择LacA蛋白氨基酸序列中的第40位的V和第57位的I,结果表明突变重组细菌素失去抑菌活性,无明显抑菌效果,推测V40和I57是重组细菌素发挥抑菌活性的关键氨基酸位点。说明这两个位点及其构成的结构域对细菌素发挥抗菌活性的必要性,需要两个关键位点和结构域同时正确,细菌素才能发挥抑菌活性。
参考文献:
[1]BOWDISH D M, DAVIDSON D J, HANCOCK R E. A re-evaluation of the role of host defence peptides in mammalian immunity[J]. Current Protein &Peptide Science, 2005, 6(1): 35-51. DOI:10.2174/1389203053027494.
[2]DEEGAN L H, COTTER P D, HILL C. Bacteriocins: biological tools for bio-preservation and shelf-life extension[J]. International Dairy Journal, 2006, 16(9): 1058-1071. DOI:10.1016/j.idairyj.2005.10.026.
[3]MESSAOUDI S, KERGOURLAY G, DALGALARRONDO M,et al. Purification and characterization of a new bacteriocin active against Campylobacter produced by Lactobacillus salivarius SMXD51[J]. Food Microbiology, 2012, 32(1): 129-134. DOI:10.1016/j.fm.2012.05.002.
[4]VALENZUELA J F, PINUER L A, CANCINO A G, et al. Metabolic fluxes in lactic acid bacteria: a review[J]. Food Biotechnology, 2015,29(2): 185-217. DOI:10.1080/08905436.2015.1027913.
[5]REVILLAGUARINOS A, GEBHARD S, ALCÁNTARA C, et al. Characterization of a regulatory network of peptide antibiotic detoxification modules in Lactobacillus casei BL23[J]. Applied &Environmental Microbiology, 2013, 79(10): 3160-3170. DOI:10.1128/AEM.00178-13.
[6]JOVANOVIĆ J N, NIKOLIĆ B, ŠEATOVIĆ S, et al. Characterization of some potentially probiotic Lactobacillus, strains of human origin[J]. Food Science and Biotechnology, 2015, 24(5): 1781-1788.DOI:10.1007/s10068-015-0232-7.
[7]AWAISHEH S S, ALNABULSI A A, OSAILI T M, et al. Inhibition of Cronobacter sakazakii by heat labile bacteriocins produced by probiotic LAB isolated from healthy infants[J]. Journal of Food Science, 2013, 78(9): 1416-1420. DOI:10.1111/1750-3841.12209.
[8]MILLETTE M, LUQUET F M, LACROIX M. In vitro growth control of selected pathogens by Lactobacillus acidophilus- and Lactobacillus casei-fermented milk[J]. Letters in Applied Microbiology, 2007, 44(3):314-319. DOI:10.1111/j.1472-765X.2006.02060.x.
[9]丹彤, 张和平. 乳酸菌细菌素的分类、生物合成及其应用[J]. 中国乳品工业, 2013, 41(3): 29-32. DOI:10.3969/j.issn.1001-2230.2013.03.007.
[10]SOBRINOLÓPEZ A, MARTÍNBELLOSO O. Use of nisin and other bacteriocins for preservation of dairy products[J]. International Dairy Journal,2008, 18(4): 329-343. DOI:10.3969/j.issn.1001-2230.2013.03.007.
[11]YANG E, FAN L, JIANG Y, et al. Antimicrobial activity of bacteriocinproducing lactic acid bacteria isolated from cheeses and yogurts[J]. AMB Express, 2012, 2(1): 1-12. DOI:10.1186/2191-0855-2-48.
[12]ZAVISIC G, PETRICEVIC S, RADULOVIC Z, et al. Probiotic features of two oral Lactobacillus isolates[J]. Brazilian Journal of Microbiology,2012, 43(1): 418-428. DOI:10.1590/S1517-83822012000100050.
[13]YANG E, FAN L, JIANG Y, et al. Antimicrobial activity of bacteriocinproducing lactic acid bacteria isolated from cheeses and yogurts[J]. AMB Express, 2012, 2(1): 1-12. DOI:10.1186/2191-0855-2-48.
[14]ROY S, MAHESHWARI N, CHAUHAN R, et al. Structure prediction and functional characterization of secondary metabolite proteins of Ocimum[J]. Bioinformation, 2011, 6(8): 315-319.DOI:10.6026/97320630006315.
[15]SALAHUDDIN P, KHAN A U. Structure function studies on different structural domains of nucleoprotein of H1N1 subtype[J].Bioinformation, 2010, 5(1): 28-30. DOI:10.6026/97320630005025.
[16]CALL E K, GOH Y J, SELLE K, et al. Sortase-deficient lactobacilli:effect on immunomodulation and gut retention[J]. Microbiology, 2015,161(2): 311-321. DOI:10.1099/mic.0.000007.
[17]XIA Y, CHU W, QI Q, et al. New insights into the quik change TM process guide the use of phusion DNA polymerase for sitedirected mutagenesis[J]. Nucleic Acids Research, 2014, 43(2): e12.DOI:10.1093/nar/gku1189.
[18]刘友华. 新型琼脂扩散法检测食品中天然防腐剂ε-聚赖氨酸含量[J].食品科学, 2015, 36(8): 225-230. DOI:10.7506/spkx1002-6630-201508042.
[19]李南薇, 李宁. 乳酸菌代谢产物对大肠杆菌和金黄色葡萄球菌抑制作用的研究[J]. 中国酿造, 2009, 28(5): 49-52. DOI:10.3969/j.issn.0254-5071.2009.05.015.
[20]ROJOBEZARES B, SÁENZ Y, NAVARRO L, et al. Cocultureinducible bacteriocin activity of Lactobacillus plantarum strain J23 isolated from grape must[J]. Food Microbiology, 2007, 24(5): 482-491. DOI:10.1016/j.fm.2006.09.003.
[21]李芬, 孙大庆, 张丽萍. 植物乳杆菌LY-78乳酸脱氢酶基因的生物信息学分析[J]. 食品科学, 2017, 38(8): 102-106. DOI:10.7506/spkx1002-6630-201708017.
[22]PEREA M, LUGTENBURG I, MAYOL E, et al. TMalphaDB and TMbetaDB: web servers to study the structural role of sequence motifs in α-helix and β-barrel domains of membrane proteins[J]. BMC Bioinformatics, 2015, 16(1): 266. DOI:10.1186/s12859-015-0699-5.
[23]NISSENMEYER J, OPPEGARD C, ROGNE P, et al. Structure and modeof-action of the two-peptide (class-IIb) bacteriocins[J]. Probiotics and Antimicrobial Proteins, 2010, 2(1): 52-60. DOI:10.1007/s12602-009-9021-z.
[24]OPPEGARD C, SCHMIDT J, KRISTIANSEN P E, et al. Mutational analysis of putative helix-helix Interacting GxxxG-motifs and tryptophan residues in the two-peptide bacteriocin lactococcin G[J].Biochemistry, 2008, 47(18): 5242-5249. DOI:10.1021/bi800289w.
[25]WIECKOWICZ M, SCHMIDT M, SIP A, et al. Development of a PCR-based assay for rapid detection of class IIa bacteriocin genes[J].Letters in Applied Microbiology, 2011, 52(3): 281-289. DOI:10.1111/j.1472-765X.2010.02999.x.
[26]MACWANA S J, MURIANA P M. A ‘bacteriocin PCR array’ for identification of bacteriocin-related structural genes in lactic acid bacteria[J]. Journal of Microbiological Methods, 2012, 88(2): 197-204.DOI:10.1016/j.mimet.2011.11.008.
[27]BORRERO J, JIMÉNEZ J J, GÚTIEZ L, et al. Protein expression vector and secretion signal peptide optimization to drive the production, secretion, and functional expression of the bacteriocin enterocin A in lactic acid bacteria[J]. Journal of Biotechnology, 2011,156(1): 76-86. DOI:10.1016/j.jbiotec.2011.07.038.
[28]OPPEGARD C, ROGNE P, KRISTIANSEN P E, et al. Structure analysis of the two-peptide bacteriocin lactococcin G by introducing D-amino acid residues[J]. Microbiology, 2010, 156(6): 1883-1889.DOI:10.1099/mic.0.038430-0.
[29]NISSENMEYER J, ROGNE P, OPPEGARD C, et al. Structurefunction relationships of the non-lanthionine-containing peptide(class II) bacteriocins produced by gram-positive bacteria[J].Current Pharmaceutical Biotechnology, 2009, 10(1): 19-37.DOI:10.2174/138920109787048661.
[30]雒丽娜, 王盛, 王玉炯. 利用重叠延伸PCR技术进行定点突变研究[J].安徽农业科学, 2012, 40(10): 719-722. DOI:10.3969/j.issn.1009-4229-B.2012.04.009.
