基于16S rRNA高通量测序方法比较新疆冷水鱼肠道中微生物多样性
2018-05-23黄丽丽倪永清
黄丽丽,张 艳,周 红,倪永清*
(石河子大学食品学院,新疆 石河子 832000)
脊椎动物的肠道内寄居着大量微生物,形成了复杂的微生物区系,这是动物长期进化的结果[1]。而鱼类是世界上最古老的脊椎动物,胃肠道上皮黏膜附着各种复杂的微生物[2-3],对鱼类的消化吸收、生产性能和免疫系统起着至关重要的作用[4-5]。刚孵化鱼的胃肠道通常是无菌的[6],但从鱼卵中孵化后的仔鱼慢慢开始捕食和饮水[6-7],由此食物、水以及水底淤泥等生存环境中的微生物就得以进入鱼类的消化道[6,8-9],成为鱼类消化道微生物区系的最初来源。然而,鱼肠道内的环境却又不同于鱼类栖息的环境,只有能适应鱼肠道环境和宿主免疫系统的一些微生物才能够定植于鱼肠道内[10]。在鱼类生长发育的过程中,鱼肠道逐渐形成了一个由好氧菌、厌氧菌组成的动态菌群,是一个复杂的微生态系统[11-12]。研究证明仔鱼的免疫系统尚未完全发育,形成了不稳定的微生物区系,相对而言,成鱼的消化系统已发育成熟,肠道微生物区系比较稳定[13]。正常情况下,鱼肠道中的各种微生物生长良好,有益微生物菌群占主导地位以维持肠道的微生态平衡。但当鱼类受到某种刺激(如饵料或环境变更)时,这种平衡状态将会受到破坏,通常会导致细菌性疾病的暴发[1,14],特别是在仔鱼时期。鱼类肠道中微生物菌群是由鱼类、微生物和环境彼此相互依赖、相互作用形成的统一整体,所以鱼肠道微生物群落的结构与组成容易受到外界环境、饮食和种系关系等众多因素的影响[9,15-17]。
肠道菌群是宿主不可或缺的一部分[2],也一直是科学界研究的热点。过去几十年,许多研究用传统的分离与培养、构建克隆文库、变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、温度梯度凝胶电泳(temperature gradient gel electrophoresis,TGGE)、末端限制性片段长度多态性(terminal restriction fragment length polymorphism,T-RFLP)、荧光原位杂交(fluorescence in situ hybridization,FISH)等方法揭示肠道中微生物的多样性[18-21],但是这些方法极可能在很大程度上低估肠道微生物的物种组成并高估其丰度,特别是鱼肠道微生物区系复杂多样且需厌氧环境。随着科学技术的日益发展,相对于传统的微生物鉴别和分析方法,高通量测序技术作为新一代的测序方法,具有较高准确性和灵敏度等优势[17,22-23],被广泛应用到肠道微生物等研究领域,从而极大地提高了对样品中微生物多样性分析的深度和广度,加深人们对肠道微生物菌群的结构和功能的了解。
额尔齐斯河流经新疆阿勒泰地区,是我国唯一一条流入北冰洋的外流河,也是新疆的第二大河流,地处北纬45°以上的中高纬度地区,属于中温带大陆性气候,生态环境复杂。其水量主要靠冰雪融水补给,水温常年在20 ℃以下,河流中栖居着丰富的冷水性鱼类。由于常年低温,使得河流中冷水鱼的肠道形成了独特的低温环境,为肠道内的低温微生物提供了良好的生存保障,也为有关冷水鱼肠道低温微生物的研究提供了有利的条件。目前关于肠道微生物的报道主要集中在陆生脊椎动物的肠道微生物,对鱼肠道微生物菌群的研究较少,尤其是新疆特色的野生冷水鱼。本研究以新疆额尔齐斯河流域的3 种野生冷水鱼为样本,通过揭示其肠道微生物组成差异及多样性,以期为额尔齐斯河冷水鱼肠道微生态相关研究和冷水鱼肠道低温微生物资源开发利用提供理论支持。
1 材料与方法
1.1 材料与试剂
样品1:哲罗鱼(Hucho taimen),编号ZL;样品2:细鳞鱼(Qinling lenok),编号XL;样品3:黑斑狗鱼(Esox reicherti),编号BG,3 种冷水鱼都属于鲑形目。
DNA提取试剂盒 德国Qiagen公司;相关生理生化试验试剂均购自天津市巴斯夫化学试剂厂。
1.2 仪器与设备
Fresco21型高速冷冻离型机、NanoDrop 2000超微量紫外分光光度计 美国Thermo公司;Gel DOC XR凝胶成像系统 美国Bio-Rad公司。
1.3 方法
1.3.1 样品采集及前处理
2014年8月从新疆额尔齐斯河流域采集冷水鱼样品,每种鱼2~3 条(体质量(2.00±0.5)kg,年龄约3 a),标记后置于4 ℃车载冰箱内冷藏,12 h内运回实验室。将运回实验室的冷水鱼样品立即用70%乙醇溶液漂洗后,在超净工作台上用灭菌的手术刀进行解剖,完整地取出鱼肠道。再次用70%乙醇溶液擦拭鱼肠道外表面,待乙醇晾干后获取冷水鱼肠道的内容物和黏膜液(标记为C、M),并置于4 ℃条件下保存备用。
1.3.2 总DNA提取与检测
冷水鱼肠道样品先用灭菌的磷酸盐缓冲液(phosphate buffer saline,PBS(pH 7.4))充分洗涤,洗涤液在4 ℃、2 000 r/min条件下离心5 min,之后取上清液于4 ℃、10 000 r/min离心10 min,收集沉淀。收集的沉淀再次用PBS充分洗涤,并移入干净的离心管中,按照TIANamp Stool DNA Kit试剂盒要求进行操作,完成冷水鱼肠道样品中细菌DNA的提取,分别使用0.8%琼脂糖凝胶电泳和超微量紫外分光光度计检测提取的 DNA,最后将DNA溶液于-20 ℃保存。
1.3.3 16S rDNA聚合酶链式反应(polymerase chain reaction,PCR)扩增及测序
针对16S rDNA V4区片段进行特异性PCR扩增,提取的DNA作为模板,以515F(5’-GTGCCAGCMGCCGCGGTAA-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)为引物,下游引物引入barcode。PCR扩增体系:5Prime Hot Master Mix 10 μL,引物各0.5 μL,DNA模板1 μL,最后补足ddH2O至25 μL。反应条件:94 ℃预变性3 min;94 ℃ 45 s,50 ℃ 60 s,72 ℃ 90 s,35 次循环;72 ℃终延伸10 min[23]。
每个样品PCR扩增3 份,扩增产物物通过琼脂糖凝胶电泳,TEANgel Midi Purification Kit试剂盒回收,用超微量紫外分光光度计上精确定量,按物质的量比例混合后在Miseq测序平台进行测序。对于测序的原始序列,利用QIIME软件进行去杂、修剪得到可精确分析的优化序列,并与Silva数据库进行相似性比对,在97%的相似性水平上归类和确定分类单位(operational taxonomic unit,OTU)数量,比较分析样品中微生物群落结构的结构及组成。
2 结果与分析
2.1 冷水鱼肠道中DNA提取与定量
提取3 种冷水鱼肠道内容物和黏膜的DNA,测定其质量浓度与纯度,并观察OD260nm/OD280nm值(表1),以便用于分析冷水鱼肠道的细菌多样性。
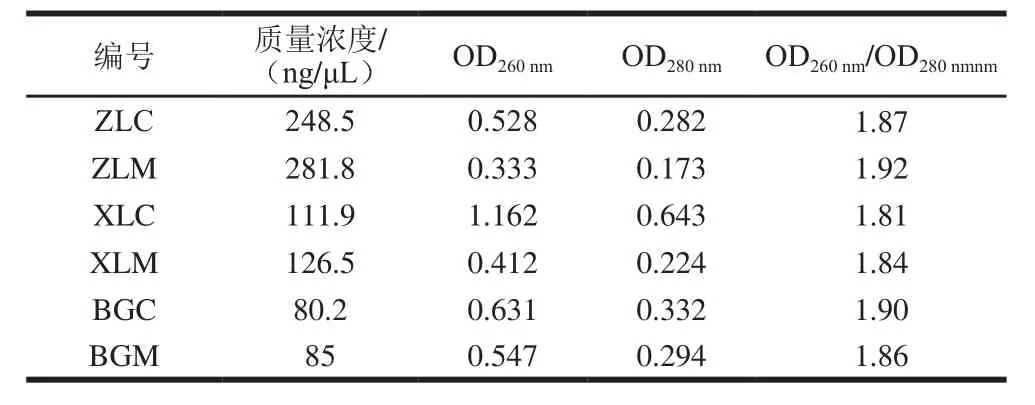
表1 冷水鱼肠道DNA质量浓度分析Table 1 DNA concentration from the intestinal tract of six cold-water fishes
2.2 冷水鱼肠道中微生物群落组成分析
从6 个样品共得到高质量质控序列58 328 条。在97%的相似水平上进行OTU序列分类,总共聚成242 个OTU。依据划分的OTU,比较冷水鱼样品中细菌群落结构的相似性。由图1可知,不同种类的冷水鱼样品聚集在不同的区域内。哲罗鱼和细鳞鱼聚集在图1的右侧,而黑斑狗鱼在左侧部分,构成一个独立的组群,表明黑斑狗鱼和哲罗鱼、细鳞鱼具有明显的差异性。而主成分2反映出每种冷水鱼样品的肠道内容物和黏膜均呈正相关,这与聚类结果一致,表明冷水鱼肠道内容物和黏膜样本尽管存在差异,但具有一定的相似性。
从6 个冷水鱼样品中共鉴定出17 个细菌门,分别是酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、蓝菌门(Cyanobacteria)、厚壁菌门(Firmicutes)、梭菌门(Fusobacteria)、硝化螺菌门(Nitrospira)、浮霉菌门(Planctomycetes)、α-变形菌门(Alphaproteobacteria)、β-变形菌门(Betaproteobacteria)、δ-变形菌门(Deltaproteobacteria)、ε-变形菌门(Epsilonproteobacteria)、丙型变形菌门(Gammaproteobacteria)、互养菌门(Synergistetes)、疣微菌门(Verrucomicrobia)、未分类菌群(Others)。如图2所示,相对丰度较高且6 个样品均有分布的主要属门类群有厚壁菌门、丙型变形菌门、α-变形菌门和拟杆菌门。不同的冷水鱼样品之间肠道微生物菌群明显不同,其中哲罗鱼肠道内容物和黏膜中厚壁菌门是最占优势的菌群门,占各自样本序列的比例高达94.11%和87.34%,而细鳞鱼和黑斑狗鱼的肠道内容物和黏膜中丙型变形菌门占主要优势,分别占各自样品序列的28.79%、24.35%、33.35%和23.92%。β-变形菌门是细鳞鱼中第二大优势菌群,在黑斑狗鱼肠道内容物中厚壁菌门的相对丰度仅次于丙型变形菌门,而黏膜中拟杆菌门所占的比例居第2位,β-变形菌门在细鳞鱼肠道内容物和黏膜中的相对丰度基本相同。对于同种冷水鱼而言,尽管肠道内容物和黏膜中微生物群落组成相似,但相对丰度却略有差异。
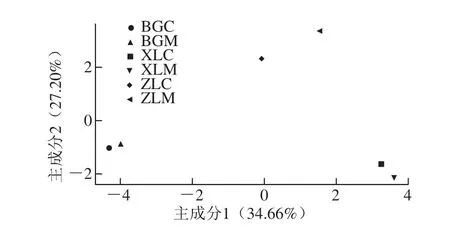
图1 冷水鱼肠道和黏膜中细菌群落结构的主成分分析图Fig. 1 Principal component analysis of bacterial community composition in intestinal contents and mucosa
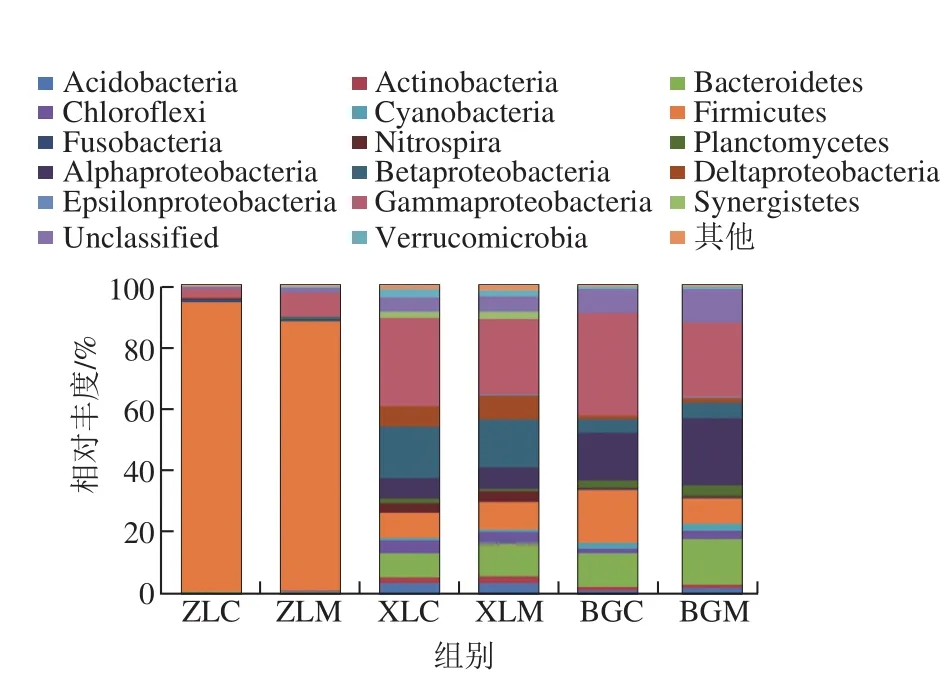
图2 冷水鱼肠道细菌门水平菌群分布Fig. 2 Frequency of phylum in microbial communities from fish samples

图3 冷水鱼肠道细菌属水平分布及丰度Fig. 3 Frequency and abundance of genus in microbial communities from fish samples
如图3所示,6 个样品共聚为两个大分支,细鳞鱼和黑斑狗鱼样品中细菌群落结构相似性较高,聚在一个分支里,哲罗鱼独自聚为一类。属水平上哲罗鱼肠道内容物和黏膜中主要以肉食杆菌属(Carnobacterium)为主,占各自样品序列的比例高达97.26%和97.15%。细鳞鱼和黑斑狗鱼样品中微生物菌群比较复杂,细鳞鱼肠道内容物中嗜冷杆菌属(Psychrobacter)和嗜酸硫杆菌属(Acidithiobacillus)所占比例较高,达到14.70%左右,而黏膜中主要以嗜酸硫杆菌属和不动杆菌属(Acinetobacter)等细菌种属为主,黑斑狗鱼样品中主要以梭菌属(Clostridium sensu stricto)、硫球形菌属(Thalassobius)和嗜甲基菌属(Methylophilus)为主,嗜酸硫杆菌属、硫球形菌属、嗜甲基菌属等属在湖泊、污泥和矿坑等环境中比较常见,这与冷水鱼生活的环境有关。
2.3 冷水鱼肠道微生物组成多样性差异
通过计算样品的多样性指数发现,哲罗鱼的Chao1指数和ACE指数是最小的,这说明细鳞鱼和黑斑狗鱼中的物种丰度明显高于哲罗鱼。同种类冷水鱼之间进行比较分析,哲罗鱼和黑斑狗鱼肠道内容物的Chao1指数和ACE指数低于其黏膜,而细鳞鱼例外(图4a、b)。哲罗鱼样品的Shannon指数低于细鳞鱼和黑斑狗鱼,而Simpson指数最高,这表明哲罗鱼样品中群落结构较简单。此外,细鳞鱼肠道内容物和黏膜的Shannon指数和Simpson指数几乎一致,差异不显著(图4c、d),而哲罗鱼和黑斑狗鱼的肠道内容物和黏膜的Shannon指数和Simpson指数相差较大。
如图5所示,Heatmap图可以通过颜色梯度及相似程度来反映多个样品在各分类水平上群落组成的相似性及差异性,这样能直观地比较分析不同样本中的某些属或同一属之间的差异。与图3结果相似,不同种类的冷水鱼微生物群落组成有所不同。哲罗鱼的细菌多样性最低,主要有肉食杆菌属、肠球菌属、支原体属和明串珠菌属等,黑斑狗鱼样品中有梭菌属、交替假单胞菌属、不动杆菌属、丛毛单胞菌属和嗜甲基菌属等,而细鳞鱼中有嗜冷杆菌属、螺菌属、不动杆菌属、固氮弧菌属、假单胞菌属、丛毛单胞菌属和脱硫弧菌属等。支原体属仅在哲罗鱼样品中检测到,是哲罗鱼特有的属,没有检测到丛毛单胞菌属,而黑斑狗鱼和细鳞鱼样品中都有丛毛单胞菌属的存在。哲罗鱼肠道内容物和黏膜中的微生物群落组成也不相同,在哲罗鱼肠道内容物中没有检测到嗜冷杆菌属、红小梨形菌属、梭菌属、嗜甲基菌属。在黑斑狗鱼和细鳞鱼中检测到相同属,但每个属在肠道内容物和黏膜中所占有的比例不同。
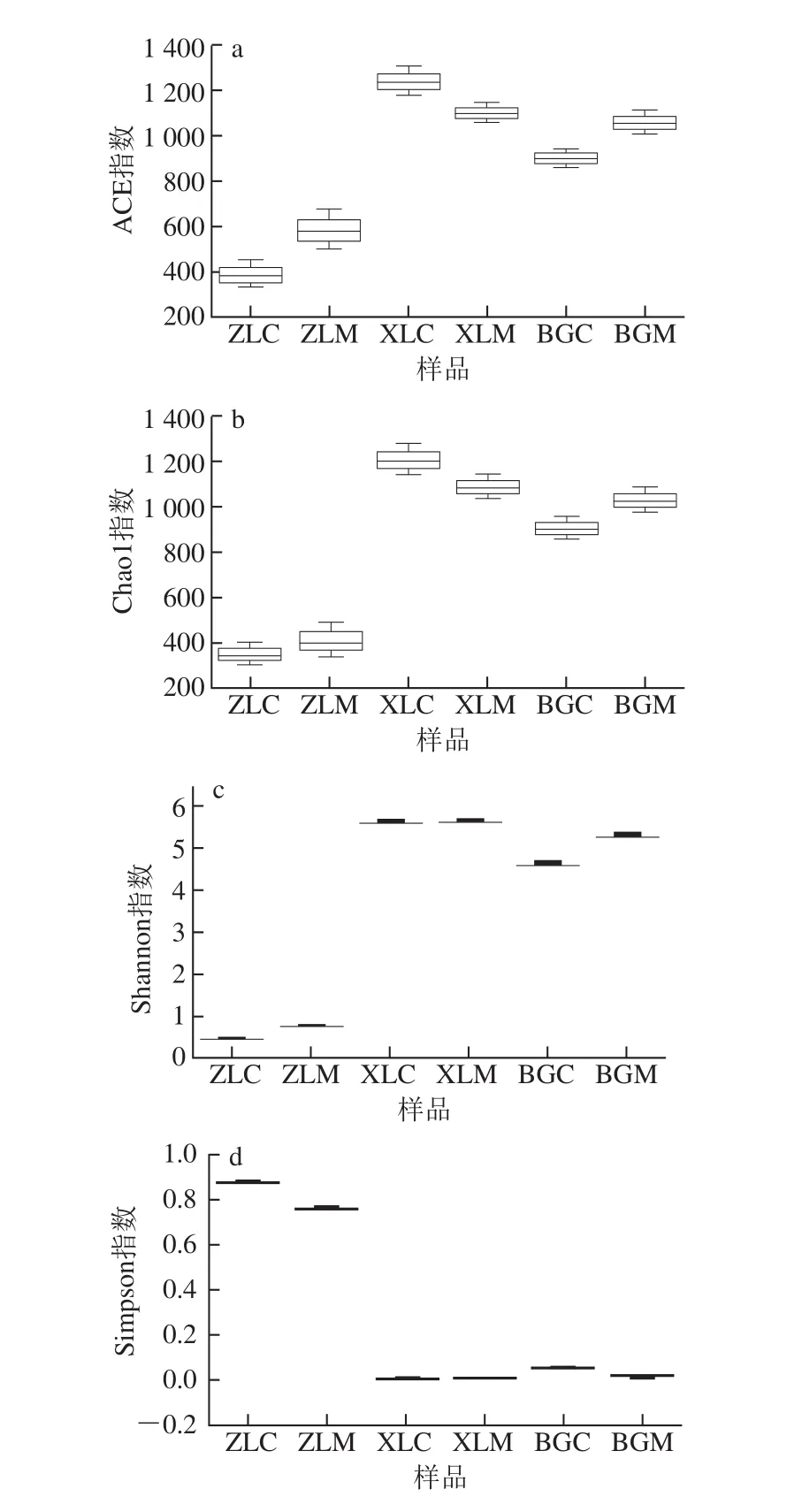
图4 冷水鱼样品中微生物多样性指数Fig. 4 Microbial diversity indexes of cold-water fishes
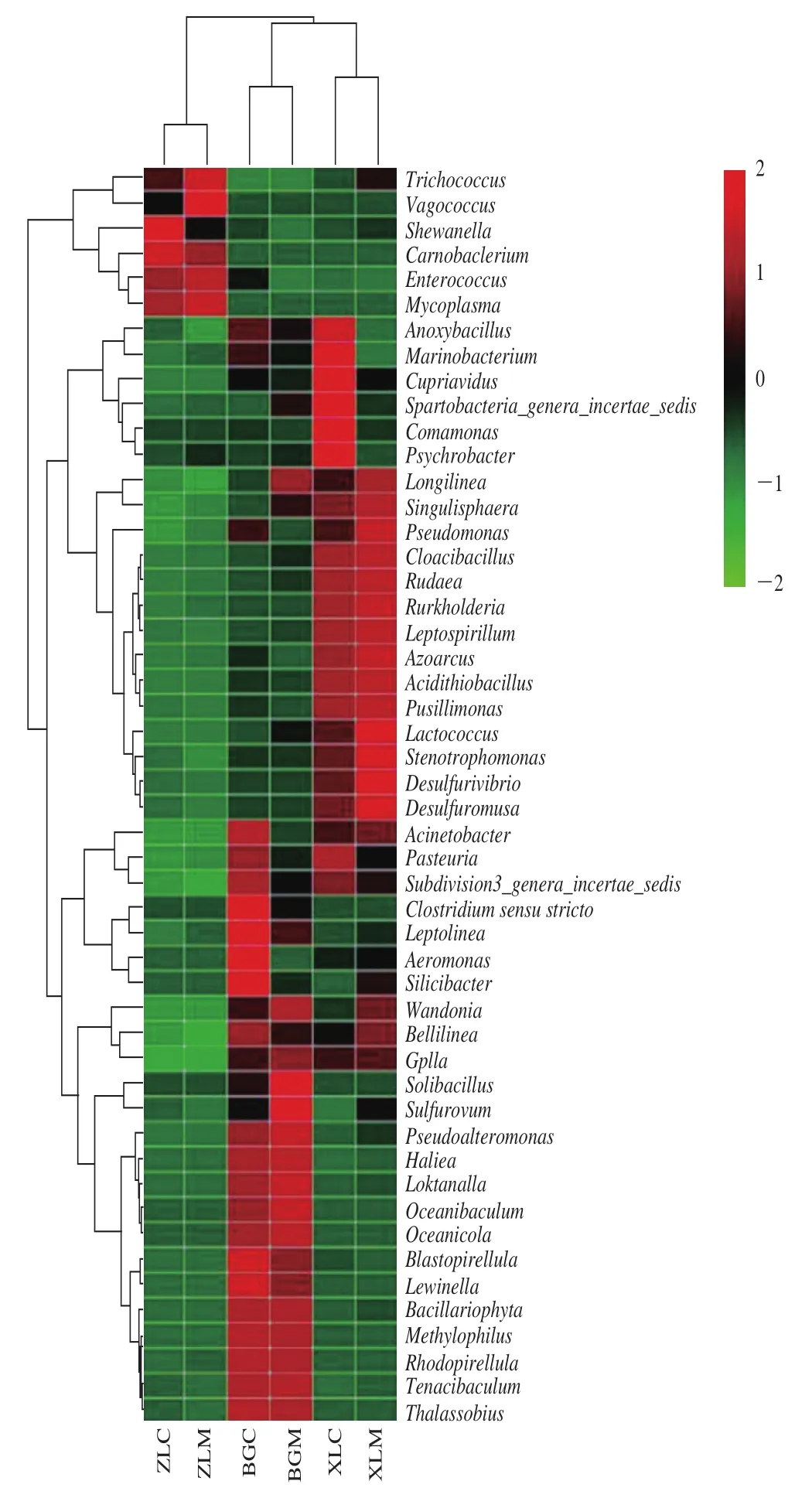
图5 冷水鱼肠道属水平群落结构Heatmap图Fig. 5 Heat map of intestinal bacterial community structure of cold-water fishes at genus level
3 讨 论
有研究结果表明,鱼肠道中细菌的数量级为105~108[24]。据报道,厚壁菌是大多数脊椎动物肠道中的优势菌群[17],而在鱼消化道中常见的微生物主要有厚壁菌、变形菌、假单胞菌、不动杆菌[25-26],并且其中一些肠道微生物在鱼类的生长、代谢过程中起着关键作用[27]。本研究发现同种冷水鱼肠道内容物和黏膜细菌群落结构差异并不明显;但不同种类的冷水鱼肠道中微生物群落组成差异显著,即使在每个样品中共同存在的一些微生物,在不同样品中其丰度也各有所异。相较于细鳞鱼和黑斑狗鱼,哲罗鱼肠道内微生物组成较为简单,主要以肉食杆菌和希瓦氏菌为主;细鳞鱼肠道中嗜冷杆菌、嗜酸硫杆菌、丛毛单胞菌和固氮弧菌所占比例较高,而梭菌、硫球形菌属和嗜甲基菌是黑斑狗鱼肠道内的优势菌群。Clements等[28]研究发现在3 种不同的海水鱼(Kyphosus sydneyanus、Odax pullus和Aplodactylus arctidens)后肠中以厚壁菌和梭菌为主。梭菌在哺乳动物的肠道中比较常见,是兼性厌氧微生物,能酵解多糖和蛋白质产生乙醇和短链脂肪酸[17];短链脂肪酸是鱼肠道中重要的有机酸,为肠道内壁细胞基本的代谢提供能量来源[29]。本研究从黑斑狗鱼肠道样品中也检测到较多的梭菌,而相较于黑斑狗鱼,在哲罗鱼和细鳞鱼样品中梭菌则几乎为零,这可能是由于黑斑狗鱼肠道较长[30],食物在消化道内发酵比较充分造成的。此外,有研究证明,从鱼肠道中分离出来的一些微生物能够有效地抑制一些病原菌[31]。邓梅等[32]从冷水鱼肠道筛选出产细菌素的耐低温肠球菌Enterococcus sp. MB2-1,其最适生长温度在24 ℃左右,能抑制李斯特氏菌(Listeria monocytogenes)、金黄色葡萄球菌(Staphylococcus aureus)、枯草芽孢杆菌(Bacillus subtilis)以及大肠杆菌(Escherichia coli)的生长。Robertson等[33]从大马哈鱼肠道中分离出肉食杆菌,能抑制米勒链球菌(Streptococcus milleri)、嗜水气单胞菌(Aeromonas hydrophila)和嗜鳃黄杆菌(Flavobacterium branchiophilum)等病原微生物的生长。实验也从冷水鱼肠道样品中检测到了肉食杆菌和肠球菌的存在,其在哲罗鱼肠道中的丰度最高,占到97%左右,而在细鳞鱼和黑斑狗鱼中相对丰度较低。嗜冷杆菌是细鳞鱼肠道内容物中的优势菌,嗜冷杆菌属于低温菌,即使在冷藏环境下也具有很强的生长活性,这对低温微生物开发具有很大的应用价值。
众所周知,脊椎动物肠道内的各类微生物与宿主的关系密切,对宿主的生长、发育和健康等有重要影响[34]。肠道群落结构的失衡可能会引起宿主发生疾病,因此,一些科学家提出肠道微生物相当于宿主的一个额外的“器官”[11,35]。反过来,肠道微生物群落的结构与组成可能会受到宿主的基因型、饮食、年龄和栖息环境等因素的影响[36]。许多有关脊椎动物肠道微生物的研究发现不同食性(草食性、杂食性和肉食性等)的脊椎动物肠道菌群有所不同。Ward等[9]发现杂食性日本鲫(Carassius cuvieri)的肠道菌群比那些肉食性鱼类较为复杂。Ni Jiajia等[37]利用DGGE技术对不同生存环境草鱼(Ctenopharyngodon idellus)的肠道微生物进行对比发现,鱼类的生活环境也会影响其肠道微生物群落。哲罗鱼喜欢潜伏在深水或两岸隐蔽处,常以其他鱼类和水中活动的蛇、蛙及水鸟为食,但在夏季水温稍高时,食欲较差,甚至会有停食现象出现;细鳞鱼多在水流湍急、大型砾石底的河段活动,主要以水生脊椎动物为食,也食被风吹落的陆生昆虫,一年四季都活跃摄食;黑斑狗鱼喜栖息于水温较低江河的缓流和水草丛生的沿岸带,以鱼、虾和水禽的幼鸟为食,在鱼饵不足时甚至会自相残食[30]。丛毛单胞菌常见于贝类和昆虫的体表;假单胞菌广泛存在于土壤、水中,且严格好氧;漫游球菌、肉食杆菌是兼性厌氧菌,且肉食杆菌对一些致病菌有抑制作用。本研究中只在细鳞鱼样品中检测到丰度较高的丛毛单胞菌,这些菌有可能是通过鱼类摄食进入鱼肠道的;假单胞菌大量存在于细鳞鱼和黑斑狗鱼肠道内,漫游球菌、肉食杆菌几乎只在哲罗鱼肠道内被检测到,这可能跟鱼类在河流中的垂直分布有关,哲罗鱼多在深水处活动,肠道内处于微氧状态,适合兼性厌氧菌大量生存,而假单胞菌等好氧菌则无法长期存活。由此推断本研究中冷水鱼肠道微生物群落的组成可能和鱼在河流中的分布和摄食类型有关。本实验对3 种冷水鱼肠道微生物群落结构与组成及多样性进行了研究,发现同种鱼肠道内容物和黏膜之间群落结构组成相似,无显著差异;不同种鱼之间肠道微生物群落构成及多样性存在明显差异,这可能与摄食种类、环境、分布、生活习性等因素有关。通过本研究,对额尔齐斯河鱼肠道微生物种群结构有了初步了解,也为冷水鱼肠道微生态的进一步探究和冷水鱼肠道低温微生物资源的开发提供科学依据。
参考文献:
[1]BJÖRKSTÉN B. The gut microbiota: a complex ecosystem[J].Clinicaland Experimental Allergy, 2006, 36(10): 1215-1217.DOI:10.1111/j.1365-2222.2006.02579.x.
[2]PÉREZ T, BALCAZAR J L, RUIZ-ZARZUELA I, et al. Hostmicrobiota interactions within the fish intestinal ecosystem[J]. Mucosal Immunol, 2010, 3(4): 355-360. DOI:10.1038/mi.2010.12.
[3]SHIINA A, ITOI S, WASHIO S, et al. Molecular identification of intestinal microflora in Takifugu niphobles[J]. Comparative Biochemistry and Physiology (Part D), 2006, 1(1): 128-132.DOI:10.1016/j.cbd.2005.10.001.
[4]FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria: potential for new insights from genomic analysis[J]. Nature Reviews Microbiology, 2008, 6(2): 121-131.DOI:10.1038/nrmicro1817.
[5]RAY A K, GHOSH K, RINGØ E. Enzyme-producing bacteria isolated from fish gut: a review[J]. Aquaculture Nutrition, 2012, 18: 465-492.DOI:10.1111/j.1365-2095.2012.00943.x.
[6]RINGØ E, BIRKBECK T H. Intestinal microflora of fish larvae and fry[J]. Aquaculture Research, 1999, 30(2): 73-93.
[7]RINGØ E, BIRKBECK T H, MUNRO P D, et al. The effect of early exposure to Vibrio pelagius on the aerobic bacterial flora of turbot, Scophthalmus maximus (L.) larvae[J]. Journal of Applied Microbiology, 1996, 81(2): 207-211. DOI:10.1111/j.1365-2672.1996.tb04502.x.
[8]ROMERO J, NAVARRETE P. 16S rDNA-based analysis of dominant bacterial populations associated with early life stages of coho salmon(Oncorhynchus kisutch)[J]. Microbial Ecology, 2006, 51(4): 422-430.DOI:10.1007/s00248-006-9037-9.
[9]WARD N, STEVEN B, PENN K, et al. Characterization of the intestinal microbiota of two Antarctic notothenioid fish species[J]. Extremophiles,2009, 13(4): 679-685. DOI:10.1007/s00792-009-0252-4.
[10]GARY L, SHERWOOD L. The normal human microflora composition[J]. Digestive Diseases and Sciences, 1986, 31(9):147-162. DOI:10.1007/BF01295996.
[11]ECKBURG P B, BIK E M, BERNSTEIN C N, et al. Diversity of the human intestinal microbial Flora[J]. Science, 2005, 308:1635-1638.
[12]NICHOLSON J K, HOLMES E, WILSON I D. Gut microorganisms,mammalian metabolism and personalized health care[J]. Nature Review on Microbiology, 2005, 3(5): 431-438. DOI:10.1038/nrmicro1152.
[13]CAMPBELL A C, BUSWEL J A. The intestinal microflora of farmed Dover sole (Solea solea) at different stages of fish development[J].Journal of Applied Microbioogy, 1983, 55(2): 215-223. DOI:10.1111/j.1365-2672.1983.tb01318.x.
[14]SUKANTA K N. Role of gastrointestinal microbiota in fish[J].Aquaculture Research, 2010, 41: 1553-1573. DOI:10.1111/j.1365-2109.2010.02546.x.
[15]SOU M, DAVID K, ULRICH S. Diet strongly influences the gut microbiota of surgeonfishes[J]. Molecular Ecology, 2015, 24(3): 656-672. DOI:10.1111/mec.13050.
[16]LEY R, LOZUPONE C, HAMADY M, et al. Worlds within worlds:evolution of the vertebrate gut microbiota[J]. Nature Reviews Microbiology, 2008, 6(10): 776-788. DOI:10.1038/nrmicro1978.
[17]FJELLHEIM A J, PLAYFOOT K J, SKJERMO J, et al. Vibrionaceae dominates the microflora antagonistic towards Listonella anguillarum in the intestine of cultured Atlantic cod (Gadus morhua L.)larvae[J]. Aquaculture, 2007, 269(1/2/3/4): 98-106. DOI:10.1016/j.aquaculture.2007.04.021.
[18]KIM D, BRUNT J, AUSTIN B. Microbial diversity of intestinal contents and mucus in rainbow trout (Oncorhynchus mykiss)[J].Journal of Applied Microbiology, 2007, 102(6): 1654-1664.DOI:10.1111/j.1365-2672.2006.03185.x.
[19]POND M J, STONE D M, ALDERMAN D J. Comparison of conventional and molecular techniques to investigate the intestinal microflora of rainbow trout (Oncorhynchus mykiss)[J]. Aquaculture,2006, 261(1): 194-203. DOI:10.1016/j.aquaculture.2006.06.037.
[20]NAVARRETE P, ESPEJO R T, ROMERO J. Molecular analysis of microbiota along the digestive tract of juvenile Atlantic salmon (Salmo salar L.)[J]. Microbial Ecology, 2009, 57(3): 550-561. DOI:10.1007/s00248-008-9448-x.
[21]LEY R, HAMADY M, LOZUPONE C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320: 1647-1651.DOI:10.1126/science.1155725.
[22]米其利, 李雪梅, 管莹, 等. 高通量测序在食品微生物生态学研究中的应用[J]. 食品科学, 2016, 37(23): 302-307. DOI:10.7506/spkx1002-6630-201623049.
[23]CAPORASOA J, LAUBERB C, WALTERS W, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J].PANS, 2011, 108(1): 4516-4522. DOI:10.1073/pnas.1000080107.
[24]VESTA S. Enzymatic activity of intestinal bacteria in roach Rutilus rutilus L.[J]. Fisheries Science, 2007, 73(4): 964-966. DOI:10.1111/j.1444-2906.2007.01421.x.
[25]VESTA S, SRUOGA A, BUTKAUSKAS D, et al. Phylogenetic analysis of intestinal bacteria of freshwater salmon Salmo salar and sea trout Salmo trutta trutta and diet[J]. Fisheries Science, 2008, 74(6):1307-1314. DOI:10.1111/j.1444-2906.2008.01656.x.
[26]SULLAM K, ESSINGER S, LOZUPONE C, et al. Environmental and ecological factors that shape the gut bacterial communities of fish: a meta-analysis[J]. Molecular Ecology, 2012, 21(13): 3363-3378.DOI:10.1111/j.1365-294X.2012.05552.x.
[27]CLEMENTS K D, ANGERT E R, MONTGOMERY W L, et al.Intestinal microbiota in fishes: what’s known and what’s not[J].Molecular Ecology, 2014, 23: 1891-1898. DOI:10.1111/mec.12699.
[28]CLEMENTS K D, PASCH I, MORAN D, et al. Clostridia dominate 16S rRNA gene libraries prepared from the hindgut of temperate marine herbivorous fishes[J]. Marine Biology, 2007, 150(6): 1431-1440. DOI:10.1007/s00227-006-0443-9.
[29]MOUNTFORT D, CAMPBELL J, CLEMENTS K. Hindgut fermentation in three species of marine herbivorous fish[J]. Applied Environment Microbiology, 2002, 68(3): 1374-1380. DOI:10.1128/AEM.68.3.1374-1380.2002.
[30]张觉民. 黑龙江省鱼类志[M]. 哈尔滨: 黑龙江科学技术出版社,1995: 50-68.
[31]SUGITA H, SHIBUYU K, SHIMOOKA H, et al. Antibacterial abilities of intestinal bacteria in freshwater cultured fish[J]. Aquaculture, 1996,145(1): 195-203. DOI:10.1016/S0044-8486(96)01319-1.
[32]邓梅, 王俊钢, 高阳, 等. 耐低温肠球菌Enterococcus sp. MB2-1产细菌素的生物学特性[J]. 食品科学, 2013, 34(19): 170-175.DOI:10.7506/spkx1002-6630-201319036.
[33]ROBERTSON P, DOWD C, BURRELS C, et al. Use of Carnobacterium sp. as a probiotic for Atlantic salmon (Salmo salar L.)and rainbow trout (Oncorhynchus mykiss, Walbaum)[J]. Aquaculture,2000, 185(3): 235-243. DOI:10.1016/S0044-8486(99)00349-X.
[34]HOOPER L, GORDON J. Commensal host-bacterial relationships in the gut[J]. Science, 2001, 292: 1115-1118. DOI:10.1126/science.1058709.
[35]FUJIMURA K E, SLUSHER N A, CABANA M D, et al. Role of the gut microbiota in defining human health[J]. Expert Review of Anti-Infective Therapy, 2010, 8(4): 435-454. DOI:10.1586/eri.10.14.
[36]NAVARRETE P, MAGNE F, ARANEDA C, et al. PCR-TTGE analysis of 16S rRNA from rainbow trout (Oncorhynchus mykiss) gut microbiota reveals host-specific communities of active bacteria[J].PLoS ONE, 2012, 7(2): e31335. DOI:10.1371/journal.pone.0031335.
[37]NI J J, YU Y H, ZHANG T L, et al. Comparison of intestinal bacterial communities in grass carp, Ctenopharyngodon idellus, from two different habitats[J]. Chinese Journal of Oceanology and Limnology,2012, 30(5): 757-765. DOI:10.1007/s00343-012-1287-4.
