基于微型DNA条形码的多种动物源性成分的鉴定
2018-05-23潘艳仪邱德义杨维东岳巧云
潘艳仪,邱德义,陈 健,杨维东,岳巧云,*
(1.暨南大学生命科学技术学院,广东 广州 510632;2.中山出入境检验检疫局检验检疫技术中心,广东 中山 528403)
近年来,国际贸易及国内市场上各种肉类假冒以及以次充好的现象时有发生[1]。经过2013年欧盟的“马肉风波”,人们对食品成分的真实性越来越关注[2]。食品掺假造假问题严重危害了人类健康和广大消费者的利益,引发宗教矛盾,甚至影响国家之间的关系[3]。随着工业技术的发展,食品掺假技术不断翻新,迫使食品成分真实性的鉴定技术不断地发展和改善。由此,可靠、有效的动物源性物种鉴定方法、技术是识别食品掺假造假的有力技术保障。
在过去的20年,DNA的分子检测技术快速发展并应用,如分子指纹图谱技术和聚合酶链式反应(polymerase chain reaction,PCR)技术。PCR技术中的实时荧光定量PCR、单链构象多态性PCR、限制性内切酶片段长度多态性PCR和巢式PCR技术等在食品成分物种鉴定领域也得到了广泛的应用[4-7]。其中,普遍使用的是具有高灵敏度的实时荧光定量PCR技术。但因为该技术使用特异的探针和引物,只能猜测性地鉴定特定某个物种,不适合于复杂未知的动物源性多成分的鉴定,而市场加工食品中可能同时混有多种成分。此外,有些物种的特异性探针和引物难以设计,从而成为了多种动物源性成分鉴定的应用瓶颈。
自2003年,加拿大动物学家Paul Hebert教授首次提出DNA条形码的概念,分子分类学方法以及分子鉴别技术在生物物种鉴定方面发挥了重要作用[8-9]。近年来,随着DNA条形码数据库的不断完善,该技术广泛应用于医学媒介生物的物种鉴定[10],并开始应用于动物源性食品成分的鉴定[11-15]。
DNA条形码是源于线粒体基因组中细胞色素氧化酶I(cytochrome oxidase I ,COI)基因的一段658 bp的DNA序列。此外,DNA条形码的适用范围很广(从低等的节肢动物到高等的哺乳动物),有较大的通用性,可以同时适用于多种动物种类的鉴定。而一些深加工食品的DNA会存在不同程度的降解[16],其中某些物种成分可能会由于DNA高度降解而不能被检出。为了适用加工食品尤其是深加工食品中动物源性成分的检测,微型的DNA条形码的研发是该技术提升的一个突破口。2008年,DNA的微型条形码被首次提出,对久置的生物标本实现了高效的物种鉴定[8]。继而,也有研究者使用coi基因和12S、16S核糖体RNA基因的短片段对动物膳食和饲料样品进行了物种鉴定[11],但各研究者所使用的短片段不如标准的DNA条形码片段那样非常统一,况且目前仍没有标准的微型DNA条形码用于动物源性食品成分的鉴定[17]。因此,设计具有适度保守性和通用性的微型DNA条型码应用于食品成分(尤其是深加工食品)的鉴定很有必要。
本研究针对常见的经济动物物种,基于coi基因序列设计了一对通用引物,结合微型DNA条形码和克隆测序技术对混合的肉样品成分实现一次性准确、高效的鉴定。以便进一步应用于未知成分的肉糜制品或深加工肉类食品的多种肉源性成分的检出。
1 材料与方法
1.1 材料与试剂
1.1.1 样本的选取和种类的确定
选取了11 种常见的经济物种以验证引物的适用性和通用性。这11 个物种的新鲜肉类样品从广东中山市某市场购得,并初步判定为鲶鱼、草鱼、马头鱼、蓝圆鲹、家鸡、鹧鸪、黄牛、水牛、家猪、绵羊和对虾。
使用DNA条形码的分子鉴定技术确定样品的物种[10]。用coi通用引物(LCO1490、HCO2198)[18]扩增出目的片段后,通过Sanger测序获得658 bp目的序列,再于BOLD数据库(生命条形码数据库http://www.boldsystems.org/)进行序列比对,以与参考序列达到99.8%相似度为标准确定样品物种。
1.1.2 试剂
DNA提取试剂盒(目录号DP304)、琼脂糖凝胶回收试剂盒(目录号DP210)、克隆试剂盒(目录号VT202-02)、重组菌落鉴定试剂盒(目录号VI102)、dNTP、Loading buffer、DNA Marker II、SYBR Green I天根生化科技(北京)有限公司;Ex-Taq DNA聚合酶大连宝生物工程有限公司;引物由Thermo Fisher Scientific Inc广州公司合成。
1.2 仪器与设备
ThermoPico 17微量台式离心机 美国Thermo Fisher公司;DYY-6C电泳仪 北京科学深蓝科技有限公司;Veriti 96梯度PCR仪 美国Applied Biosystems公司;MD-02N-220金属浴 美国Major Science公司;Alliance 4.7 Thermo UVITEC凝胶成像仪 英国Uvitec公司;量程可调移液器。
1.3 方法
1.3.1 引物设计
从BOLD和GenBank(美国国立生物技术信息中心https://www.ncbi.nlm.nih.gov/)下载了140 条源于动物coi基因的序列,包括了28 个属(石鸡属、原鸡属、鸭属、雁属、鲶鱼属、鲇鱼属、黄鳝属、马头鱼属、鲳鱼属、非鲫属、丽鲷属、圆鯵属、鲈鱼属、草鱼属、鲢鱼属、鲤鱼属、鲫鱼属、泥鳅属、对虾属、龙虾属、白虾属、长臂虾属、疣猪属、猪属、黄牛属、水牛属、山羊属和羊属)和51个动物物种(25 种鱼、6 种虾、10 种家禽、3 种猪、3 种牛和4 种羊)。选取的物种基本上都是常见的作为商品肉食的经济动物及其近缘种,每个物种有2或3条源于不同个体的序列。目的是寻找具有较小同种个体间差异和较大种间差异的区域,而且引物的位点要有足够的保守性和通用性。用MEGAv. 6.0.6(分子进化遗传分析软件)进行多序列比对[19],结合引物设计的一般原则,选取了一段长度为136 bp目的片段。使用Oligo 7.0对所选引物的特性(引物二聚体、发夹结构,在其他位点错误引发的效率等)进行理论上的分析和评价[20]。再使用BLAST(基本局部比对搜索工具https://blast.ncbi.nlm.nih.gov/Blast.cgi)检验引物的特异性。经过综合分析,最终确定了最佳的一对引物序列如下:正向引物为5’-GGTCAACAAATCATAAAGATATTGG-3’,即标准DNA条形码的正向引物LCO1490[18];反向引物为5’-ACT ATAAAGAAGATTATTACAAAGGC-3’。
1.3.2 引物通用性分析
由于本实验的正向引物源于标准条形码的正向引物序列,其引物本身对多种动物物种都具有较高的通用性[8]。为评估这一对引物对所选物种的通用性,对引物的保守位点和引物与各属内物种的同源性进行分析。使用MEGAv. 6.0.6软件对各属物种序列的引物区域与反向引物序列进行多序列比对,对引物位点的核酸碱基组成情况进行整合分析。
1.3.3 样品前处理和DNA的提取
为了防止不同肉之间的交叉污染,样品处理前,每把剪刀都经高温高压处理,并紫外照射30 min,而且在同一次混合操作中,一把剪刀对应一种肉。每种肉样品都要用无菌的剪刀剪取其内部肌肉组织。肉类样品设为A和H两组:A组是11 个物种的肉独立样品,H组是由上述的11 个物种的生鲜肉等比例混合的样品。对11 个物种的生鲜肉样各剪取0.03 g组成A组的独立样品。再以同样的方式对11 种肉类样品各剪取0.01 g,充分磨碎并混匀形成H组的混合肉样品。H组中的混合样品设置了3 个平行。按照血液/细胞/组织基因组DNA提取试剂盒说明书指导提取全基因组DNA。
1.3.4 PCR扩增条件
A组和H组的样品经DNA提取后,按以下扩增条件获取目的片段:94 ℃预变性3 min、94 ℃变性30 s、48 ℃退火30 s、68 ℃延伸30 s,共30 个循环;最后68 ℃延伸7 min。PCR反应体系(50 μL):10 倍PCR缓冲液5 μL;正向引物(20 nmol/μL)1 μL;反向引物(20 nmol/μL)1 μL;dNTP(10 nmol/μL)2 μL;Ex-Taq(5 U/μL)1 μL;模板DNA(50 ng/μL)3 μL;无菌水定容到50 μL。
1.3.5 直接测序
把A组11 个独立的物种样品的PCR产物送交上海立菲生物科技有限公司测序,测序引物同扩增引物。使用DNA MAN软件[21]和MEGA软件对测序公司返回的序列进行序列拼接和序列处理(去除引物和碱基校对),再进行蛋白质翻译校对无误后,在GenBank数据库中进行比对和相似度分析,对序列进行物种注释。
1.3.6 DNA纯化
把H组混合肉类样品的PCR产物进行纯化。采用琼脂糖凝胶DNA回收试剂盒,按照试剂盒说明书对PCR产物进行纯化。
1.3.7 克隆测序
采用pGM-T连接试剂盒,按照试剂盒的使用手册对H组样品的纯化PCR产物进行连接和转化。转化后把产物置于37 ℃进行菌液扩大培养1 h,再把菌液均匀涂布于LB固体培养基平板上,置于37 ℃过夜培养。经过蓝白斑筛选,随机挑取白色菌落进行阳性克隆检测,以确定克隆的有效性。最终每个样品随机挑选130个阳性菌株送交上海立菲生物科技有限公司测序。以上述方法1.3.5节同样的方式对测序结果进行序列分析和物种注释。
1.3.8 比对微型条形码与标准条形码的对物种鉴定的效果
基于140 条、51 个物种的coi基因的DNA序列(序列来源与1.3.1节所述的一致),序列分别处理为长度为136 bp的微型条形码序列和长度为658 bp的标准条形码序列,再用MEGA v.6.0.6软件,选用邻接(Neighbor joining,NJ)法构建进化树及其默认参数值(P-distance模式、检测次数为500等),分别构建微型条形码和标准条形码的系统发育树,以比较微型条形码与标准条形码的对物种鉴定的效果。
2 结果与分析
2.1 引物的通用性分析
对所选的28 个属、140 条序列的反向引物区域的同源性分析情况如表1所示。反向引物序列区域中共有17 个完全保守位点,分别以“*”标记。28 个属中,共有14 个属具有较高同源性(少于2 个差异位点):猪属、雁属、鲇鱼属、马头鱼属、非鲫属、鲤鱼属、石鸡属、鲶鱼属、圆鯵属、草鱼属、鲢鱼属、鲫鱼属、长臂虾属和黄牛属。有4 个属具有较低同源性(4~5个差异位点):黄鳝属、对虾属、龙虾属和白虾属。其余的10 个属具有中等的同源性(3 个差异位点)。为了进一步检验引物的适用性和通用性,选取源于3 种不同的同源性水平的11 个物种(表1中以上标“1”标记)进行后续的实验验证,扩增的目的片段如图1所示。

表1 28 个属内物种与引物的同源性Table 1 Sequence homology with primers among species of 28 genera
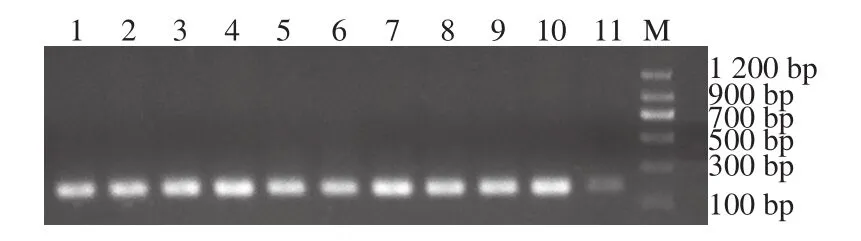
图1 11 个物种的目的片段Fig. 1 Target fragments for 11 species
2.2 样品物种的鉴定
经DNA条形码分子鉴定,11 个样品的目的序列都能与BOLD条形码数据库的参考序列达99.8%以上的相似度,达到物种鉴定的标准,确定样品的物种如下:鲶鱼、草鱼、马头鱼、蓝圆鲹、原鸡、石鸡、黄牛、水牛、家猪、绵羊和凡纳滨对虾。
2.3 微型DNA条形码鉴定物种的准确性
A组的11 个物种的基因组DNA都能被成功提取并高效扩增出长度为136 bp的目的片段,如图1所示。测序后返回的DNA序列经序列处理和分析,并蛋白质翻译校对无误后,结合BOLD数据库的参考序列和GenBank的匹配序列,所有目的片段和对应的两种序列都有100%的相似度,即对每个物种的鉴定都准确地达到了种的水平[8,22](表2)。
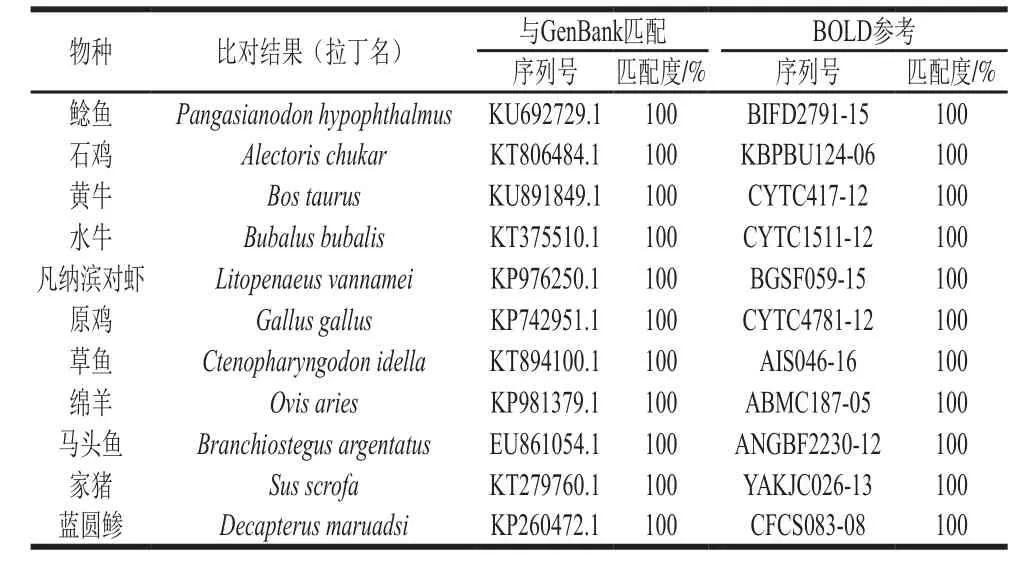
表2 11 个样品物种的微型DNA条形码的鉴定结果Table 2 Identification of 11 species based on mini-DNA barcode
2.4 混合肉样品的克隆测序结果
H组3 个平行的混合肉样品中的390 个阳性克隆经过Sanger测序后,其中绝大部分的阳性克隆都能被成功测序并获得高质量的测序结果,只有3.6%的阳性克隆测序失败或者序列比对结果不佳,说明本实验的克隆测序法的阳性克隆具有较高的有效性。经序列处理和序列分析后,各物种的微型条形码序列(136 bp)几乎都能与数据库中的参考序列达到100%的相似度,说明微型条形码与克隆测序法结合后,对物种鉴定依然具有较高的准确度。综合3 个平行样品,除了原鸡和蓝圆鲹,其余9 个物种皆一并检出(图2)。进一步分析各个物种的序列丰度,不同物种之间被检出的概率差异较大。混合样品中鲶鱼、石鸡、黄牛和绵羊物种的检出概率较高,其检出概率的范围为7.7%~54.0%,而其余物种的检出概率的范围为0%~1.5%(按3 个平行样品物种序列数的均值计算)。

图2 混合样品各物种被检出的阳性克隆数Fig. 2 Number of detected positive clones of each specie from mixed meat samples
2.5 微型条形码与标准条形码的NJ树的比较分析
基于140 条51 个常见动物经济物种的coi基因序列,构建出微型条形码(136 bp)与标准条形码(658 bp)的NJ树见图3。由图3a可见,28 个不同的属(每个属已在方法的1.3.1节中一一列举)都能得到明确的区分。除了原鸡属、雁属、亚口鱼属、罗非鱼属和绵羊属内的物种序列之间有较高的相似度,其他的21个属内各物种的种间距离基本都大于2%,达到了物种区分的界限[22]。属内种间差异较小的物种有:原鸡和灰原鸡,雪雁、豆雁、灰雁和白额雁,莫桑比克罗非鱼和尼罗罗非鱼,黑鲫和鲫鱼,扁吻亚口鱼和圣安娜亚口鱼,银鲳鱼和翎鲳鱼以及落基山脉大角羊和白大角羊(图3已标注)。另一方面,除银鲳鱼外,其余的50 个物种种内的不同个体的序列之间都拥有高于98%的相似度,即个体差异小于种间差异,达到种内保守。标准条形码的NJ树(图3b)的各属内物种差异较小的物种较少,但原鸡属、雁属及绵羊属内的种间差异也小于2%(图3已标注)。种内个体差异较大的物种较多,包括了银鲳鱼、阿氏六须鲶和鲢鱼。
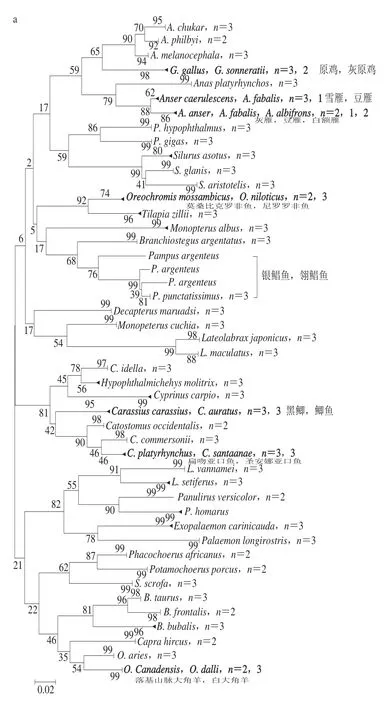

图3 微型条形码(a)和标准条形码(b)的NJ进化树(P-distance模式)Fig. 3 Neighbor joining trees of mini-barcode and standard barcode(P-distance model)
比对分析微型条形码与标准的条形码的NJ树,两者在区分所选物种的种间和种内的能力上有高度的一致性。说明微型条形码尽管片段较短,也依然能保留标准条形码基本的物种分类能力,能对所选的大部分的物种实现较好的物种分类和鉴定。
3 讨 论
3.1 引物的通用性
经140 条序列的反向引物区域同源性的分析,26 个引物位点中有17 个完全保守位点。虽然引物3末端的5 个碱基不是连续的完全保守位点,但以G、C碱基(完全保守位点)为末端的引物一般与模板结合时具有较高的稳定性,能增加引物与模板的总体结合力。而且后续的验证引物的适用性和通用性的实验表明引物能对不同引物同源性水平的11 种个肉类物种(猪、牛、羊、鸡、鱼和虾)的目的序列进行高效的扩增,并得到高质量的序列测序结果。虽然引物的同源性分析反映了该引物对所选的51 个动物经济物种有一定的通用性,且对所选的11 个物种序列有良好的扩增效率,但仍需进一步的实验以对引物通用性进行更全面的检验。
3.2 克隆测序法的检测效果
在混合样品中9 个物种一并检出并得到准确鉴定,物种成分的检测在总体达到了较高的检出效率,但仍然有2 个物种未能检出。由于克隆法自身的局限性,难以对多组分样品的成分进行全面检出。进一步分析各物种被检出的序列丰度,不同物种之间的检出概率差异较大,这可能源于混合样品的各个成分中在PCR扩增过程中存在竞争效应[23-24]。对于那些具有相对较低的扩增效率的物种,则呈现较低的检测概率。另一方面,克隆测序法对混合样品成分的检测具有一定的随机性,而导致了检测结果的稳定性较低。这与挑取阳性克隆的相对丰度以及选取阳性克隆的随机性有关。
3.3 微型DNA条形码的适用性和前景性
选取的11 个物种皆能通过本实验设计的通用引物成功扩增出136 bp的目的片段并得到准确的物种鉴定(皆与数据库的目的物种序列有100%的相似度),说明该微型DNA条形码可准确鉴定这11 种肉类成分,可进一步应用于深加工食品或复杂成分食品的多种动物源性成分的检测。
此外,经过微型条形码和标准条形码的邻接树的对比分析,微型条形码对所选的51 个常见经济物种的分类鉴定能力与标准条形码高度相似。由于微型条形码的序列仅有136 bp,对某些在coi基因片段中具有极少差异位点的近缘物种(2.5节)则难以区分。除去种间差异较小的8 个物种和种内个体差异较大的1 个物种,该微型条形码对其余的42 个物种都具有较好的种间和种内的区分能力。针对克隆测序法难以对于含有较多成分样品达到全面检出的情况,近年来,已有一些研究者开始借助二代测序平台对多种动植物源性成分进行鉴定[25-27]。由于现今市场上常用的二代测序平台有读长长度限制[28-29],尽管采用PE300的测序策略,最多只能检测长度为550 bp的扩增片段。所以,标准的条形码658 bp由于长度过长而不能直接应用于二代测序平台。因此,也有一些研究者结合一种或多种微型条形码和二代测序技术,进而对复杂的、未知的样品成分的进行检测和鉴定,如蜂蜜成分的鉴定,花粉混合物的物种来源鉴定及糖果中动物物种成分分析等[29-31]。由于本实验的微型条形码和标准条形码在动物物种分类的能力上有高度的一致性,而且长度符合二代测序的读长要求,所以能进一步与二代测序平台结合,进而对复杂、未知的食品中多种动物源性成分进行全面的检测和鉴定。
4 结 论
单个样品的Sanger测序和混合样品的克隆测序结果皆显示,本实验研发的微型条形码对于多种动物源性物种成分,包括鱼、虾、鸡、猪、牛和羊的鉴定有高度的准确性。克隆测序法能对混合样品的物种成分达到较高的检测效率,但同时也存在一些局限性。基于微型条形码与标准条形码在物种分类上高度的一致性,把微型条形码与二代测序技术相结合,是未来在复杂食品成分快速鉴定的领域中进一步的研究方向。
参考文献:
[1]GALAL-KHALLAF A, ARDURA A, MOHAMMED-GEBA K, et al.DNA barcoding reveals a high level of mislabeling in Egyptian fish fillets[J]. Food Control, 2014, 46: 441-445. DOI:10.1016/j.foodcont.2014.06.016.
[2]BALLIN N Z. Authentication of meat and meat products[J]. Meat Science, 2010, 86(3): 577-587. DOI:10.1016/j.meatsci.2010.06.001.
[3]SYNTESA H L. Communicating food safety, authenticity and consumer choice. Field experiences[J]. Recent Patents on Food Nutrition & Agriculture, 2013, 5(1): 19-34. DOI:10.2174/2212798411 305010005.
[4]OKUMA T A, HELLBERG R S. Identification of meat species in pet foods using a real-time polymerase chain reaction (PCR) assay[J].Food Control, 2015, 50: 9-17. DOI:10.1016/j.foodcont.2014.08.017.
[5]SARRI C, STAMATIS C, SARAFIDOU T, et al. A new set of 16S rRNA universal primers for identification of animal species[J]. Food Control, 2014, 43: 35-41. DOI:10.1016/j.foodcont.2014.02.036.
[6]HAIDER N, NABULSI I, ALSAFADI B. Identification of meat species by PCR-RFLP of the mitochondrial coi gene[J]. Meat Science,2012, 90(2): 490-493. DOI:10.1016/j.meatsci.2011.09.013.
[7]ZHANG C. Semi-nested multiplex PCR enhanced method sensitivity of species detection in further-processed meats[J]. Food Control, 2013,31(2): 326-330. DOI:10.1016/j.foodcont.2012.11.002.
[8]HEBERT P D N, CYWINSKA A, BALLl S L, et al. Biological identifications through DNA barcodes[J]. Proceedings Biological Sciences, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218.
[9]HEBERT P D N, STOECKLE M Y, ZEMLAK T S, et al.Identification of birds through DNA Barcodes[J]. PLoS Biology, 2004,2(10): e312. DOI:10.1371/journal.pbio.0020312.
[10]岳巧云, 邱德义, 胡佳, 等. DNA条形码: 医学媒介生物快速准确鉴定的利器[J]. 检验检疫学刊, 2013, 23(5): 60-63. DOI:10.3969/j.issn.1674-5354.2013.05.017.
[11]MALGORZATA N W, PIOTR KRZYSCIN, AGATA P K.The species identification of bovine, porcine, ovine and chicken components in animal meals, feeds and their ingredients, based on COX I analysis and ribosomal DNA sequences[J]. Food Control, 2013,34: 69-78. DOI:10.1016/j.foodcont.2013.04.014.
[12]MARALIT B A, AGUILA R D, VENTOLERO M F H, et al. Detection of mislabeled commercial fishery by-products in the Philippines using DNA barcodes and its implications to food traceability and safety[J]. Food Control, 2013, 33: 119-125. DOI:10.1016/j.foodcont.2013.02.018.
[13]GALIMBERTI A, MATTIA F D, LOSA A, et al. DNA barcoding as a new tool for food traceability[J]. Food Research International, 2013,50: 55-63. DOI:10.1016/j.foodres.2012.09.036.
[14]邱德义, 胡佳, 刘德星, 等. DNA条形码技术在肉品防欺诈鉴别中的应用[J]. 肉类研究, 2013, 27(4): 40-43.
[15]陈健, 岳巧云, 邱德义. DNA条形码技术检测调理肉制品掺杂[J]. 肉类研究, 2016, 30(6): 35-39.
[16]叶可萍, 周光宏, 徐幸莲, 等. 转基因大豆不同加工过程DNA降解研究[J]. 食品科学, 2010, 31(13): 312-315.
[17]STAATS M, ARULANDHU A J, GRAVENDEEL B, et al. Advances in DNA metabarcoding for food and wildlife forensic species identification[J]. Analytical & Bioanalytical Chemistry, 2016, 408(17):4615-4630. DOI:10.1007/s00216-016-9595-8.
[18]FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Molecular Marine Biology &Biotechnology, 1994, 3(5): 294-299.
[19]KUMAR S, NEI M, DUDLEY J, et al. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences[J]. Briefings in Bioinformatics, 2008, 9(4): 299-306. DOI:10.1093/bib/bbn017.
[20]RYCHLIK W. OLIGO 7 primer analysis software[J]. Methods in Molecular Biology, 2007, 402: 35-59. DOI:10.1007/978-1-59745-528-2_2.
[21]EDWARDS D J, GRODEVANT N W, LEE P J, et al. DNA-MAN:dynamic natural attributes for synthetic military forces[C]//Systems and Information Engineering Design Symposium, IEEE, 2007: 1-5.DOI:10.1109/SIEDS.2007.4374030.
[22]HEBERT P D, RATNASINGHAM S, DEWAARD J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings: Biological Sciences, 2003, 270(1):96-99. DOI:10.1098/rsbl.2003.0025.
[23]PENG Q, SATYA R V, LEWIS M, et al. Reducing amplification artifacts in high multiplex amplicon sequencing by using molecular barcodes[J]. BMC Genomics, 2015, 16(1): 589-593. DOI:10.1186/s12864-015-1806-8.
[24]BERTOLINI F, GHIONDA M C, D'ALESSANDRO E, et al. A next generation semiconductor based sequencing approach for the identification of meat species in DNA mixtures[J]. PLoS ONE, 2015,10(4): e0121701. DOI:10.1371/journal.pone.0121701.
[25]TILLMAR A O, DELLl’AMICO B, WELANDER J, et al. A universal method for species identification of mammals utilizing next generation sequencing for the analysis of DNA mixtures[J]. PLoS ONE, 2013,8(12): e83761. DOI:10.1371/journal.pone.0083761.
[26]CHENG X, SU X, CHEN X, et al. Biological ingredient analysis of traditional Chinese medicine preparation based on high-throughput sequencing: the story for Liuwei Dihuang Wan[J]. Scientific Reports,2014, 4: 5147. DOI:10.1038/srep05147.
[27]QUAIL M A, SMITH M, COUPLAND P, et al. A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers[J]. BMC Genomics, 2012,13: 341-353. DOI:10.1186/1471-2164-13-341.
[28]SHOKRALLA S, GIBSON J F, NIKBAKHT H, et al. Next-generation DNA barcoding: using next-generation sequencing to enhance and accelerate DNA barcode capture from single specimens[J]. Molecular Ecology Resources,2014, 14(5): 892-901. DOI:10.1111/1755-0998.12236.
[29]PROSSER S W J, HEBERT P D N. Rapid identification of the botanical and entomological sources of honey using DNA metabarcoding[J]. Food Chemistry, 2017, 214: 183-191. DOI:10.1016/j.foodchem.2016.07.077.
[30]POMON A, ESCARAVAGE N, BURRUS M, et al. Using metabarcoding to reveal and quantify plant-pollinator interactions[J].Scientific Reports, 2016, 6: 27282. DOI:10.1038/srep27282.
[31]MUNOZ-COLMENERO M, MARTINEZ J L, ROCA A, et al. NGS tools for traceability in candies as high processed food products: ion torrent PGM versus conventional PCR-cloning[J]. Food Chemistry,2017, 214: 631-636. DOI:10.1016/j.foodchem.2016.07.121.
