扇贝核糖体ITS1和5S rDNA序列的遗传变异及亲缘关系分析
2018-05-16廖德杰曹善茂童金苟俞小牧
廖德杰 曹善茂 童金苟 周 颖, 俞小牧 刘 阳 王 潇
(1. 大连海洋大学辽宁省贝类良种繁育工程技术研究中心, 大连 116023; 2. 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室, 武汉 430072; 3. 中国科学院大学, 北京 100049)
真核生物核糖体DNA是由高度串联重复序列组成的多基因家族, 与其他蛋白编码核基因不同,核糖体核DNA主要编码核糖体RNA, 具有不同速度的进化区域, 如保守的编码基因18S rDNA、5.8S rDNA和28S rDNA等, 以及基因之间进化较快的转录间隔区域(ITSs), 还包括独立的5S rDNA[1]。其中, 核糖体内转录间隔区域(Internal transcribed spacer, ITSs)属于非编码区, 包括ITS1和ITS2两个片段, 中间由5.8S rDNA隔开, 因其具有选择压力小、位点信息丰富、进化速度相对较快等特点[2], 目前被广泛应用于动物、植物、真菌等类群的系统发育与分类研究[3—7]。5S rDNA与其他核糖体基因相对独立, 位于不同的转录单位[3], 由高度保守的串联重复序列组成, 每个重复单元包含编码区和非转录间隔区(Non transcribed spacer, NTS)。编码区具有高度保守性, 而NTS由于不发生转录, 导致物种在其进化过程中更容易积累其所发生的变异, 因而在不同物种间表现出明显差异[8]。因此, 5S rDNA NTS多态性作为可靠的分子标记同样广泛应用于动植物的分子进化遗传学和系统发育等相关研究[9,10]。
扇贝养殖是我国海水养殖业的主导产业之一,目前国内主要养殖的品种有北方的栉孔扇贝(Chlamys farreri)、南方的华贵栉孔扇贝(Mimachlamys nobilis)以及从美国引进的海湾扇贝(Argopecten irradians)和从日本引进的虾夷扇贝(Patinopecten yessoensis)。然而, 伴随着扇贝养殖产业的迅猛发展, 纵观整个产业链条从苗种生产到成贝养殖均存在诸多问题, 如种质衰退严重、疾病灾害频发、死亡率升高、品质下降等[11]。因此, 新物种的引进可作为一种快速且有效的方法来应对目前扇贝养殖生产中所面临的严峻问题。岩扇贝Rock scallop(Crassadoma gigantea)隶属于软体动物门(Mollusca)、瓣鳃纲(Lamellibranchia)、珍珠贝目(Pterioida)、扇贝科(Pectinidae), 原产于北美太平洋沿岸, 具有个体大、生长速度快、抗逆性强、肉质鲜美等优点, 在国际市场倍受青睐。因此, 大连海洋大学曹善茂教授于2012年开始从加拿大引进岩扇贝种贝, 并于2015年成功完成了全人工室内育苗试验[12], 目前处于养殖阶段。然而, 有关岩扇贝的研究现主要集中在生理生态、生产养殖等方面[13—15],其遗传学背景的研究在国内外却鲜见报道。作为外来引进新物种, 对其遗传学及种质资源的评估资料仍处短缺状态, 这将给生产应用和育种以及推广带来很大困扰。因此, 掌握品种资源的起源及其亲缘关系, 是进行种质创新的基础。
本研究拟通过对岩扇贝、虾夷扇贝、栉孔扇贝及海湾扇贝的核糖体ITS1序列和5S rDNA序列的测定, 分析其遗传变异趋势, 计算种间遗传距离,并结合GenBank数据库中其他扇贝的相应基因序列构建系统发育树, 从分子水平上探讨岩山贝的遗传背景及其系统进化地位, 以期为岩扇贝的种质资源保护及今后的遗传育种研究工作提供部分基础信息。
1 材料与方法
1.1 实验材料
本研究所用的岩扇贝、虾夷扇贝、栉孔扇贝及海湾扇贝样品信息如表 1。所有个体经形态学鉴定后取闭壳肌保存于无水乙醇中备用。GenBank数据库中获得的其他贝类的信息如表 2。
1.2 实验方法
基因组DNA的提取参考Sambrook等[16]的酚/氯仿抽提法并略做修改。取溶解后的2 μL基因组DNA进行琼脂糖凝胶电泳, 检测基因组DNA的完整性, 并取1 μL在NanoDrop 2000微量分光光度计上测定浓度, 最后稀释成50 ng/μL的工作液于4℃条件下保存备用。
引物实验所用引物由上海生物工程技术服务有限公司合成(武汉)。ITS1区域扩增引物序列参考丁小雷等[17]的引物序列, 正向引物F: 5′-GTTCC CCATGAACGAGGAATTCC-3′, 反向引物R: 5′-CGCATTTCGCTGCGTTCTTC-3′; 5S基因扩增引物序列参考José López-Piñón等[6]的引物序列, 包括2对引物, 分别为A (AF: 5′-AACACCGGTTCTCG TCCGATC-3′和AR: 5′-CAACGTGATATGGTC GTAGAC-3′)和B (BF: 5′-AGCCCGGTTAGT ACTTGG-3′和BR: 5′-CGACGTTGCTTAACTTCG-3′)。

表 1 扇贝样品采集信息Tab. 1 Sample collection information of four scallops

表 2 从GenBank中获得的其他物种序列的信息及GenBank登录号Tab. 2 The information of related species and login numbers downloaded from the GenBank database
PCR扩增和测序PCR反应总体系为25 μL,包括1 μL模板DNA(50 ng/μL), 2.5 μL 10×reaction buffer, 1.0 μL dNTP MIX (2.5 mmol/L), 1.0 μL上下游混合引物(各10 μmol/L), 0.2 μL 1 UTaqDNA聚合酶, 18.8 μL超纯水; PCR反应条件是: 94℃预变性5min; 94℃变性35s, 50℃退火40s, 72℃延伸40s, 循环数为38; 最后72℃充分延伸10min。PCR产物于1%琼脂糖凝胶电泳检测, 经BioSpin GelExtraction Kit纯化回收后, 连接到pMD18-T载体上, 再转化到大肠杆菌感受态细胞DH5α, 挑选阳性克隆送天一辉远(武汉)生物科技有限公司进行双向测序。
数据分析用BioEdit7.0进行序列拼接, 采用ClustalX 1.83 软件进行编辑、校对和排序。单倍型数(h)、单倍型多样性指数(Hd) 、核苷酸多样性指数(π)、平均核苷酸差异数(k)等遗传多样性参数使用DNAsp4.10软件进行分析计算, 使用MEGA5.1软件计算种间遗传距离, 并应用邻接法Neighbor-Joining(NJ)构建系统发育树, 系统树中节点的自举置信水平应用Bootstrap(重复次数1000)。
2 结果
2.1 序列分析
四种扇贝ITS1序列分析4种扇贝的ITS1和部分18S的测序结果及GenBank登入号如表 3。首先, 通过PCR扩增测序, 4种扇贝均获得长度为246 bp的部分18S序列和长度不等的ITS1全长序列。ITS1序列长度在229—277 bp, 其中海湾扇贝的序列最长, 而岩扇贝最短。其次, 碱基组成结果显示, 4种扇贝部分18S序列的A+T含量均为48.4%, 低于G+C含量, 无种间差异; 而ITS1序列的A+T含量在54.5%—58.1%, 均高于G+C含量。此外, 4种扇贝ITS1序列的遗传变异参数如表 4, 海湾扇贝、栉孔扇贝、岩扇贝和虾夷扇贝分别获得2、6、2、5个单倍型, 4个种无共有单倍型; 核苷酸变异位点数分别为1、12、1、4, 其中栉孔扇贝和虾夷扇贝均含有2个插入/缺变异位点, 其余均为转换和颠换变异;4种扇贝的单倍型多样性指数、核苷酸多样性指数以及平均核苷酸差异值分别在0.425—0.800、0.00081—0.00486以及0.366—2.442, 所有参数的最高值和最低值均分别为栉孔扇贝和岩扇贝。
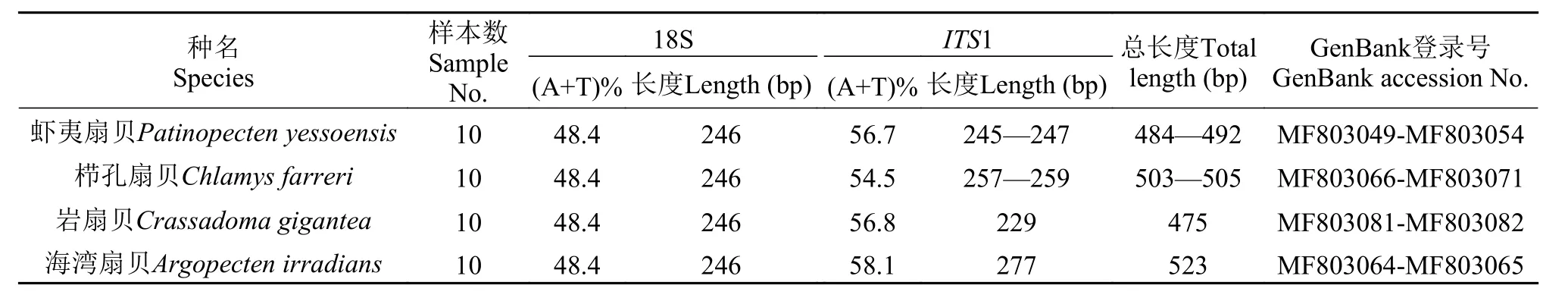
表 3 四种扇贝18S-ITS1测序结果及GenBank登录号Tab. 3 The sequencing results of 18S-ITS1 and GenBank accession No. of four scallops

表 4 四种扇贝ITS1序列的遗传变异分析Tab. 4 Genetic variation analysis of the ITS1 sequences in four scallop
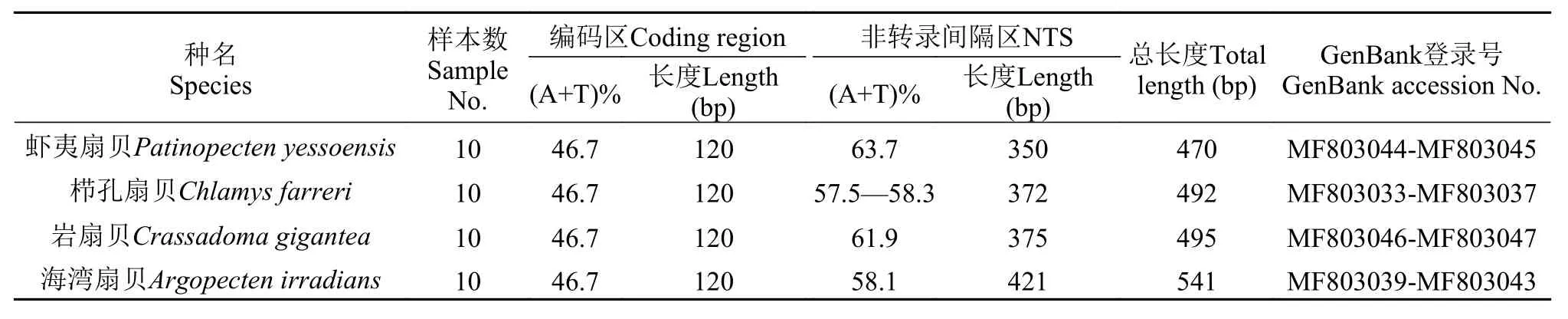
表 5 四种扇贝5S rDNA序列测序结果及GenBank登录号Tab. 5 The sequencing results of 5S rDNA and GenBank accession No. of four scallops
四种扇贝5S序列分析4种扇贝5S序列的测序结果及GenBank登录号如表 5。首先, 通过PCR扩增测序, 4种扇贝均获得长度为120 bp的编码区序列和长度不等的NTS全长序列。NTS序列总长度在350—421, 序列最长和最短的分别为海湾扇贝和虾夷扇贝。其次, 碱基组成结果显示, 4种扇贝的5S rDNA编码区序列的A+T含量均为46.7%, 低于G+C含量; 而在NTS序列中的A+T含量在57.5%—63.7%, 均高于G+C含量。此外, 4种扇贝5S rDNA NTS序列的遗传变异参数如表 6, 海湾扇贝、栉孔扇贝、岩扇贝和虾夷扇贝分别获得5、5、2、3个单倍型, 4个种无共有单倍型; 核苷酸变异位点数分别为12、14、1、4, 所有变异位点均为转换和颠换变异; 4种扇贝的单倍型多样性指数、核苷酸多样性指数以及平均核苷酸差异值分别在0.533—0.842、0.00108—0.01058、0.533—5.726,所有参数的最高值和最低值均分别为海湾扇贝和岩扇贝。
2.2 种间遗传距离
利用获得的所有ITS1以及5Sr DNA序列分别对4种扇贝进行种间遗传距离计算, 结果如表 7。首先, 对角线上方的数值为基于5Sr DNA序列计算所得, 4种扇贝相互之间的遗传距离在0.043—0.457,其中最大值为海湾扇贝与栉孔扇贝之间的遗传距离, 最小值为岩扇贝与虾夷扇贝之间的遗传距离,而岩扇贝与栉孔扇贝和海湾扇贝的遗传距离分别为0.096和0.413。其次, 对角线下方数值为基于ITS1序列计算所得, 4种扇贝相互之间的遗传距离在0.040—0.131, 其中最大值为海湾扇贝与栉孔扇贝之间的遗传距离, 最小值为岩扇贝与虾夷扇贝之间的遗传距离, 而岩扇贝与栉孔扇贝和海湾扇贝的遗传距离分别为0.062和0.121。总体上看, 基于2种方法所得结果的趋势较为一致。但也存在一定的差异, 如基于ITS1序列的结果显示海湾扇贝与虾夷扇贝间的遗传距离(0.124)略微大于海湾扇贝与岩扇贝间的遗传距离(0.121), 而基于5S rDNA序列的计算结果却与之相反, 但数值上同样相差0.003。
2.3 系统发育树
本研究利用获得的所有ITS1以及5S rDNA的单倍型序列, 并结合GenBank数据库中其他扇贝的相应序列, 利用邻接法Neighbor-Joining (NJ) 构建系统发育树, 选取牡蛎科的两个种作为外源群。目前GenBank数据库中能够获得的扇贝科物种的相关数据较少, 本研究仅对获得的部分数据进行分析。如图 1和图 2所示, 首先, 基于ITS1序列构建的系统进化树结果显示(图 1), 除外源群牡蛎科的两个物种外, 扇贝科的10个物种明显分为2个单系群, 置信度在90%以上, 其中1个单系群包括Patinopecten、Crassadoma、Chlamys以及Mimachlamys四个扇贝属, 该单系群又分为2个分支, 其中本研究所得虾夷扇贝的5个单倍型、岩扇贝的2个单倍型以及海湾扇贝的6个单倍型分别聚为一支, 虾夷扇贝与岩扇贝聚类之后再与栉孔扇贝聚类, 但Chlamys中的一个种却与Mimachlamys中的2个聚为了另一支; 另一个单系群包括Argopecten、Pecten以及Aequipecten三个扇贝属, 其中本研究所得海湾扇贝的2个单倍型与紫扇贝聚为一支, 再与女王扇贝聚类, 最后与欧洲扇贝聚为一个单系群。此外, 基于5S序列构建的系统进化树结果显示(图 4), 其不同扇贝属间的聚类结果与基于ITS1序列构建的系统进化树结果完全一致, 但置信度值存在一定差异。
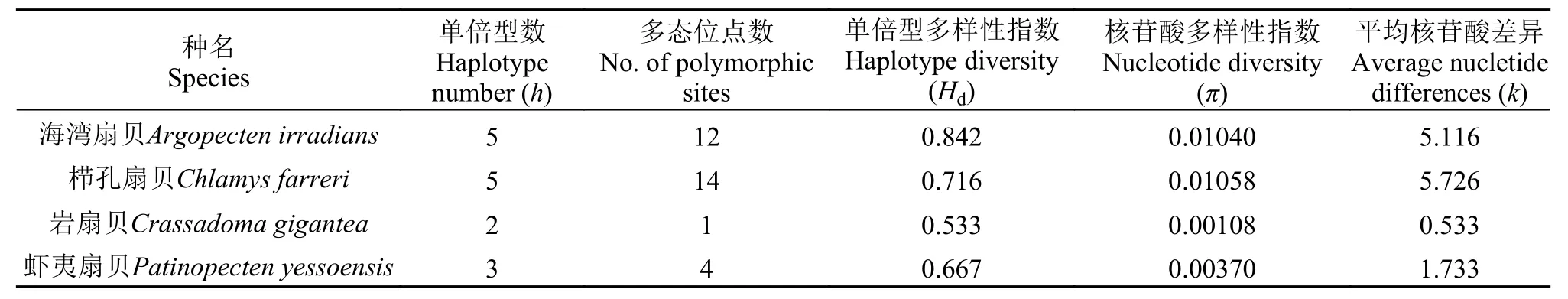
表 6 四种扇贝5S rDNA序列的遗传变异分析Tab. 6 Genetic variation analysis based on the sequences of 5S rDNA in four scallops
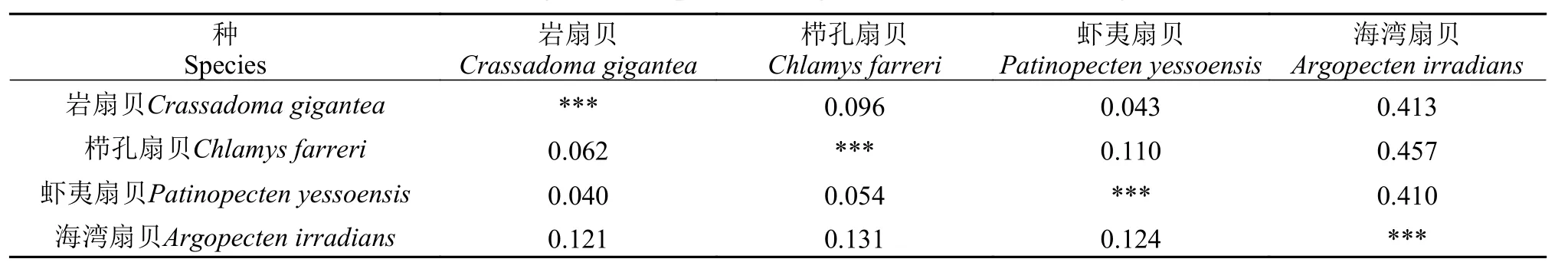
表 7 四种扇贝的种间遗传距离(对角线下方为ITS1; 对角线上方为5S rDNA)Tab. 7 Genetic distance among four scallops(below diagonal is for ITS1; above diagonal is for 5S rDNA)

图 1 应用邻接法(NJ)构建基于ITS1序列的扇贝科系统进化树Fig. 1 The Neighbor-joining (NJ) phylogenetic tree in Pectinidae constructed with the ITS1 sequences

图 2 应用邻接法(NJ)构建基于5S rDNA序列的扇贝科系统进化树Fig. 2 The Neighbor-joining (NJ) phylogenetic tree in Pectinidae constructed with the 5S rDNA sequences
3 讨论
3.1 四种扇贝ITS1序列特征
核糖体DNA转录间隔区ITS1, 具有变异较大、多态性较高等特点, 适合亲缘关系较近的物种遗传多样性研究[18], 同时已广泛应用于物种鉴定和系统发育研究[19—21]。本研究通过测序, 获得了4种扇贝部分18S rDNA序列和ITS1全序列。首先, 所得的18S rDNA序列经Clustal X比对分析发现, 4种扇贝具有高度同源性, 且A+T含量均为48.37%, 低于G+C含量, 无物种间差异, 该研究结果与丁小雷等[17]对双壳类的相关研究结论一致, 因此学者们普遍认为18S rDNA比较适合于动植物高级阶元类的遗传差异及系统发育等相关研究[4,22]。而4种扇贝ITS1序列的种间同源性较低, 有包括转换、颠换以及插入/缺失在内的核苷酸变异, 且存在明显的种间长度多态。有报道,ITS1序列最短的仅有70 bp(鹿茸珊瑚Acropora longicyathus)[23], 而最长的达760 bp(克氏原螯虾Procambarus clarkii)[24], 因此许多学者认为ITS1可作为物种鉴定的有效分子标记[1,5]。此外, 本研究所得4种扇贝ITS1序列的碱基组成具有一定差异, 其A+T含量在54.48%—58.1%, 均高于G+C含量, 该趋势与Insua等[6]在其他扇贝中的研究结果一致。Freire等[25]曾报道, 在双壳类动物中,ITS1序列的A+T含量一般在34%—55%, 但在随后相关研究却表明双壳类的A+T含量普遍接近该范围的最高值, 甚至更高[6]。
3.2 四种扇贝5S rDNA序列特征
5S rDNA因拷贝数多且具有高度保守性, 以及在核基因组不同拷贝间趋于相近等特点, 通过FISH定位可水生生物类的染色体鉴别、倍性鉴定、遗传标记开发等提供新的有效途径, 目前在双壳类中已有许多相关研究报道, 如Insua等[26]利用5S rDNA对欧洲土著的2种扇贝(Pecten maximus和Mimachlamys varia)进行了核型及染色体定位研究,Huang等[27—29]利用5S rDNA分别对栉孔扇贝、虾夷扇贝以及海湾扇贝进行了染色体定位等相关研究。此外, 5S rDNA NTS由于具有高度变异的特点,已作为可靠的分子标记被广泛应用于动植物的遗传进化及系统发育相关研究, 尤其在植物方面有大量的研究报道[10,30]。本研究通过测序, 获得了4种扇贝的5S rDNA全序列, 其编码区序列均为120 bp,序列经Clustal X比对发现具有高度的种间同源性,同时与José López-Piñón等[9]获得的4种欧洲扇贝5S rDNA编码区序列也具有高度同源性。然而4种扇贝NTS序列的种间同源性较低, 同时具有转换、颠换和插入/缺失变异, 且存在明显的种间长度多态,该结果与José López-Piñón等[9]对其他扇贝的相关研究结果一致。碱基组成结果显示, 4种扇贝NTS序列的A+T含量(57.5%—63%)明显高于G+C含量, 该趋势与其他扇贝的结果一致[9], 但与谢明树等[31]对鱼类研究所得的结果却相反, 在哺乳类中也存在类似的现象(G+C>60%)[32,33], 对于该现象的解释, 至今尚未有学者进行详细报道, 但笔者猜测这可能是不同物种通过对环境长期适应适应的机制而造成的差异。
3.3 四种扇贝ITS1和5S rDNA序列的遗传变异分析
目前, 核苷酸多样性指数(π)和单倍型多样性指数(Hd)通常是评估种群遗传多样性的两个重要指标, 它们反映了物种遗传多样性水平的高低[34]。但由于单个碱基的变异就能生成一种新的单倍型, 因此Hd值可在短时间内积累变异而快速提高, 但对π的影响却很小,π值的提高需要长时间的积累, 因而π值在衡量种群遗传多样性时比Hd值更具有代表性[35]。首先, 本研究中基于ITS1序列的分析结果表明, 栉孔扇贝和虾夷扇贝的Hd最高(均为0.800), 岩扇贝最低, 并与海湾扇贝相差不大, 但虾夷扇贝的π值明显低于栉孔扇贝。因此, 鉴于郝桂英等[35]的结论, 笔者认为本研究采样区的4种扇贝中, 栉孔扇贝的遗传多样性最高(0.00486), 其次是虾夷扇贝(0.00252), 而岩扇贝的遗传多样性最低。而基于5S序列的分析结果显示, 栉孔扇贝的遗传多样最高(π=0.01058), 其次海湾扇贝是(π=0.01040), 但两者的遗传多样性水平较接近, 而岩扇贝的遗传多样性依然最低(π=0.00108)。通常来说, 种群遗传多样性是种群活力的基础, 是物种适应环境变化、维持生存和进化的重要保证[36]。综合本研究中ITS1序列和5S rDNA序列的分析结果看, 尽管2种基因的分析结果存在一定的差异, 但笔者认为采样区的海湾扇贝、虾夷扇贝和栉孔扇贝均表现出较低的遗传多样性(π<0.01)[36], 将不利物种适应环境变化而维持生存, 这可能与实验样本均为养殖群体有关。至于造成2种基因的结果差异, 一方面原因可能是实验样本量不够大以及采样地区的限制, 另一方面则是不同基因的进化速度本身存在差异。而引进种岩扇贝则表现出更低的遗传多样性, 这可能是由于实验样本为F1代个体, 仅由少数几个引进亲本近交繁育而来, 其遗传背景相对单一, 胡丽萍等[37]曾在其他扇贝中也报道了类似现象。
3.4 四种扇贝的遗传距离及系统发育分析
本研究通过获得的所有ITS1序列和5S rDNA序列, 计算了4种扇贝相互之间的遗传距离。首先, 基于ITS1序列的遗传距离分析结果表明, 引进种岩扇贝与虾夷扇贝间的遗传距离最近(0.040), 紧接着是栉孔扇贝(0.062), 与海湾扇贝之间的遗传距离最远(0.121); 而其余3种扇贝则是虾夷扇贝与栉孔扇贝之间的遗传距离最近(0.054), 此部分结果与秦艳杰等[38]应用AFLP技术的研究结果一致。同时, 在本研究中基于5S序列的遗传距离分析结果与基于ITS1序列的分析结果一致, 这进一步证实了4种扇贝间亲缘关系的远近。
此外, 本研究还利用获得的所有ITS1和5S rDNA单倍型序列以及GenBank数据库中其他扇贝的相应序列, 构建系统发育树, 以探讨不同扇贝间的亲缘关系。结果显示, 基于ITS1序列和5S rDNA序列的系统发育树聚类结果完全一致, 引进种岩扇贝先与虾夷扇贝聚为一支, 再与栉孔扇贝进行聚类,而海湾扇贝则与其他扇贝聚为另一个单系群。目前基于ITS1和5S rDNA序列在扇贝系统进化方面的研究鲜有报道, 仅有Insua等[6]和José López-Piñón等[9]对欧洲的4种扇贝进行了相关研究。大量文献资料却显示, 扇贝系统发育及进化等相关的研究主要是基于线粒体基因序列进行开展(COI、16S、12S等), 如Canapa 等[39]通过对6种扇贝16S序列的比较分析, 探讨了这些扇贝物种的系统发生关系;Barucca等[40]利用16S和12S序列的比较分析, 构建了系统进化树; Puslednik等[41]同时采用线粒体基因(16S和12S )以及核基因(H3 组蛋白)的部分序列构建了46种扇贝的系统进化树, 包括引进种岩扇贝在内, 其结果表明虾夷扇贝先与冰岛扇贝(Chlamys islandica)聚为一个分支, 再与岩扇贝进行聚类, 而与海湾扇贝相距较远, 但文中未对栉孔扇贝(C. farreri)的亲缘关系进行报道。
4 结论
综上所述, 本研究通过测序获得了岩扇贝、海湾扇贝、虾夷扇贝、栉孔扇贝的核糖体内转录间隔区ITS1及5S rDNA的序列, 首先对其序列的碱基组成及遗传多样性进行了分析, 相比4种扇贝的分析结果看栉孔扇贝的遗传多样性最高, 岩扇贝的遗传多样性最低, 但总体来看, 4种扇贝均表现出较低的多样性水平; 其次, 利用获得的所有ITS1序列和5S rDNA序列分别对4种扇贝之间的遗传距离进行了计算, 2种方法的结果均表明岩扇贝与虾夷扇贝的遗传距离最近, 而与海湾扇贝的遗传距离最远;最后, 利用获得的4种扇贝的单倍型序列以及Ge-Bank中其他扇贝的相应序列, 基于NJ法构建了系统进化树, 基于ITS1序列和5S rDNA序列分别构建的系统发育树聚类结果完全一致, 引进种岩扇贝先与虾夷扇贝聚为一支, 再与栉孔扇贝进行聚类, 而海湾扇贝则与别的扇贝聚为另一个单系群。该研究结果将为岩扇贝的引种与养殖以及今后的遗传育种研究工作提供一些基础数据。
参考文献:
[1] Ferreira I A, Oliveira C, Venere P C,et al. 5S rDNA variation and its phylogenetic inference in the genusLeporinus(Characiformes: Anostomidae) [J].Genetica, 2007,129(3): 253—257
[2] Hillis D M, Dixon M T. Ribosomal DNA: molecular evolution and phylogenetic inference [J].The Quarterly Review of Biology, 1991, 66(4): 411—453
[3] Zhu M, Liu Z H, Kuang Y C. The applications of nuclear genes in fish molecular systematic [J].Journalof Chongqing Normal University, 2013, 30(2): 10—16 [朱敏, 刘智皓, 旷毓婵, 等. 核基因在鱼类分子系统学研究中的应用. 重庆师范大学学报(自然科学版), 2013,30(2): 10—16]
[4] Fan J, Bai Y, Shu M Y. ITS1 and ITS2 sequences analysis of seven medicinal plants inPolygalaL [J].Chinese Traditional & Herbal Drugs, 2015, 46(4): 562—565 [樊杰, 白妍, 束明月, 等. 远志属7种药用植物ITS1和ITS2序列分析. 中草药, 2015, 46(4): 562—565]
[5] Parengul.Rahat, Jiao L, Yue C. Molecular identification ofDiplostomum paracaudumonEsox luciusin Irtysh River [J].Acta Hydrobiologica Sinica, 2016, 40(5):992—996 [番林古丽.热哈提, 焦丽, 岳城. 额尔齐斯河白斑狗鱼假尾复口吸虫的分子鉴定. 水生生物学报,2016, 40(5): 992—996]
[6] Insua A, López-Piñón M J, Freire R,et al. Sequence analysis of the ribosomal DNA internal transcribed spacer region in some scallop species (Mollusca: Bivalvia: Pectinidae) [J].Genome, 2003, 46(4): 595—604
[7] Guo P H, Liu X L, Cui Y P,et al. The value of universal fungal primers Its1 and Its4 in the clinical identification of filamentous fungi [J].Chinese Journal of Microecology, 2013, 25(8): 922—924 [郭鹏豪, 刘秀丽, 崔颖鹏,等. 真菌通用引物Its1和Its4在丝状真菌鉴定中的价值评价. 中国微生态学杂志, 2013, 25(8): 922—924]
[8] Qin Q, Wang Y, Wang J,et al. The autotetraploid fish derived from hybridization ofCarassius auratusred var.(female)×Megalobrama amblycephala(male) [J].Biology of Reproduction, 2014, 91(4): 93, 1—11
[9] José López-Piñón M, Freire R, Insua A,et al. Sequence characterization and phylogenetic analysis of the 5S ribosomal DNA in some scallops (Bivalvia: Pectinidae) [J].Hereditas, 2008, 145(1): 9—19
[10] Zhang M, Zhang D Z, Xu X H,et al. 5S rRNA gene spacer sequences from ligularia medicinal plants and the identification of HPAs-containing species [J].Chinese Journal of Natural Medicines, 2005, 3(1): 38—40 [张勉,张达治, 许翔鸿, 等. 橐吾属药用植物5S rRNA基因间隔区序列与含HPAs植物的鉴别. 中国天然药物, 2005,3(1): 38—40]
[11] Kou L X. Introduction of four scallop species mainly cultured in China [J].Hebei Fisheries, 2012, (6): 56—59 [寇凌霄. 我国四个主要扇贝品种介绍及养殖发展对策. 河北渔业, 2012, (6): 56—59]
[12] Cao S M, Wang J, Wang Q,et al. Artificial breeding of introduced rock scallopCrassadoma gigantea[J].Journal of Dalian Ocean University, 2017, 32(1): 1—6 [曹善茂, 汪健, 王谦, 等. 岩扇贝人工育苗的初步研究. 大连海洋大学学报, 2017, 32(1): 1—6]
[13] MacDonald B A, Bourne N F. Growth of the purple-hinge rock scallop,Crassadoma giganteaGray, 1825 under natural conditions and those associated with suspended culture [J].Journal of Shellfish Research, 1989, 8(1):179—186
[14] Beitler M K. Toxicity of adductor muscles from the purple hinge rock scallop (Crassadoma gigantea) along the Pacific coast of North America [J].Toxicon, 1991,29(7): 889—894
[15] Culver C S, Richards J B, Page H M. Manipulation of the cementing process of the purple-hinge rock scallop,Crassadoma gigantea[J].Journal of Shellfish Research,2000, 19(6): 651—652
[16] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning:a Laboratory Manual [M]. Beijing: Science Press. 1995,27—29 [萨姆布鲁克 J, 弗里奇 E F, 曼尼阿蒂斯 T. 分子克隆实验指南. 北京: 科学出版社. 1995, 27—29]
[17] Ding X L, He M X, Deng F J,et al. 18S-ITS1 sequence of rRNA in bivalves and its application in phylogenetic analysis [J].Hereditas, 2004, 26(3): 319—324 [丁小雷,何毛贤, 邓凤姣, 等. 双壳纲动物核糖体RNA 18SITS1序列及其在分子系统发育研究中的应用. 遗传,2004, 26(3): 319—324]
[18] Yu D H, Li Y NI, Wu K C. Analysis on sequence variation ofITS2 rDNA inPinctada fucatafrom China, Japan and Australia [J].South China Fisheries Science, 2005,1(2): 1—6 [喻达辉, 李有宁, 吴开畅. 中国, 日本和澳大利亚珍珠贝的ITS2序列特征分析. 南方水产科学,2005, 1(2): 1—6]
[19] Chu K H, Li C P, Ho H Y. The first internal transcribed spacer (ITS-1) of ribosomal DNA as a molecular marker for phylogenetic and population analyses in Crustacea [J].Marine Biotechnology, 2001, 3(4): 355—361
[20] Chen C A, Chen C P, Fan T Y,et al. Nucleotide sequences of ribosomal internal transcribed spacers and their utility in distinguishing closely relatedPerinereis polychaets(Annelida; Polychaeta; Nereididae) [J].Marine Biotechnology, 2002, 4(1): 17—29
[21] Lopez-Pinon M J, Insua A, Mendez J. Identification of four scallop species using PCP and restriction analysis of the ribosomal DNA internal transcribed spacer region [J].Marine Biotechnology, 2002, 4(5): 495—502
[22] Liu D F, Jiang G F. The application of nuclear genes sequences in insect molecular systematic [J].Zoological Systematics, 2005, 30(3): 484—492 [刘殿锋, 蒋国芳. 核基因序列在昆虫分子系统学上的应用. 动物分类学报,2005, 30(3): 484—492]
[23] Odorico D M, Miller D J. Variation in the ribosomal internal transcribed spacers and 5.8S rDNA among five species of Acropora (Cnidaria; Scleractinia): patterns of variation consistent with reticulate evolution [J].Molecular Biology & Evolution, 1997, 14(5): 465
[24] Harris D J, Crandall K A. Intragenomic variation withinITS1 andITS2 of freshwater crayfishes (Decapoda: Cambaridae): Implications for phylogenetic and microsatellite studies [J].Molecular Biology & Evolution, 2000, 17(2):284
[25] Freire, R. Análisis de secuencias de ADN ribosómico en berberechos y mejillones de la costa europea. PhD. thesis,Universidade da Coruña, Coruña, Spain. 2002
[26] Insua A, López-Piñón M J, Freire R,et al. Karyotype and chromosomal location of 18S-28S and 5S ribosomal DNA in the scallopsPecten maximusandMimachlamys varia(Bivalvia: Pectinidae) [J].Genetica, 2006, 126(3):291—301
[27] HUANG, Xiaoting, Zhenmin,et al. Chromosomal localization of the major ribosomal RNA genes in scallopChlamys farreri[J].Acta Oceanologica Sinica, 2006,25(3): 108—115
[28] Huang X, Hu X, Hu J,et al. Mapping of ribosomal DNA and (TTAGGG)n telomeric sequence by FISH in the bivalvePatinopecten yessoensis(Jay, 1857) [J].Journal of Molluscan Studies, 2007, 73(4): 393—398
[29] Huang X, Hu J, Hu X,et al. Cytogenetic characterization of the bay scallop,Argopecten irradians irradians, by multiple staining techniques and fluorescencein situhybridization [J].Genes & Genetic Systems, 2007, 82(3):257
[30] Tan R, Ma D Q, Ding Y. Comparative analysis of sequences of the 5S rDNA NTS in wild close relatives of barley from Tibet of China [J].Journal of Genetics and Genomics, 2005, 32(10): 1094—1100 [谭睿, 马得泉, 丁毅. 中国西藏近缘野生大麦 5S rDNA NTS 序列分析.遗传学报, 2005, 32(10): 1094—1100]
[31] Xie M S, Sun B, Zhang B,et al. Chromosome mapping of 5S rDNA in olive flounder (Paralichthys olivaceus) and half-smooth tongue-sole (Cynoglossus semilaevis) and molecular systematics analysis of five flatfishes [J].Journal of Fisheries of China, 2012, 36(8): 1159—1166[谢明树, 孙冰, 张博, 等. 牙鲆和半滑舌鳎5S rDNA基因的染色体定位及鲽形目5种鱼类的分子系统学分析. 水产学报, 2012, 36(8): 1159—1166]
[32] Suzuki H, Moriwaki K, Sakurai S. Sequences and evolutionary analysis of mouse 5S rDNAs [J].Molecular Biology & Evolution, 1994, 11(4): 704—710
[33] Suzuki H, Sakurai S, Matsuda Y. Rat 5S rDNA spacer se-quences and chromosomal assignment of the genes to the extreme terminal region of chromosome 19 [J].Cytogenetic & Genome Research, 1996, 72(1): 1—4
[34] Wu S G, Wang G T, Xi B W,et al. Population genetic structure of the parasitic nematodeCamallanus cottiinferred from DNA sequences ofITS1 rDNA and the mitochondrialCOI gene [J].Veterinary Parasitology, 2009,164(2): 248—256
[35] Hao G Y, Yang Y D, Gu X B,et al. Population genetic diversity ofTaenia multicepsisolated from Sichuan based on mitochondrial cox1 and Cytbgene [J].Chinese Journal of Animal & Veterinary Sciences, 2014, 45(4): 631—638 [郝桂英, 杨应东, 古小彬, 等. 基于线粒体cox1和Cytb基因对四川地区多头带绦虫的种群遗传多样性研究. 畜牧兽医学报, 2014, 45(4): 631—638]
[36] Gu X B, Zhu J Y, Wang B J,et al. Genetic variation in mitochondrial cox2 ofHeterakis gallinarumfrom poultry in Sichuan, China [J].Acta Veterinaria et Zootechnica Sinica, 2016, 47(4): 796—804 [古小彬, 朱俊扬, 王保健,等. 四川地区鸡异刺线虫核糖体转录间隔区(ITS1/2)序列的遗传变异分析. 畜牧兽医学报, 2016, 47(4): 796—804]
[37] Hu L P, Jiang L M, Huang X T,et al. Phylogenetic position ofArgopecten purpuratusin genusArgopecteninferred by mitochondrial 16S rDNA gene sequences [J].Oceanologia et Limnologia Sinica, 2016, 47(6):1149—1157 [胡丽萍, 姜黎明, 黄晓婷, 等. 基于16S rDNA基因序列探讨引进物种紫扇贝(Argopecten purpuratus)在海湾扇贝属(Argopecten)中的分类地位. 海洋与湖沼, 2016, 47(6): 1149—1157]
[38] Qin Y, LI X, Zhou B,et al. Genetic relationship ofPlacopecten magellanicusand three scallop species by AFLP[J].Journal of Northeast Agricultural University, 2009,40(10): 69—74 [秦艳杰, 李霞, 周伯文, 等. 利用AFLP技术分析海扇贝与三种扇贝的亲缘关系. 東北農業大學學報, 2009, 40(10): 69—74]
[39] Canapa A, Barucca M, Marinelli A,et al. Molecular data from the 16S rRNA gene for the phylogeny of Pectinidae(Mollusca: Bivalvia) [J].Journal of Molecular Evolution,2000, 50(1): 93—97
[40] Barucca M, Olmo E, Schiaparelli S,et al. Molecular phylogeny of the family Pectinidae (Mollusca: Bivalvia)based on mitochondrial 16S and 12S rRNA genes [J].Molecular Phylogenetics and Evolution, 2004, 31(1):89—95
[41] Puslednik L, Serb J M. Molecular phylogenetics of the Pectinidae (Mollusca: Bivalvia) and effect of increased taxon sampling and outgroup selection on tree topology[J].Molecular Phylogenetics and Evolution, 2008, 48(3):1178—1188
