大鲵短型肽聚糖识别蛋白PGRP-S的克隆及其功能研究
2018-05-16任诗思齐志涛昌鸣先
任诗思 齐志涛 昌鸣先
(1. 中国科学院水生生物研究所, 农业部水产养殖病害防控重点实验室, 淡水生态与生物技术国家重点实验室, 武汉 430072;2. 中国科学院大学, 北京 100049; 3. 盐城工学院海洋与生物工程学院, 盐城 224051)
肽聚糖识别蛋白(Peptidoglycan recognition protein, PGRPs)是一类广泛存在于无脊椎动物和脊椎动物中, 能特异地识别细菌细胞壁肽聚糖(Peptidoglycan, PGN)成分的高度保守的模式识别受体。PGRP最早是在家蚕的血淋巴和角质层中被发现[1]。之后, 随着黑腹果蝇(Drosophila elanogaster)、冈比亚按蚊(Anopheles gambiae)及人类基因组测序的发展, 相继在哺乳动物和昆虫中发现了PGRPs家族的众多成员[2—4]。此后, 在其他的无脊椎动物(如软体动物、棘皮动物)和脊椎动物(鱼类、两栖类、鸟类)中也相继有PGRPs基因被发现和鉴定[5—9]。
昆虫PGRPs家族成员根据其mRNA转录本的大小分为短型PGRP (PGRP-S)和长型PGRP (PGRPL)。哺乳动物的4个PGRPs成员, 最初按照mRNA转录本的大小命名为短型PGRP (PGRP-S)、中型PGRP (PGRP-I, 包括PGRP-Iα/PGRP-Iβ)和长型PGRP (PGRP-L)[4], 后更名为PGLYRP-1、PGLYRP-2、PGLYRP-3、PGLYRP-4。短型PGRP(PGRP-S), 大约含有200个氨基酸残基, 蛋白分子量19—20 kD左右, 其含有的PGRP结构域占据其绝大部分序列。有的中型PGRP (如哺乳动物PGLYRP-3、PGLYRP-4)或长型的PGRP (如果蝇PGRPLF)则含有2个PGRP结构域。此外, 长型PGRP除了C端的PGRP结构域外, 还包含一段长度可变的N端序列; 且该N端序列在PGRPs家族成员中不保守, 赋予每个PGRP独特性和功能多样性。PGRPs家族成员大多数为分泌蛋白(如果蝇PGRP-SA和PGRPSD、哺乳动物PGLYRP1等), 少部分是跨膜蛋白(如果蝇PGRP-LF)和胞内蛋白(如果蝇PGRP-LE)。
在天然免疫系统中, PGRPs主要作为模式识别受体、调节子和效应分子发挥作用。在不同的物种中PGRPs的功能具有一定的保守性, 但也存在着明显的差异。昆虫PGRPs功能主要包括激活酚氧化酶原级联反应、激活Toll信号通路、激活IMD信号通路以及具有酰胺酶活性[5]。哺乳动物PGRPs的功能主要包括具有酰胺酶活性和直接的杀菌活性2个方面[10]。鱼类作为低等脊椎动物, 进化上处于昆虫和哺乳动物之间, 它们的PGRPs却同时兼具酰胺酶活性和直接的杀菌活性[8]。两栖动物在进化上介于鱼类和哺乳动物之间, 目前关于两栖类的PGRPs报道甚少。而大鲵(Andrias davidianus, 俗称“娃娃鱼”), 作为现存个体最大的两栖动物, 存在已有三亿五千多万年的历史, 有“活化石”之称, 具有极高的学术科研价值、医用和食用等经济价值。近年来, 由于自然环境的破坏, 非法捕杀、走私贩卖等原因, 野生大鲵资源量急剧下降; 而人工养殖大鲵随着养殖规模的扩大, 大鲵的各种病害也接踵而来, 其细菌性以及病毒性疾病成为大鲵产业发展的限制瓶颈。
我们以往的研究揭示了硬骨鱼类短型的PGRPSC或者PGRP5、长型的PGRP6的选择性剪接及其免疫功能[11—14]。本研究构建了两栖动物大鲵短型肽聚糖识别蛋白PGRP-S的真核表达质粒, 并研究了其抗菌活性、PGN结合活性、酰胺酶活性以及对NF-κB启动子活性的影响。该研究为揭示脊椎动物PGRPs在进化中的功能异同以及大鲵的免疫防治奠定了重要的理论基础。
1 材料与方法
1.1 细胞系、细菌菌株及主要试剂
HEK293T (Human embryonic kidney cell)和迟缓爱德华氏菌(Edwardsiella tardaPPD130/91)由本实验室保存并培养。Lys-type PGN (No.77140)或Dap-type PGN (No. 69554)购买于Sigma公司。
1.2 真核表达质粒构建
大鲵PGRP-S的真核表达质粒由盐城工学院海洋与生物工程学院的齐志涛博士克隆并提供。真核表达的上游引物为CCCAAGCTTATGGGTGA GTGCTTGTTAC; 下游引物为CGGGATCCGGGCA GCAGAGTCGGCGTTTTTTCTGTG, 下划线部分为限制性内切酶位点。
1.3 生物信息学分析
大鲵PGRP-S的结构域通过NCBI网站CD-search (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测; 使用SignalP 3.0 (http://www.cbs.dtu.dk/services/SignalP/)预测蛋白的信号肽; 多序列比对使用ClustalW1.83分析; 不同物种同源基因的相似性用MatGat2.02分析[15]; 大鲵PGRP-S和其他脊椎动物PGRPs的进化关系采用MEGA4.0软件的邻接法(Neighbor-Joining, NJ)构建的系统进化树来分析,设置1000次bootstraps进行评估。
1.4 NF-κB活性分析
将HEK293T细胞接种24孔板(3×105/孔), 37℃5% CO2细胞培养箱中培养过夜。将200ng的pNF-κB-Luc报告质粒和1 ng Rellina内参质粒分别与不同量的p3xFLAG或PGRP-S-FLAG质粒共转染HEK293T细胞。48h后收集并裂解细胞, 用Promega公司的Dual-Luciferase Reporter Assay System检测样品的荧光素酶活性。
1.5 抗菌活性分析
胞内抗菌活性分析 将HEK293T细胞接种24孔板并培养过夜, 分别将p3xFLAG或PGRP-SFLAG转染到细胞中(0.5 μg/孔)。随后用迟缓爱德华氏菌(MOI=10)感染过表达的细胞(170×g, 离心5min; 25℃, 感染1h), 用1×PBS轻轻洗涤细胞3次,加入100 μg/mL庆大霉素的DMEM高糖培养基杀胞外菌1h, 随后再更换为16 μg/mL庆大霉素的DMEM高糖培养基。分别在感染3h和6h后用1×PBS缓冲液洗涤细胞3—5次, 再用含1%Triton-X100的PBS缓冲液裂解细胞并将细胞裂解产物梯度稀释后涂布平板, 计数。
胞外抗菌活性分析 细胞种板及感染步骤同上, 不加入含有庆大霉素的DMEM培养基。感染3h和6h后, 分别取细胞培养上清梯度稀释后涂布平板, 计数。
1.6 肽聚糖结合活性分析
将HEK293T细胞接种6孔板(1×106/孔)并培养过夜, 分别将pTurbo-GFP、p3xFLAG或PGRP-SFLAG转染到细胞中(2.5 μg/孔), 36—48h后提取细胞总蛋白。分别将40 μg不溶的Lys-type PGN或Dap-type PGN与100 μL总蛋白低温孵育3—4h后,离心除去未结合的蛋白, 重悬洗涤4次后通过Western-blotting检测蛋白结合情况。
1.7 酰胺酶活性分析
HEK293T细胞接种到25cm2的细胞培养瓶(5×106—1×107/瓶)并在培养过夜后, 将10 μg p3×FLAG或PGRP-S-FLAG转染到细胞之中,36—48h后提取细胞总蛋白。分别将40 μg不溶的Lys-type PGN或Dap-type PGN与50 μg p3xFLAG或PGRP-S-FLAG总蛋白加入到Tris-ZnCl2缓冲液中(20 mmol/L Tris-HCl, 150 mmol/L NaCl, 10 μmol/L ZnCl2, pH7.2), 室温孵育120min, 每10min用全波长分光光度计读取λ540的吸光度值。
2 结果
2.1 大鲵PGRP-S 开放阅读框及其编码的氨基酸序列
大鲵PGRP-S的ORF长495bp, 编码165个氨基酸, 在其53—161氨基酸处含有一个保守的PGRP结构域。SignalP软件分析, 大鲵PGRP-S的N端不含有信号肽。通过ClustalX1.8对大鲵PGRP-S和其他物种的PGRPs氨基酸序列进行同源性比较分析, 发现大鲵PGRP-S具有2个相距较近的半胱氨酸残基, 而这2个半胱氨酸对于PGRP的功能和结构完整性都是十分重要的[5,10,16]。此外, 大鲵PGRP-S具有保守的2个Zn2+结合位点(H83和Y117)。
MatGat2.0相似性比对显示大鲵PGRP-S与其他物种的PGRPs相似性在19.1%—50.5%, 其中与非洲爪蟾PGLYRP1相似性最高, 为50.5%; 其次是热带爪蟾和牛的PGLYRP1, 分别为45.1%和42.1%; 与鲤科鱼类短型PGRP5的相似性为29.8%—36.1%; 与其他脊椎动物长型PGRP2和PGRP6的相似性为19.1%—22.4% (表 1)。
2.2 大鲵PGRP-S系统进化树分析
使用MEGA4.0软件邻接法构建大鲵PGRP-S和其他脊椎动物PGRPs的系统进化树, 结果表明大鲵PGRP-S与两栖动物爪蟾的短型肽聚糖识别蛋白PGLYRP1的亲缘关系最近; 同时哺乳动物短型PGLYRP1与中间型PGLYRP-3和PGLYRP-4聚为一个大的分支(图 1)。
2.3 大鲵PGRP-S真核表达产物免疫印迹检测
由于HEK293T细胞高的转染效率, 我们以HEK293T细胞作为模型研究大鲵PGRP-S的功能。用重组质粒PGRP-S-FLAG以及相应的空质粒p3xFLAG转染HEK293T细胞36h后提取蛋白, 经SDS-PAGE电泳后, 转PVDF膜, Western-blotting分析结果显示HEK293T细胞的胞外培养基以及细胞溶胞产物中在15—25 kD有特异性条带, 与目的蛋白理论分子量大小一致(18 kD左右), 而空质粒p3xFLAG转染组无特异性条带, 表明目的蛋白在HEK293T细胞胞外和胞内都有表达。

表 1 大鲵PGRP-S与其他物种的PGRPs氨基酸序列相同性/相似性比较Tab. 1 Amino acid identities/similarities of Andrias davidianus PGRP-S and those of other species
2.4 大鲵PGRP-S的NF-κB活性分析
NF-κB信号通路是肽聚糖识别蛋白识别病原微生物继而产生抗菌效应的重要通路, 果蝇PGRPs家族许多成员均可通过此信号通路诱导抗菌肽的产生[17,18]。为了研究大鲵PGRP-S是否能激活NF-κB信号通路, 我们将大鲵PGRP-S及空质粒分别与NF-κB启动子报告质粒共转染到HEK293T细胞中,48h后裂解细胞。该荧光素酶检测的结果表明, 大鲵PGRP-S能显著激活NF-κB启动子活性(图 2)。
2.5 大鲵PGRP-S的抗菌作用
为了研究大鲵PGRP-S对胞外以及胞内迟缓爱德华氏菌增殖的影响, 我们将PGRP-S-FLAG质粒及空质粒p3xFLAG转染到HEK293T细胞, 然后用迟缓爱德华氏菌感染过表达的细胞, 分别在感染后3h和6h收集细胞培养基或者裂解物, 通过平板计数法来计算各组细胞内或者细胞外迟缓爱德华氏菌的数量。结果显示, 在迟缓爱德华氏菌感染3h和6h后, 实验组的细菌数量显著低于对照组(图 3)。
2.6 大鲵PGRP-S的肽聚糖结合活性
肽聚糖识别蛋白, 作为天然免疫系统中的一类模式识别受体, 因其能特异性地识别细菌细胞壁组分PGN而得名。为了研究大鲵的PGRP-S与PGN的结合情况, 我们在HEK293T细胞中分别过表达大鲵PGRP-S的FLAG标签融合蛋白以及GFP蛋白, 并将胞浆蛋白提取物分别与Lys-type和Dap-type PGN共孵育, 再通过Western-blotting技术检测大鲵PGRP-S与这2种PGN的结合情况。结果表明, 大鲵PGRP-S能结合Lys-type和Dap-type PGN (图 4)。
2.7 大鲵PGRP-S的酰胺酶活性
肽聚糖识别蛋白家族的部分成员与T7溶菌酶相似, 具有酰胺酶活性, 通过切割MurNAc与LAla之间的酰胺键水解细菌细胞壁的PGN。为了研究大鲵PGRP-S是否具有酰胺酶活性, 我们在HEK293T细胞中过表达大鲵PGRP-S的重组质粒, 并将蛋白提取物分别与Lys-type和Dap-type PGN在Tris-Zn-Cl2缓冲液中共孵育, 用全波长分光光度计检测OD540的变化。结果表明, 大鲵PGRP-S没有酰胺酶活性, 不能水解Lys-type和Dap-type PGN (图 5)。
3 讨论
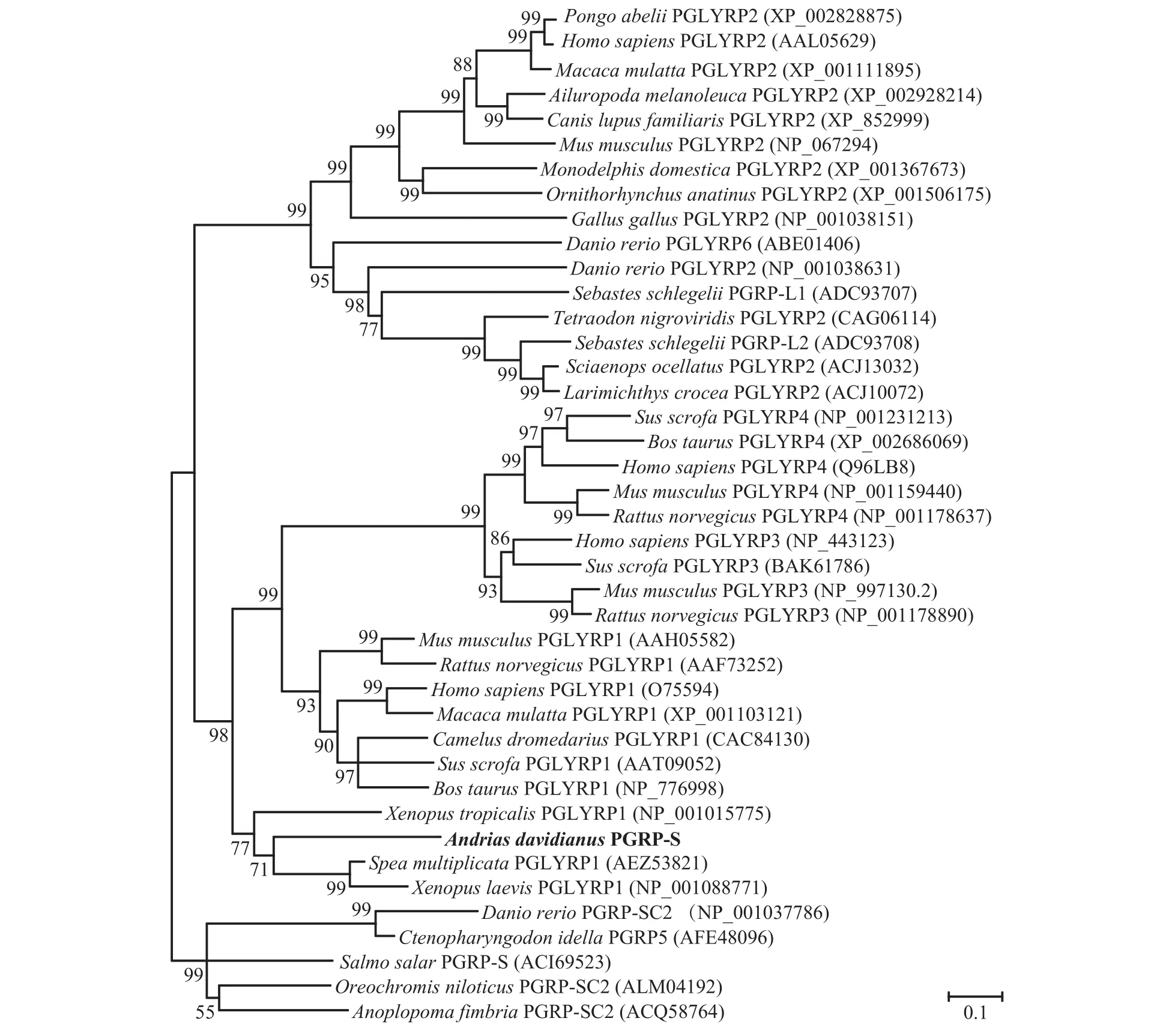
图 1 邻接法构建大鲵PGRP-S与其他物种PGRPs的系统进化树Fig. 1 Phylogenetic tree of Andrias davidianus PGRP-S with those of other species

图 2 大鲵PGRP-S对NF-κB启动子活性的影响Fig. 2 The effect of Andrias davidianus PGRP-S on the NF-κB activity
肽聚糖识别蛋白家族成员既可编码分泌蛋白,又可编码膜蛋白或者胞内蛋白。在果蝇中, 几乎所有短型PGRPs均为分泌蛋白; 其中PGRP-SA和PGRP-SD起着模式识别受体的作用, PGRP-SB和PGRP-SC则具有水解PGN的酰胺酶活性[19]。哺乳动物的4个PGRPs均为分泌蛋白, 它们分别表达于不同的组织, 参与该组织的免疫应答[4]。在鱼类中,斑马鱼的PGLYRP2、PGLYRP5和PGLYRP6及草鱼的PGLYRP6也均能分泌到胞外[8,13]。本研究中的大鲵PGRP-S尽管没有信号肽, 但也能分泌到胞外, 这与斑马鱼PGLYRP5的情况类似[8]。这表明PGRPs家族成员具有不依赖于信号肽的分泌机制。

图 3 大鲵PGRP-S对胞内(A)以及胞外(B)迟缓爱德华氏菌增殖的影响Fig. 3 The effect of Andrias davidianus PGRP-S on the intracellular (A) and extracellular (B) E.tarda’s growth in HEK293T cells
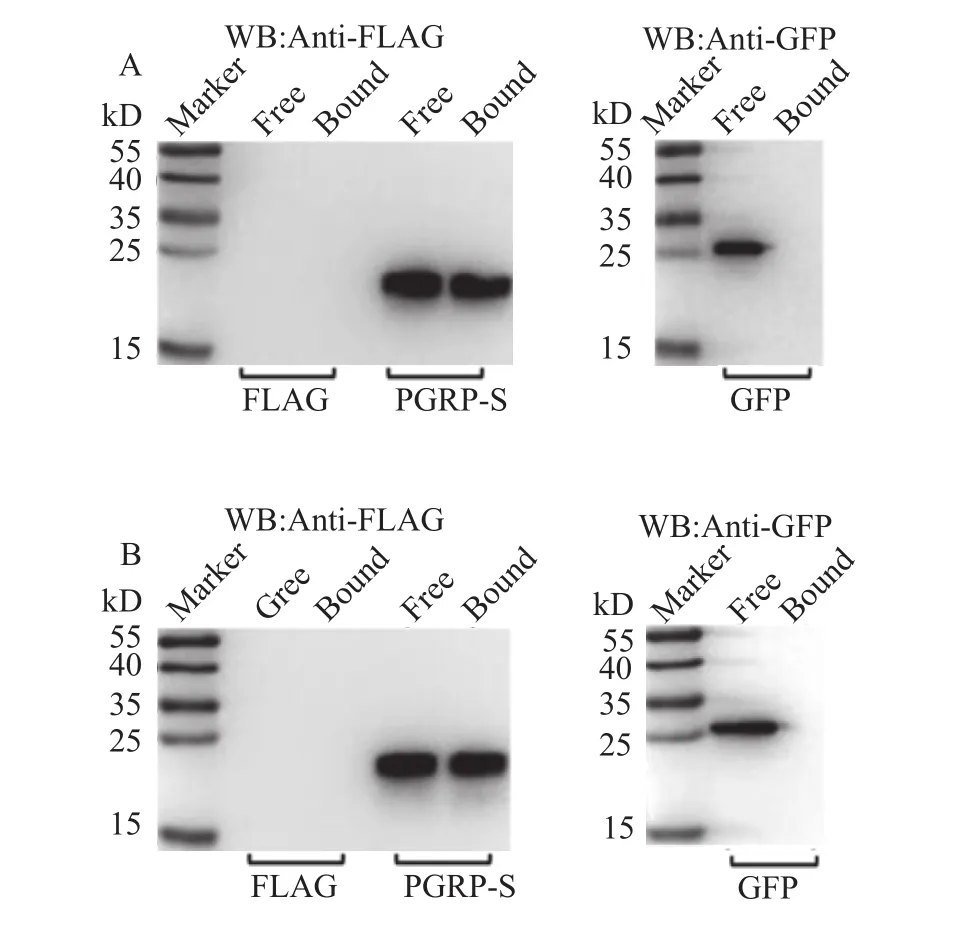
图 4 大鲵PGRP-S与PGN的结合作用Fig. 4 The binding activity of Andrias davidianus PGRP-S with PGN
肽聚糖识别蛋白最初因能识别细菌细胞壁的PGN而得名, 目前发现的大多数PGRPs均具有PGN结合能力, 并且这种结合活性可能并不依赖于PGRP结构域的完整性[14]。已有的研究显示, PGRPs对于Lys-type和Dap-type PGN的结合能力存在差异;它们区别Lys和Dap型PGN主要是通过2个途径:(1)通过区别不同类型肽聚糖的四肽尾之间的肽桥;(2)通过肽聚糖结合大沟内的3个重要的氨基酸的组成区别不同类型的肽聚糖[20], 如人类的PGLYRP1(Gly89、Trp90和Arg109GWR)识别Dap型PGN; PGLYRP3(Asn236、Phe237和Val256NFV)识别Lys型PGN[5], 草鱼PGLYRP5(Gly73、Phe74和Arg93GFR)识别2种类型的PGN[12]。在本研究中, 大鲵PGRP-S (Lys111、Arg112和Arg131KRR)也识别2种类型的PGN。此外, 在其他的物种中这3个位点还存在其他不同的氨基酸组成, 这些不同的氨基酸组成与PGN结合特异性之间的关系还有待进一步的研究。
目前发现的PGRPs成员几乎都参与天然免疫应答, 它们的功能概括起来主要包括三个方面:(1)作为模式识别受体(如果蝇的PGRP-SA), 激活下游的免疫应答信号通路, 介导抗菌肽等效应分子的释放; (2)作为效应分子, 具有直接的杀菌作用, 如哺乳动物的PGLYRP1、PGLYRP3、PGLYRP4; (3)作为调节分子, 调节免疫信号通路的应答水平: 包括具有酰胺酶活性的PGRPs (如PGRP-LB)和一些PGRPs分子的异构体(如rPGRP-LC)[16,19,21]。本研究发现在HEK293T细胞中过表达大鲵PGRP-S能抑制E. tarda在胞内的增殖, 这与之前Li等[13]和Yu等[14]在草鱼CIK细胞中对草鱼PGRP6及其异构体的研究结果类似, 与之不同的是我们还发现大鲵PGRP-S对E.tarda的胞外增殖也具有抑制作用, 这表明大鲵的PGRP-S在抵抗胞内和胞外细菌感染的免疫应答中都具有重要作用。
NF-κB作为无脊椎动物和脊椎动物的天然免疫系统乃至适应性免疫系统中重要的转录调控因子, 参与多个免疫信号通路的激活。昆虫中2个重要的抗菌天然免疫信号通路(Toll和IMD)以及脊椎动物中其他的PRRs (如TLRs和NODs)介导的免疫应答信号通路都涉及NF-κB的活化。Li等[13]和Yu等[14]研究发现, 在草鱼头肾细胞中过表达草鱼PGRP6及其异构体能激活NF-κB信号通路。而本研究发现, 在HEK293T细胞中过表达大鲵的PGRPS也能诱导NF-κB启动子的活性, 我们推测大鲵PGRP-S可能和鱼类一样, 通过激活NF-κB相关的免疫信号通路发挥抗菌功能。

图 5 大鲵PGRP-S的酰胺酶活性分析Fig. 5 Amidase activity of Andrias davidianus PGRP-S
在肽聚糖识别蛋白家族中, 有的PGRPs具有酰胺酶活性, 如果蝇PGRP-SB1、PGRP-LB, 哺乳动物PGLYRP2等[22—24]。目前发现的所有具有酰胺酶活性的PGRPs均含有4个保守的Zn2+结合位点, 如斑马鱼PGLYRP5(His98、Tyr132、His206和Cys214,HYHC)[8]。Zn2+在PGRPs水解PGN的过程中作为亲电催化剂, 促进N-乙酰胞壁酸和L-丙氨酸之间的酰胺键的水解[25]。因此, 这4个Zn2+结合位点对具有催化活性的PGRPs至关重要, 人类PGLYRP2C530S突变及果蝇PGRP-SC1b C168A和C168S突变均失去酰胺酶活性[26,27]。本研究发现, 只有2个Zn2+结合位点的大鲵PGRP-S不具有酰胺酶活性, 这表明所克隆的大鲵PGRP-S类似于哺乳动物的PGLYRP1、PGLYRP3和PGLYRP4, 能结合但不能直接降解细菌的肽聚糖; 其杀菌机制有可能是因为与细菌细胞壁的相互作用抑制了肽聚糖的合成, 进而杀死细菌或者抑制细菌的增殖[28,29]。
总之, 本研究克隆了大鲵的一个短型肽聚糖识别蛋白PGRP-S, 在其氨基酸序列中只含有2个保守的Zn2+结合位点。功能研究发现, 大鲵PGRP-S无酰胺酶活性, 但能识别并结合Lys型及Dap型PGN, 对胞内及胞外的细菌具有显著的抑菌作用, 这说明本研究中所克隆的大鲵PGRP-S在功能上更类似于哺乳动物的短型PGRP。
参考文献:
[1] Yoshida H, Kinoshita K, Ashida M. Purification of a peptidoglycan recognition protein from hemolymph of the silkworm,Bombyxmori[J].Journal of Biological Chemistry, 1996, 271(23): 13854—13860
[2] Werner T, Liu G, Kang D,et al. A family of peptidoglycan recognition proteins in the fruit flyDroso-phila melanogaster[J].Proceedings of the National Academy of Sciences, 2000, 97(25): 13772—13777
[3] Christophides G K, Zdobnov E, Barillas-Mury C,et al.Immunity-related genes and gene families inAnopheles gambiae[J].Science, 2002, 298(5591): 159—165
[4] Liu C, Xu Z, Gupta D,et al. Peptidoglycan recognition proteins a novel family of four human innate immunity pattern recognition molecules [J].Journal of Biological Chemistry, 2001, 276(37): 34686—34694
[5] Dziarski R, Gupta D. The peptidoglycan recognition proteins (PGRPs) [J].Genome Biology, 2006, 7(8): 232
[6] Ni D, Song L, Wu L,et al. Molecular cloning and mRNA expression of peptidoglycan recognition protein (PGRP)gene in bay scallop (Argopectenirradians, Lamarck 1819)[J].Developmental & Comparative Immunology, 2007,31(6): 548—558
[7] Coteur G, Mellroth P, De Lefortery C,et al. Peptidoglycan recognition proteins with amidase activity in early deuterostomes (Echinodermata) [J].Developmental &Comparative Immunology, 2007, 31(8): 790—804
[8] Li X, Wang S, Qi J,et al. Zebrafish peptidoglycan recognition proteins are bactericidal amidases essential for defense against bacterial infections [J].Immunity, 2007,27(3): 518—529
[9] Qi Z T, Zhang Q H, Wang Z S,et al. Cloning and expression analysis of a long type peptidoglycan recognition protein (PGRP-L) fromXenopus tropicalis[J].Zoological Research, 2011, 32(4): 371
[10] Dziarski R, Gupta D. Review: Mammalian peptidoglycan recognition proteins (PGRPs) in innate immunity [J].Innate Immunity, 2010, 16(3): 168
[11] Chang M X, Nie P, Wei L L. Short and long peptidoglycan recognition proteins (PGRPs) in zebrafish,with findings of multiple PGRP homologs in teleost fish[J].Molecular Immunology, 2007, 44(11): 3005
[12] Li J H, Chang M X, Xue N N,et al. Functional characterization of a short peptidoglycan recognition protein,PGRP5 in grass carpCtenopharyngodon idella[J].Fish &Shellfish Immunology, 2013, 35(2): 221—230
[13] Li J H, Yu Z L, Xue N N,et al. Molecular cloning and functional characterization of peptidoglycan recognition protein 6 in grass carpCtenopharyngodon idella[J].De-velopmental & Comparative Immunology, 2014, 42(2):244—255
[14] Yu Z L, Li J H, Xue N N,et al. Expression and functional characterization of PGRP6 splice variants in grass carpCtenopharyngodon idella[J].Developmental & Comparative Immunology, 2014, 47(2): 264—274
[15] Campanella J J, Bitincka L, Smalley J. MatGAT: an application that generates similarity/identity matrices using protein or DNA sequences [J].BMC Bioinformatics,2003, 4(1): 29
[16] Royet J, Dziarski R. Peptidoglycan recognition proteins:pleiotropic sensors and effectors of antimicrobial defences [J].Nature Reviews Microbiology, 2007, 5(4):264—277
[17] Takehana A, Katsuyama T, Yano T,et al. Overexpression of a pattern-recognition receptor, peptidoglycan-recognition protein-LE, activates imd/relish-mediated antibacterial defense and the prophenoloxidase cascade inDrosophila larvae[J].Proceedings of the National Academy of Sciences, 2002, 99(21): 13705—13710
[18] Gobert V, Gottar M, Matskevich A A,et al. Dual activation of the Drosophila toll pathway by two pattern recognition receptors [J].Science, 2003, 302(5653): 2126—2130
[19] Royet J, Gupta D, Dziarski R. Peptidoglycan recognition proteins: modulators of the microbiome and inflammation [J].Nature Reviews Immunology, 2011, 11(12):837—851
[20] Swaminathan C P, Brown P H, Roychowdhury A,et al.Dual strategies for peptidoglycan discrimination by peptidoglycan recognition proteins (PGRPs) [J].Proceedingsof the National Academy of Sciences, 2006, 103(3):684—689
[21] Neyen C, Runchel C, Schüpfer F,et al. The regulatory isoform rPGRP-LC induces immune resolution via endosomal degradation of receptors [J].Nature Immunology,2016, 17(10): 1150—1158
[22] Zhang Y, Van Der Fits L, Voerman J S,et al. Identification of serum N-acetylmuramoyl-L-alanine amidase as liver peptidoglycan recognition protein 2 [J].Biochimicaet Biophysica Act (BBA)-Proteins and Proteomics,2005, 1752(1): 34—46
[23] Mellroth P, Steiner H. PGRP-SB1: an N-acetylmuramoyl L-alanine amidase with antibacterial activity [J].Biochemical and Biophysical Research Communications,2006, 350(4): 994—999
[24] Zaidman-Rémy A, Hervé M, Poidevin M,et al. TheDrosophila amidasePGRP-LB modulates the immune response to bacterial infection [J].Immunity, 2006, 24(4):463—473
[25] Guan R, Roychowdhury A, Ember B,et al. Structural basis for peptidoglycan binding by peptidoglycan recognition proteins [J].Proceedings of the National Academy of Sciences, 2004, 101(49): 17168
[26] Mellroth P, Karlsson J, Steiner H. A scavenger function for a drosophila peptidoglycan recognition protein [J].Journal of Biological Chemistry, 2003, 278(9): 7059—7064
[27] Wang Z M, Li X, Cocklin R R,et al. Human peptidoglycan recognition protein-L is an N-acetylmuramoyl-L-alanine amidase [J].Journal of Biological Chemistry,2003, 278(49): 49044—49052
