基于G-SSR和EST-SSR标记的鲫6个群体遗传结构分析
2018-05-16甘宝江庞美霞俞小牧童金苟
甘宝江 庞美霞 俞小牧 童金苟
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
鲫属(Carassius)隶属鲤科(Cyprinidae)中的鲤亚科(Cyprininae), 该属通常被划分为3个种: 黑鲫(Carassius carassius)、鲫(Carassius auratus)和银鲫(Carassius auratus gibelioBloch)。目前的研究表明, 黑鲫主要分布于中欧、东欧, 亚洲西伯利亚勒那河和我国新疆; 鲫主要分布于中国、日本和朝鲜半岛[1]; 银鲫因其多倍体起源[2]、独特的多重生殖方式[3], 已逐渐被视为一个独立的物种[4], 其广泛分布于亚欧大陆[5]。鲫有三倍体银鲫和二倍体鲫之分[6], 在我国分布很广泛, 拥有的地理种群繁多, 遍布我国各大湖泊及水系, 且拥有很强的环境适应能力。在不同水系环境中生活的鲫, 其形态、行为、生活史、体色、肉质等方面出现了一定的变异和分化, 展现出丰富的遗传多样性。因此, 鲫在我国淡水生态系统和水产养殖中占据重要地位, 对其进行种群遗传多样性的研究具有较大的理论和应用价值。
遗传多样性和遗传结构是物种长期生存的关键因素, 包括对环境变化的适应[7], 并在野生动物保护和管理中发挥关键作用。通过对物种遗传多样性和遗传结构的分析, 可以确定哪些种群应该被优先保护[8], 而且其分析结果可以反应物种或种群的进化历史, 估算其进化潜力和育种能力, 为保护稀有或濒危物种资源提供参考, 还可以为鱼类经济性状的改良和种质资源的保护奠定基础[9]。目前, 在水产动物遗传学研究中常使用的遗传标记主要包括线粒体DNA (Mitochondrial DNA, mtDNA)、限制性片段长度多态性(Restriction fragment length polymorphism, RFLP)、随机扩增多态性DNA (Random amplified polymorphic DNA, RAPD)、扩增片段长度多态性(Amplified fragment length polymorphism, AFLP)、小卫星DNA (Minisatellite DNA)、微卫星(Microsatellite DNA)、单核苷酸多态性(Single nucleotide polymorphism, SNP)以及表达序列标签(Expressed Sequence tags, ESTs)等[10]。由于各种遗传标记的特性各不相同, 它们适用的遗传学研究领域也不尽相同。其中微卫星标记(也称简单重复序列, Simple sequence repeat, SSR), 由于其具有稳定性好、多态性高、数量丰富、呈共显性遗传且广泛而均匀地分布于基因组等优点, 已被广泛应用于水产动物的遗传多样性及遗传结构分析[11—14]。
SSR按照其来源可分为基因组SSR (G-SSR)和表达序列标签SSR (EST-SSR)。其中G-SSR标记来源于基因组序列, 一般通过经典的构建与筛选基因组文库、微卫星富集、省略筛库和数据库搜索等[15]方法进行开发, 该类标记极少能定位到基因上。EST-SSR标记则来源于转录序列, 可以定位到相关功能基因上, 因此, EST-SSR标记可以检测种群基因功能的遗传多态性, 而且能够在近源物种中进行跨种扩增[16], 相较于G-SSR标记更有助于提高标记辅助育种的效率[17,18]。近年来, 利用多种分子标记对鲫群体遗传多样性已进行了初步探讨, 如蒋芳芳等[19]利用形态学标记、细胞学标记、mtDNA标记及转铁蛋白等标记分析了二倍体和三倍体鲫群体的遗传多样性; 杨林等[20]利用转铁蛋白和同工酶对银鲫克隆系亲缘关系及遗传多样性进行了分析。然而, 利用微卫星标记分析鲫野生群体遗传多样性的研究鲜有报道。
本研究分别利用8个FIASCO (Fast Isolation by AFLP of Sequences Containing Repeats)微卫星富集技术开发的G-SSR标记和8个本实验室开发的ESTSSR标记, 以来自黑龙江、长江、奉化江及淮河水系共6个鲫野生二倍体群体作为研究对象, 通过全自动DNA测序仪分析技术对这些鲫野生群体进行遗传多样性分析, 目的是为开展鲫种质资源的遗传评价和保护以及鲫经济性状遗传改良等基础和应用研究提供群体遗传学参考信息; 2种不同来源的微卫星标记的遗传多样性分析和比较结果, 也为鲫或其他鱼类微卫星标记的开发和分子遗传分析与应用提供一定的指导意义。
1 材料与方法
1.1 实验材料
本研究所使用的6个鲫野生群体(图 1、表 1)样本于2006—2016年分别采集于黑龙江水系的镜泊湖(JPH,n=45)和杜尔伯特蒙古族自治县的九河乡(JHX,n=35), 奉化江水系的宁波(NB,n=41)、长江水系的牛山湖(NSH,n=34)和太湖(TH,n=40), 及淮河水系的洪泽湖(HZH,n=36)。每尾样本鱼剪取尾鳍约0.5 g浸泡于无水乙醇中保存备用, 采用经典的酚–氯仿法提取基因组DNA[21]。
1.2 倍性鉴定
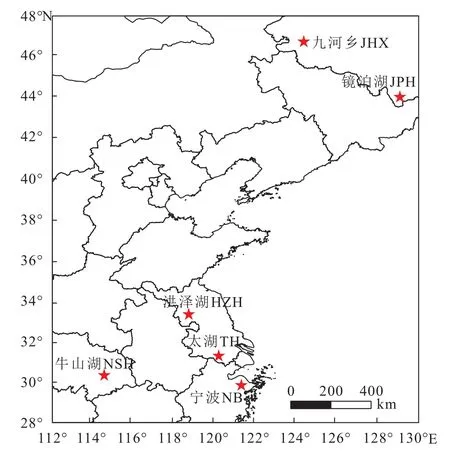
图 1 鲫6个群体的采样点示意图(用五角星表示)Fig. 1 Distribution of sampling sites for six Carassius auratus populations of China (the star dots stand for sampling sites)
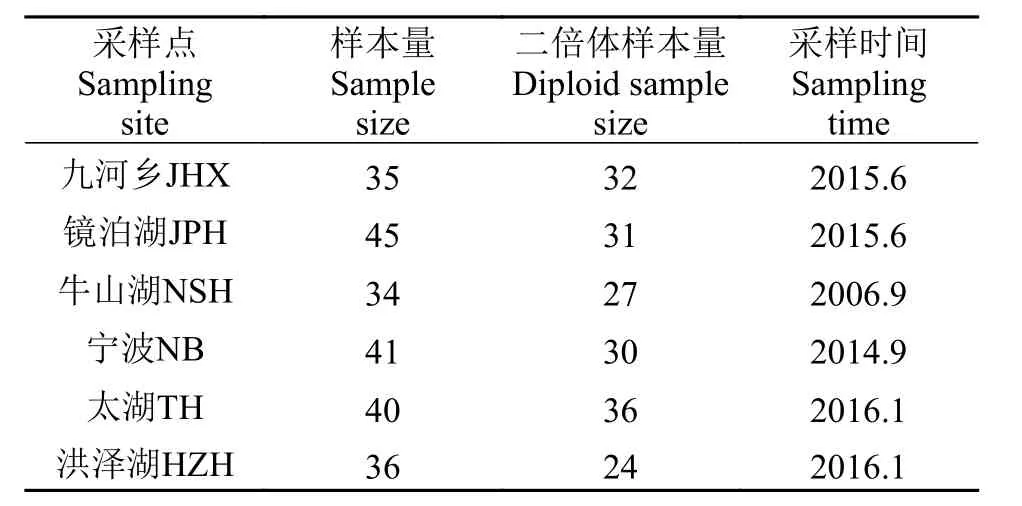
表 1 本研究中所用的鲫样本信息Tab. 1 Sample information of six crucian carp populations used in this study
参照Mishina等[22]利用微卫星标记鉴定鲫染色体倍性的方法, 本实验室筛选出能高效、准确地鉴定鲫倍性的微卫星标记[23,24](YJ0004、HLJYJ018、HLJYJ029、HLJYJ049), 并用流式细胞仪检测结果进行了验证(庞美霞等, 未发表资料)。本文对采集的共计231尾鲫复合种样本的基因组DNA进行上述4个SSR位点的PCR扩增, PCR产物用10%的非变性聚丙烯酰氨凝胶进行电泳分型, 用溴化乙锭(EB)对凝胶进行染色, 并用JS-A380凝胶成像仪(上海培清科技公司)扫描并拍照。统计每个个体在每个位点上的等位基因数并判断鲫样本的染色体倍性。综合4个位点的等位基因数目信息, 只要有1个及以上位点上同时出现3个等位基因则认为该个体是三倍体(或称为六倍体)鲫; 从中挑选出二倍体(或称为四倍体)鲫野生样本进行后续的群体遗传学研究。鉴定结果发现二倍体鲫个体总计180尾, 占总数的77.6%。其中, JHX、JPH、NSH、NB、TH、HZH的二倍体鲫的样本数分别为32、31、27、30、36和24尾。
1.3 引物选取及PCR扩增
本研究使用的G-SSR标记来自于已发表的8个HLJYJ微卫星标记[18], EST-SSR标记来自于本实验室根据鲫EST序列设计的8个GCE微卫星标记(未发表的数据), 每对微卫星标记的正向引物5′端使用FAM或HEX荧光染料进行标记。
PCR反应总体积为15 μL, 反应混合物包含以下组分: 36—60 ng模板DNA, 0.48 UTaqDNA聚合酶, 1.5 μL 10×PCR buffer, 0.48 μL dNTP (2.5 mmol/L), 0.48 μL正反向混合引物(各2.5 μmol/L), 11.22 μL灭菌去离子水。PCR扩增条件如下: 94℃预变性5min; 扩增32个循环, 每个循环94℃变性35s, 48—60℃退火35s (退火温度见表 2), 72℃延伸40s; 72℃终延伸10min。PCR产物采用3730型DNA测序仪(ABI, USA)测序分型, 微卫星荧光引物合成以及PCR扩增产物测序分型均由武汉天一辉远生物科技有限公司完成。
1.4 数据统计及分析
使用GeneMarker 2.2软件读取3730型DNA测序仪测序结果, 进行微卫星片段大小的统计。利用软件PopGene 32[25]计算观测杂合度(Ho)、期望杂合度(He)、等位基因数(Na)、有效等位基因数(Ne)以及Nei’s遗传距离[26]。Microsoft-toolkit插件计算多态信息含量(PIC)。根据Nei’s遗传距离, 使用MEGA 4软件[27]对6个鲫野生群体进行聚类分析。Arlequin 3.1软件[28]计算各群体间的分化系数Fst, 同时用于进行各群体的哈迪-温伯格平衡(Hardy-Weinberge quilibrium)检验。进行多重比较时, 概率的显著性水平需要进行Bonferroni校正[29]。利用分子变异分析(AMOVA)方法[30]计算群体遗传结构, 利用Structure 2.2[31]软件中的贝叶斯方法[32]混合模型来构建群体遗传结构图。
2 结果
2.1 微卫星位点多态性及群体多样性分析
统计8个G-SSR标记在所有鲫样品中的多态性信息发现8个G-SSR (HLJYJ028、HLJYJ075、HLJYJ120、HLJYJ033、HLJYJ048、HLJYJ029、HLJYJ124、HLJYJ049)的Na和Ne分别为9—31和4.7—20.2, HLJYJ124位点Na最小(9), HLJYJ028最大(31)。8个G-SSR位点的He、Ho以及PIC分别为0.789—0.953、0.659—0.878和0.757—0.948, 而8个EST-SSR (GCE092、GCE489、GCE359、GCE416、GCE451、GCE460、GCE464、 GCE474)的Na、Ne、He、Ho以及PIC分别为12—35、4.1—22.4、0.756—0.958、0.511—0.906和0.726—0.953。在HLJYJ引物和GCE引物中分别检测到173和155个等位基因, HLJYJ和GCE微卫星位点的平均Na、Ne、He、Ho以及PIC分别为22、12.9、0.769、0.893、0.879, 19、9.5、0.728、0.870和0.855。G-SSR和EST-SSR标记均为较高多态性的位点, 对比发现,HLJYJ微卫星位点的各平均遗传多样性参数均高于GCE微卫星(表 3、表 4), 表明鲫EST-SSR标记多态性稍低于G-SSR标记。
6个鲫野生群体(JHX、JPH、NSH、NB、TH、HZH)在各位点的多样性详细信息见表 4。在共计180尾样本中, 共检测到328个等位基因, 其中黑龙江水系JHX和JPH群体分别检测到194和188个等位基因, 奉化江水系NB群体检测到212个等位基因, 长江水系的NSH和TH群体分别检测到211和232个等位基因, 而淮河水系的HZH群体检测到210个等位基因。利用G-SSR和EST-SSR检测每个群体的平均Na和平均Ne分别在12—15、7.6—10.6;11—14、5.5—7.7变化(表 5), 各个群体的平均He、平均Ho以及平均PIC分别介于0.816—0.896、0.739—0.869和0.786—0.864; 0.801—0.867、0755—0.856和0.761—0.833。2种微卫星标记在6个鲫野生群体中的检测结果均显示, 属于黑龙江水系的JHX和JPH两群体的各平均多样性参数都相对较小, 表明黑龙江水系群体遗传多样性偏低; 而淮河、奉化江与长江水系鲫野生群体间遗传多样性无明显差异。
2.2 群体间的遗传结构
AMOVA分析发现, 群体间的遗传变异占4.47%,群体内的遗传变异占95.53%, 说明鲫遗传变异主要来自群体内部个体间。8个G-SSR在6个鲫群体的遗传分化分析结果显示, 鲫群体间Fst值介于0.008—0.085 (表 6), 表明各群体间存在不同程度的遗传分化。隶属黑龙江水系的JHX和JPH两群体间Fst值较低, 而这2群体与其余水系群体间的Fst值均较高。其中JPH与长江、奉化江和淮河水系的群体两两间Fst值为0.060—0.085, 遗传分化均极显著(P<0.01);JHX群体与奉化江、长江和淮河水系的群体两两间Fst值为0.044—0.081之间, 与NSH以及NB群体间的Fst值达到了0.081和0.060(P<0.01)。位于奉化江水系的NB、长江水系下游的TH和淮河水系HZH两两群体之间Fst值较小(0.008—0.017), 而这3个群体与位于长江水系中游的NSH群体间Fst值则相对较大(0.028—0.040)。利用软件Structure2.2对群体内的各个体进行混合模型分析(图 2), 本研究选取K值为2—6, 重复计算20次, 根据公式ΔK=m([L″K])/s[L(K)][33]计算出最佳理论群数ΔK=2。假设K=2时, 可以明显看出6个鲫群体分属于2个类群,其中来自黑龙江水系的JHX、JPH群体与其余水系的群体间的遗传结构分化明显, 聚为一个类群, 而其他群体相对独立为另一类群。各个鲫群体间的Nei’s遗传距离介于0.203—0.701(表 6), 以TH和HZH群体之间的遗传距离最小, 而JPH与NSH群体之间的遗传距离最大, 遗传距离与地理距离之间成正相关关系。依据计算出来的Nei’s遗传距离(表6)对6个鲫群体进行了聚类分析(图 3), 发现来自黑龙江水系的JHX与JPH群体聚为一枝, 其余群体聚为另一大枝, 该分枝中长江水系下游的TH、奉化江水系NB与淮河水系的HZH群体聚为一枝, 再与位于长江水系中游的NSH群体聚为一大枝。
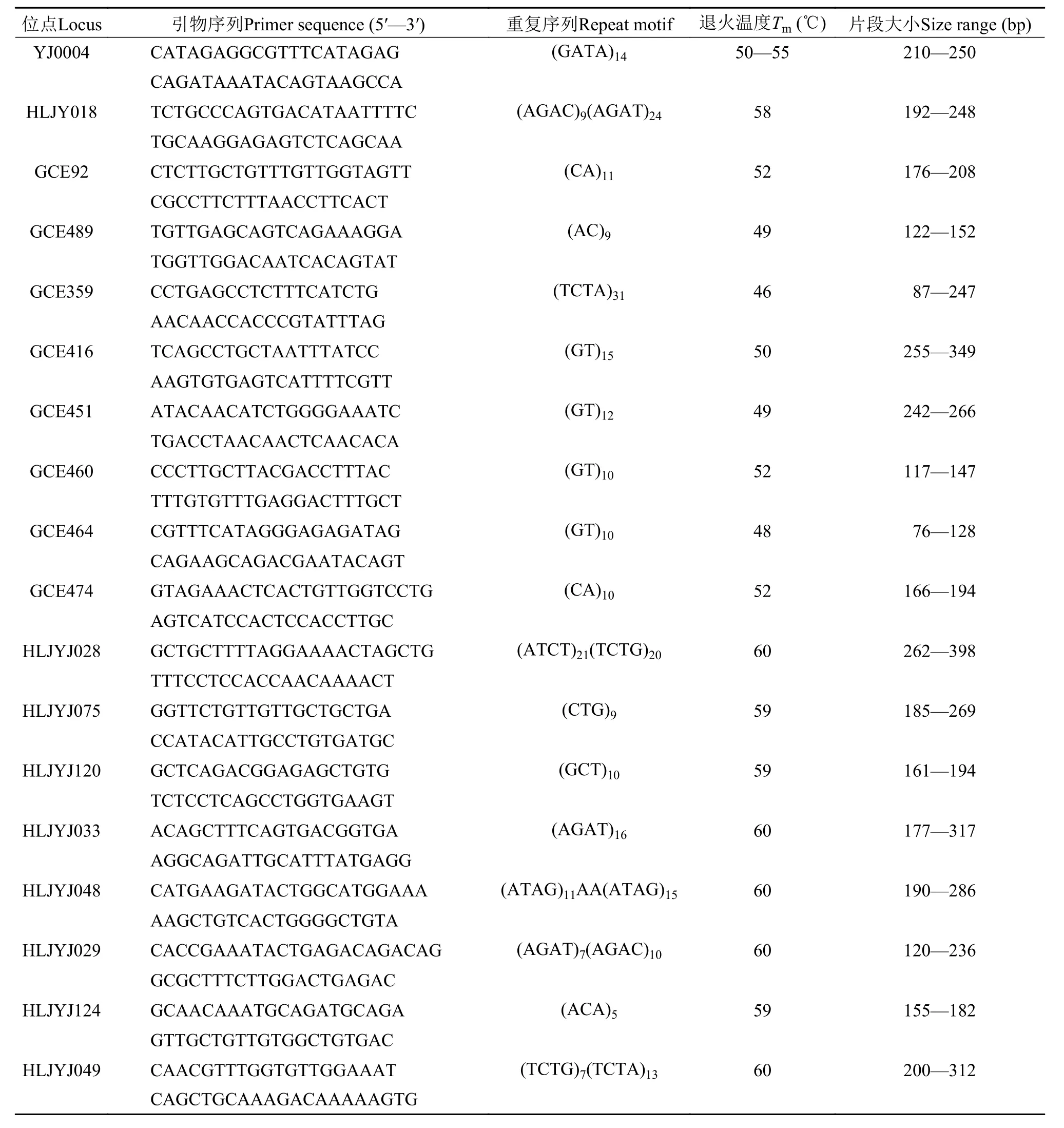
表 2 本研究中所用鲫微卫星标记的基本信息Tab. 2 Information of Carassius auratus microsatellite markers used in this study
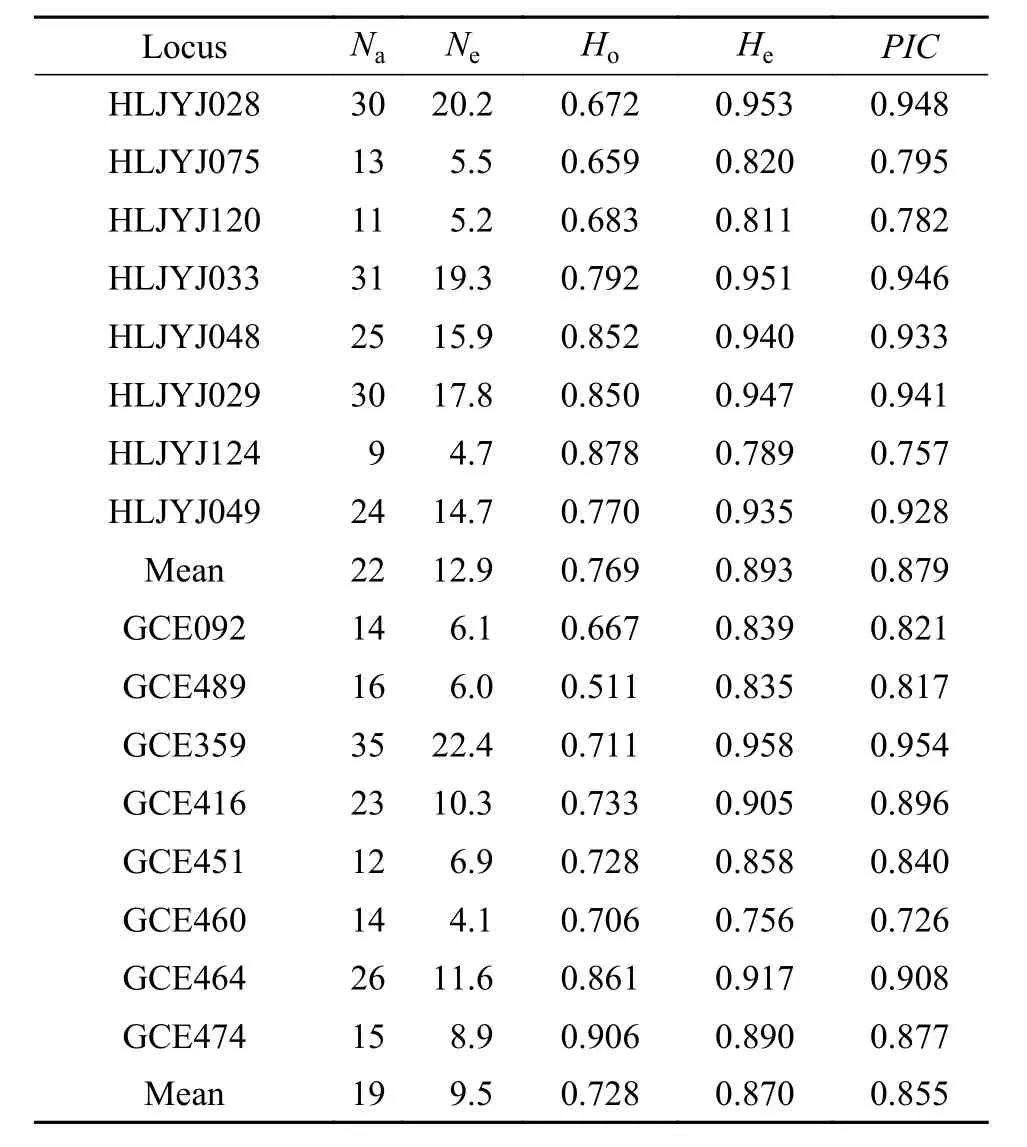
表 3 16个微卫星标记的遗传参数信息表Tab. 3 Genetic parameters at 16 microsatellite markers
利用8个EST-SSR计算6个鲫野生群体间群体间Fst值、Nei’s遗传距离分别介于0.203—0.701和0.117—0.683(表 6), 贝叶斯分析根据公式ΔK=m([L″K])/s[L(K)][33]计算出最佳理论群数ΔK=2。与G-SSR分析结果相比, 基于EST-SSR标记的UPGMA聚类分析和贝叶斯分析也将6个鲫群体划为两大类群: 黑龙江水系群体, 奉化江、长江和淮河水系群体(图 3), 尽管EST-SSR与G-SSR标记的Fst值、Nei’s遗传距离数值不同, 但两类SSR标记揭示了相似的鲫群体遗传结构和分化格局。
3 讨论
3.1 鲫野生群体遗传多样性
一般来说, 微卫星位点的核心重复次数至少为5次才可能检测出遗传多态性[36], 而且重复次数越高, 其等位基因数也就相应越多[37]。本文所使用的16个SSR标记的核心序列重复次数均在5—31次(表2), 在6个鲫群体中的多态信息含量(PIC)介于0.725—0.953, 表明它们均为高度多态性座位[38](PIC>0.5, 表 3)。PIC是反映群体遗传多样性的度量之一, 其数值与基因丰度成正相关[39]。在本研究中, 2种来源的SSR被用于鲫群体遗传多样性的研究, 在不同水系来源的6个群体中均检测出较高的多态性(表 4、表 5)。贾志英等[40]利用12个微卫星标记对黑龙江水系6个鲫野生群体进行遗传多样性分析发现, 平均PIC为0.596—0.648, 其鲫群体遗传多样性相较本研究中黑龙江水系2个鲫野生群体偏低; 鲁双庆等[41]利用9个微卫星标记对4个鲫群体研究发现, 平均PIC为0.530—0.670, 同样低于本研究中的6个鲫群体遗传多样性。这些PIC差异可能是由以下两方面的原因造成的: 一方面可能是由于使用的微卫星标记的种类和具体位点不同; 另一方面可能是由于不同分辨率的检测方法所产生的遗传多态性差异[42,43]。与传统方法的聚丙烯酰胺凝胶电泳后银染显色相比, 使用DNA测序仪检测到的等位基因数要比前者多得多。例如, 利用相同的8个微卫星标记研究草鱼(Ctenopharyngodon idellus)群体遗传多样性, 李家乐等[44]利用测序仪分型检测技术和付建军等[45]利用传统聚丙烯酰胺电泳技术所检测到的等位基因数分别为7—12个和2—6个。本研究还发现利用2种不同来源微卫星标记, 均发现黑龙江水系鲫群体的各遗传参数明显低于其余3个水系。在利用微卫星标记研究黑龙江、长江和珠江水系草鱼[45]、鲢(Hypophthalmichehys molitrix)[46]群体遗传多样性时, 不同作者也发现黑龙江水系这2种鱼类的群体遗传多样性偏低。产生以上现象的原因可能是黑龙江水系位于高纬度地区, 由于饵料及气候环境等[45]因素造成鱼类资源比较匮乏; 而长江水系水面大、饵料丰富和气候环境适宜等使其能够容纳更丰富的鱼类资源, 因而长江水系鱼类群体具有较高遗传多样性。Nevo[47]也认为微卫星等分子遗传标记的多态性位点数目与环境温度呈正相关。李思发等[48]进一步证实同种鱼类不同种群的多态位点比例有随纬度的升高而降低的趋势, 黑龙江水系鱼类种群遗传多样性相对较低。
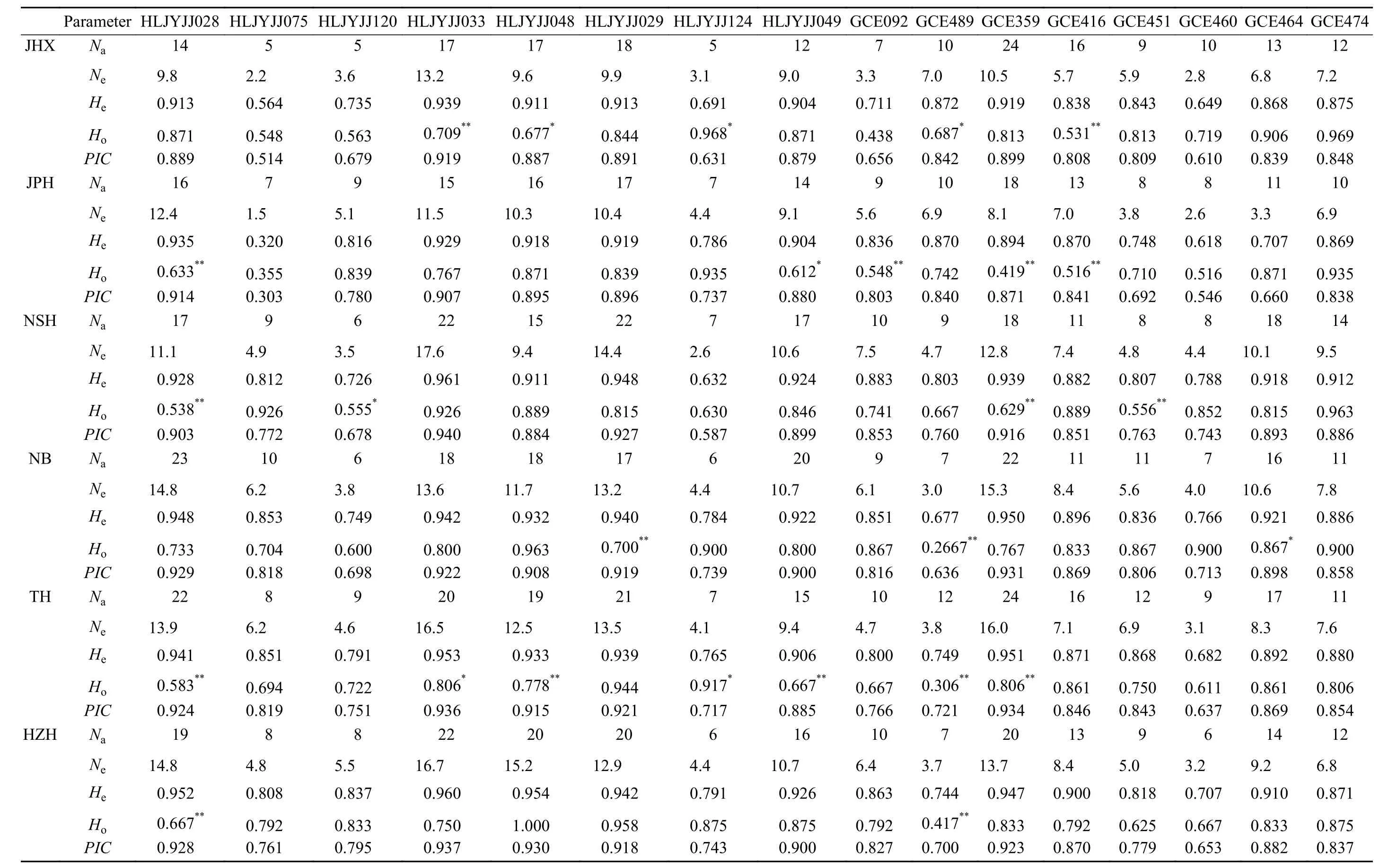
表4 六个鲫群体在16个核微卫星位点的多态性信息Tab. 4 Major statistics of the analysis at 16 Carassius auratus microsatellite loci
3.2 鲫群体结构
群体遗传分化系数Fst是反映群体间遗传分化程度的常用指标, 数值越大则表示两群体间的遗传分化程度越高[49]。一般认为,Fst值在0.05以上时群体间的遗传分化程度达到中等或显著性[50]。本研究中8个G-SSR标记检测群体间基因分化系数显示(表 6), 来自黑龙江水系的2个群体与其余水系的所有群体两两间Fst值介于0.044—0.085, 均达到或接近于中等程度的遗传分化, 表明黑龙江鲫群体与其他水系群体间的遗传结构存在较明显的差异。李思发等[48]在对长江、珠江、黑龙江的鲢、鳙(Aristichthys nobilis)和草鱼原种种群的生化遗传结构与变异的研究中发现, 同种鱼在不同水系种群之间存在着明显的生化遗传差异, 尤其是黑龙江水系群体与其他水系群体差异最大, 与本研究结果相似。这可能是因为黑龙江水系群体处于相对独立的边缘地区[51], 与其余水系群体形成地理隔离导致生殖隔离所致; 来自长江、奉化江和淮河水系的群体两两间Fst值均小于0.05, 属于轻微程度的遗传分化或无明显分化, 这是由于这些水系之间存在一定的连通, 或由于地理距离较近存在人为迁移和运输因素,导致属于这些水系的鱼类群体之间可能具有一定程度的基因交流。聚类分析也显示(图 3), 黑龙江水系两群体单独聚为一枝, 其余水系群体聚为另一枝, 表明鲫群体间遗传距离与群体间的地理距离成正相关。王伟伟等[52]发现相近的斑鳜(Siniperca scherzeri)地理群体之间的遗传关系较近。杨慧荣等[53]对珠江和长江水系赤眼鳟(Squaliobarbus curriculus)线粒体DNA D-loop分析后, 也发现相近的地理群体的遗传结构相似。这些研究均支持较大的地理距离与遗传距离正相关的结论。
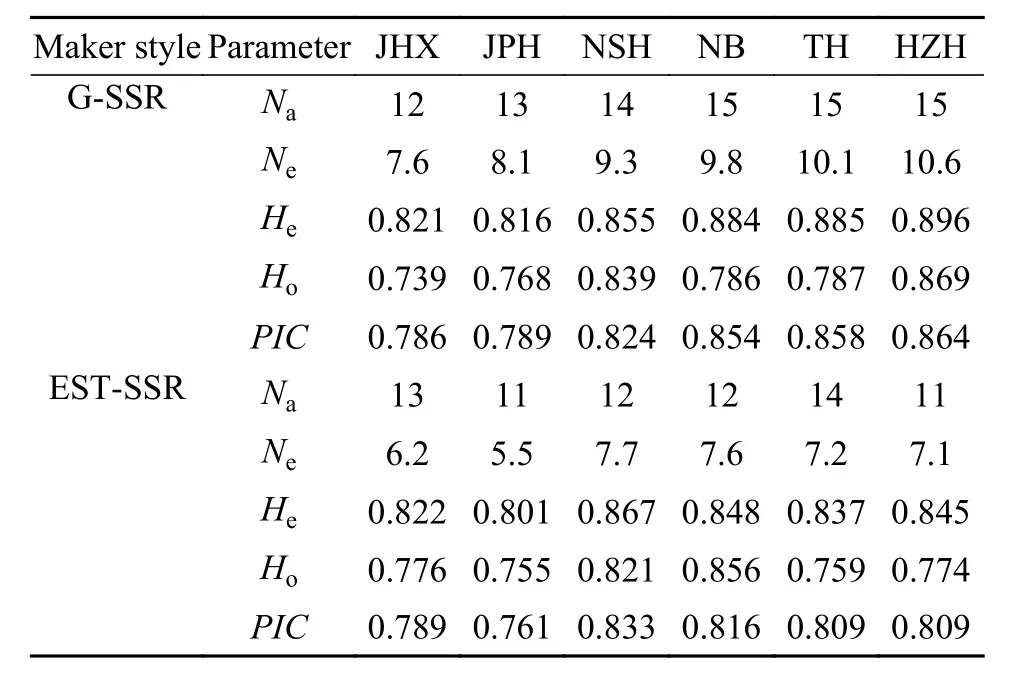
表 5 六个鲫群体的平均遗传参数Tab. 5 Mean genetic parameters in 6 Carassius auratus populations
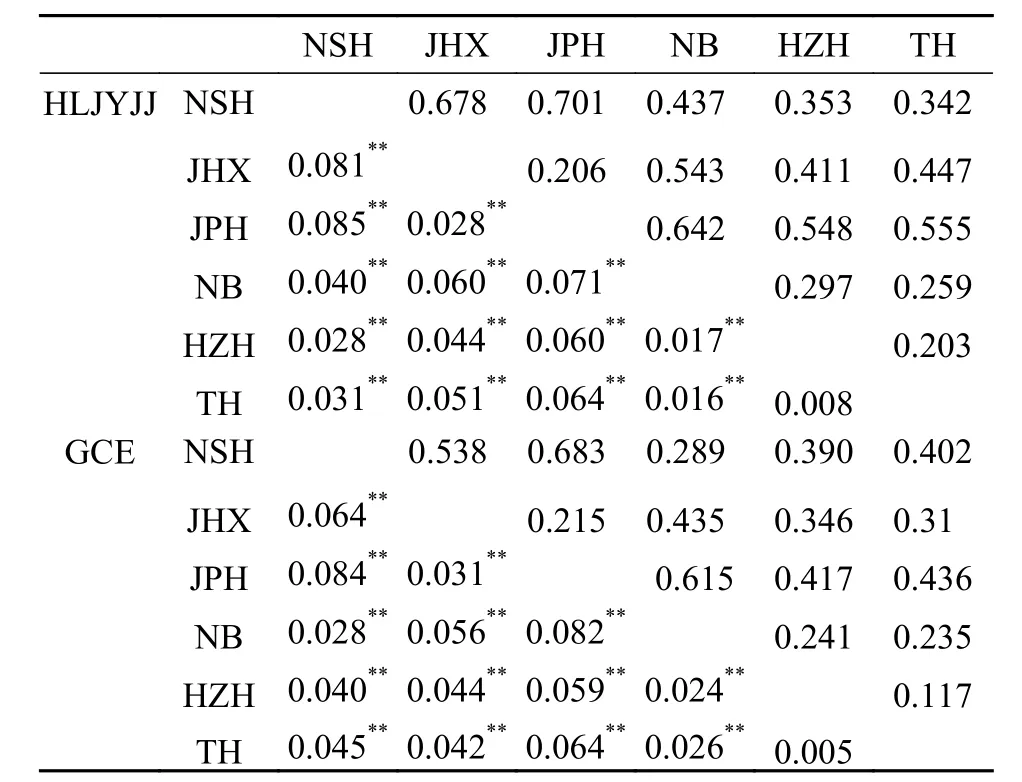
表 6 六个鲫群体间的Fst值(对角线下方)及遗传距离(对角线上方)Tab. 6 Nei's genetic distance (above diagonal) and pairwise Fst values (below diagonal) among 6 Carassius auratus populations
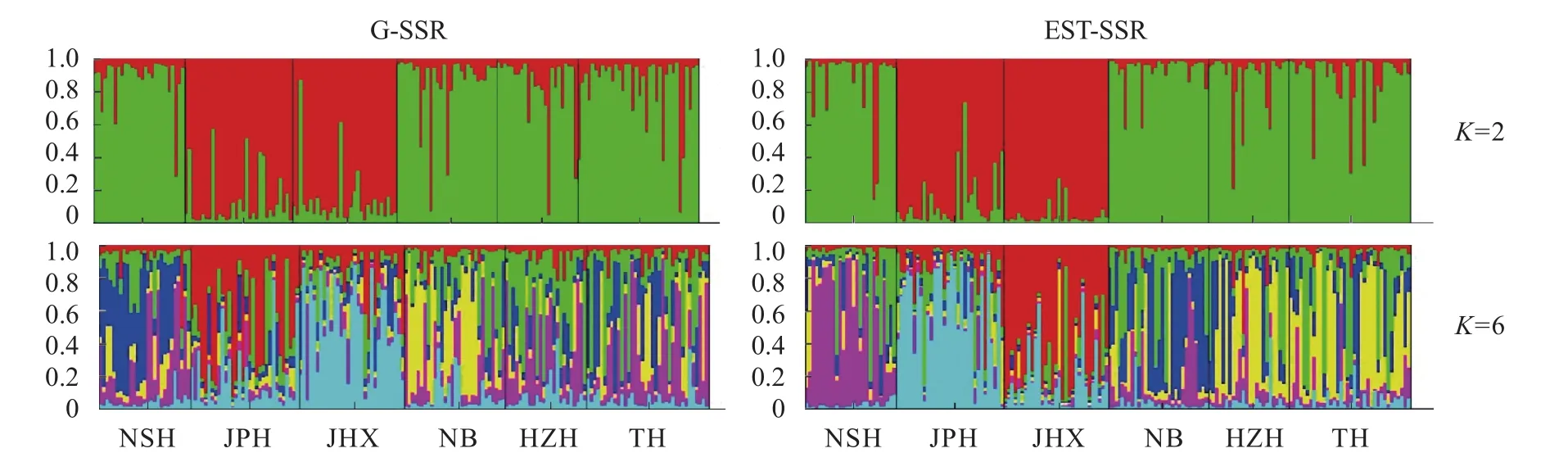
图 2 六个鲫群体个体在不同假设K值的遗传结构Fig. 2 Genetic structure of assuming different K values for individuals of six Carassius auratus populations
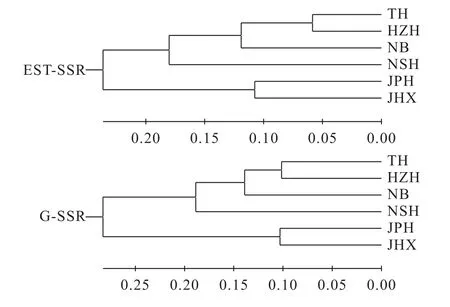
图 3 基于Nei’s遗传距离的6个鲫群体聚类图Fig. 3 The dendrogram of the six Carassius auratus populations based on Nei’s genetic distance using UPGMA method
迄今还未有同时利用G-SSR和EST-SSR标记对同一种鱼进行群体遗传多样性和遗传结构研究的报道。为了进行比较研究, 本研究分别利用8个GSSR和8个EST-SSR标记对6个鲫野生群体间进行贝叶斯分析和聚类分析发现(图 2、图 3), 基于ESTSSR与G-SSR标记的UPGMA聚类分析和贝叶斯分析结果相似, 两类SSR标记揭示了相似的鲫群体遗传结构和分化格局。此结果与丁西朋等[54]研究GSSR和EST-SSR标记在柱花草(Stylosanthes)种间的遗传差异研究结果相同; 孔令锋等[55]基于G-SSR和EST-SSR标记研究野生和养殖的太平洋牡蛎(Crassostrea gigas)遗传多样性时, 2种标记的聚类分析结果不一致; 这有可能是太平洋牡蛎中的养殖群体经历近交、选择和遗传漂变[56]而导致群体多样性下降, 无效等位基因的存在以及稀有等位基因流失等原因造成的。
3.3 鲫野生群体G-SSR和EST-SSR多态性比较
SSR的核心序列重复单位以2个核苷酸为主,少数为3个核苷酸的重复, 极少数重复单位为4个或更多个核苷酸[35]。本研究所使用的G-SSR标记核心序列是三或四碱基重复, EST-SSR标记中只有1个为四碱基重复, 其余7个均为两碱基重复。动植物微卫星标记的研究表明: 在一般情况下G-SSR标记的多态性要明显高于EST-SSR标记[57], 在小麦(Triticum aestivum)[58,59]和太平洋牡蛎[55]的遗传多样性研究中均是如此。其原因主要是因为GSSR标记主要来源于基因组的非编码区段, 而ESTSSR标记则是来源于编码区内或靠近编码区[60]。在物种进化历程中, 位于编码区的EST-SSR标记相对稳定、突变几率小[61]。本研究结果中G-SSR标记的平均PIC仅略高于EST-SSR标记的平均PIC(表3), 这可能是由于以二碱基为主的8个EST-SSR标记平均重复单元次数为13.5, 而以三、四碱基为主的8个G-SSR标记平均重复单元次数为18, 重复单元为两碱基的微卫星标记具有较高的突变率, 其次为三碱基和四碱基以及复合型重复类型的微卫星标记[62]。
EST-SSR标记的优势在于其作为基因的一部分而拥有特殊功能的等位基因[63], 其侧翼序列在物种之间高度保守, 相对于基因组来源的SSR标记,EST-SSR在物种之间有较好的通用性[64]; ESTSSR标记具有突变率低且较稳定的特点还可被用于重建较为久远的进化事件[60]; 随着高通量测序技术的快速发展, 越来越多EST-SSR标记将会被发掘和利用[65]。因此, 研究EST-SSR标记与功能基因的相关性, 不仅可以检测近缘物种该功能基因的多态性, 还可以构建鱼类的遗传进化史。多态性较低是EST-SSR标记的特性之一。本研究根据EST序列设计的GCE359微卫星标记是一个重复单元为四碱基、重复次数多达31次、呈现较高水平多态性的EST-SSR标记, 这表明提高EST-SSR标记多态性还是有迹可循的。可能提高EST-SSR标记多态性的方法包括: (1)选择EST-SSR标记时, 尽量靠近3′或5′端非翻编译区段, 因为基于这些区域的位点变异性可能较高[65]; (2)改进分析手段(例如等位基因分型采用DNA测序仪分析技术); (3)尽可能利用3个或4个以上核苷酸重复以及重复次数高的位点, 可有效提高EST-SSR标记的多态性。
本研究通过对6个鲫群体的遗传分析, 发现鲫野生群体整体上具有较高的遗传多样性, 但黑龙江2个群体遗传多样性明显低于其他水系; 由于地理隔离, 来自黑龙江水系的鲫野生群体遗传结构与其余群体(长江、淮河、奉化江)分化显著。另一方面, 尽管EST-SSR标记的多态性略低于G-SSR标记,但这两类SSR标记揭示了相似的鲫群体遗传结构和分化格局。EST-SSR标记反映了基因转录部分的遗传多态性, 通常代表着某种基因功能的变化,因此若将其与之相关的功能联系起来, 将会在转录组图谱的绘制、功能基因的发掘和利用领域发挥重要作用。本文的这些研究结果将为鲫种质资源的保护和利用以及评价EST-SSR标记在鱼类群体遗传学研究中的价值提供新的参考信息。
致谢:
感谢本实验室的冯秀、刘海洋、王新华、刘雪丽、周颖等以及中国水产科学研究院珠江水产研究所李新辉研究员实验室为鲫样本采集和分子遗传分析提供的帮助。
参考文献:
[1] Zhou Q B, Zheng Y, Zhou L,et al. Genetic diversity of crucian carp (Carassius auratus) from Yili River of Sinkiang District [J].Acta Hydrobiologica Sinica, 2009,33(4): 690—695 [周秋白, 郑宇, 周莉, 等. 新疆伊犁河鲫鱼遗传多样性初步研究. 水生生物学报, 2009, 33(4):690—695]
[2] Li X Y, Zhang X J, Li Z,et al. Evolutionary history of two divergentDmrt1 genes reveals two rounds of polyploidy origins in gibel carp [J].Molecular Phylogenetics and Evolution, 2014, 78: 96—104
[3] Wang Z W, Zhu H P, Wang D,et al. A novel nucleocytoplasmic hybrid clone formed via androgenesis in polyploid gibel carp [J].BMC Research Notes, 2011,4(1): 82
[4] Li X Y, Zhang Q Y, Zhang J,et al. Extra microchromosomes play male determination role in polyploid gibel carp [J].Genetics, 2016, 203(3): 1415—1424
[5] Gui J F, Zhou L. Genetic basis and breeding application of clonal diversity and dual reproduction modes in polyploidCarassius auratus gibelio[J].Science China Life Sciences, 2010, 53(4): 409—415
[6] Jiang F F. Studies on distribution pattern and genetic diversity of gibel carp, Carassius auratus gibelio across mainland China [D]. Thesis for Doctor of Science. Institute of Hydrobiology, the Chinese Academy of Sciences,Wuhan. 2012 [蒋芳芳. 银鲫在全国主要水域的生态分布格局及其不同地理种群的遗传多样性研究. 博士学位论文, 中国科学院水生生物研究所, 武汉. 2012]
[7] Taberlet P, Fumagalli L, Wust-Saucy A G,et al. Comparative phylogeography and postglacial colonization routes in Europe [J].Molecular Ecology, 1998, 7(4): 453—464
[8] Newton A C, Allnutt T R, Gillies A. Molecular phylogeography, intraspecific variation and the conservation of tree species [J].Trends in Ecology & Evolution, 1999,14(4): 140
[9] Li G Q, Wu Y Y, Qin Z F,et al. Studies on the genetic diversity of fishes: a review [J].Fisheries Science, 2004,23(8): 42—44 [李国庆, 伍育源, 秦志峰, 等. 鱼类遗传多样性研究. 水产科学, 2004, 23(8): 42—44]
[10] Gao Z X, Wang W M, Zhou X Y. DNA marker technologies and their applications in aquaculture genetics [J].Biotechnology Bulletin, 2007, (2): 108—113 [高泽霞, 王卫民, 周小云. DNA分子标记技术及其在水产动物遗传上的应用研究. 生物技术通报, 2007, (2): 108—113]
[11] Rezaee S, Farahmand H, Nematollahi M A. Genetic diversity status of Pacific white shrimp (Litopenaeus vannamei) using SSR markers in Iran [J].Aquaculture International, 2016, 24(2): 479—489
[12] Araneda C, Lam N, Iturra P,et al. Utility of five SSR markers for genetic diversity and paternity exclusion analysis in the Patagonian toothfish [J].Latin American Journal of Aquatic Research, 2017, 45(1): 188
[13] Achrem M, Skuza L, Kirczuk L,et al. Genetic variation ofSalmo truttaL. populations from the catchment areas of the Rega, Parseta and Wieprza rivers evaluated by RAPD and SSR markers [J].Folia Biologica, 2015,63(1): 1—7
[14] Sundaray J K, Rasal K D, Chakrapani V,et al. Simple sequence repeats (SSRs) markers in fish genomic research and their acceleration via next-generation sequencing and computational approaches [J].Aquaculture International,2016, 24(4): 1089—1102
[15] Li M F, Zheng X Q. Research progress of methods of SSR primers development [J].Hereditas, 2004, 26(5):769—776 [李明芳, 郑学勤. 开发SSR引物方法之研究动态. 遗传, 2004, 26(5): 769—776]
[16] Pashley C H, Ellis J R, McCauley D E,et al. EST databases as a source for molecular markers: lessons from Helianthus [J].Journal of Heredity, 2006, 97(4): 381
[17] Gupta P K, Rustgi S. Molecular markers from the transcribed/expressed region of the genome in higher plants[J].Functional & Integrative Genomics, 2004, 4(3):139—162
[18] Skaala Ø, Høyheim B, Glover K,et al. Microsatellite analysis in domesticated and wild Atlantic salmon (Salmo salarL.): allelic diversity and identification of individuals [J].Aquaculture, 2004, 240(1): 131—143
[19] Jiang F F, Wang Z W, Zhou L,et al. High male incidence and evolutionary implications of triploid form in northeast AsiaCarassius auratuscomplex [J].Molecular Phylogenetics and Evolution, 2013, 66(1): 350—359
[20] Yang L, Yang S T, Wei X H,et al. Genetic diversity among different clones of the gynogenetic silver crucian carp,Carassius auratus gibelio, revealed by transferring and isozyme markers [J].Biochemical Genetics, 2001,39(5): 213—225
[21] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning:a Laboratory Manual [M]. Cold Spring Harbor Laboratory Press. 1989
[22] Mishina T, Takada M, Takeshima H,et al. Molecular identification of species and ploidy of Carassius fishes in Lake Biwa, using mtDNA and microsatellite multiplex PCRs [J].Ichthyological Research, 2014, 61(2):169—175
[23] Guo W, Gui J F. Microsatellite marker isolation and cultured strain identification inCarassius auratus gibelio[J].Aquaculture International, 2008, 16(6): 497—510
[24] Zheng X H, Lu C Y, Zhao Y Y,et al. A set of polymorphic trinucleotide and tetranucleotide microsatellite markers for silver crucian carp (Carassius auratus gibelio) and cross-amplification in crucian carp [J].Biochemical Genetics, 2010, 48(7-8): 624—635
[25]Nei M, Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases [J].Proceedings of the National Academy of Sciences, 1979,76(10): 5269—5273
[26] Nei M, Li W H. Mathematical model for studying genetic variation in terms of restriction endonucleases [J].Proceedings of the National Academy of Sciences, 1979,76(10): 5269—5273
[27] Tamura K, Dudley J, Nei M,et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0 [J].Molecular Biology and Evolution, 2007, 24(8):1596—1599
[28] Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J].Evolutionary Bioinformatics, 2005,1: 47—50
[29] Rice W R. Analyzing tables of statistical tests [J].Evolution, 1989, 43(1): 223—225
[30] Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data [J].Genetics, 1992, 131(2):479—491
[31] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data [J].Genetics, 2000, 155(2): 945—959
[32] Falush D, Stephens M, Pritchard J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies [J].Genetics, 2003,164(4): 1567—1587
[33] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study [J].Molecular Ecology, 2005, 14(8):2611—2620
[34] He P. Abundance, polymorphism and applications of microsatellite in eukaryote [J].Hereditas, 1998, 20(4):42—47 [何平. 真核生物中的微卫星及其应用. 遗传,1998, 20(4): 42—47]
[35] Li M, Qi X. Genomic Progress on Microsatellites of Eukaryotes [J].Journal of Tianjin Agricultural University,2006, 13(1): 47—51 [李明, 亓小红. 微卫星标记的基因组学研究进展. 天津农学院学报, 2006, 13(1): 47—51]
[36] Valdes A M, Slatkin M, Freimer N B. Allele frequencies at microsatellite loci: the stepwise mutation model revisited [J].Genetics, 1993, 133(3): 737—749
[37] Ma Z Q, Röder M, Sorrells M E. Frequencies and sequence characteristics of di-, tri-, and tetra-nucleotide microsatellites in wheat [J].Genome, 1996, 39(1): 123—130
[38] Botstein D, White R L, Skolnick M,et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J].American Journal of Human Genetics, 1980, 32(3): 314
[39] Jia Z, Sun X, Liang L,et al. Isolation and characterization of microsatellite markers from Fangzheng silver crucian carp,Carassius auratus gibelio(Bloch), and crossamplification in the closely related species crucian carp,Carassius auratus auratus(Linnaeus) [J].Molecular Ecology Notes, 2007, 6(4): 1141—1143
[40] Jia Z Y, Shi L Y, Liu X F,et al. The genetic diversity of diploid and triploid crucian carp from six populations in Heilongjiang River System [J].Hereditas, 2008, 30(11):1459—1465 [贾智英, 石连玉, 刘晓峰, 等. 黑龙江水系不同倍性鲫鱼的遗传多样性. 遗传, 2008, 30(11):1459—1465]
[41] Lu S Q, Liu Z, Liu H Y,et al. Microsatellite DNA analysis of genetic diversity and the phylogenetic relationships of four breed varieties ofCarassiussp. [J].Journal of Fishery Sciences of China, 2005, 12(4): 371—376 [鲁双庆, 刘臻, 刘红玉, 等. 鲫鱼 4 群体基因组DNA遗传多样性及亲缘关系的微卫星分析. 中国水产科学, 2005,12(4): 371—376]
[42] Sun X W, Lu C Y, Liang L Q. Isolation of microsatellite with enrichment by magnetic beads in grass carp,Ctenopharyngodon idella[J].Journal of Fisheries of China,2005, 29(4): 482—486 [孙效文, 鲁翠云, 梁利群. 磁珠富集法分离草鱼微卫星分子标记. 水产学报, 2005, 29(4):482—486]
[43] Liu F, Xia J H, Bai Z Y,et al. High genetic diversity and substantial population differentiation in grass carp (Ctenopharyngodon idella) revealed by microsatellite analysis[J].Aquaculture, 2009, 297(1): 51—56
[44] Li J L, Zhu Z Y, Wang G L,et al. Isolation and characterization of 17 polymorphic microsatellites in grass carp [J].Molecular Ecology Resources, 2007, 7(6): 1114—1116
[45] Fu J J, Li J L, Shen Y B,et al. Genetic variation analysis of wild populations of grass carp (Ctenopharyngodon idella) using microsatellite markers [J].Hereditas, 2013,35(2): 192—201 [傅建军, 李家乐, 沈玉帮, 等. 草鱼野生群体遗传变异的微卫星分析. 遗传, 2013, 35(2):192—201]
[46] Ji C H, Gu J J, Mao R X,et al. Analysis of genetic diversity among wild silver carp (Hypophthamlichthys molitrix) populations in the Yangtze, Heilongjiang and Pearl Rivers using microsatellite markers [J].Journal of Fisheries of China, 2009, 33(3): 364—371 [姬长虹, 谷晶晶,毛瑞鑫, 等. 长江, 珠江, 黑龙江水系野生鲢遗传多样性的微卫星分析. 水产学报, 2009, 33(3): 364—371]
[47] Nevo E. Genetic variation in natural populations: patterns and theory [J].Theoretical Population Biology, 1978,13(1): 121—177
[48] Li S F, Wang Q, Chen Y L. Biochemical genetic structures and variations in natural populations of silver carp,bighead and grass carp in Changjiang River, Zhujiang River and Heilongjiang River [J].Journal of Fisheries of China, 1986, 10(4): 351—372 [李思发, 王强, 陈永乐. 长江、珠江、黑龙江三水系的鲢、鳙、草鱼原种种群的生化遗传结构与变异. 水产学报, 1986, 10(4):351—372]
[49] Xu T J, Wang R X, Sun Y N,et al. Genetic differentiation and structure of four cultured stock ofMarsupenaeus japonicusin China [J].Oceanologia et Limnologia Sinica,2009, 40(6): 786—792
[50] Wright S. The genetical structure of populations [J].Annals of Eugenics, 1949, 15(1): 323—354
[51] Li S Z, Fang F. On the geographical distribution of the four kinds of pond-cultured carps in China [J].Acta Zoologica Sinica, 1990, 36(3): 244—250 [李思忠, 方芳. 鲢,鳙, 青, 草鱼地理分布的研究. 动物学报, 1990, 36(3):244—250]
[52] Wang W W, Zhao J L, Li S F,et al. Genetic variation ofSiniperca scherzeriSteindachner from different geographical populations by AFLP analysis [J].Acta Hydrobiologica Sinica, 2009, 33(2): 304—309 [王伟伟, 赵金良, 李思发, 等. 斑鳜不同地理群体遗传变异的AFLP分析. 水生生物学报, 2009, 33(2): 304—309]
[53] Yang H R, Zhao H H, Liu L,et al. Assessing genetic diversity of mitochondrial D-loop sequences ofSqualiobarbus curriculusinYangtze River and Pearl River [J].Journal of Shanghai Ocean University, 2012, 21(3):337—343 [杨慧荣, 赵会宏, 刘丽, 等. 珠江和长江水系赤眼鳟D-loop基因序列遗传变异分析. 上海海洋大学学报, 2012, 21(3): 337—343]
[54] Ding X P, Luo X Y, Shao C G,et al. Comparative analysis of genetic diversity revealed bygenomic-SSR and EST-SSR makers in interspecies ofStylosanthes[J].Guangdong Agricultural Sciences, 2015, 42(14):106—113 [丁西朋, 罗小燕, 邵辰光, 等. Genomic-SSR和EST-SSR在柱花草种间的遗传差异. 广东农业科学, 2015, 42(14): 106—113]
[55] Kong L F, Bai J, Li Q. Comparative assessment of genomic SSR, EST-SSR and EST-SNP markers for evaluation of the genetic diversity of wild and cultured Pacific oyster,Crassostrea gigasThunberg [J].Aquaculture,2014, 420: S85—S91
[56] Mignon-Grasteau S, Boissy A, Bouix J,et al. Genetics of adaptation and domestication in livestock [J].Livestock Production Science, 2005, 93(1): 3—14
[57] Ellis J R, Burke J M. EST-SSRs as a resource for population genetic analyses [J].Heredity, 2007, 99(2): 125—132
[58] Eujayl I, Sorrells M E, Baum M,et al. Assessment of genotypic variation among cultivated durum wheat based on EST-SSRs and genomic SSRs [J].Euphytica, 2001,119(1-2): 39—43
[59] Eujayl I, Sorrells M E, Baum M,et al. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat [J].Theoretical and Applied Genetics, 2002, 104(2): 399—407
[60] Wren J D, Forgacs E, Fondon J W,et al. Repeat polymorphisms within gene regions: phenotypic and evolutionary implications [J].The American Journal of Human Genetics, 2000, 67(2): 345—356
[61] Li Y Q, Li H W, Gao L F,et al. Prograss of simple sequence repeats derived from expressed sequence tags [J].Journal of Plant Genetic Resources, 2004, 5(1): 91—95[李永强, 李宏伟, 高丽锋, 等. 基于表达序列标签的微卫星标记(EST-SSRs)研究进展. 植物遗传资源学报, 2004,5(1): 91—95]
[62] Gao H, Yu F, Luan S,et al. Relationships between polymorphism and types of SSR motif in the genome of Chinese shrimp Fenneropenaeus chinensis [J].Acta Hydrobiologica Sinica, 2009, 33(1): 94—102 [高焕, 于飞,栾生, 等. 中国明对虾基因组微卫星重复单元类型与其多态性关系. 水生生物学报, 2009, 33(1): 94—102]
[63] Katti M V, Sami-Subbu R, Ranjekar P K,et al. Amino acid repeat patterns in protein sequences: Their diversity and structural-functional implications [J].Protein Science, 2000, 9(6): 1203—1209
[64] Röder M S, Korzun V, Wendehake K,et al. A microsatellite map of wheat [J].Genetics, 1998, 149(4): 2007—2023
[65] Scott K D, Eggler P, Seaton G,et al. Analysis of SSRs derived from grape ESTs [J].TAG Theoretical and Applied Genetics, 2000, 100(5): 723—726
