中国地鼠口腔颊黏膜癌中miRNA与mRNA表达谱的关联分析
2018-05-04李莉红庞文彪常凯阎小艳高继萍宋国华
李莉红,庞文彪,常凯,阎小艳,高继萍,宋国华
(山西医科大学实验动物中心,实验动物与人类疾病动物模型山西省重点实验室,太原 030001)
口腔鳞状细胞癌(OSCC)已经成为众多癌症中最常见的一种头颈部恶性肿瘤,其发病率呈现逐年上升的趋势,并且趋于年轻化[1]。研究发现,OSCC的发生发展是一个多层面调控过程,包括从DNA水平、转录水平、转录后水平及蛋白表达水平都参与病变过程[2]。miRNA不仅与肿瘤发生、发展、诊断、治疗及预后密切相关,而且与许多细胞信号转导系统共同构成复杂的调控网络,发挥多种生物学作用,成为近年来肿瘤研究领域的热点。miRNA与靶mRNA完全或不完全结合在转录后水平上通过调控基因的表达、细胞增殖凋亡、上皮间质转化等过程参与口腔癌的发生发展[3-5]。因此,为进一步了解OSCC发生和发展相关miRNA参与的生物学过程,为疾病的分级、早期诊断及治疗提供重要的信息,本文将采用新一代高通量测序技术,构建中国地鼠口腔颊黏膜癌中miRNA和mRNA表达谱,筛选差异表达的miRNA和mRNA并进行二者关联分析,探讨miRNA在OSCC发生发展中的靶向调控作用,为寻找新的更好的肿瘤标志物提供实验依据。
1 材料与方法
1.1 实验动物与分组
60只8~10周龄清洁级中国地鼠,雄性,体重 25~35 g,由山西医科大学实验动物中心提供【SCXK(晋)2015-0001】,饲养于屏障环境【SYXK(晋)2015-0001】。随机分为三组,分别为空白对照组(24只)、溶剂对照组(12只)、模型组(24只)。隔天上午用小头棉签在模型组中国地鼠双侧颊囊涂擦0.5% DMBA丙酮液,并禁食禁水2 h。分别在第6、9、12、15周进行处死取颊囊组织,一部分低温保存,一部分进行病理学检测。病理学结果详见文献[6]。本实验应用中国地鼠期间按照山西医科大学实验动物管理委员会的要求进行实验操作,并通过伦理审核。本实验选取15周鉴定为鳞癌的3个冻存组织和3个正常组织作为本实验的研究材料。
1.2 方法
1.2.1 构建差异miRNA表达谱
分别从所取得样本中提取总RNA,经质检合格后,对总RNA片段化处理,分离胶技术回收18~35nt的RNA片段,加接头逆转录成cDNA,得到组织样本的测序文库。采用Illumina HiSeq 2500 高通量测序平台[7]对组织样本的miRNA进行测序,得到原始数据之后进行去接头、去低质量以及片段化选择等处理,获得目标序列。根据miRBase数据库(Release 21)中的基因注释信息,与已知miRNA进行比对[8],采用miRDeep2软件预测新miRNA[9],构建OSCC组织和正常组织的miRNA表达谱。分别将两组织的miRNA的表达量归一化,计算出RPM值(每百万条测序序列中出现的目标序列的数量),比较分析两组织中显著差异表达的miRNA [筛选条件:padj<0.05且|log2(Fold Change)|≥ 1 (Fold Change表示鳞癌组和正常组表达量的倍数变化)]。
1.2.2 构建差异mRNA表达谱
总RNA质检合格后用带有Oligo(dT)的磁珠富集mRNA,加入fragmentation buffer片段化,用六碱基随机引物合成cDNA第一链,加入缓冲液、dNTPs、RNaseH和DNA聚合酶I合成cDNA第二链,用QIAQuick PCR试剂盒纯化后加EB缓冲液、双联cDNA末端修复、加碱基A、加测序接头、2%琼脂糖凝胶电泳回收、PCR扩增,得到组织样品测序文库。采用Illumina HiSeq 2500 高通量测序平台提供的PE100测序策略对组织样本的mRNA进行测序。同miRNA一样得到Clean Reads后采用TopHat软件(v2.0.12)[10]和Bowtie2软件[11]与UCSC (University of California Santa Cruz) 基因组数据库中的Cricetulus griseus参考基因组比对分析,构建mRNA表达谱。分别将两组织的mRNA的表达量归一化,计算出RPKM值[12],采用DEGseq[13]进行基因差异表达分析[筛选条件:P<0.05且|log2(Fold Change)|≥ 1]。
1.2.3 OSCC和正常组织中差异表达miRNA/mRNA对接
应用Gene Ontology功能富集和KEGG Pathway分析,将1.2和1.3的结果关联分析,预测与OSCC发生发展相关性较高的miRNA和mRNA。
2 结果
2.1 OSCC和正常组织中差异miRNA表达谱
2.1.1 已知miRNA表达谱
对照OSCC和正常组织中已知的miRNA,共有268个miRNA差异表达,其中137个表达上调,131个表达下调,结果见图1所示。进一步对差异miRNA进行深度分析,显著差异的miRNA共有11个,其中上调的miRNA有8个,下调的有3个,具体结果见表1。
2.1.2 新的miRNA表达谱
经过分析统计共发现了208个新miRNA,其中有112个表达上调,96个表达下调,结果见图2。进一步深度分析,得到显著差异的miRNA共有3个,其中上调的miRNA有1个,下调的有2个。与正常组相比,上调的miRNA有Novel-118,padj(padj为校正之后的P值)为9.63E-06;下调的有Novel-117和Novel-135,padj值均为0.0412。

表1 口腔颊黏膜鳞癌组织和正常组织中显著差异表达的已知miRNATab.1 Significant differentially expressed known miRNA between OSCC and normal groups
注:padj为校正之后的P值;Fold Change表示鳞癌组和正常组表达量的倍数变化。
Note. Padj is the correctedPvalue. Fold change indicates the multiple changes in the expression of OSCC and normal group.
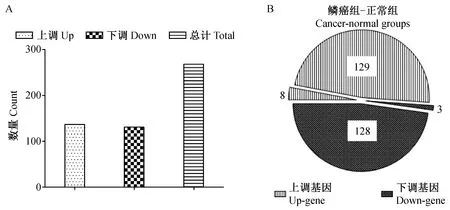
图1 口腔颊黏膜鳞癌组织和正常组织的差异表达miRNA柱状图和饼状图Fig.1 Histogram (A) and pie chart (B) of differentially expressed known miRNA between OSCC and normal groups

图2 口腔颊囊黏膜鳞癌组织和正常组织的新miRNA差异表达柱状图和饼状图Fig.2 Histogram (A) and pie chart (B) of differentially expressed novel miRNA between the OSCC and normal groups
2.2 OSCC和正常组织中差异mRNA表达谱
对照OSCC和正常组织中的mRNA进行分析,筛选后共检测出194个基因表达差异,其中128个基因表达上调,66个基因表达下调,结果见图3。口腔颊囊黏膜鳞癌组织和正常颊囊黏膜组织中差异表达前30位的基因见表2。
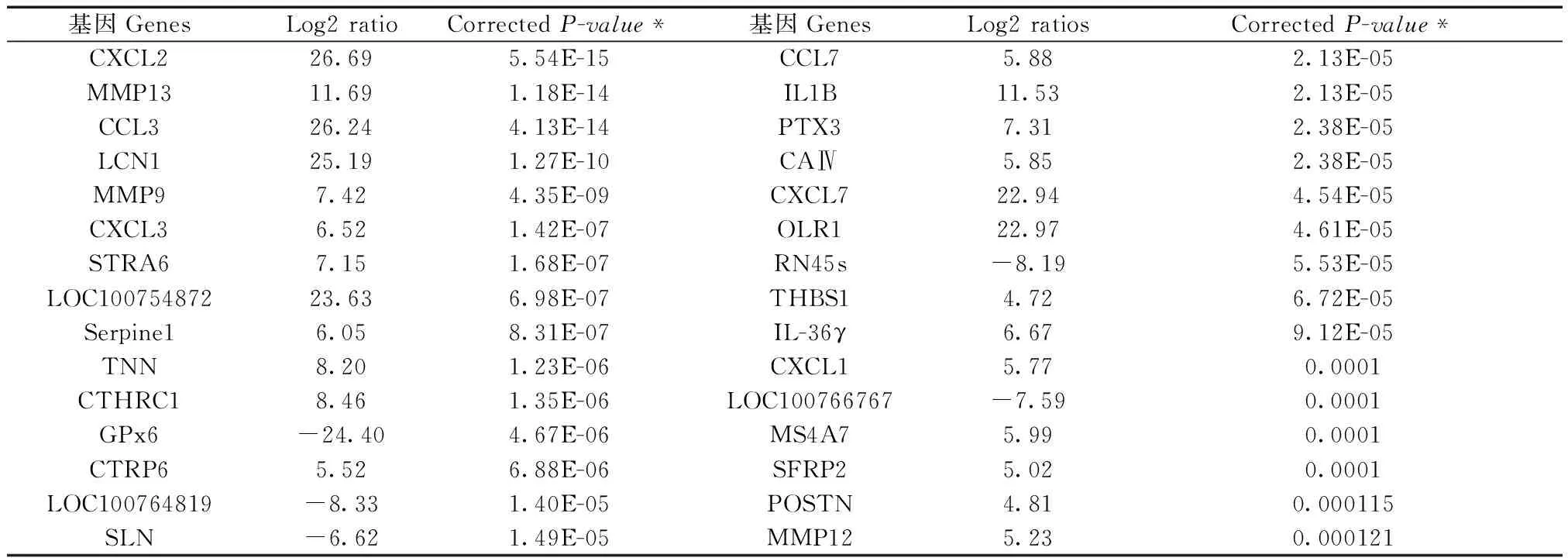
表2 口腔颊囊黏膜鳞癌组织和正常组织中前30位差异表达基因Tab.2 Differentially expressed genes in the top 30 between OSCC and normal groups
注:CorrectedP-value*为校正之后的P值;Ratio为Fold Change;Fold Change表示鳞癌组和正常组表达量的倍数变化。
Note. CorrectedP-value* is the correctedPvalue; Ratio is Fold Change; Fold Change indicates the multiple changes in the expression of OSCC and normal groups.

图3 口腔颊囊黏膜鳞癌组织和正常组织的差异表达基因柱状图和饼状图Fig.3 Histogram (A) and pie chart (B) of differentially expressed genes between OSCC and normal groups
2.3 OSCC组织中差异表达miRNA/mRNA的对接研究
大量的研究证实,miRNA通过转录后调节mRNA降解或抑制其翻译。据此,miRNA的表达水平应与其靶mRNA的表达水平相反,利用这一调节机制我们将转录组差异基因库和miRNA测序获得的差异的靶基因库做交集,找出两库重合基因,然后根据上调miRNA得到下调差异靶基因,下调miRNA得到上调差异靶基因。关联分析结果表明,中国地鼠口腔颊囊黏膜鳞癌组织与正常组织中显著差异表达的11个 miRNA 中有8个关联到差异靶基因,其中有 5 个 miRNA 上调(cgr-miR-21-3p,cgr-miR-34b-5p,cgr-miR-542-3p,cgr-miR-130-3p,cgr-miR-34c-5p),3 个 miRNA 下调(cgr-miR-499-5p,cgr-miR-486-3p,cgr-miR-504)。
但是,由于miRNA 与mRNA靶向调控关系是一对多,多对一的关系,即单个miRNA 可以靶向调控多个基因,多个miRNA靶向调控某个基因,因此,并不是上调的每个miRNA所靶向的每个基因都是呈现低表达,下调的每个miRNA所靶向的每个基因都是呈现高表达。最终,我们得到靶向调控的5个基因下调(Ttn,Ryr1,Tgm7,Pfkm,LOC100766767),靶向参与上调的基因有 14 个(Slc1a1,Islr2,Hoxa1,Thbs1,Inhbb,Palm,Gpr176,Fkbp10,Fam71f2,C1qtnf6,Krt19,LOC100767496,LOC100765213,LOC100762478)(如图4所示)。因新预测出的显著差异的miRNA在与基因进行关联分析时未发现靶基因,故后续不再研究。

图4 显著差异表达的已知miRNA及其差异表达靶基因统计Fig.4 Summary of significantly differentially expressed miRNA and its differentially expressed target genes
进一步根据显著差异表达的miRNA和基因表达的相关性构建miRNA-基因作用网络(如图5所示)。椭圆形表示基因,菱形表示miRNA,相互作用用直线表示。
3 讨论

图5 显著差异表达miRNA与差异表达靶基因的通路图Fig.5 Pathway map between significantly differentially expressed miRNA and differentially expressed target genes
OSCC是一种严重威胁生命的恶性肿瘤。由于广泛危险因素的联系,以及不可预测的治疗结果使OSCC成为最复杂的癌症之一。目前尽管已有许多治疗方式,但是在过去的40年里,OSCC患者的5年生存率并没有超过50%[14-15]。大量的证据表明,miRNA不仅可以作为癌症的诊断和预后标记,而且还能提供靶向治疗的靶点[16]。其在转录后水平上使靶mRNA降解,也可以与靶mRNA不完全互补在蛋白质翻译水平上抑制其表达[17]。新一代高通量测序技术是近十年来逐步从新技术成长为主流的测序技术,以其简单、快速、高分辨率和高通量的特点,在肿瘤研究领域得到广泛应用[18],但是在口腔癌的研究中应用鲜少。本研究首次在中国地鼠中成功构建口腔颊黏膜癌miRNA和mRNA的表达谱,对研究口腔颊黏膜癌机制具有参考意义。
大量研究表明,miRNA通过靶向mRNA参与肿瘤的发生发展,在许多肿瘤中miR-21[19],miR-130[20],miR-142[21]表达上调,而miR-486[22]表达下调。我们此次miRNA测序结果显示,cgr-miR-21-3p、cgr-miR-21-5p、cgr-miR-130b-3p、cgr-miR-142-5p表达量升高,cgr-miR-486-3p降低,与口腔黏膜正常组织比较具有统计学意义(P<0.05)。这充分显示与OSCC相关的miRNA的表达水平与已有研究一致,证实了测序结果可信。在转录组测序中,我们得到了194差异表达的基因。从前30位差异表达的基因看出,这些基因主要是促炎与抗炎因子、促瘤与抑瘤因子,其中基质金属蛋白酶(MMPs)家族中的MMP9、MMP13在多项口腔鳞癌研究中已经证实显著高表达[23-24];趋化因子CXCL2、CXCL3由Peng[25]在大鼠口腔鳞癌微阵列芯片分析中检测出显著高表达。这些研究表明:与口腔癌相关的差异表达基因的表达水平与本实验一致,在OSCC的发生、侵袭、转移等方面发挥重要作用。
从联合分析的结果来看,一个miRNA可调控多个靶基因,如:cgr-miR-21-3p、cgr-miR-542-3p、cgr-miR-34c-5p、cgr-miR-486-3p、cgr-miR-504。同样,一个基因被多个miRNA调控,如:Ttn、C1qtnf6、Ryr 1。据报道,功能失调的miR-21-3p在卵巢癌细胞中通过靶向基因RBPMS、RCBTB1、ZNF608和NAV3促进癌细胞增殖和侵袭[26-27]。miR-542-3p在结肠直肠癌中靶向基因OTUB1、CTTN抑制肿瘤细胞的增殖、迁移[28-29]。Gandla等[30]研究恶性癌症疼痛时,深入的生物信息学分析显示,在miR-34c-5p的高度靶向的目标基因中,有Cav2.3、12 P2rx6、Oprd1和Oprm1。至于miR-486-3p,目前没有直接的研究指明其在口腔鳞状细胞癌的发生发展中发挥的作用以及如何调控OSCC机制,但是有肺癌中miR-486-5p通过下调促癌基因Arhgap5和胰岛素生长因子信号转导的肿瘤抑制基因被确定为肿瘤抑制基因,其表达在血浆样品中也得到验证[31-33]。MiR-504通过直接结合肿瘤抑制基因TP53的3′-UTR和靶基因FOXP1影响细胞凋亡、细胞周期和细胞的生长[34-35]。以上研究虽未在口腔癌中充分证实,但也暗示出一个miRNA可以调控多个基因,与我们的实验结果一致。
Pfhler等[36]研究发现 TTN作为存在于黑色素瘤相关性视网膜病患者的血清抗体中的抗原,是黑色素瘤潜在的生物标记,可能具有致癌作用。C1qtnf6是重组人补体肿瘤坏死因子相关蛋白6,在卵巢癌患者血清和卵巢癌细胞系中表达显著下调,C1qtnf6可抑制 IL-8 /VEGF通路从而抑制卵巢癌细胞的增殖和转移[37]。兰尼碱受体1 (ryanodine receptors 1, Ryr 1)是一种同种四聚体钙离子通道蛋白,有学者采用基因沉默技术,使肺癌细胞中的 Ryr1基因表达下调,证实Ryr1基因沉默可使细胞增殖能力下降,使细胞凋亡率显著增高,进而证实Ryr1基因沉默可使肺癌细胞生物学活性减低,使肿瘤的生长受到抑制[38]。虽然目前还没有基因Ttn、C1qtnf6、Ryr1被多个miRNA调控的报道,但是已有许多生物测序实验如:廖江铨等[39]在冠心病血瘀证相关lncRNA-miRNA-mRNA调控网络研究时得到,基因CTSB可以被hsa-miR-106a-5p、hsa-miR-106b-5p和hsa-miR-20b-5p调节,而RTKN2可被hsa-miR-3158-3p和novel-m9052-5p调控,暗示我们的miRNA-mRNA调控网络分析选出的基因Ttn、C1qtnf6、Ryr1被多个miRNA调控的结果是可信的。
本实验通过对OSCC和正常组织中miRNA和mRNA表达谱的建立以及二者联合分析,使我们更加深入的了解到:一个miRNA可调控多个mRNA,同时一个mRNA可由多个miRNA调控,它们相互作用形成复杂的调控网络,在口腔癌的发生发展中发挥重要作用,为口腔癌的发生、发病机制,及临床治疗及预后判断提供理论依据,同时也为我们筛选口腔癌标志物提供了理论依据。
参考文献(References)
[1] 徐佳瑛, 杨丽丽, 马超, 等. IL-17、COX-2 在口腔扁平苔藓和口腔鳞癌中的表达分析[J]. 实用癌症杂志, 2013, 28(5): 480-482.
Xu JY, Yang LL, Ma C, et al. Analysis of expression of COX-2 and IL-17 in the oral lichen planus and oral squamous cell carcinoma [J]. Pract J Cancer, 2013, 28(5): 480-482.
[2] Williams HK. Molecular pathogenesis of oral squamous carcinoma [J]. Mol Pathol, 2000, 53(4): 165-172.
[3] Li J, Huang H, Sun L, et al. MiR-21 indicates poor prognosis in tongue squamous cell carcinomas as an apoptosis inhibitor [J]. Clin Cancer Res, 2009, 15(12): 3998- 4008.
[4] Chang CJ, Hsu CC, Chang CH, et al. Let-7d functions as novel regulator of epithelial-mesenchymal transition and chemoresistant property in oral cancer [J]. Oncol Rep, 2011, 26(4): 1003-1010.
[5] Long XB, Sun GB, Hu S, et al. Let-7a microRNA functions as a potential tumor suppressor in human laryngeal cancer [J]. Oncol Rep, 2009, 22(5): 1189-1195.
[6] 皇甫冰, 庞文彪, 宋国华, 等. 中国地鼠口腔颊黏膜癌模型的建立及癌变的动态观察 [J]. 癌变·畸变·突变, 2016, 28(1): 56-65.
Huangfu B, Pang WB, Song GH, et al. Development of oral buccal pouch mucosa cancer in Chinese hamsters and dynamic observation of their carcinogenesis [J]. Carcinog Teratog Mutag, 2016, 28(1): 56-65.
[7] Markus Hafner, Pablo Landgraf, Janos Ludwig, et al. Identification of microRNAs and other small regulatory RNAs using cDNA library sequencing [J]. Methods, 2008, 44(1): 3-12.
[8] Quinlan AR, Hall IM. BEDTools: a flexible suite of utilities for comparing genomic features [J]. Bioinformatics. 2010, 26(6): 841-842.
[9] Friedlander MR, Mackowiak SD, Li N, et al. miRDeep2 accurately identifies known and hundreds of novel microRNA genes in seven animal clades [J]. Nucleic Acids Res, 2012, 40(1): 37-52.
[10] Trapnell C, Pachter L, Salzberg SL. TopHat: discovering splice junctions with RNA-seq [J]. Bioinformatics, 2009, 25(9): 1105-1111.
[11] Langmead B, Trapnell C, Pop M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome [J]. Genome Biol, 2009, 10(3): R25.
[12] Wagner GP, Kin K, Lynch VJ. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples [J]. Theory Biosci, 2012, 131(4): 281-285.
[13] Wang L, Feng Z, Wang X, et al. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data [J]. Bioinformatics, 2010, 26(1): 136-138.
[14] Ferlay J, Soerjomataram I, Dikshit R, et al. Cancer incidence and mortality worldwide: sources, methods and major patterns in GLOBOCAN 2012 [J]. Int J Cancer. 2015, 136(5): E359-86.
[15] Lippman SM, Hong WK. Molecular markers of the risk of oral cancer [J]. N Engl J Med, 2001, 344(17): 1323-1326.
[16] Vandenboom Ii TG, Li Y, Philip PA, et al. MicroRNA and cancer: tiny molecules with major implications [J]. Curr Genomics, 2008, 9(2): 97-109.
[17] Lau NC, Lim LP, Weinstein EG, et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans [J]. Science, 2001, 294(5543): 858-862.
[18] 肖岚.新一代测序技术及其在肿瘤研究中的应用 [J]. 国际病理科学与临床杂志, 2013, 33(4): 322-326.
Xiao L. Next generation sequencing technology and its application in cancer research [J]. Int J Pathol Clin Med, 2013, 33(4): 322-326.
[19] Li SJ, Zhou J, Zhang L, et al. The effect of miR-21 on SWOZ2 glioma cells and its biological mechanism [J]. J BUON, 2017, 22(2): 468-473.
[20] Jiang H, Yu WW, Wang LL, et al. miR-130a acts as a potential diagnostic biomarker and promotes gastric cancer migration, invasion and proliferation by targeting RUNX3 [J]. Oncol Rep, 2015, 34(3): 1153-1161.
[21] Liu LQ, Liu SC, Duan QX, et al. MicroRNA-142-5p promotes cell growth and migration in renal cell carcinoma by targeting BTG3 [J]. Am J Transl Res, 2017, 9(5): 2394-2402.
[22] Wang J, Tian X, Han R, et al. Downregulation of miR-486-5p contributes to tumor progression and metastasis by targeting protumorigenic ARHGAP5 in lung cancer [J]. Oncogene, 2014, 33(9): 1181-1189.
[23] 张瑛, 钱忠义, 普苹, 等. VEGF、MMP-9、TIMP-2在口腔颌面部鳞癌中的表达研究 [J]. 口腔医学研究, 2012, 28(11): 1141-1148.
Zhang Y, Qian ZY, Pu P, et al. Expression and significance of VEGF, MMP-9 and TIMP-2 in OSCC [J]. J Oral Sci Res, 2012, 28(11): 1141-1148.
[24] 苏颖颖, 朱恩新, 马卫东, 等. c-fos及基质金属蛋白酶13在人口腔鳞状细胞癌发生中的表达及病理意义 [J]. 口腔医学研究, 2009, 25(1): 23-25.
Su YY, Zhu EX, Ma WD, et al. Expression and pathological significance of c-fos and MMP13 in human oral squamous cell carcinoma [J]. Oral Sci Res, 2009, 25(1): 23-25.
[25] Peng X, Li W, Johnson WD, et al. Overexpression of lipocalins and pro-inflammatory chemokines and altered methylation of PTGS2 and APC2 in oral squamous cell carcinomas induced in rats by 4-nitroquinoline-1-oxide [J]. PLoS One, 2015, 10(1): e0116285.
[26] Pink RC, Samuel P, Massa D, et al. The passenger strand, miR-21-3p, plays a role in mediating cisplatin resistance in ovarian cancer cells [J]. Gynecol Oncol, 2015, 137(1): 143-151.
[27] Báez-Vega PM, Echevarría Vargas IM, Valiyeva F, et al. Targeting miR-21-3p inhibits proliferation and invasion of ovarian cancer cells [J]. Oncotarget, 2016, 7(24): 36321-36337.
[28] Yuan L, Yuan P, Yuan HJ, et al. miR-542-3p inhibits colorectal cancer cell proliferation, migration and invasion by targeting OTUB1 [J]. Am J Cancer Res, 2017, 7(1): 159-172.
[29] Long HC, Gao X, Lei CJ, et al. miR-542-3p inhibits the growth and invasion of colorectal cancer cells through targeted regulation of cortactin[J]. Int J Mol Med, 2016, 37(4): 1112-1118.
[30] Gandla J, Lomada SK, Lu J, et al. miR-34c-5p functions as pronociceptive microRNA in cancer pain by targeting Cav2.3 containing calcium channels [J]. Pain, 2017, 158(9): 1765-1779.
[31] Wang J, Tian X, Han R, et al. Downregulation of miR-486-5p contributes to tumor progression and metastasis by targeting protumorigenic ARHGAP5 in lung cancer [J]. Oncogene, 2014, 33: 1181-1189.
[32] Peng Y, Dai Y, Hitchcock C, et al. Insulin growth factor signaling is regulated by microRNA-486, an underexpressed microRNA in lung cancer [J]. Proc Natl Acad Sci U S A, 2013, 110(37): 15043-15048.
[33] Mozzoni P, Banda I, Goldoni M, et al. Plasma and EBC microRNAs as early biomarkers of non-small-cell lung cancer [J]. Biomarkers, 2013, 18(8): 679-686.
[34] Hu W, Chan CS, Wu R, et al., Negative regulation of tumor suppressor p53 by microRNA miR-504 [J]. Mol Cell, 2010, 38(5): 689-699.
[35] Kikkawa N, Kinoshita T, Nohata N, et al. MicroRNA-504 inhibits cancer cell proliferation via targeting CDK6 in hypopharyngeal squa mous cell carcinoma [J]. Int J Oncol, 2014; 44(6): 2085-2092.
[36] Pfohler C, Preuss KD, Tilgen W, et al. Mitofilin and titin as target antigens in melanoma-associated retinopathy [J].Int J Cancer, 2007, 120(4): 788-795.
[37] 王林, 刘子冬, 段利利, 等. C1q/肿瘤坏死因子相关蛋白 6(CTRP6) 抑制卵巢癌细胞的增殖和迁移 [J]. 细胞与分子免疫学杂志, 2015, 31(12): 1664-1668.
Wang L, Liu ZD, Duan LL, et al. C1q tumor necrosis factor-related protein 6 (CTRP6) inhibits the proliferation and migration of ovarian cancer cells [J]. Chin J Cell Mol Immunol, 2015, 31(12): 1664-1668.
[38] 薄龙, 潘泓, 赵冰冰, 等. Ryr1基因沉默对人肺癌细胞生物学活性的影响 [J]. 广西医科大学学报, 2013, 30(6): 856-859.
Bo L, Pan H, Zhao BB, et al. Effect of Ryr1 gene silencing on biological activity of human lung cancer cells [J]. J Guangxi Med Univ, 2013, 30(6): 856-859.
[39] 廖江铨, 王阶. 冠心病血瘀证相关lncRNA-miRNA-mRNA调控网络研究 [D]. 北京中医药大学,2017.
Liao JQ, Wang J. Study on related lncRNA-miRNA-mRNA regulatory network in CHD Blood Stasis syndrome [D]. Beijing University of Chinese Medicine, 2017.
