早期乳腺癌患者循环肿瘤细胞TP53、磷脂酰肌醇-3激酶基因突变分析及其与患者临床病理特征的相关性
2018-05-04贾淞淋吴雅妮刘超乾苏东玮李恒宇
林 健, 贾淞淋, 方 敏, 吴雅妮, 刘超乾, 苏东玮, 于 跃, 李恒宇, 盛 湲
海军军医大学附属长海医院甲状腺乳腺外科,上海 200433
乳腺癌是女性最常见的恶性肿瘤之一,晚期预后较差。因此早期监测肿瘤负荷对评判治疗效果和预防疾病进展非常重要[1]。Cristofanilli等[2]采用CellSearch法对外周血循环肿瘤细胞(circulating tumor cells,CTC)进行筛选计数,证实CTC是转移性乳腺癌进展和患者生存的独立预后因素,CTC数量升高(>5个/7.5 mL)与预后不良及乳腺癌进展密切相关。肿瘤患者体内来自肿瘤细胞的DNA会持续释放入血或其他体液形成循环肿瘤DNA(circulating tumor DNA,ctDNA),是反映转移性乳腺癌治疗反应特异性和敏感性最高的生物学指标[1]。ctDNA包含肿瘤特异性的基因突变,可作为一种特异性的基因标志或生物标志物[3]。
乳腺癌最常见的突变基因是TP53和磷脂酰肌醇-3激酶(phosphatidylinositol-3 kinase,PI3K)。抑癌基因TP53突变与多种人类肿瘤发生和发展密切相关[4]。乳腺癌患者TP53的突变率为20%~50%[5],TP53常见的突变位点有p.R175H、p.R248Q和p.R273H等,这些突变常导致下游基因的激活功能丢失[6],基因组稳定性降低[7]。PI3K信号通路异常激活与乳腺癌的发生和发展密切相关。约25%的乳腺癌中发现PI3K突变,其常见突变位点是PI3KCA基因编码螺旋形区域的p.E545K和p.E542K位点及编码激酶区域的p.H1047R位点。这些突变会导致下游信号增强,促肿瘤发生和发展[8]。TP53和PI3K基因不同位点的突变与肿瘤不同的发生、发展和预后相关[9-10]。因此,TP53/PI3K突变检测常被用于各类肿瘤治疗管理[8, 11-12]。
Dawson等[1]发现转移性乳腺癌中ctDNA TP53、PI3K突变位点的检测能更敏感、更早期地预测乳腺癌的治疗反应和疾病进展情况。因此,本研究选择TP53基因的p.R248Q、p.R273H、p.R175H位点及PI3K基因的p.E545K、p.H1047R位点进行检测。已有研究[13-15]采用检测尿液ctDNA特定基因突变诊断列腺癌或膀胱癌甚至监测非小细胞肺癌,但尚无利用尿液来监测早期乳腺癌的报道。因此,本研究尝试采用尿液作为检测标本来源。
为了进一步探讨TP53、PI3K突变在早期乳腺癌中的作用,本研究收集了患者的血液和尿液标本,采用CellSearch法采集血中CTC并检测其数目,并采用微滴式数字PCR(ddPCR)技术检测患者血、CTC及尿标本中TP53(p.R248Q)、TP53(p.R273H)、TP53(p.R175H)、PI3K(p.E545K)和PI3K(p.H1047R)共5种基因位点的突变情况,同时收集所有患者的临床资料,分析基因突变和临床病理特征间的相关性,探讨CTC TP53/PIK基因突变检测对乳腺癌诊疗和预后的判断作用,以期在早期乳腺癌防治上寻找更有价值的生物学指标。
1 资料与方法
1.1 一般资料 选择2015年1月—2月海军军医大学附属长海医院收治的女性乳腺癌患者和良性肿瘤患者共30例。纳入标准:(1)无其他肿瘤病史;(2)术前未进行过任何抗肿瘤治疗;(3)病理证实为乳腺癌或乳腺良性肿瘤;(4)术前检查无转移相关证据;(5)无其他严重系统性疾病或感染。其中26例乳腺癌初诊患者接受手术治疗;3例良性乳腺肿瘤患者接受肿块切除;1例患者入院前已接受过乳腺肿块切除手术,其免疫组织化学检测结果未携带,因此未被纳入本研究。
所有患者发病年龄为30~79岁,平均(54.2±12.8)岁;其中26例乳腺癌患者平均年龄为(54.6±13.1)岁。26例乳腺癌患者按分子分型:Luminal A型3例(11.5%),Luminal B型14例(53.9%),HER-2阳性型7例(26.9%),三阴性2例(7.7%);根据临床分期:最多的是ⅡA期9例(34.6%),其余分别为0期3例(11.5%),ⅠA期4例(15.4%),ⅡB期2例(7.7%),ⅢA期4例(15.4%),ⅢC期4例(15.4%);按病理类型:浸润性导管癌19例(73.1%),导管原位癌3例(11.5%),非特殊性浸润癌2例(7.7%),多形性癌1例(3.8%),髓样特征的浸润性癌1例(3.8%)。3例良性肿瘤患者均为乳腺纤维腺瘤。
所有患者的临床信息均从医院电子病历管理系统中提取,本研究经医院伦理委员会审核批准。参与研究的患者均签署了正式的知情同意文件。
1.2 病理学检查 乳腺癌的组织类型、组织分级、雌激素受体(ER)、孕激素受体(PR)、HER-2和Ki-67水平等均来自患者的病理报告,其中ER、PR、HER-2和Ki-67指数等来自免疫组织化学染色结果。HER-2表达是免疫组织化学检测和(或)荧光原位杂交(FISH)结果。HER-2阳性是指免疫组织化学检测结果为和(或)FISH结果>2.2;HER-2阴性是指免疫组织化学检测结果为0~+和(或)FISH<1.8[5]。
1.3 CTC计数 术前抽取7.5 mL静脉全血于含EDTA抗凝及细胞防腐剂的CellSave管中,室温保存,72 h内。采用CellSearch系统(Veridex)分离和计数CTC。采用CellSearch Epithelial Cell Kit处理标本,结果用CellSpitter Analyzer分析。荧光染色特性为EpCAM(+)、CK(+)、DAPI(+)和CD45(-)的细胞即为CTC[16]。
1.4 血浆、尿液的收集及DNA抽提 术前使用BCT管抽取至少10 mL全血,并留取20 mL尿液。血标本抽取后,3 h内分别以1 600×g和16 000×g,4℃,离心10 min,去除血细胞,每2~4 mL上清液分装到50 mL离心管中,-80℃冻存;然后根据操作手册,使用QIAamp Circulating Nucleic Acid Kit(德国Qiagen公司产品)抽提血浆中ctDNA。尿液、CTC的DNA抽提参考既往文献[13,17]进行。
1.5 基因突变检测 按照QX200TM微滴式数字PCR(droplet digital PCR,ddPCR)系统(美国Bio-Rad公司产品)操作说明进行基因突变检测。ddPCR反应体系共20 μL,包括10 μL 2×ddPCR SuperMix、5 μL DNA模板、2 μL引物 mix和3 μL去离子水。将20 μL反应体系依次加入96孔板,随后加入70 μL微滴发生油,在自动微滴发生器中生成微滴。微滴生成结束后,将96孔板放入热封仪中封铝膜,随后放入PCR扩增仪进行PCR扩增。PCR反应程序:95℃ 5 min;94℃ 30 s,55℃ 1 min,40个循环;最后98℃ 10 min。扩增结束后采用QX200 Droplet Reader软件进行分析,并用QuantaSoft分析软件计算突变等位基因浓度(copies/μL,CMUT)。引物由生工生物工程(上海)股份有限公司合成,引物序列信息参考以往文献[11,18-19]报道。

2 结 果
2.1 CTC计数 共30例女性乳腺癌或良性肿瘤患者接受了CTC筛查和血、尿TP53/PI3K基因检测,其中1例乳腺癌患者因就诊前已在外院接受乳腺肿块切除术而缺乏免疫组织化学检测结果,因此被排除出研究。纳入研究的29例患者中,良性肿瘤患者3例,均未检测到CTC;乳腺癌患者26例,其中6例(23.1%)未检测到CTC,20例(76.9%)检测到CTC,CTC>5个者4例(15.4%),CTC数值范围为0~12,平均数为2.846±3.133,中位数为2。按临床分期,除ⅢC期患者外,其余均检测到CTC;其中,ⅠA和ⅢA期患者全部检测到CTC,ⅡB期患者检测到CTC较多,平均数目为4。按分子分型,Luminal A和三阴性乳腺癌全部检测到CTC;其中CTC检测数目最高的为三阴性乳腺癌,平均CTC检测数为4。>40岁的患者CTC检出率较高(P=0.028)。CTC与患者其他临床病理特征的相关性无统计学意义。
2.2 TP53、PI3K基因突变
2.2.1 总体突变情况 29例患者血、CTC和尿标本,共获得435个基因检测结果,5种检测的基因位点总共有82个发生突变(18.9%)。各基因突变率不同,最高的是TP53(p.R175H),有29个(6.7%)突变结果;TP53(p.R248Q)、TP53(p.R273H)和PI3K(p.H1047R)分别有25(5.7%)、23(5.3%)和4(0.9%)个突变结果;最低为PI3K(p.E545K),仅1个(0.2%)发生突变。5种基因位点间突变率差异有统计学意义(χ2=50.133,P<0.001)。
2.2.2 三类标本总体基因突变情况 血、CTC和尿三类标本中检测到5种基因突变分别为29个(6.7%)、21个(4.8%)和3个(7.4%),突变率差异无统计学意义(χ2=2.915,P=0.233)。
2.2.3 良恶性肿瘤5种基因突变情况 结果显示每类标本的每种基因突变在良恶性肿瘤间突变构成情况无差异。82个基因突变结果中,乳腺癌的TP53 (p.R248Q、p.R273H、p.R175H)及PI3K (p.E545K、p.H1047R)突变数分别为22、21、28、1、3个,良性肿瘤的这5种基因突变数分别为3、2、1、0、1,统计分析显示在良恶性肿瘤间5种基因突变构成差异无统计学意义(P=0.359)。
2.2.4 三类标本中5种基因突变情况 血、CTC、尿三类标本中5种基因突变分布情况差异有统计学意义(χ2=50.133,P<0.001)。其中,29份CTC标本中,突变率最高的是TP53(p.R175H),有7份(24.1%)发生突变;TP53(p.R248Q)和TP53(p.R273H)均有6份发生突变(20.7%),PI3K(p.H1047R)有2份(6.9%),PI3K(p.E545K)无突变;5种基因突变率差异有统计学意义(P=0.007)。29份血标本中,突变率最高的是TP53(p.R175H),有11份(37.9%)发生突变;TP53(p.R248Q)和TP53(p.R273H)分别有10份(34.5%)和8份(27.6%)发生突变,PI3K(p.E545K)和PI3K(p.H1047R)均无突变;5种基因突变率差异有统计学意义(χ2=25.172,P<0.001)。29份尿标本中,突变率最高的是TP53(p.R175H),有11份发生突变(37.9%);TP53(p.R248Q)和TP53(p.R273H)均有9份(31.0%)发生突变,PI3K(p.H1047R)有2份(6.9%)发生突变;最低的是PI3K(p.E545K),仅1份(3.4%)发生突变;5种基因突变率差异有统计学意义(χ2=16.681,P=0.002)。
2.3 TP53、PI3K突变与乳腺癌临床病理特征的相关性 结果(表1)表明:乳腺癌分子分型和Ki-67指数与CTC中TP53(p.R248Q)突变相关(Fisher Exact Test=10.839,P=0.003;P=0.028),其中Luminal A型和Ki-67指数小于20%的乳腺癌TP53(p.R248Q)突变率较高,提示在治疗Luminal A型乳腺癌时需要考虑TP53(p.R248Q)基因突变带来的内分泌药物抵抗等因素;腋窝淋巴结转移可能与CTC标本中TP53(p.R175H)基因突变有关(P=0.058),其中发生淋巴结转移的乳腺癌患者TP53(p.R175H)未突变较多;未在血标本中检测到Luminal A型患者有基因突变,而且年龄小于40岁的乳腺癌患者未检测到CTC标本有基因突变。其余临床病理特征与基因突变间无明显相关性。
3 讨 论
CTC是转移性乳腺癌进展和生存的独立预后因素[2]。众多围绕CTC数量、表型甚至单细胞测序等开展的研究进一步证实,CTC对乳腺癌转移具有监测作用[20-21],并已被应用于晚期乳腺癌的疗效评估和预后判断[22]。然而针对早期乳腺癌,CTC的作用还不确切。
本课题组前期针对早期非转移性乳腺癌检测CTC时发现,肿瘤负荷重和激素受体阴性的乳腺癌患者往往CTC数量高,HER-2阳性和三阴性乳腺癌中CTC较少表达角蛋白,提示激素受体阴性可能与CTC上皮-间质转化有关[23]。本研究再次证实,临床分期较高或三阴性乳腺癌患者的CTC数量较多。
本研究进一步将CTC与ctDNA基因突变相结合来分析乳腺癌患者的临床病理特征。通过ddPCR检测CTC中TP53和PI3K基因的突变情况,分析结合CTC和TP53/PI3K突变检测对乳腺癌肿瘤负荷和疗效监测及预后评估的作用。结果发现,在早期乳腺癌中分子分型和肿瘤指标Ki-67与CTC标本中TP53(p.R248Q)基因突变相关,腋窝淋巴结转移情况可能与TP53(p.R175H)基因突变有关,提示这两种基因突变检测对确定早期乳腺癌的治疗方案及判断患者预后具有很好的指导作用。由于样本量有限,本研究尚未发现早期乳腺癌CTC与其他临床病理特征的相关性,这与之前的研究[16,23]结果一致。但通过检测CTC可预先评估疗效,判断特定患者可能出现的预后情况,或用来监控病情变化。
在乳腺癌的诊疗全程管理中,要充分考虑PI3K基因突变可能带来的影响。一项最新的大数据汇总分析表明,约32%的早期乳腺癌患者发生PI3K突变,编码螺旋形区域(p.E545K、p.E542K)与编码激酶区域(p.H1047R)的突变分别占52%和39%;PI3K突变更多见于较大年龄、ER阳性、低级别和小肿瘤,且PI3K突变与较好的临床结局(无浸润生存、无远处转移生存和总生存)显著相关[24]。以往研究[25]还提示,PI3K(p.H1047R)突变与淋巴结转移阴性有关,PI3K(p.E545K)突变与较大年龄有关。本研究结果与之前报道一致,PI3K(p.H1047R)突变共3例,2例发生于淋巴结阴性乳腺癌患者,而1例PI3K(p.E545K)突变发生在年龄大于40岁的乳腺癌患者。因此,今后治疗乳腺癌不仅依据是否发生PI3K突变,还要根据特定的突变位点来设计诊疗方案。
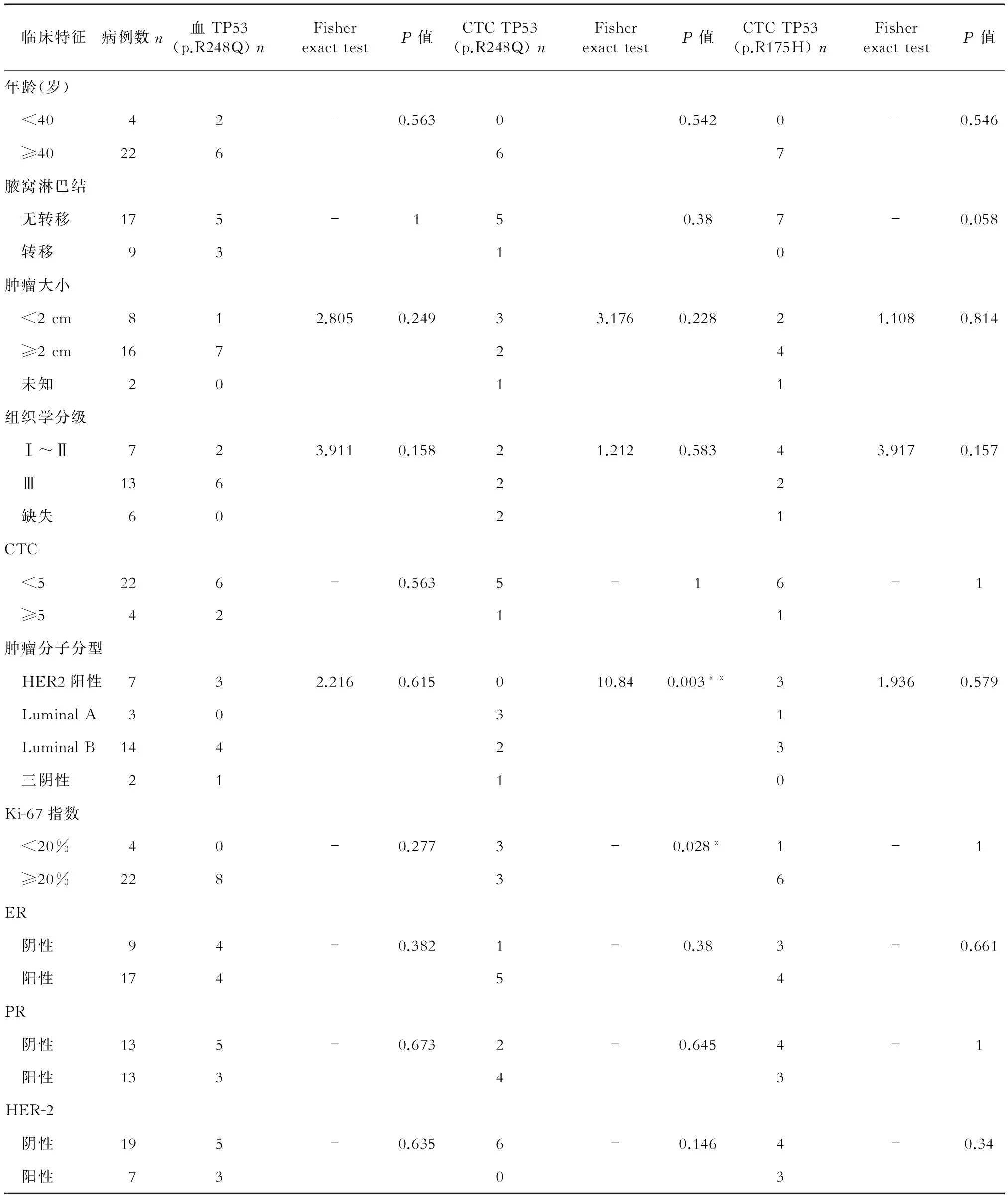
表1 乳腺癌患者血TP53(p.R248Q)、CTC TP53(p.R248Q)、CTC TP53(p.R175H)基因突变与临床病理特征的相关性
**P<0.01,*P<0.05
TP53突变与原发肿瘤和复发肿瘤突变的一致率高达90%[12],在血或其他体液中检测到TP53突变比常用的肿瘤监测指标CA125能更早发现肿瘤进展[18]。相比无突变的野生型TP53乳腺癌患者,TP53突变患者对药物治疗的反应和疾病预后更差[26]。BIG02-98临床试验也提示,TP53不同位点突变对预后的作用不同[27]。本研究涉及的TP53突变位点密码子转录后,翻译为不同的氨基酸,影响整个p53蛋白功能。因此不同部位TP53突变会产生不同的生物学影响。本研究检测的血、CTC和尿三类标本基因总体突变情况无差异,提示三类标本均可作为突变检测标本来源,但各标本内不同位点突变有差异,均是TP53(p.R175H)突变率最高;但TP53(p.R175H)在三类标本中的突变情况与患者临床病理特征无显著相关,仅在CTC标本中提示腋窝淋巴结转移可能与TP53(p.R175H)基因突变有关(P=0.058)。这可能与本研究纳入样本量较少有关。有研究指出,胚系TP53突变与乳腺癌诊断年龄和HER-2状态有关,诊断乳腺癌时年龄每增加1岁,TP53突变率减少5%,而年轻女性HER-2阳性乳腺癌的TP53突变风险增加近7倍[5]。但本研究结果与之有差别,年龄>40岁组患者TP53突变有21例(84.0%),HER-2阴性组有18例(72.0%)。
TP53的预后价值与淋巴结转移和肿瘤大小有关[26]。本研究中肿瘤增殖指标Ki-67指数与CTC中TP53(p.R248Q)突变相关(P=0.028),且腋窝淋巴结转移可能与CTC标本中TP53(p.R175H)基因突变有关(P=0.058)。这些结果提示,TP53突变与肿瘤预后可能相关,尤其是TP53(p.R175H)基因突变的患者发生淋巴结转移的可能性较小,预后较好。而一项针对2 000多例乳腺癌患者的基因和转录分析表明,按乳腺癌分子分型来看,TP53突变最容易发生在三阴性乳腺癌(34%),其次是HER-2阳性乳腺癌(22%)、Luminal B(13%)和Luminal A(5%)型乳腺癌[28]。本研究同样揭示,乳腺癌分子分型与CTC标本中TP53(p.R248Q)突变相关(P=0.003),提示乳腺癌不同分子分型中TP53突变率不同;但本研究各基因突变分布与既往研究不同。另外,汇总三类标本5种基因位点的总体突变情况,发现Luminal A型患者的血标本中未检测到突变,年龄<40岁的CTC标本中未检测到突变,这可能与Luminal A型患者较少有关。本研究中CTC在年龄>40岁组患者中检测率高,这可能影响了40岁以下组CTC标本的基因突变检出率。
综上所述,本研究采用CellSearch方法对早期乳腺癌患者进行CTC检测,并收集血、CTC和尿三类标本,通过ddPCR方法来检测TP53和PI3K基因的突变情况,并结合CTC和TP53/PI3K突变检测分析其对乳腺癌肿瘤负荷、疗效和预后的监测作用。结果提示,在早期乳腺癌中,乳腺癌分子分型和肿瘤指标Ki-67与CTC标本中TP53(p.R248Q)基因突变相关,腋窝淋巴结转移可能与TP53(p.R175H)基因突变有关,这为早期乳腺癌治疗方案的选择和预后判断提供了新的依据。尽管本实验中,CTC和TP53/PI3K突变情况与早期乳腺癌的大部分临床病理特征无明显相关,但根据既往文献[19, 25-27]报道,了解掌握TP53/PI3K基因突变情况有助于针对乳腺癌患者开展个性化用药、疗效监测和预后判断。另外,本研究首次采用尿液作为早期乳腺癌检测标本,扩展了乳腺癌检测有效生物标志的来源,且本研究发现三类标本的基因突变检测结果无明显差异,提示可以根据临床需要获取合适的检测标本。由于CTC汇聚了多种肿瘤疗效和预后监测的生物指标,因此检测CTC突变基因的效能更强,更有可能在早期肿瘤中发现阳性结果。未来建立个体化的多生物指标联合分析方法可能成为一种更有效地发现早期肿瘤进展和预后的方法。
[ 1 ] DAWSON S J, TSUI D W, MURTAZA M, et al. Analysis of circulating tumor DNA to monitor metastatic breast cancer[J]. N Engl J Med, 2013,368(13):1199-1209.
[ 2 ] CRISTOFANILLI M, BUDD G T, ELLIS M J, et al. Circulating tumor cells, disease progression, and survival in metastatic breast cancer[J]. N Engl J Med, 2004,351(8):781-791.
[ 3 ] WAN J C M, MASSIE C, GARCIA-CORBACHO J, et al. Liquid biopsies come of age: towards implementation of circulating tumour DNA[J]. Nat Rev Cancer, 2017,17(4):223-238.
[ 4 ] LEROY B, ANDERSON M, SOUSSI T. TP53 mutations in human cancer: database reassessment and prospects for the next decade[J]. Hum Mutat, 2014,35(6):672-688.
[ 5 ] MELHEM-BERTRANDT A, BOJADZIEVA J, READY K J, et al. Early onset HER2-positive breast cancer is associated with germline TP53 mutations[J]. Cancer, 2012,118(4):908-913.
[ 6 ] IMAI H, KATO S, SAKAMOTO Y, et al. High throughput RNAi screening identifies ID1 as a synthetic sick/lethal gene interacting with the common TP53 mutation R175H[J]. Oncol Rep, 2014,31(3):1043-1050.
[ 7 ] SAMASSEKOU O, BASTIEN N, LICHTENSZTEJN D, et al. Different TP53 mutations are associated with specific chromosomal rearrangements, telomere length changes, and remodeling of the nuclear architecture of telomeres[J]. Genes Chromosomes Cancer, 2014,53(11):934-950.
[ 8 ] BASELGA J. Targeting the phosphoinositide-3 (PI3) kinase pathway in breast cancer[J]. Oncologist, 2011,16 Suppl 1:12-19.
[ 9 ] CHAUSSADE C, CHO K, MAWSON C, et al. Functional differences between two classes of oncogenic mutation in the PIK3CA gene[J]. Biochem Biophys Res Commun, 2009,381(4):577-581.
[10] LI J, YANG L, GAUR S, et al. Mutants TP53 p.R273H and p.R273C but not p.R273G enhance cancer cell malignancy[J]. Hum Mutat, 2014,35(5):575-584.
[11] RIVA F, BIDARD F C, HOUY A, et al. Patient-specific circulating tumor DNA detection during neoadjuvant chemotherapy in triple-negative breast cancer[J]. Clin Chem, 2017,63(3):691-699.
[12] VAN GINKEL J H, DE LENG W W, DE BREE R, et al. Targeted sequencing reveals TP53 as a potential diagnostic biomarker in the post-treatment surveillance of head and neck cancer[J]. Oncotarget, 2016,7(38):61575-61586.
[13] MOCHIZUKI H, SHAPIRO S G, BREEN M. Detection of BRAF mutation in urine DNA as a molecular diagnostic for canine urothelial and prostatic carcinoma[J]. PLoS One, 2015,10(12):e0144170.
[14] CHRISTENSEN E, BIRKENKAMP-DEMTRÖDER K, NORDENTOFT I, et al. Liquid biopsy analysis of FGFR3 and PIK3CA hotspot mutations for disease surveillance in bladder cancer[J]. Eur Urol, 2017,71(6):961-969.
[15] CHEN S, ZHAO J, CUI L, et al. Urinary circulating DNA detection for dynamic tracking of EGFR mutations for NSCLC patients treated with EGFR-TKIs[J]. Clin Transl Oncol, 2017,19(3):332-340.
[16] SHENG Y, WANG T, LI H, et al. Comparison of analytic performances of Cellsearch and iFISH approach in detecting circulating tumor cells[J].Oncotarget,2017,8(5):8801-8806.
[17] REID A L, FREEMAN J B, MILLWARD M, et al. Detection of BRAF-V600E and V600K in melanoma circulating tumour cells by droplet digital PCR[J].Clin Biochem, 2015, 48(15):999-1002.
[18] PARKINSON C A, GALE D, PISKORZ A M, et al. Exploratory analysis of TP53 mutations in circulating tumour DNA as biomarkers of treatment response for patients with relapsed high-grade serous ovarian carcinoma: a retrospective study[J]. PLoS Med, 2016,13(12):e1002198.
[19] TAKESHITA T, YAMAMOTO Y, YAMAMOTO-IBUSUKI M, et al. Prognostic role of PIK3CA mutations of cell-free DNA in early-stage triple negative breast cancer[J]. Cancer Sci, 2015,106(11):1582-1589.
[20] YU M, BARDIA A, WITTNER B S, et al. Circulating breast tumor cells exhibit dynamic changes in epithelial and mesenchymal composition[J]. Science, 2013,339(6119):580-584.
[21] MOSTERT B, SIEUWERTS A M, KRAAN J, et al. Gene expression profiles in circulating tumor cells to predict prognosis in metastatic breast cancer patients[J]. Ann Oncol, 2015,26(3):510-516.
[22] GIORDANO A, EGLESTON B L, HAJAGE D, et al. Establishment and validation of circulating tumor cell-based prognostic nomograms in first-line metastatic breast cancer patients[J]. Clin Cancer Res, 2013,19(6):1596-1602.
[23] XU L, JIA S, LI H, et al. Characterization of circulating tumor cells in newly diagnosed breast cancer[J]. Oncol Lett, 2018,15(2):2522-2528.
[24] ZARDAVAS D, TE MARVELDE L, MILNE R L, et al. Tumor PIK3CA genotype and prognosis in early-stage breast cancer: a pooled analysis of individual patient data[J]. J Clin Oncol, 2018:JCO2017748301.
[25] KALINSKY K, JACKS L M, HEGUY A, et al. PIK3CA mutation associates with improved outcome in breast cancer[J]. Clin Cancer Res, 2009,15(16):5049-5059.
[26] DOBES P, PODHOREC J, COUFAL O, et al. Influence of mutation type on prognostic and predictive values of TP53 status in primary breast cancer patients[J]. Oncol Rep, 2014,32(4):1695-1702.
[27] FERNANDEZ-CUESTA L, OAKMAN C, FALAGAN-LOTSCH P, et al. Prognostic and predictive value of TP53 mutations in node-positive breast cancer patients treated with anthracycline- or anthracycline/taxane-based adjuvant therapy: results from the BIG 02-98 phase Ⅲ trial[J]. Breast Cancer Res, 2012,14(3):R70.
[28] CURTIS C, SHAH S P, CHIN S F, et al. The genomic and transcriptomic architecture of 2,000 breast tumours reveals novel subgroups[J]. Nature, 2012,486(7403):346-352.
