鼠伤寒沙门菌质粒毒力基因对耐药表型的影响
2018-04-02朱春晖王文杏陈庆法吴春雪刘晓艳赖华毅余晓君
朱春晖,王文杏,陈 强,陈庆法,吴春雪,刘晓艳,赖华毅,余晓君
1)江西省儿童医院感染科 南昌 330006 2)江西省儿童医院内科 南昌 330006 3)江西省儿童医院检验科 南昌 330006
沙门菌是一组寄生于人类和动物肠道内生化反应和抗原构造相似的革兰阴性杆菌,除可以引起伤寒、副伤寒外,也是人类细菌性食物中毒、腹泻、菌血症和败血症的主要病原体。菌血症和败血症严重威胁着人类健康,在免疫功能未发育完善的儿童中病死率极高。据不完全统计,沙门菌每年造成全球约750万儿童感染病例,在非洲地区沙门菌感染在儿童中的死亡率甚至高达24%[1]。本课题组前期研究[2]结果表明,江西地区腹泻儿童大便中沙门菌分离率高,其中鼠伤寒沙门菌为主要血清型,耐药现象严重。2013世界经济论坛全球风险报告将抗生素耐药细菌列为人类健康最大危害之一。因此,沙门菌耐药机制的研究显得尤为重要。据此,本课题组构建稳定的沙门菌表达载体,并在前期使用同源重组法制备鼠伤寒沙门菌质粒毒力基因(Salmonellaplasmid virulence genes,spv)基因缺陷株[3-5]基础上构建鼠伤寒沙门菌spv回补株,分析菌株间耐药表型的差异,并探讨其可能机制,为逆转鼠伤寒沙门菌耐药提供新的分子靶标。
1 材料与方法
1.1质粒、菌株、酶类和主要试剂野生型鼠伤寒沙门菌株STM.211为前期从临床腹泻患者粪便中分离并由江西省儿童医院微生物室保存的临床分离株(药敏试验提示对阿莫西林克拉维酸钾、头孢噻肟、头孢他啶、头孢吡肟、头孢唑啉、庆大霉素、环丙沙星耐药);spvB基因缺陷株STM.211-△-spvB、spvC基因缺陷株STM.211-△-spvC和spvB及spvC基因共同缺陷株STM.211-△-spvBC(课题组前期研究构建,并保存);大肠埃希菌Top10购自天根生化科技(北京)有限公司;pMD19-T克隆载体购自大连TaKaRa公司;pACYC184克隆载体购自南通麦杰生物科技有限公司;pAZ44-H质粒由华中农业大学农业微生物学国家重点实验室郭爱珍教授惠赠。限制性内切酶SalⅠ、SphⅠ、T4 DNA连接酶、EcoRⅠ、EcoRⅤ、DMEM培养基及胎牛血清购自Hyclone公司,质粒提取试剂盒、DNA 胶 回收试剂盒、RT-PCR试剂盒购自Promega 公司,细菌基因组 DNA 提取试剂盒(离心柱)购自天根生化科技(北京)有限公司,DNA 聚合酶试剂盒购自Qiagen 公司,脂质体Lipofectamine及总RNA提取试剂购自Invitrogen公司,PCR引物由TaKaRa合成,DNA 分子质量标准Marker 购自生工生物工程(上海)股份有限公司。
1.2eGFP基因的合成及克隆根据GenBank中的增强绿色荧光蛋白(enhanced green fluroscent protein,eGFP)基因序列,在上游增加Trc Promoter启动子序列,下游增加rrnB ribosomal终止子序列,位点上游增加XhoⅠ和XbaⅠ酶切位点,下游增加NheⅠ酶切位点。设计引物序列,由生工生物工程(上海)股份有限公司完成合成。以设计合成的引物E1至E26各10 μL混合物为模板,进行PCR预扩增。取PCR产物为模板,用E1和E26进行PCR扩增,获得含启动子和终止子的eGFP基因,割胶纯化产物,用XhoⅠ、NheⅠ和SalⅠ、XbaⅠ对PCR产物和pACYC184载体进行双酶切,电泳分离目的基因,对目的条带割胶纯化后用T4连接酶进行定向连接以构建pACYC184-eGFP,转至Top10感受态细菌,氯霉素筛选阳性克隆,PCR检测并测序鉴定。
1.3hok/sok-parDE(HOK)基因的调取及克隆以pAZ44-H质粒为模板,用HOK正反义引物进行PCR扩增获取aphA-hok-parDE基因,割胶纯化产物后用NheⅠ和XbaⅠ对产物和pACYC184-eGFP载体进行酶切,电泳分离目的基因后,割胶纯化,用T4连接酶进行定向连接构建pACYC184-eGFP-HOK载体,转化至E.coli Top10感受态细菌,氯霉素、卡那霉素双抗筛选阳性克隆,PCR检测并测序鉴定证实载体构建的正确性。
1.4spvB及spvC基因的调取及克隆根据GenBank中公布的鼠伤寒沙门菌spvB、spvC基因的上、下游核苷酸序列,以沙门菌基因组DNA为模板进行PCR扩增,获取spvB、spvC基因,割胶纯化产物,分别克隆至pMD19-T获得pMD-spvB和 pMD-spvC质粒,转至Top10感受态细菌,筛选阳性克隆后用BglⅡ、NotⅠ和SalⅠ、NotⅠ进行双酶切验证并测序鉴定。
使用限制性内切酶对质粒pMD-spvB、pMD-spvC及pACYC184-eGFP-HOK进行酶切,电泳分离目的基因后进行割胶纯化,用T4连接酶进行定向连接,构建pACYC184-spvB-eGFP-HOK和pACYC184-spvC-eGFP-HOK载体,转至Top10感受态细菌,氯霉素、卡那霉素双抗筛选阳性克隆,PCR检测并测序鉴定。
1.5spvB及spvC回补株STM.211-c-spvBC的构建及鉴定将pACYC184-eGFP、pACYC184-spvB-eGFP-HOK、pACYC184-spvC-eGFP-HOK质粒DNA与电感受态STM.211-△-spvBC轻吸混匀制备混合物,加入预冷的电击杯中,排除气泡,使用2 100 V电击混合物,然后迅速加入无抗LB培养液中,吹吸混匀并转移到1.5 mL离心管中,取重悬菌液均匀涂布于含50 mg/L卡那霉素和氯霉素的LB固体培养基平板上,倒置平板,37 ℃ 20 h。重组菌经PCR测序鉴定后命名为STM.211-c-spvBC、STM.211-c-spvBC-HOK。
1.6质粒稳定性评价分别挑取STM.211-c-spvBC、STM.211-c-spvBC-HOK单菌落接种于含有100 mg/L卡那霉素的LB液体培养基,37 ℃ 200 r/ min振摇培养,使菌液的A600 nm达到0.1~0 .3,取10 μL菌液转接到10 mL无抗性LB的液体培养基中,37 ℃振荡培养。每12 h传代1次,连续培养6 d,转接的同时将菌液进行倍比稀释。取适当稀释度的菌液分别涂布在LB固体平板和卡那霉素/氯霉素双抗琼脂平板后统计菌落数,每个平板涂10 μL菌液,每个稀释度重复3次,计数LB平板菌落数,计数双抗平板带质粒的菌落数,二者比值为抗性菌落百分数,用以评价质粒稳定性。实验重复3 次。采用标准碱裂解法抽提质粒确定质粒存在。
1.7药物敏感性实验根据美国临床实验室标准委员会(NCCLS)标准应用琼脂平板稀释法检测菌株(STM.211、STM.211-△-spvB、STM.211-△-spvC、STM.211-△-spvBC、STM.211-c-spvBC)对临床常用抗生素的最小抑菌浓度(minimal inhibit concentration,MIC),参照NCCLS2004年标准判断结果。质控菌为大肠埃希菌ATCC25922。
1.8外排泵相关蛋白AcrA和AcrB的Westernblot检测待毒力基因spv缺陷株、毒力基因spv回补株及野生菌株的菌液生长至A600 nm=0.6~0.8,离心收集细胞,沉淀用离解液[20 nmol/L Tri-盐酸(pH=8.0),100 mmol/L氯化钠,30%甘油,AEBSF, 1 mmol/L依地酸(EDTA,pH=8.0)]悬浮,超声破细胞。用Bradford法测定总蛋白浓度,小牛血清白蛋白定标,行SDS-PAGE,将蛋白在25 V 电压下电转移30 min(检测AcrB时,采用电压20 V,20 min),然后移至硝酸纤维素膜上,再先后与抗AcrA抗体或抗AcrB抗体(1200)及HRP标记的抗兔 IgG 二抗杂交,化学发光试剂显色。
2 结果
2.1eGFP基因的合成、克隆及鉴定含启动子及终止子的eGFP基因PCR结果见图1。XhoⅠ及NheⅠ对eGFP基因片段进行双酶切,SalⅠ及XbaⅠ对pACYC184载体进行双酶切。切胶纯化回收后T4连接酶链接eGFP片段和pACYC184线性载体合成重组质粒pACYC184-eGFP,转化入Top10感受态大肠埃希菌。挑斑于含有35 mg/L氯霉素的LB液体培养基中摇菌培养,设计测序引物,PCR扩增产物测序鉴定,结果正确。
2.2hok/sok-parDE(HOK)基因的调取及克隆以pAZ44-H质粒为模板,PCR扩增获得HOK基因片段(图2),重组入pACYC184-eGFP质粒并转化入感受态大肠埃希菌。挑取阳性克隆进行抗性培养并PCR扩增,产物送测序,结果正确。
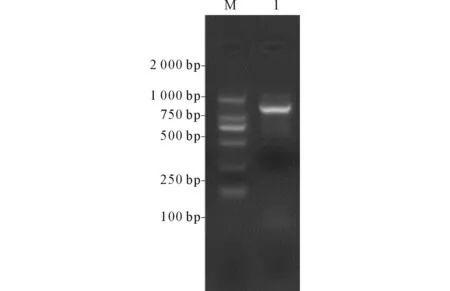
M:Marker;1:多重PCR得到的eGFP基因片段图1 eGFP基因(含启动子终止子)PCR扩增结果
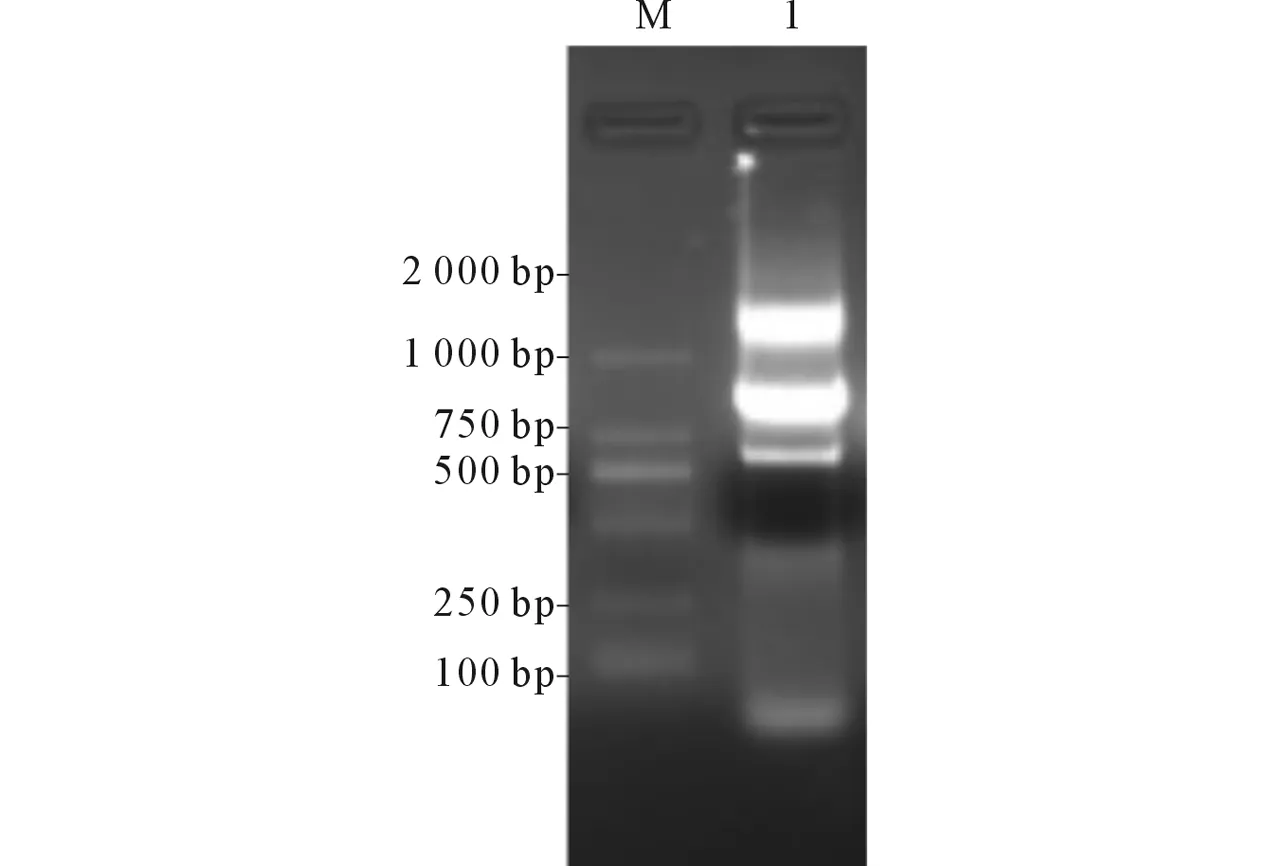
M:Marker;1:HOK基因片段图2 HOK基因PCR扩增结果
2.3spvB及spvC基因的调取及克隆以沙门菌基因组DNA为模板,PCR扩增获得spvB和spvC基因片段(图3)。切胶纯化回收PCR产物。T4连接酶链接spvB、spvC与pMD19-T载体得到pMD-spvB及pMD-spvC,分别转化入感受态大肠埃希菌,挑斑于含氨苄青霉素抗性的LB培养基中培养。挑取阳性克隆提取重组质粒,NotⅠ单酶切产生一条环状条带,一条略大些的线性DNA,NotⅠ/BglⅡ双酶切pMD-spvB产生大约1.8 kb的条带,SalⅠ/NotⅠ双酶切pMD-spvC产生大约0.72 kb的条带(见图4)。切胶纯化回收spvB、spvC基因片段及质粒进行测序鉴定,鉴定正确后,将回收的spvB、spvC基因片段分别和pACYC-eGFP-HOK线性载体重新连接,合成重组质粒后转化入感受态细菌。挑取阳性克隆于含氯霉素抗性的LB培养基摇菌培养,设计测序引物,PCR扩增产物测序鉴定。
2.4平板计数法测量质粒稳定性结果见图5、6。携带质粒的细菌STM.211-c-spvBC培养第2到3代质粒开始丢失,第9到10代完全丢失。而携带质粒STM.211-c-spvBC-eGFP-HOK的细菌均无明显的质粒丢失。实验结果提示,重组质粒pACYC184-spvBC-eGFP-HOK因携带hok/sok-parD/E质粒从而可稳定保留,稳定性高于重组质粒pACYC184-spvBC-eGFP。
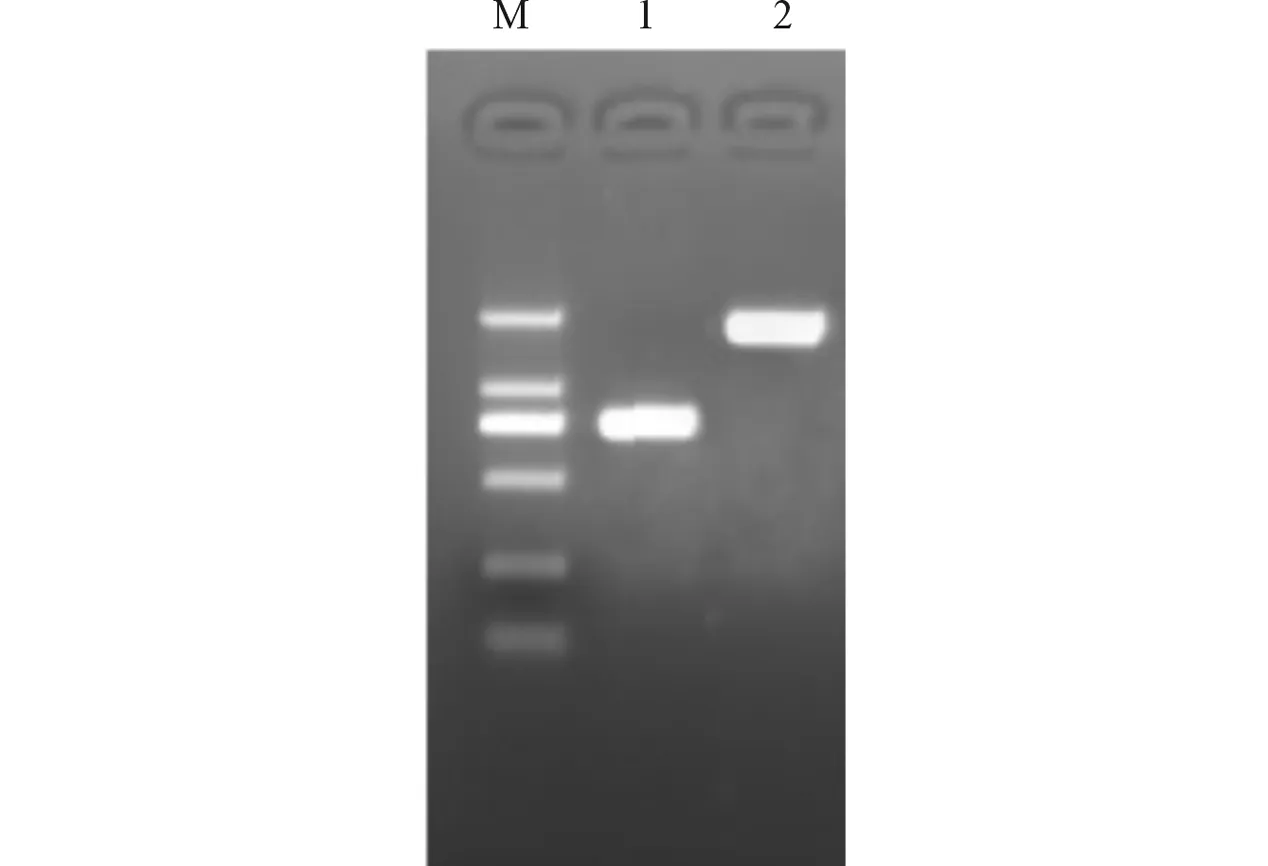
M:Marker;1:spvC基因片段;2:spvB基因片段图3 spvB、spvC基因PCR扩增结果
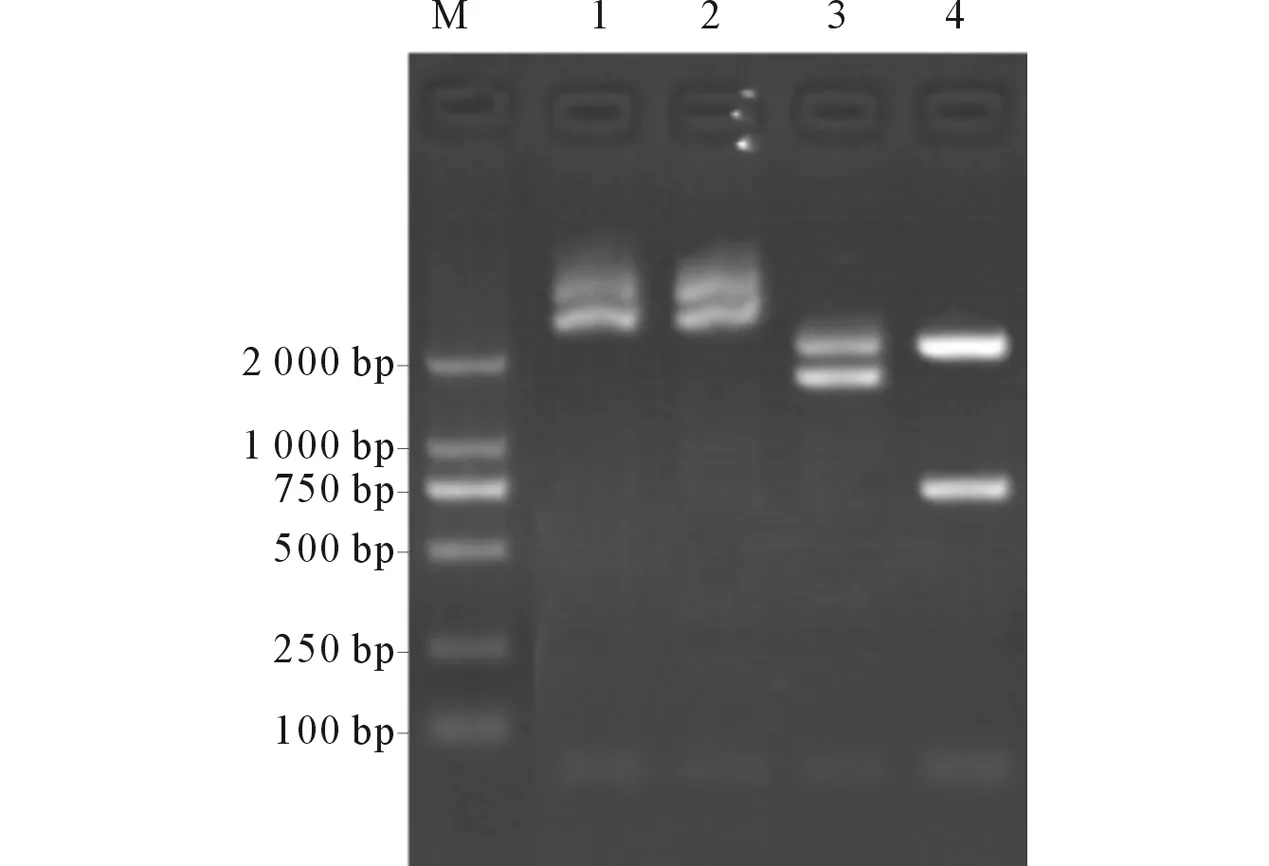
M:Marker;1:单酶切pMD-spvB产物;2:单酶切pMD-spvC产物;3:双酶切pMD-spvB产物;4: 双酶切pMD-spvC产物图4 重组质粒pMD-spvB及pMD-spvC的酶切结果
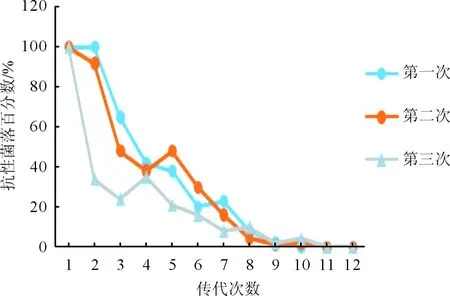
图5 STM.211-c-spvB培养6 d抗性菌落百分比
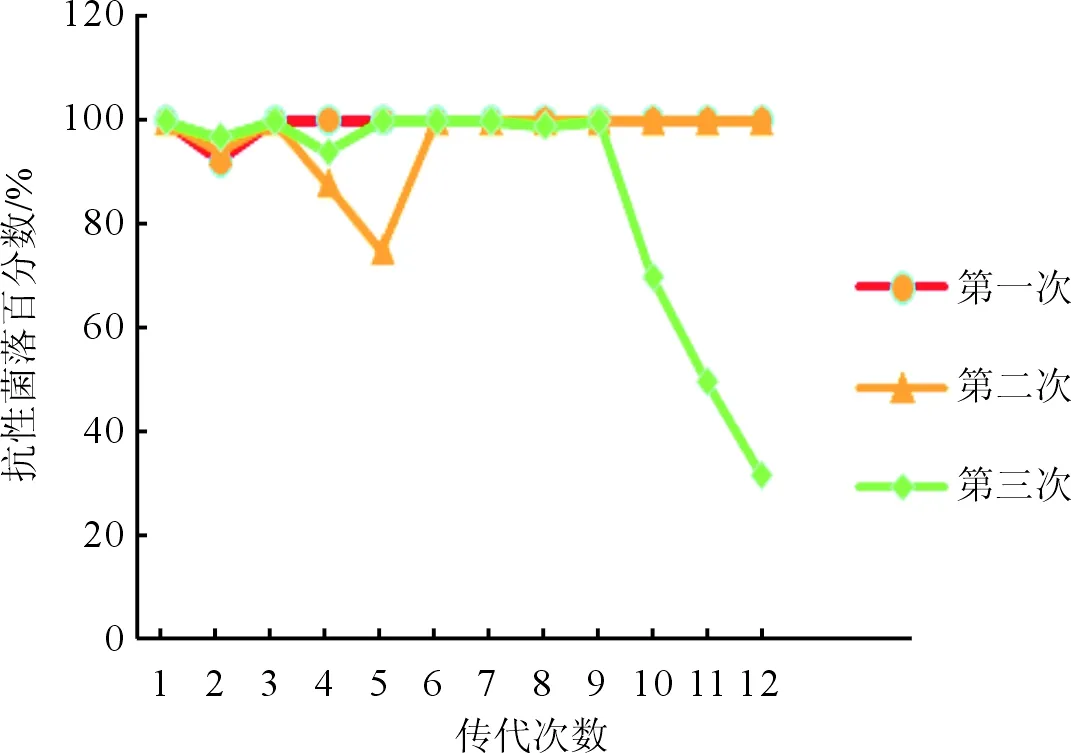
图6 STM.211-c-spvBC-eGFP-HOK培养6 d抗性菌落百分比
2.5药物敏感性检测结果见表1。STM.211-△-spvBC对阿莫西林克拉维酸钾、头孢噻肟、头孢他啶、头孢吡肟、头孢唑啉、庆大霉素及环丙沙星敏感性优于STM.211。所有菌株均对亚胺培南敏感。
2.6外排泵相关蛋白AcrA、AcrB的表达见图7和表2。STM.211-△-spvBC的AcrA蛋白水平低于STM.211及STM.211-c-spvBC(P<0.05);STM.211-△-spvB、STM.211-△-spvC与STM.211-△-spvBC间差异无统计学意义(P>0.05)。STM.211、STM.211-△-spvB、STM.211-△-spvC、STM.211-△-spvBC、STM.211-c-spvBC间AcrB的表达差异无统计学意义(P>0.05)。
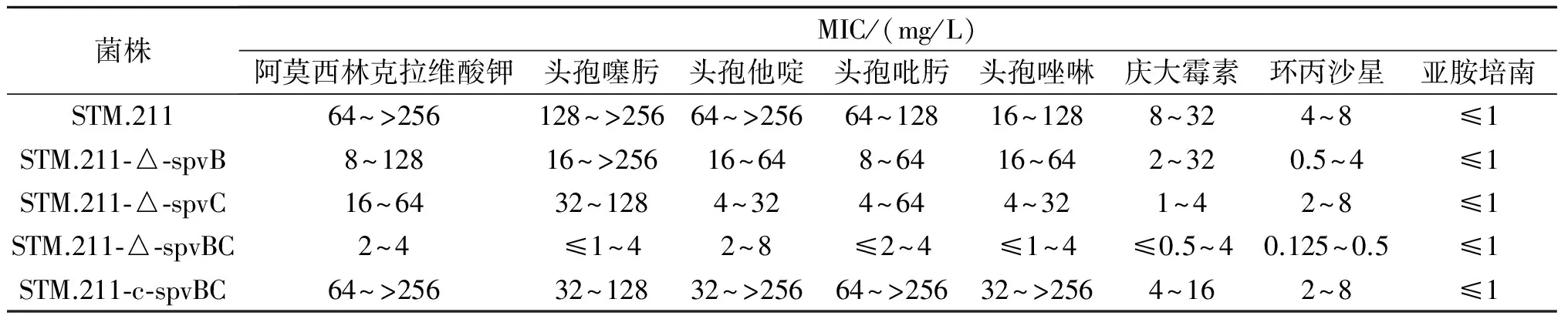
表1 不同抗生素对菌株的MIC值
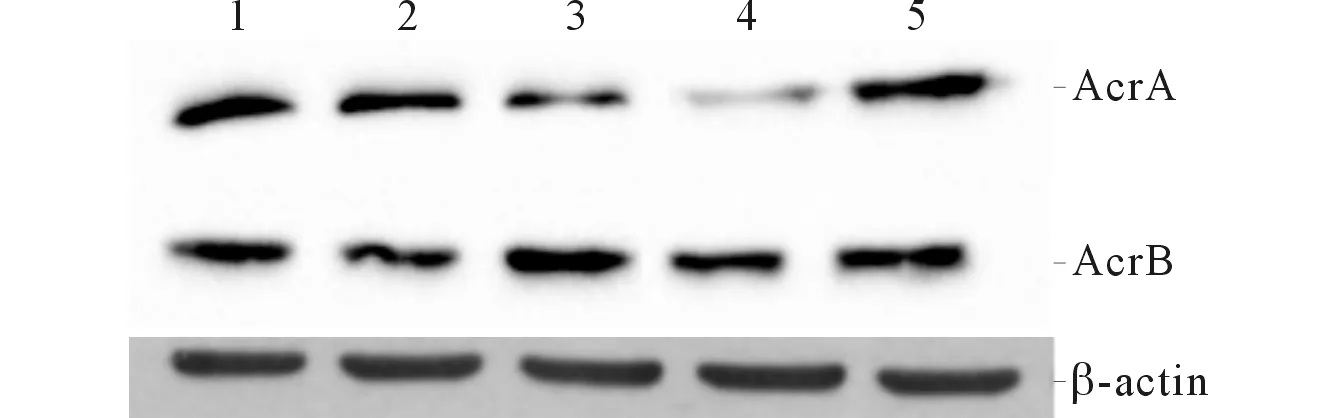
1:STM.211;2:STM.211-△-spvB;3:STM.211-△-spvC;4:STM.211-△-spvBC;5:STM.211-c-spvBC图7 菌株外排泵相关蛋白AcrA、AcrB的表达
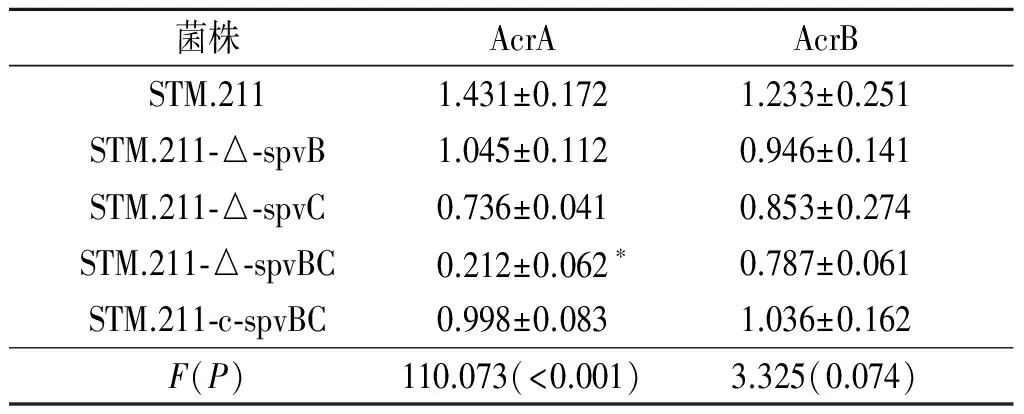
表2 菌株外排泵相关蛋白AcrA、AcrB表达量的比较(n=6)
*:与STM.211、STM.211-c-spvBC相比,P<0.05
3 讨论
沙门菌作为一种革兰阴性杆菌,感染人类和动物后常常引起菌血症、伤寒热和肠炎等临床表现,据报道,欧洲每年至少约350 000例感染患者[6],而在美国每年约200 000例,而实际上,感染的真正人群则高出这个数字3倍[7]。沙门菌的耐药现象日益严峻,增加了临床治疗的难度。既往研究集中在研究细菌耐药机制上,近年来,在人类微生物群日益受到关注的大背景下,科学家们重拾用噬菌体来治疗细菌感染的兴趣,但通过改变细菌毒力来逆转细菌耐药表型目前尚未见报道。课题组前期以从腹泻儿童粪便中分离的耐药鼠伤寒沙门菌为研究对象,利用自杀质粒构建沙门菌spvBC缺陷菌株并测序鉴定,体内外实验表明spvBC缺陷后鼠伤寒沙门菌毒力下降。本研究中作者使用电转化技术构建spvBC回补株并比较菌株间耐药性差异及耐药相关蛋白表达的异同。
在重组细菌中,高拷贝质粒因为同源重组后复制扩增导致“二聚体灾难”形成质粒二聚体,代谢符合增加导致稳定性下降[8]。低拷贝质粒能稳定表达目的基因组,但拷贝数低,分离过程不能稳定持有。启动子转录翻译过程中启动子功能可能受到抑制[9],在转录下游增加转录终止子可以增加mRNA的稳定性,因此,本课题组采用Trp及Lac启动子杂合拼接成的Trc启动子和高效表达目的基因所必需的rrnB ribosomal 终止子人工合成eGFP以获得更高转录效率。构建重组质粒过程中,往往因编码基因增加宿主细胞的代谢负荷导致质粒易于丢失稳定性差,目前可使用以下3种途径来增加质粒稳定性:以抗生素作为选择压力的质粒载体系统(人、动物体内无法实现且有导致抗药菌扩散的风险);染色体-质粒平衡致死系统(对培养基、细菌种属、基因型有所要求而限制其使用);质粒解离后致死系统(post segregational killing system,PSK )。本实验使用的hok/sok-parDE质粒稳定系统即是PSK中的经典之作,hok/sok和parD/E分属两个独立的平衡致死系统,hok基因和parE基因分别编码两种毒力蛋白,sok和parD能拮抗他们的毒性作用。在含质粒的细胞中,sok RNA通过与hok mRNA结合抑制hok mRNA的表达,parD能抑制parE的转录、翻译及其毒力效应,细胞维持了稳态。当细胞质粒丢失,sok RNA及parD逐渐降解,Hok和ParE在细胞中聚集,产生毒力蛋白使细胞裂解死亡。hok/sok和parDE组合系统比hok/sok或parDE单一系统具有更强的质粒稳定作用[10]。本研究使用hok/sok-parDE质粒稳定系统将融合基因插入编码eGFP蛋白的质粒中得到重组质粒pACYC184-eGFP-HOK,然后向预留的酶切位点中插入目的基因spvBC,重组质粒pACYC184-spvB-eGFP-HOK 和pACYC184-spvC-eGFP-HOK成功构建后将其电转化入感受态细菌构建回补株,对重组质粒稳定性研究表明,含hok/sok-parDE质粒的菌株所携带的质粒稳定持有,质粒无明显丢失,而不含该系统的质粒在传2至3代后出现质粒丢失的现象。经基因测序证实鼠伤寒沙门菌荧光表达载体pACYC184-eGFP-HOK、pACYC184-spvB-eGFP-HOK、pACYC184-spvC-eGFP-HOK及spvBC缺陷株的回补株均成功构建。
解决重组细菌的技术难题后,构建菌株及野生菌株的药敏试验显示,鼠伤寒沙门菌spvBC缺陷后菌株对β-内酰胺类、氟喹诺酮类、氨基糖甙类的代表药物敏感性均明显增强。既往研究[11]发现宿主抗性因子Nramp1能上调致病岛-2毒力基因的表达,而AcrD的灭活导致与基础代谢、毒力和应激应答在内的基本过程相关的403种基因表达的改变[12]:细菌的耐药与毒力间并非“各行其道”而是相互影响,细菌的耐药机制中主要的外排泵除能将抗生素泵出外也能将机体遭受细菌感染后分泌的胆汁、激素及宿主防御因子泵出以利于细菌对宿主细胞的侵袭[13],故宿主抗性因子及外排泵的存在有助于细菌在宿主体内的存活、侵袭、运动等生物特性。本研究结果显示:spv的缺陷能改变鼠伤寒沙门菌的耐药表型,非伤寒沙门菌中普遍存在质粒spv,大小约50~90 kb,与细菌黏附、运动、血清抗性及定居有关[14]。黄瑞等[15]研究表明,分离自伤寒沙门菌的耐药质粒pRsT98上的spvR和spvB与鼠伤寒沙门菌的spv有大于99%的同源性。spv作为细菌某些耐药机制的上游发挥作用抑或spv与耐药相关因子有相同的作用位点目前尚未明了。本研究随后对导致细菌耐药外排泵中研究得最多的AcrA/B蛋白进行检测,结果发现spvBC基因缺陷菌株的AcrA蛋白水平低于野生菌株及spvBC缺陷回补株,这表明spvBC的缺陷能下调AcrA蛋白的表达,而spvBC的回补能解除对AcrA蛋白的表达的抑制。
众所周知,作为一种典型的外流泵系统,AcrA/B-TolC外排泵系统能将代谢产物或抗生素等有毒物质泵出细胞外并抑制孔蛋白表达,使抗生素无法进入细菌内发挥作用以应对环境压力适者生存[16]。spv作为一种毒力基因是通过何种途径来影响AcrA蛋白的表达?与此同时,spv表达的改变在影响AcrA蛋白的表达的同时AcrB蛋白的表达却未受到影响等均有待进一步的研究。下一步课题组将检测AcrAB的外流通道外膜蛋白TolC及调控基因acrR、ramA、marA等的表达情况,以进一步探讨毒力基因spv改变耐药表型的机制,为将spv作为逆转鼠伤寒沙门菌耐药的分子靶标打下实验基础。
[1] BRENT AJ,OUNDO JO,MWANGI I,et al.Salmonellabacteremia in Kenyan children[J].Pediatr Infect Dis,2006,25(3):230
[2] 陈强,余晓君,李俏俏,等.引起儿童腹泻的沙门菌属临床分离株的耐药特点及分子流行病学研究[J].中华检验医学杂志,2011,34(3):249
[3] 吴春雪,陈强,李红,等.鼠伤寒沙门菌spvB基因缺陷变异株的制备及其抗酸能力检测[J].华中科技大学学报(医学版),2014,43(4):399
[4] 陈强,吴春雪,余晓君,等.鼠伤寒沙门菌spvC基因缺陷变异株的制备[J].江苏大学学报(医学版),2014,24(3):235
[5] 刘晓艳,陈强,李红,等.spvB/spvC基因对沙门菌毒力及宿主免疫功能的影响[J].南方医科大学学报,2015,35(11):1649
[6] FISHER IST.International trends inSalmonellaserotypes 1998-2003: a surveillance report from the enter-net internationalsurveillance network[J].Euro Surveill 2004,9(11):9
[7] HARVEY RR,FRIEDMAN CR,CRIM SM,et al.Epidemiology ofSalmonellaenterica serotype dublin infections among Humans, United States, 1968-2013[J].Emerg Infect Dis,2017,23(9):1493
[8] FIELD CM,SUMMERS DK.Multicopy plasmid stability:revisiting the dimer catastrophe[J].J Theor Biol,2011,291:119
[9] LOBATO-MRQUEZ D,MOLINA-GARCA L,MORENO-CRDOBA I,et al.Stabilization of the virulence plasmid pSLT ofSalmonellatyphimuriumby three maintenance systems and its evaluation by using a new stability test[J].Front Mol Biosci,2016,17(3):66
[10]GONG CC,KLUMPP S.MODELING sRNA-regulated plasmid maintenance[J].PLoS One,2017,12(1):e0169703
[11]ZAHARIK ML,VALLANCE BA,PUENTE JL,et al.Host-pathogen interactions:host resistance factor Nramp1 up-regulates the expression ofSalmonellapathogenicity island-2 virulence genes[J].Proc Natl Acad Sci USA,2002,99(24):15705
[12]BUCKNER MM,BLAIR JM,LA RAGIONE RM,et al.Beyond antimicrobial resistance:evidence for a distinct role of the AcrD efflux pump inSalmonellabiology[J].MBio,2016,7(6):16
[13]CHEN Y,HU DX,ZHANG QJ,et al.Efflux pump overexpression contributes to tigecycline heteroresistance inSalmonellaenterica serovar Typhimurium[J].Front Cell Infect Microbiol,2017,7:37
[14]WU SY,WANG LD,LI JL,et al.Salmonellaspv locus suppresses host innate immune responses to bacterial infection[J].Fish Shellfish Immunol,2016,58:387
[15]黄瑞,吴淑燕,张学光.伤寒杆菌耐药质粒pRsags毒力基因的研究[J].中华微生物学和免疫学杂志,2003,23(5):393
[16]WESTON N,SHARMA P,RICCI V,et al.Regulation of the AcrAB-TolC efflux pump inEnterobacteriaceae[J].Res Microbiol,2017.doi:10.1016/j.resmic.2017.10.005
