牛副流感3型NP融合蛋白原核表达及间接ELISA检测方法的建立
2018-03-29王苗苗张玲玲冉旭华闻晓波
王苗苗,张玲玲,张 峣,冉旭华,闻晓波
(黑龙江八一农垦大学动物科技学院,黑龙江 大庆 163319)
牛副流感病毒3型(Bovine parainfluenza virus type 3,BPIV3)为副黏病毒科、呼吸道病毒属成员,与牛病毒性腹泻病毒(Bovine viral diarrhea virus,BVDV)、牛传染性鼻气管炎病毒(Infectious bovine rhinotracheitis virus,IBRV)、牛呼吸道合胞体病毒(Bovine respiratory syncytial virus,BRSV)共同构成了引发牛呼吸道疾病综合征(Bovine respiratory disease complex,BRDC)的主要病毒性病原,常导致持续性感染,是目前引发反刍动物急性呼吸道疾病的主要病原,也是引起世界范围内的舍饲牛发病及死亡的主要原因之一,对世界养牛业造成的经济损失较大[1-4]。自1959年Reisinger等首次从犊牛肾脏中分离到BPIV3,之后世界上许多国家也相继报道分离出BPIV3,目前该病毒已呈现世界性广泛分布的趋势[5-10]。我国于2008年由刘鹏等在黑龙江省分离出该病毒[11],此后在多地也分离到该病毒[3,12]。经血清学调查表明,养牛业发达的地区感染该病毒情况较重,已在我国广泛性分布[13]。
BPIV3具有核衣壳结构和囊膜[14],与仙台病毒等其他副粘病毒相似。目前为止,已发现BPIV3有A、B、C 3种基因型[15]。编码M蛋白、磷蛋白P、F蛋白、N蛋白、NP蛋白、L蛋白及D、V、C 3种非结构蛋白[14,16]。NP蛋白为BPIV3的核衣壳蛋白,保守性较高[16],因此本研究拟选取NP基因构建融合蛋白表达载体,建立间接ELISA方法来检测BPIV3抗体水平,为牛副流感3型提供高效便捷的检测方法。
1 材料和方法
1.1 质粒、菌种、病毒、血清 原核表达质粒pET-28a、 菌 株 E.coli BL21(DE3)及 E.coli DH5α、BPIV3c NX49分离株、BPIV3阳性血清及阴性血清、BVDV阳性血清、BHV-I阳性血清均由黑龙江八一农垦大学预防实验室保存。
1.2 主要试剂 TRIzol、Ni-NTA Purification System( 美 国 Invitrogen); RevertAid First Strand cDNA Synthesis Kit、蛋白质Marker、限制性内切酶Nde I、BamH I、Sac I、HindⅢ(美国Thermo Fisher Scientific); Wizard®SV Gel and PCR Clean-Up System、GoTaq® Green Master Mix(美国Promega);辣根过氧化物酶(HRP)标记的兔抗牛IgG(Sigma);PrimeSTARTMHS DNA Poymerase及DNA Marker(日本TaKaRa);其他试剂均为国产分析纯。
1.3 抗原表位区域的筛选 利用DNASTAR中Protean程序对NX49株NP基因进行抗原表位分析,所选区域命名为 NP-N(8~156 aa)及 NP-C(368~507 aa)并设计2对特异性引物进行扩增,引物由北京六合华大基因科技股份有限公司合成。引物序列如表1所示。
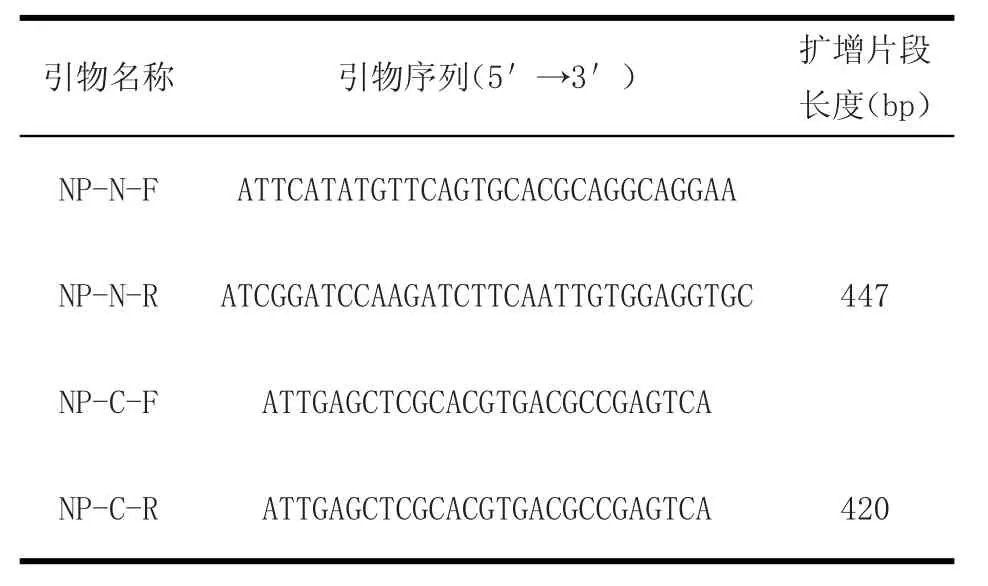
表 1 引物序列Table 1 Sequences of primer
1.4 目的基因的扩增及纯化 参照TRIzol使用说明书提取BPIV3 RNA,参照Thermo反转录试剂盒说明书进行反转录获得cDNA,以cDNA为模板进行PCR扩增,并以ddH2O为阴性对照,反应体系如下:5×PrimeSTAR®Buffer 10 μL,dNTPs 5 μL,Prime-STAR®HS 0.5 μL,上、下游引物各1 μL,cDNA 3 μL,补加ddH2O至50 μL。PCR反应条件:98℃预变性3 min;98℃变性10 s,60℃退火10 s,72℃延伸1 min,共35个循环;72℃最终延伸10 min,PCR产物以1%琼脂糖凝胶进行电泳,并通过Wizard®SV Gel and PCR Clean-Up System试剂盒进行回收纯化。
1.5 重组质粒ppEETT--2288aa--NNPP--NN--CC的构建 将NP-N纯化产物及载体pET-28a分别经NdeI、BamH I双酶切,回收并纯化酶切产物,以T4 DNA连接酶16℃连接12 h,将连接产物转化至感受态E.coli DH5α,于37℃培养12 h,挑取单菌落接种至2 mL含35 μg/mL卡那霉素的LB液体培养基中培养,以NP-N-F/R、NP-C-F/R、T7 Promoter/Terminator为引物对重组菌进行鉴定,以ddH2O为阴性对照。通过碱裂解法提取重组菌质粒,并以NdeI、BamH I双酶切做进一步鉴定。将NP-C纯化产物与重组质粒pET-28a-NP-N分别经SacI、HindⅢ双酶切,以T4 DNA连接酶连接、转化、鉴定方法同上。将鉴定正确的重组菌送往北京六合华大基因科技股份有限公司测序,将测序正确的重组质粒命名为pET-28a-NP-N-C。
1.6 重组蛋白的诱导表达及可溶性分析 将重组质粒pET-28a-NP-N-C及空载体pET-28a分别转化至E.coliBL21(DE3)感受态中,并将重组菌1%转接至含有35 μg/mL的LB培养液中,37℃培养至对数生长期,加入终浓度为1 mM的IPTG,诱导4 h,收集菌体并以PBS洗涤,Bug Baster®Master Mix裂解菌体,分别收集裂解后上清及沉淀,经SDS-PAGE电泳分析重组蛋白的表达形式。
1.7 重组蛋白的纯化及鉴定 收集2 L诱导表达后的重组菌,超声裂解菌体,转化至E.coliBL21(DE3)感受态中收集裂解沉淀,采用Ni-NTA Purification System纯化重组蛋白,具体方法参照说明书,对纯化后的目的蛋白进行复性处理。重组蛋白以SDS-PAGE电泳后,转膜并以脱脂乳封闭,加入BPIV3阳性血清(1:80倍稀释),4℃孵育过夜。加入HRP标记的兔抗牛IgG(1:5 000倍稀释),37℃孵育1 h。加入DAB显色3 min,以ddH2O终止反应。
1.8 间接EELLIISSAA方法的优化 将融合蛋白NP-N-C以CBS倍比稀释至0.5~8 μg/mL,包被ELISA板,100 μL/孔。对BPIV3阴、阳性血清也分别倍比稀释至1:40~1:320,采用棋盘滴定试验,确定最佳抗原包被浓度及血清最佳稀释度。设置4℃包被过夜,37℃包被1 h,37℃包被2 h,37℃包被1 h后4℃过夜,共4组不同的包被条件,确定抗原最佳包被条件。分别以2.5%、5%、10%3个浓度脱脂乳作为封闭液,37℃封闭,封闭时间设置为30、60、90和120 min,对封闭液最佳浓度及时间进行优化。分别以1:3 000、1:5 000、1:7 000 3个不同的稀释度对HRP标记的兔抗牛IgG进行稀释,37℃孵育,孵育时间设置为30、45和60 min,对二抗最佳稀释倍数及孵育时间进行优化。以TMB显色液显色,显色时间分别设置为5、10、15和20 min,对显色时间进行优化。测定阳性血清、阴性血清的OD450并计算P/N值,以P/N值最大且阳性血清OD450≥1.0、阴性血清OD450<0.2作为间接ELISA优化条件的判定标准。
1.9 阴、阳性临界值的确立 随机选取85份BPIV3阳性血清及79份BPIV3阴性血清,以已建立的ELISA方法分别进行检测,测定OD450值。将测得的OD450值进行统计学分析。通过非参数法构建ROC曲线,以Youden指数最大的切点作为阴阳性判断的临界点,并确定该方法的特异性及敏感性。
1.10 重复性试验 取一块同批次包被的酶标板,用其检测4份不同效价的阳性血清和2份阴性血清,每份血清做3个平行重复,以ELISA方法筛选出的最优条件来对稀释后血清进行检测,统计学分析OD450并计算批内重复试验变异系数,统计学分析试验结果。
用不同时间抗原包被的ELISA板,对4份不同效价的阳性血清和2份阴性血清进行3次批间重复检测,以ELISA筛选出的最优条件来对稀释后血清进行检测,统计学分析OD450并计算批间重复试验变异系数,评价该方法的批间重复性。
2 结果
2.1 目的基因NNPP--NN及NNPP--CC的扩增 PCR扩增目的基因NP-N及NP-C,扩增产物经1%琼脂糖凝胶电泳,得到447 bp及420 bp 2条特异性条带,与预期片段大小(如图1)。
2.2 原核表达重组质粒的鉴定 菌液PCR产物经1%琼脂糖凝胶电泳,扩增出片段大小为447 bp(NPN)、420 bp(NP-C)、1 146 bp(NP-N-C)的特异性条带,与预期相符(如图2)。测序结果表明,重组质粒中的目的基因与NX49株基因组序列素编码的核苷酸、氨基酸的同源性均为100%,可用于原核表达。
2.3 重组蛋白的诱导表达形式的鉴定 诱导表达产物裂解后上清及沉淀经Glycine-SDS-PAGE分析,得到大小为42 kDa的条带,与预期大小相符(如图3),并以包涵体的形式表达。同时于约35 kDa处出现一条带,需进一步鉴定。
2.4 重组蛋白的纯化 重组蛋白经Ni-NTA系统纯化后于42 kDa及35 kDa位置均出现条带(如图4)。纯化的重组蛋白浓度约为123 μg/mL。
2.5 重组蛋白的Western BBlloott鉴定 以BPIV3 NX49株为阳性对照,Western Blot结果表明重组蛋白在42 kDa及35 kDa处条带均能与BPIV3阳性血清发生特异性反应,全病毒蛋白对照组也在42 kDa及35 kDa位置处出现了2条特异性条带(如图5),说明表达的目的蛋白以2种形式存在,而且均具有良好的反应活性。
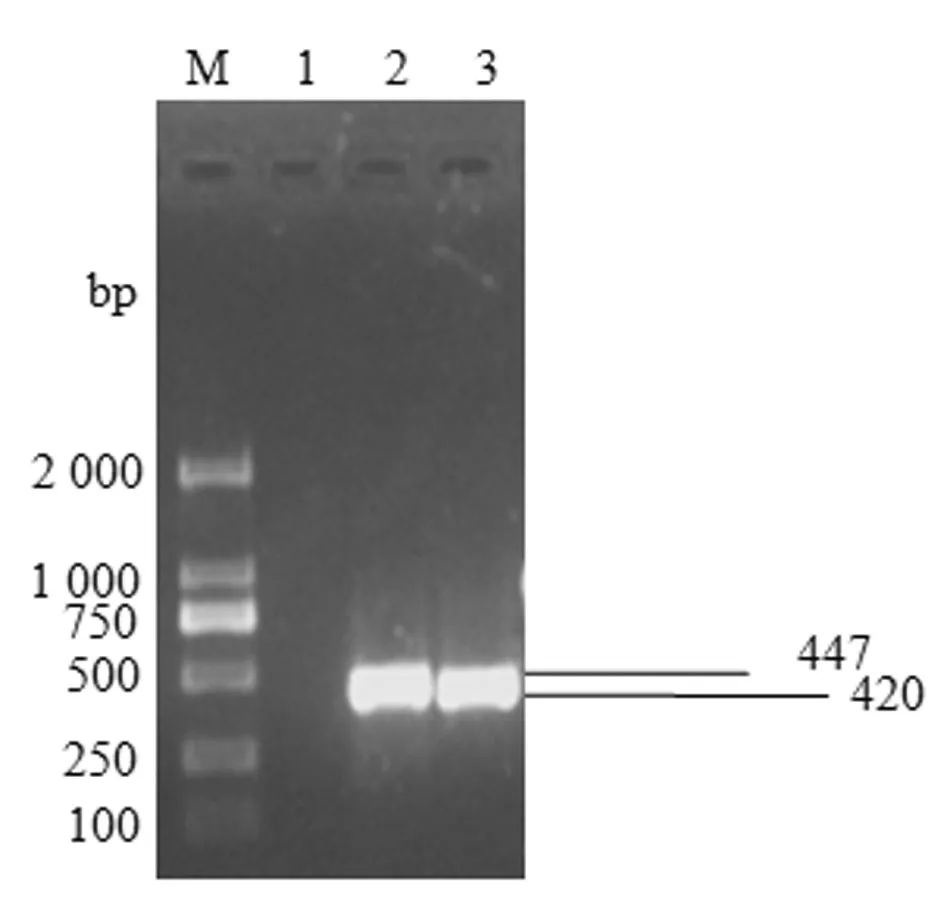
图 1 NP基因片段的RT-PCR扩增Fig.1 Amplifiation ofNPfragment by PCR
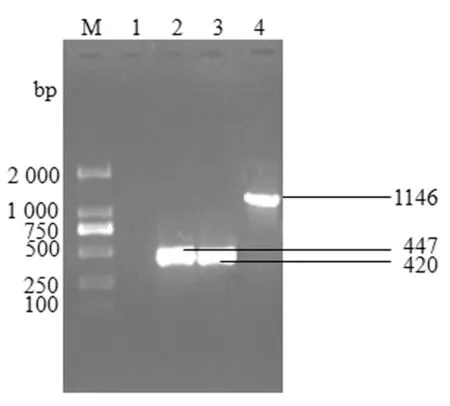
图 2 目的基因的扩增Fig.2 Amplifiation of target genes
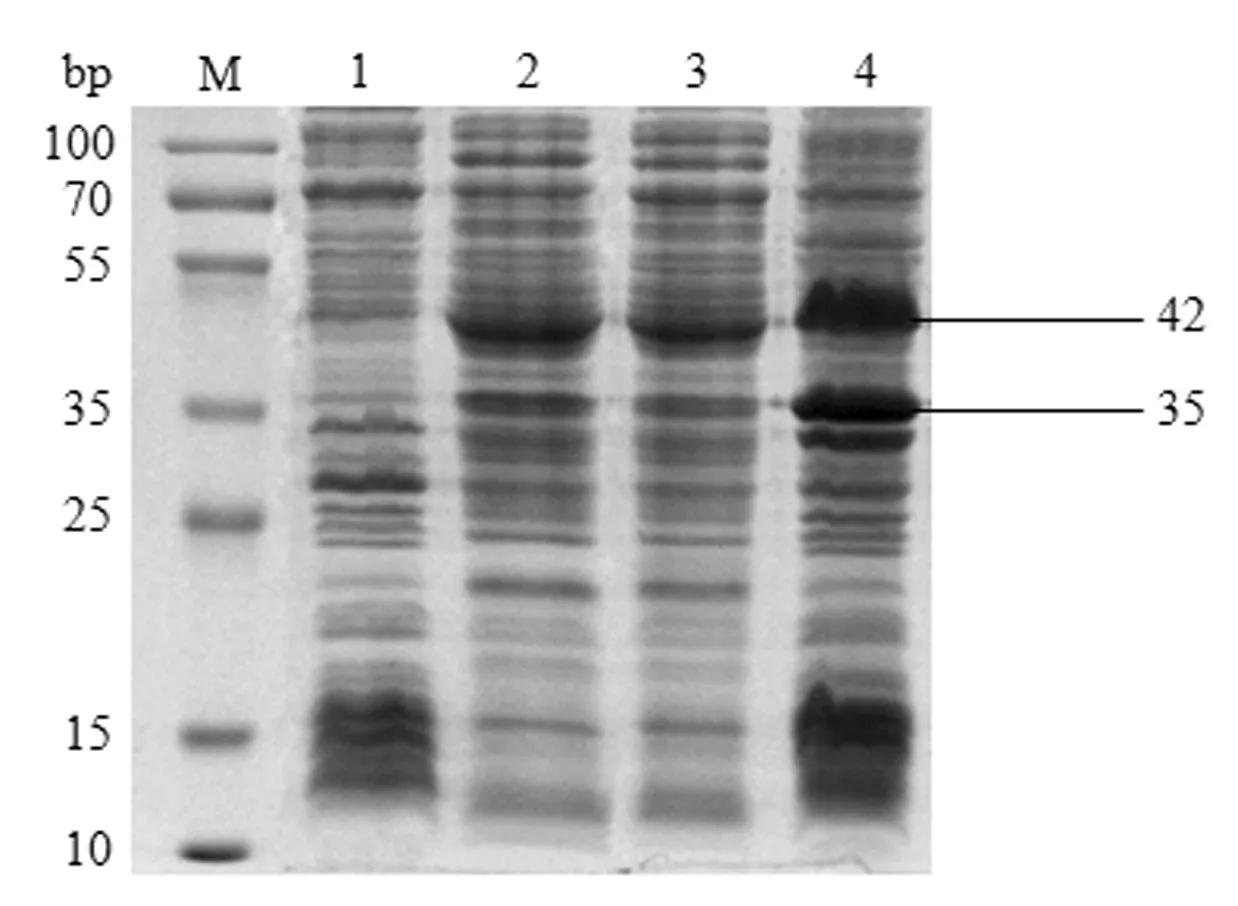
M:蛋白Marker;1:对照组诱导后裂解上清;2:对照组诱导后裂解沉淀;3:重组菌诱导后裂解上清;4:重组菌诱导后裂解沉淀
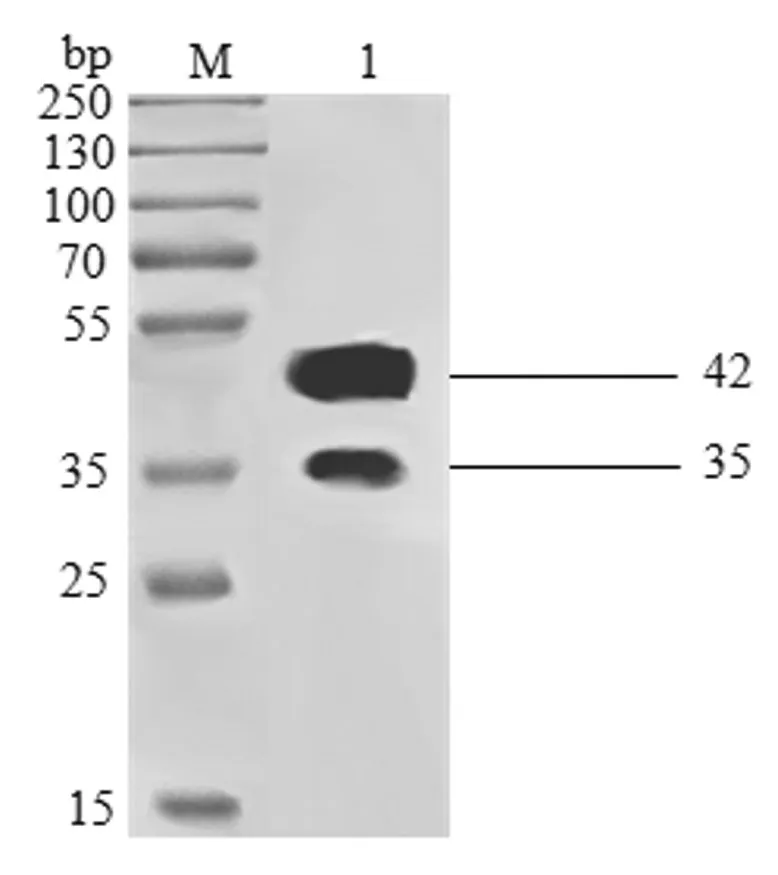
图 4 纯化后重组蛋白的Glycine-SDS-PAGE分析Fig.4 The Glycine-SDS-PAGE of purified recombinant protein
2.6 间接EELLSSIIAA方法反应条件的优化结果 对BPIV3间接ELISA反应条件的优化结果见表2。
2.7 间接EELLIISSAA临界值的确定 通过对随机选取的85份BPIV3阳性血清及79份BPIV3阴性血清检测,统计学分析OD450值,得到特异性与敏感性相关的ROC曲线(如图6),本研究中Youden指数最大为0.927,所对应的临界值为0.267,此时该方法的特异性为97.4%,灵敏性为95.3%。
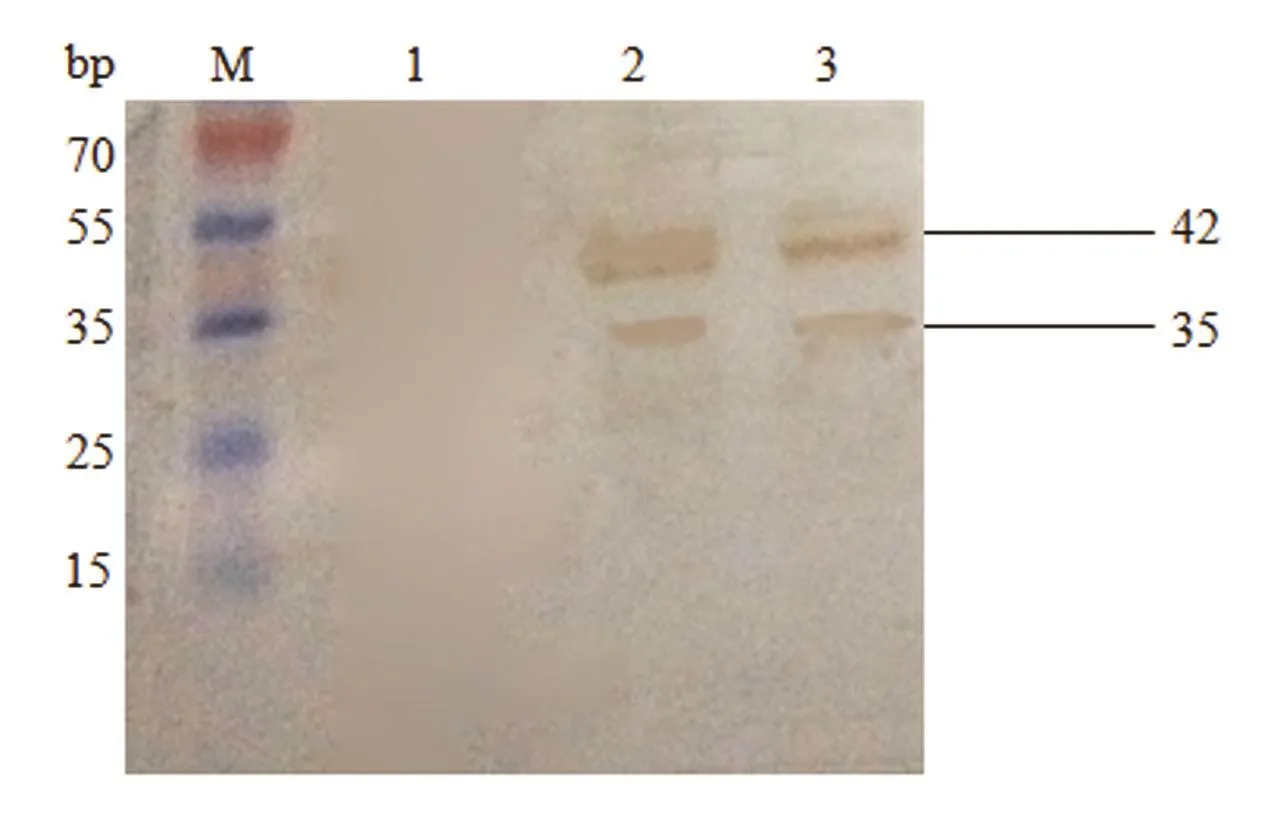
M:蛋白质;1:对照组诱导后裂解沉淀与BPIV3阳性血清的反应;2:重组蛋白与BPIV3阳性血清的反应;3:NX49株与BPIV3阳性血清的反应
2.8 重复性试验 通过对4份阳性血清及2份阴性血清进行的同批次及不同批次的重复性试验,分析统计试验结果,结果见表3,分别计算OD450平均值、标准差、变异系数,结果显示批内批间重复性试验变异系数均小于10%,证明了该间接ELISA方法重复性较好。

图 6 ROC曲线Fig.6 Receiver-operator characteristic curve
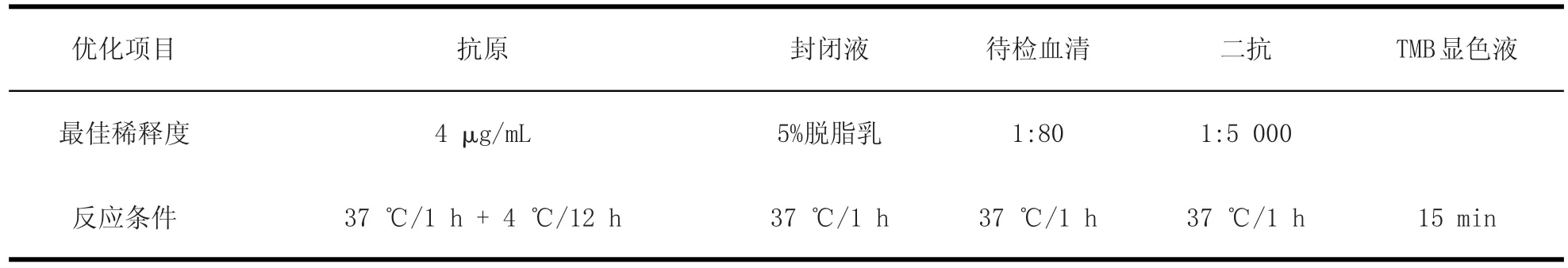
表 2 间接ELISA方法反应条件的优化Table 2 The optimized coditions of indirect ELISA

表 3 重复性试验结果Table 3 The result of repeatability test
2.9 临床样品的检测 对279份来自不同地区的血清采用本试验建立的间接ELSIA方法进行检测,检出BPIV3阳性血清223份,阴性血清56份,血清BPIV3抗体阳性率为79.9%,如表4所示。
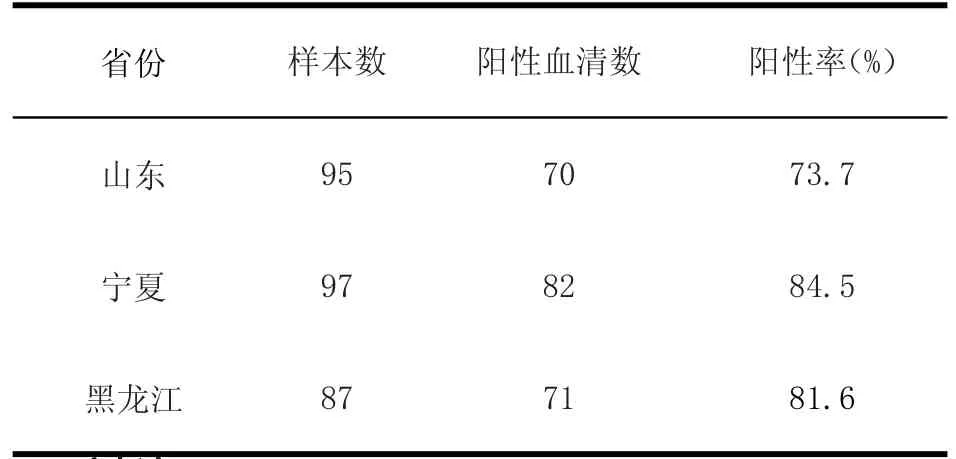
表 4 BPIV3抗体血清学调查Table 4 Serological survey of antibodies against BPIV3 in 3 provinces of China
3 讨论
BPIV3等病毒引起的BRDC发病率较高,常与其他病原体混合感染或继发感染,临床诊断较难,危害舍饲牛的健康,严重影响养牛业的经济效益,而我国针对BPIV3的检测方法的研究尚不成熟,尚无商品化的检测试剂盒[17-19]。在养牛业的发达国家,BPIV3的感染率及抗体阳性率同样处于较高水平。有研究指出20世纪80年代,墨西哥及阿根廷的BPIV3阳性率分别为85.6%和77.3%,欧美的健康牛血清中BPIV3普遍存在抗体[20-21]。澳洲仅出口牛中BPIV3的感染率及抗体阳性率达到了46%和87%[22]。国内也进行了一些BPIV3感染的流行病学调查,我国BPIV3血清阳性抗体率高达77.96%,感染率与抗体阳性率在逐渐增加[23]。因此建立针对BPIV3的诊断技术对BPIV3防控至关重要。
ELISA方法具有检测快速、操作便捷等特点,适用于流行病学调查及牛场大规模检测时的血清检测。传统的间接ELISA检测方法一般以全病毒或病毒的某个蛋白作为包被抗原,全病毒抗原难以纯化且成本较高。本研究选取NP基因中的抗原表位区域构建了pET-28a-NP-N-C原核表达载体,转化至E.coli BL21(DE3)感受态中,以纯化的重组蛋白作为抗原与待检BPIV3抗体相结合,建立的间接ELISA方法重复性好、特异性强和敏感性高,可用于BPIV3抗体的检测,为BPIV3检测试剂盒的开发铺设了条件。
[1]Horwood P F,Mahony T J.Multiplex real-time RT-PCR detection of three viruses associated withthebovinerespiratorydiseasecomplex[J].J Virol Methods,2011,171(2):360-363.
[2]Fulton R W,Purdy C W,Confer A W,et al.Bovine viral diarrhea viral infections in feeder calves with respiratory disease:interactions with Pasteurella spp.,parainfluenza-3 virus,and bovine respiratory syncytial virus[J].Can J Vet Res,2000,64(3):151-159.
[3]Zhu Y M,Shi H F,Gao Y R,et al.Isolation and genetic characterization of bovine parainfluenza virus type 3 from cattle in China[J].Vet Microbiol,2011,149(3-4):446-451.
[4]Bukreyev A,Skiadopoulos M H,Murphy B R,et al.Nonsegmentednegative-strandvirusesas vaccine vectors[J]. J Virol, 2006, 80(21):10293-10306.
[5]Marshall R G.Isolation of bovine parainfluenza-3 virus in chick embryos[J].J Bacteriol,1964,88:267-268.
[6]Sobhy N M,Mor S K,Mohammed M E,et al.Isolation and molecular characterization of bovine enteroviruses in Egypt[J].Vet J,2015,206(3):317-321.
[7]Konishi M,Ohkura T,Shimizu M,et al.Complete genome sequence of the first isolate of genotype C bovine parainfluenza virus type 3 in Japan[J]. Genome Announc, 2014, 2(6):e01215-01214.
[8]Ohkura T,Kokuho T,Konishi M,et al.Complete Genome Sequences of Bovine Parainfluenza Virus Type 3 Strain BN-1 and Vaccine Strain BN-CE[J].Genome Announc,2013,1(1):e00247-00212.
[9]Newcomer B W,Neill J D,Galik P K,et al.Serologic survey for antibodies against three genotypes of bovine parainfluenza 3 virus in unvaccinated ungulates in Alabama[J]. Am J Vet Res,2017,78(2):239-243.
[10]Oem J K,Lee E Y,Lee K K,et al.Molecular characterization of a Korean bovine parainfluenza virus type 3 isolate[J].Vet Microbi-ol,2013,162(1):224-227.
[11]刘鹏,侯喜林,周玉龙,等.牛副流感病毒3型的分离鉴定[J]. 微生物学通报,2009,36(9):1384-1389.
[12]冉旭华,张峣,曹思,等.C基因型牛副流感3型病毒的分离鉴定[J]. 中国畜牧兽医,2016,43(5):1368-1373.
[13]王海勇,童钦,王炜,等.我国牛副流感病毒3型血清学调查[J]. 中国预防兽医学报,2014,36(2):154-156.
[14]Neill J D,Ridpath J F,Valayudhan B T.Identification and genome characterization of genotype B and genotype C bovine parainfluenza type 3 viruses isolated in the United States[J].BMC Vet Res,2015,11:112.
[15]冉旭华,张峣,闻晓波.牛副流感病毒3型研究概述[J]. 黑龙江八一农垦大学学报,2017,29(3):24-28.
[16]Bailly J E,McAuliffe J M,Skiadopoulos M H,et al.Sequence determination and molecular analysis of two strains of bovine parainfluenza virustype 3that are attenuatedfor primates[J].Virus Genes,2000,20(2):173-182.
[17]杨建乐,赵贵民,侯佩莉,等.牛副流感病毒3型抗体间接ELISA检测方法的建立与初步应用[J].动物医学进展,2016,37(11):19-24.
[18]张欣欣.牛副流感病毒3型实时荧光定量RT-PCR检测方法的建立[D].呼和浩特:内蒙古农业大学,2016.
[19]李丽阳,李艳婷,余丽芸,等.牛副流感病毒3型双抗体夹心ELISA检测方法的建立[J].中国预防兽医学报,2016,38(2):132-136.
[20]Solis-Calderon J J,Segura-Correa J C,Aguilar-Romero F,et al.Detection of antibodies andriskfactorsforinfectionwithbovine respiratory syncytial virus and parainfluenza virus-3 in beef cattle of Yucatan,Mexico[J].Prev Vet Med,2007,82(1-2):102-110.
[21]Maidana S S,Lomonaco P M,Combessies G,et al.Isolation and characterization of bovine parainfluenza virus type 3 from water buffaloes(Bubalus bubalis) in Argentina[J]. BMC Vet Res,2012,8:83.
[22]Moore S J,O'Dea M A,Perkins N,et al.Estimation of nasal shedding and seroprevalence of organisms known to be associated with bovine respiratory disease in Australian live export cattle[J].J Vet Diagn Invest,2015,27(1):6-17.
[23]朱远茂,马磊,杨婷,等.牛副流感的流行、危害及其防控[J]. 中国预防兽医学报,2016,38(8):667-671.
