改良Blocker PCR检测EGFR-TKI耐药后非小细胞肺癌血浆EGFR T790M突变的价值
2018-03-22
(1复旦大学附属中山医院呼吸内科 上海 200032; 2上海允英医疗科技有限公司 上海 201216)
表皮生长因子受体酪氨酸激酶抑制剂(epidermal growth factor receptor-tyrosine kinase inhibitor,EGFR-TKI)已广泛应用于EGFR基因突变的晚期非小细胞肺癌(non-small-cell lung cancer,NSCLC)患者的治疗[1-2]。然而,在应用EGFR-TKI的过程中,对初始治疗有应答的患者不可避免的在1~2年内出现获得性耐药。既往研究表明,EGFR-TKI 获得性耐药机制中约50% 归因于T790M突变。为明确耐药原因,临床常规推荐组织二次活检。然而,二次活检为创伤性操作,不仅风险大,而且由于肿瘤组织存在异质性,组织活检有时不能反映肿瘤组织整体的基因突变状况[3-5]。血浆游离DNA(cell free DNA,cfDNA)基因突变检测不仅创伤小,容易取材,避免二次活检的创伤,而且能够多次动态监测治疗过程中的基因突变状态,为不宜二次活检的肺癌患者提供持续精准治疗的依据[6-7]。
本研究采用Blocker PCR 对EGFR-TKI 获得性耐药的NSCLC患者检测血浆cfDNA中EGFR敏感突变和T790M耐药突变,并采用配对组织检测及二代测序(next generation sequencing,NGS)方法验证其可靠性,探讨Blocker PCR在获得性T790M监测中的应用价值。
材 料 和 方 法
病例收集收集2015年3月至2016年9月期间就诊于复旦大学附属中山医院呼吸内科并符合以下入组标准的病例:(1)治疗前存在EGFR敏感突变或EGFR-TKI治疗有效;(2)经过EGFR-TKI治疗初始有应答;(3) RECIST标准评估疾病出现进展。符合入组标准的病例共127例,入组病例行血检EGFR敏感突变及T790M耐药突变检测;并选取部分病例血浆标本行NGS与Blocker PCR配对比较;为了验证血液检测的可靠性,同时采用二次活检组织与血浆检测配对比较。然而,二次组织活检在患者中难以开展,本组病例中仅有11例病例经二次活检。由于二次组织活检病例较少,无法评估血检的可靠性,而据文献报道,EGFR-TKI耐药的敏感突变与原始敏感突变保持一致[8-9]。故本研究选取部分EGFR-TKI耐药后血浆检出EGFR敏感突变的病例与治疗前组织标本行Blocker PCR配对比较。
分离血浆cfDNA抽提取抗凝血约5 mL置于EDTA抗凝管中,于室温下120×g离心20 min,分离上清,于4 ℃下16 000×g离心10 min,留取上清行血浆循环肿瘤DNA (circulating tumor DNA,ctDNA)抽提。采用循环肿瘤DNA抽提试剂盒(上海允英医疗科技有限公司)进行血浆ctDNA抽提,根据说明书进行操作。抽提cfDNA,进行定量。根据 Qubit微量紫外分光光度计(美国Life Technology公司)检测浓度值,样品浓度定量在0.5~10 ng/μL。
BlockerPCR基因突变检测根据Genbank公布的人EGFR基因序列,由上海允英医疗科技有限公司设计合成EGFR第18、19、20、21外显子基因特异性引物和探针(专利申请码:201510187617.3)。PCR扩增反应体系20 μL,模板2 μL,扩增条件为95 ℃变性10 min后进入主循环:95 ℃,15 s;60 ℃,1 min;15个循环后,重复95 ℃,15 s,60 ℃,1 min;35个循环后所得PCR产物,经SDS 2.0软件系统测定。
二代测序基因检测根据VAHTS Nano DNA Library Prep Kit for Illumina 试剂盒(南京诺唯赞生物科技有限公司)操作说明书进行cfDNA文库制备,cfDNA 片段长度约为160 bp;超深度测序的靶基因谱包括23个NSCLC相关基因,靶基因的富集应用SureSelect Target富集系统(美国Agilent公司);捕获的DNA文库在NextSeq 500测序仪(美国Illumina公司)上机测序。数据处理参考Newman 等[10]的生物信息学方法,检测单核苷酸点突变及缺失突变,采用具有后处理过滤器功能的VarScan 2[11]来提高突变识别可信度(variant call confidence),对融合突变及断点的表征采用融合与染色体易位计数与恢复算法(fusion and chromosomal translocation enumeration and recovery algorithm,FACTERA)[10]。
统计学处理应用SPSS 17.0进行统计分析,计数资料以例数及百分比表示。P<0.05为差异有统计学意义。
结 果
入组病例基本情况127例经过EGFR-TKI治疗后出现进展的患者中(表1),男性56例(44.10%),女性71例(55.90%);年龄27~83岁,平均63岁;124例(97.63%)为肺腺癌患者;56例(44.09%)患者接受EGFR-TKI一线治疗,74例(55.91%)经二线或三线EGFR-TKI治疗;120例(94.49%)为肺癌Ⅳ期患者。
BlockerPCR血浆检测结果127例EGFR-TKI耐药后患者的血浆标本中,40.15% (51/127)的样本含有T790M突变,其中21.57% (11/51)为单纯T790M突变,78.43%(40/51)为T790M合并EGFR敏感突变;59.84% (76/127)的样本为T790M阴性,其中37例为单纯EGFR敏感突变,36例为野生型突变,3例为其他突变:1例为E19-DELs+c-met扩增,1例为E19-DELs+ALK融合突变,1例为L858R+HER-2 插入突变(图1)。
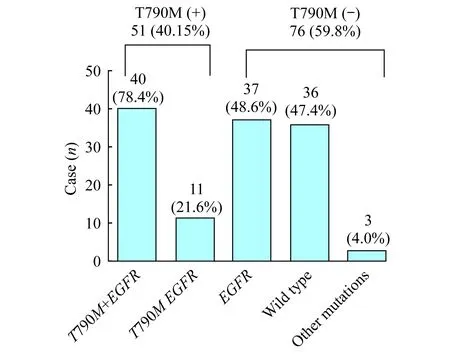
图1 Blocker PCR 在血浆中的基因突变检测结果Fig 1 EGFR sensitizing mutations and T790M mutationsdetected by Blocker PCR in plasma
组织与血浆基因检测结果比较11例EGFR-TKI耐药后的入组病例行二次活检组织和配对血浆基因检测,二次活检组织结果与血浆检测结果具有较高一致率(图2),其中仅有1例编号为3的病例检测结果不一致;在EGFR-TKI耐药后二次活检组织标本中T790M阳性例数为6例,T790M检出率为54.54% (6/11)。
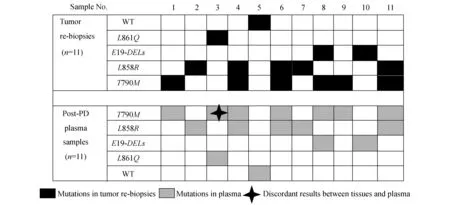
PD:Progression development;E19-DELs:Deletion of exon 19;WT:Wild type.
图2二次活检组织与配对血浆基因检测结果
Fig2EGFRmutationsinplasmaandpairedtumorrebiopsies
由于二次活检的病例较少,无法评估血浆T790M检测的准确性,因此,我们检测了21例未经EGFR-TKI治疗的首次活检组织标本及该21例病例经EGFR-TKI治疗后耐药的配对血浆标本。如图3所示,EGFR-TKI耐药后血浆携带的敏感突变类型为治疗前组织原有敏感突变类型,未出现突变亚型之间的转化,仅有2例在EGFR-TKI耐药后出现野生型。EGFR-TKI耐药后的32例血浆标本中共14例为T790M阳性(图2~3),因而血浆T790M的检出率为43.75% (14/32)。
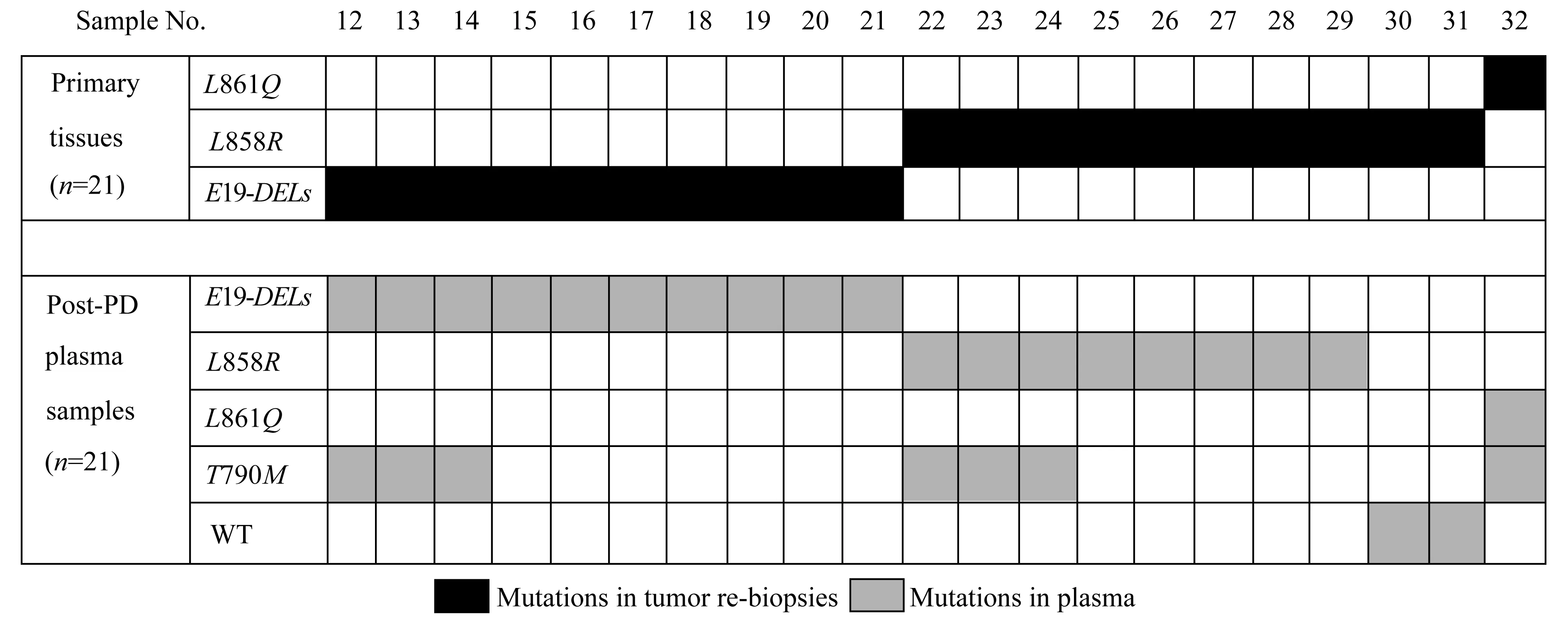
PD:Progression development;E19-DELs:Deletion of exon 19;WT:Wild type.
图3首次活检组织与耐药后配对血浆基因检测结果
Fig3EGFRmutationsinpost-PDplasmasamplesandpairedprimarytumortissues
二代测序与BlockerPCR突变检测的比较在EGFR和T790M突变位点,二代测序和Blocker PCR对18例EGFR-TKI耐药后患者的检出率完全一致,均为100%。其中 6例(33%)为EGFR敏感突变合并T790M突变;4例(22%)为单纯敏感突变;8例(22%)为野生型。二代测序还检测到文献报道的其他EGFR-TKI耐药相关突变位点,如KRAS、PTEN、PIK3CA、MET、BIM和ROS-1等(图4)。

图4 二代测序基因检测结果与Blocker PCR检测结果一致性 Fig 4 Concordance of mutations identified by Blocker PCR and NGS in plasma
讨 论
EGFR基因突变是NSCLC的驱动基因之一,目前约30%~40%的亚洲NSCLC患者在确诊时携带有EGFR基因突变,根据NCCN指南、ASCO指南及《中国原发性肺癌诊疗规范》推荐,EGFR-TKI一线治疗是EGFR突变的晚期NSCLC患者的主要治疗方法[12]。在应用EGFR-TKI的治疗过程中,对初始EGFR-TKI治疗应答良好的患者不可避免地出现疾病进展,对原始靶向药物产生获得性耐药,肿瘤基因突变状态发生改变。EGFR-TKI 获得性耐药机制中,约50%为T790M突变[13],其他耐药机制包括c-met扩增、HER-2突变、PIK3CA和KRAS等[13-14]。对一代EGFR-TKI继发性耐药并存在T790M突变的患者,三代EGFR-TKI 奥希替尼(Osimertinib,AZD9291,英国阿斯利康制药有限公司)取得非常好的疗效,治疗缓解率达到63%,PFS可达9.7个月[15]。因此,是否存在T790M突变显得尤为重要,目前推荐对进展的肿瘤行二次活检基因突变检测[16]。然而,二次活检不仅创伤大,危险性高,而且肿瘤组织存在异质性,二次活检组织不能够反映肿瘤的整体突变状况[17]。
cfDNA能够反映肿瘤的分子生物学特征(如基因突变、甲基化等),cfDNA基因检测不仅可避免二次活检的创伤,而且能反复取材,在靶向治疗的过程中,动态监测基因突变状态的改变[6,18-19]。然而,液体活检同时也面临着诸多缺陷,与组织活检相比,虽然其特异性可达90%以上,其敏感性却因采用的方法不同而有差异。例如,以组织基因检测作为金标准,采用ARMS 血浆基因检测EGFR敏感突变中,敏感性为75%[20]。采用cobas 血液检测对EGFR敏感突变的敏感性可以提高到82%[21]。采用数字PCR方法对ctDNA进行EGFR敏感基因突变检测,敏感性达到80%以上[22-23]。但据既往报告,基于PCR技术检测血浆T790M突变则不尽如人意,Kenneth等[21]比较了cobasEGFR突变检测方法、therascreenEGFRARMS方法、微滴数字PCR和BEAMing 数字 PCR在检测T790M的应用,以组织为标准,检测T790M的敏感性最高仅达到73%。据分析,T790M突变区域影响PCR引物结合,从而导致PCR方法检测此位点的敏感性不高。最新研究发现足够深度的测序技术在晚期NSCLCT790M的检测方面具有更高的检出优势,但是由于NGS成本高,耗时长,因而在临床应用存在困难[24-25]。
本研究采用Blocker PCR方法对一代EGFR-TKI耐药患者血浆T790M基因突变进行检测,T790M突变检出率为40.16%,而据文献报道,组织二次活检T790M的检出率约为50%,本研究与二次活检组织的检出率相似[26-27]。此外,本研究血浆检出T790M突变的患者中,T790M合并敏感突变占78.44%。Zheng等[8]研究发现,在117例患者的血浆检测中,T790M合并敏感突变比单纯T790M突变所占比例要大[8]。NGS是近年兴起的高通量的测序方法,其通量高,实验流程稳定,重复性好,能够检测未知的基因突变[10,28]。为了检测Blocker PCR的可靠性,本文选取18例经Blocker PCR 检测的血浆样本同时行NGS检测,无论是在EGFR敏感突变位点还是在T790M突变位点,NGS与Blocker PCR的检测结果一致率均为100%。为了验证血液检测的可靠性,通常以同期二次活检组织为金标准进行比较,但在临床上二次活检在患者中难以开展。本研究中仅有11例病例经二次活检,由于病例较少,无法评估血液检测的可靠性。据文献报道,EGFR-TKI耐药后携带的敏感突变与EGFR-TKI治疗前原始敏感突变保持一致,很少发生转化[9,29]。故本文选取EGFR-TKI耐药后的血浆检出EGFR敏感突变的病例与EGFR-TKI治疗前组织相比较,结果显示,耐药后血浆的敏感突变位点与EGFR-TKI治疗前的组织敏感突变位点完全一致。总之,通过与文献报道的组织T790M阳性率比较,同一血浆标本NGS与Blocker PCR 相验证,并通过组织与血浆相比较,均表明本研究所用的Blocker PCR检测血浆T790M具有可靠性。
本研究之所以能够提高血浆T790M检出率,使其接近二次活检组织EGFRT790M检出率,一方面是在血浆cfDNA的抽提技术方面做了改进,综合了物理和化学的方法提取更高比例的ctDNA;另一方面是采用了Blocker PCR的检测技术,Blocker PCR 在血液检测中具有较高的灵敏度,可以达到0.1%。其原理主要是通过改造 5’和 3’端探针的结构,使改造后的探针与野生型等位基因特异结合的最适温度比与突变型结合的温度高10 ℃。在温度由 95 ℃降至 60 ℃的过程中,探针与野生型等位基因特异结合从而被阻滞。当温度降至60 ℃时,大部分的野生型等位基因被探针特异性阻滞,突变型等位基因则相对富集,因此突变特异性探针能够特异性的扩增突变的DNA链而不被野生型等位基因所干扰。与此同时,通过优化T790M探针及反应条件,克服了基于PCR方法对T790M检出率低的缺陷。在前期100余例EGFR-TKI治疗前组织与治疗前配对血浆的研究中,Blocker PCR 结合改良后的抽提方法可以将敏感突变的检测敏感性提高到95%(相关研究另行投稿,数据在本文中未提供)。血浆基因检测是个体精准治疗的理想方式,但EGFR-TKI耐药后血检T790M检出率相对较低是其一大弊端。AZD9291上市的同时,FDA批准了第1个EGFR耐药突变伴随检验试剂盒cobas EGFR Mutation Test v2,而Cobas血浆检测T790M的敏感性为77%(30/41),特异性为67%(16/24)[21]。本研究通过改良抽提方法及Blocker PCR改良,使T790M检出率更加接近组织突变状态,能够提高依靠液体活检指导临床用药的可靠性。在靶向治疗的肺癌患者中,使用Blocker PCR在cfDNA中检测耐药突变,尤其是检测T790M突变,是应用AZD9291等新一代靶向药物的重要依据。此外,Blocker PCR也可以检测c-met、HER-2、KRAS等基因突变,相对弥补了其检测广度不如NGS全面的不足。无论是耗时还是花费,Blocker PCR都比NGS更有优势。
本研究的不足之处在于:(1)由于二次活检组织难以获取,因而组织配对标本量比较少;(2)研究进行时AZD9291在国内尚未批准上市,故血液检测到T790M突变的患者无法用后续靶向治疗效果验证。综上所述,经过改良的血浆cfDNA抽提方法合并Blocker PCR 检测T790M突变有望指导无法获取二次活检组织的患者后续EGFR-TKI的选用。
致谢计海婴医师、裴东妮女士和赵海莲女士在血液标本收集的过程中提供了帮助。
[1] ROSELL R,CARCERENY E,GERVAIS R,etal.Erlotinib versus standard chemotherapy as first-line treatment for European patients with advanced EGFR mutation-positive non-small-cell lung cancer (EURTAC):a multicentre,open-label,randomised phase 3 trial[J].LancetOncol,2012,13(3):239-246.
[2] MIYAUCHI E,INOUE A,KOBAYASHI K,etal.Efficacy of chemotherapy after first-line gefitinib therapy in EGFR mutation-positive advanced non-small cell lung cancer-data from a randomized Phase Ⅲ study comparing gefitinib with carboplatin plus paclitaxel (NEJ002)[J].JpnJClinOncol,2015,45(7):670-676.
[3] BAI H,WANG Z,CHEN K,etal.Influence of chemotherapy on EGFR mutation status among patients with non-small-cell lung cancer[J].JClinOncol,2012,30(5):3077-3083.
[4] GERLINGER M,ROWAN AJ,HORSWELL S,etal.Intratumor heterogeneity and branched evolution revealed by multiregion sequencing[J].NEnglJMed,2012,366(10):883-892.
[5] ZHANG J,FUJIMOTO J,ZHANG J,etal.Intratumor heterogeneity in localized lung adenocarcinomas delineated by multiregion sequencing[J].Science,2014,346(6206):256-259.
[6] MOK T,WU Y,LEE JS,etal.Detection and dynamic changes of EGFR mutations from circulating tumor DNA as a predictor of survival outcomes in NSCLC patients treated with first-line intercalated erlotinib and chemotherapy[J].ClinCancerRes,2015,21(14):3196-3203.
[7] OLSSON E,WINTER C,GEORGE A,etal.Serial monitoring of circulating tumor DNA in patients with primary breast cancer for detection of occult metastatic disease[J].EMBOMolMed,2015,7(8):1034-1047.
[8] ZHENG D,YE X,ZHANG MZ,etal.Plasma EGFR T790M ctDNA status is associated with clinical outcome in advanced NSCLC patients with acquired EGFR-TKI resistance[J].SciRep,2016,6:20913.
[9] KOSAKA T,YATABE Y,ENDOH H,etal.Analysis of epidermal growth factor receptor gene mutation in patients with non-small cell lung cancer and acquired resistance to gefitinib[J].ClinCancerRes,2006,12(19):5764-5769.
[10] NEWMAN AM,BRATMAN SV,To J,etal.An ultrasensitive method for quantitating circulating tumor DNA with broad patient coverage[J].NatureMed,2014,20(5):548-554.
[11] KOBOLDT DC,ZHANG Q,LARSON DE,etal.VarScan 2:somatic mutation and copy number alteration discovery in cancer by exome sequencing[J].GenomeRes,2012,22(3):568-576.
[12] ETTINGER DS,WOOD DE,AKERLEY W,etal.NCCN guidelines insights:non-small cell lung cancer,Version 4.2016[J].JNatlComprCancNetw,2016,14(3):255-264.
[13] OHASHI K,MARUVKA YE,MICHOR F,etal.Epidermal growth factor receptor tyrosine kinase inhibitor-resistant disease[J].JClinOncol,2013,31(8):1070-1080.
[14] GOU LY,LI AN,YANG JJ,etal.The coexistence of MET over-expression and an EGFR T790M mutation is related to acquired resistance to EGFR tyrosine kinase inhibitors in advanced non-small cell lung cancer[J].Oncotarget,2016,7(32):51311-51319.
[15] OXNARD GR,THRESS KS,ALDEN RS,etal.Association between plasma genotyping and outcomes of treatment with Osimertinib (AZD9291) in advanced non-small-cell lung cancer[J].JClinOncol,2016,34(28):3375-3382.
[16] CROSS DA,ASHTON SE,GHIORGHIU S,etal.AZD9291,an irreversible EGFR TKI,overcomes T790M-mediated resistance to EGFR inhibitors in lung cancer[J].CancerDiscov,2014,4(9):1046-1061.
[17] MCGRANHAN N,SWANTON C.Biological and therapeutic impact of intratumor heterogeneity in cancer evolution[J].CancerCell,2015,27(1):15-26.
[18] WANG Z,CHEN R,WANG S,etal.Quantification and dynamic monitoring of EGFR T790M in plasma cell-free DNA by digital PCR for prognosis of EGFR-TKI treatment in advanced NSCLC[J].PLoSOne,2014,9(11):e110780.
[19] DIAZ LA JR,BARDELLI A.Liquid biopsies:genotyping circulating tumor DNA[J].JClinOncol,2014,32(6):579-586.
[20] LIU X,LU Y,ZHU G,etal.The diagnostic accuracy of pleural effusion and plasma samples versus tumour tissue for detection of EGFR mutation in patients with advanced non-small cell lung cancer:comparison of methodologies[J].JClinPathol,2013,66(12):1065-1069.
[21] THRESS KS,BRANT R,CARR TH,etal.EGFR mutation detection in ctDNA from NSCLC patient plasma:a cross-platform comparison of leading technologies to support the clinical development of AZD9291[J].LungCancer,2015,90 (2015):509-515.
[22] YUNG TKF,CHAN KCA,MOK TSK,etal.Single-molecule detection of epidermal growth factor receptor mutations in plasma by microfluidics digital PCR in non-small cell lung cancer patients[J].ClinCancerRes,2009,15(6):2076-2084.
[23] ZHU G,YE X,DONG Z,etal.Highly sensitive droplet digital PCR method for detection of EGFR-activating mutations in plasma cell-free DNA from patients with advanced non-small cell lung cancer[J].JMolDiagn,2015,17(3):265-272.
[24] UCHIDA J,KATO K,KUKITA Y,etal.Diagnostic accuracy of noninvasive genotyping of EGFR in lung cancer patients by deep sequencing of plasma cell-free DNA[J].ClinChem, 2015,61(9):1191-1196.
[25] JIN Y,SHAO Y,SHI X,etal.Mutational profiling of non-small-cell lung cancer patients resistant to first-generation EGFR tyrosine kinase inhibitors using next generation sequencing[J].Oncotarget,2016,7(38):61755-61763.
[26] OXNARD GR,ARCILA ME,SIMA CS,etal.Acquired resistance to EGFR tyrosine kinase inhibitors in EGFR-mutant lung cancer:distinct natural history of patients with tumors harboring the T790M mutation[J].ClinCancerRes,2011,17(6):1616-1622.
[27] HATA A,KATAKAMI N,YOSHIOKA H,etal.Rebiopsy of non-small cell lung cancer patients with acquired resistance to epidermal growth factor receptor-tyrosine kinase inhibitor:comparison between T790M mutation-positive and mutation-negative populations[J].Cancer,2013,119(24):4325-4332.
[28] XU S,LOU F,WU Y,etal.Circulating tumor DNA identified by targeted sequencing in advanced-stage non-small cell lung cancer patients[J].CancerLett,2016,370(2):324-331.
[29] HATA AN,NIEDERST MJ,ARCHIBALD HL,etal.Tumor cells can follow distinct evolutionary paths to become resistant to epidermal growth factor receptor inhibition[J].NatMed,2016,22(3):262-269.
