高分子量腈水合酶工程菌生产烟酰胺的工艺建立
2018-03-16郭军玲王哲刘中美周哲敏
郭军玲,王哲,刘中美,周哲敏
(江南大学 生物工程学院,工业生物技术教育部重点实验室,江苏 无锡,214122)
腈水合酶(Nitrile hydratase,简称NHase,EC 4.2.1.84)是一种能够催化腈类物质生成酰胺类化合物的金属酶[1],主要用于工业上丙烯酰胺和烟酰胺的生产[2]。常见NHase大多数来源于红球菌属(Rhodococcus)、假诺卡氏菌属(Pseudonocardia)以及诺卡氏菌(Nocardia)[3]。根据NHase活性中心所含金属离子的不同,一般可将其分为铁型腈水合酶(Fe-NHase)和钴型腈水合酶(Co-NHase),他们的活性中心分别含有一个非血红素铁离子[4]和一个非咕啉态钴离子[1],并且金属离子都位于α亚基保守序列-Cys-X-Leu-Cys(SO2H)-Ser-Cys(SOH)上,其中包含两个被氧化的半胱氨酸残基:被深度氧化的硫酰基(—SO2—)和被浅度氧化的亚硫酰基(—SO—)[4-6]。生物体内,NHase通常与腈水解酶[7]、酰胺酶[6]这两种关键酶一起发挥作用进行腈类物质代谢。
目前很多化工产品的生产已经由传统的化学方法转变为以酶或全细胞为主的生物法[8-9],NHase就是其中最有代表性的例子,其用于酰胺类物质如丙烯酰胺、尼克酰胺以及5-氰基戊酰胺的生产已有数十年历史[2]。尼可酰胺是一种水溶性维生素,也是一种用于药物和杀虫剂合成的重要中间体,在制药行业和食品工业中有重要应用[10-11]。目前用于生产尼可酰胺的菌株为玫瑰色红球菌RhodococcusrhodochrousJ1(J1),它也是第三代工业菌株[12-13]。在J1中同时存在两种不同类型的腈水合酶,按其分子量不同分为高分子量腈水合酶(H-NHase)和低分子量腈水合酶(L-NHase)。虽然目前工业生产烟酰胺主要用J1这一生产菌,但其培养时间长,蛋白表达量低[14-15]。相比较而言,大肠杆菌表达系统具有明显的优势:菌体生长速度快,蛋白表达量高。目前,L-NHase和H-NHase已在大肠杆菌表达系统中实现高效表达[16-17],这就使得用蛋白表达量高的重组基因工程菌替代现有的工业用菌成为可能。
大肠杆菌高密度发酵技术是一种通过一定的培养方法提高菌体生物量从而提高蛋白表达量的有效手段[18-19]。目前很多表达酶蛋白或抗生素等异源物质的重组大肠杆菌都实现了高密度扩大培养[20-21]。若想真正在工业规模用大肠杆菌替代红球菌生产烟酰胺,高密度发酵技术的实现就非常有必要。
本研究对细胞培养基首先进行了摇瓶水平优化,使菌体生物量和细胞酶活得到提高;建立烟酰胺生产的催化工艺,使得表达H-NHase腈水合酶基因大肠杆菌催化烟腈生成终浓度更高的烟酰胺产物;对重组菌进行了分批补料扩大培养,实现了大肠杆菌高密度培养和重组蛋白的大量表达;最后与实际生产情况相结合,用高密度的发酵罐培养后重组菌进行烟腈催化反应,产物浓度相比摇瓶发酵细胞的催化有进一步的提高。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
基因工程菌E.coilBL21(DE3)/pET-24a(+)-nhhBrbsArbsG(BAG)[17]、E.coilBL21(DE3)/pET-24a(+)-nhhBrbsArbsG(BAE)[16],表达载体pET-24a(+)以及宿主菌E.coilBL21(DE3),以上均为实验室构建和保藏。
1.1.2 培养基
LB培养基(g/L):胰蛋白胨 10.0,酵母提取物 5.0,NaCl 10.0。
2YT培养基(g/L):胰蛋白胨 16.0,酵母提取物 10.0,NaCl 5.0,IPTG终浓度0.6 mmol/L,CoCl2·6H2O 0.1。
摇瓶优化培养基(g/L):胰蛋白胨 12.0,酵母提取物 6.0,甘油10.0,KH2PO42.31,KH2PO412.54,IPTG终浓度0.6 mmol/L,CoCl2·6H2O 0.1。
5 L发酵罐培养基(g/L):葡萄糖 12.0,KH2PO413.5,(NH4)2PO44.0,柠檬酸 1.7,MgSO41.68,微量元素 10 mL。
补料培养基(g/L):葡萄糖500.0,MgSO47.33,酵母提取物 4.0,胰蛋白胨 4.0。
诱导剂(g/100mL):乳糖 10.0,CoCl2·6H2O 0.8。
微量元素(g/100mL):FeSO4·7H2O 1.0,ZnSO4·7H2O 0.525,CuSO4·2H2O 0.3,MnSO4·4H2O 0.05,硼砂 0.023,CaCl20.2,钼酸铵 0.01。
1.1.3 试剂
3-氰基吡啶,梯希爱上海化成工业发展有限公司;尼克酰胺标准样品,日本和光纯药工业株式会社;葡萄糖、甘油、培养基中所用各种盐类、丙烯腈、丙烯酰胺,国药化学试剂有限公司;胰蛋白胨,酵母提取物,英国Oxoid公司。
1.1.4 仪器
高效液相色谱仪HPLC、色谱柱HITACHI LaChrom C18(250 ACHI LaChrom,5 μm),日立(HITACHI)公司;UV-1800PC 型紫外可见分光光度计,上海美谱达有限公司;生物传感分析仪,山东省科学院生物研究所;5 L全自动发酵罐,美国Winpact公司;蠕动泵,兰格恒流泵有限公司。
1.2 实验方法
1.2.1 摇瓶培养基的正交优化
选取甘油、酵母提取物、胰蛋白胨、Co2+这4种主要培养基组分,作为正交试验设计的因素。试验采用4因素3水平正交试验L9(34),通过实验分析它们对产酶和菌体的生长影响情况。
1.2.2 重组质粒在大肠杆菌表达系统中的表达(摇瓶水平)
将高分子量腈水合酶大肠杆菌重组菌(BAG)在含50 μg/mL卡那霉素LB固体平板上划线活化,取LB平板上单菌落,接种至5 mL含50 μg/mL卡那霉素的LB培养基中,在37 ℃,200 r/min条件下振荡培养7.5 h,取300 μL上述培养物接种至50 mL含50 μg/mL卡那霉素的2YT培养基或摇瓶优化培养基中,在37 ℃,200 r/min条件下振荡培养至菌液OD600值达到0.6~1.0时,加入IPTG至终浓度为0.6 mmol/L以及CoCl2·6H2O至终质量浓度为0.1 g/L,24 ℃诱导18 h。
1.2.3 重组型基因工程菌全细胞催化酶活测定
细胞酶活的测定方法参考之前纯酶酶活检测的方法[15],细胞干重比酶活力定义为:26 ℃条件下1 mg细胞每分钟催化产生烟酰胺的量(U/mgDCW);单位体积发酵液酶活力定义为:26 ℃条件下1 mL发酵液每分钟催化产生烟酰胺的量(U/mL)。
将100 μL,OD600=1.0的菌体(磷酸缓冲液溶解)加入400 μL,125 mmol/L烟腈溶液中,25 ℃反应10 min,500 μL乙腈终止反应,加入终止液后立即在4 ℃,12 000 r/min条件下离心1 min,吸取上清。上清稀释10倍后,用0.22 μm微孔滤膜进行过滤,采用C18色谱柱进行HPLC检测,流动相为水和乙腈[V(水)∶V(乙腈)=2∶1]的混合溶液。检测波长设定220 nm,柱温为40 ℃。流速设定为0.8 mL/min,进样体积10 μL。
1.2.4 菌体回收
对摇瓶发酵结束的菌体测定OD600,按照比例(最终OD600=4.0,8.0或160.0)计算需要离心的发酵液并离心回收,4 ℃,8 000 r/min条件下离心10 min,弃上清后将剩余菌体用水悬浮成30 mL发酵液。
1.2.5 底物流加细胞催化工艺
[22]中的方法。0.5 mol/L的烟腈经流动泵以0.38 mL/min、1.5 mL/min的流速加入30 mL,OD600=4.0 的菌液中并不断搅拌,定期取样并测定产物的积累量,待产物浓度维持不变时停止流加。流加过程中,体系温度维持在25~28 ℃范围内。
1.2.6 烟腈底物分批补料细胞催化工艺
参考文献[11, 23]的方法。烟腈固体粉末以0.2 mol/L(20.82 g/L)或0.4 mol/L(41.64 g/L)的终浓度加入到30 mL,OD600=8.0或160.0的菌液中并不断搅拌,当批底物反应完毕后再加入下一批底物,定期取样并测定产物的积累量,体系温度维持在25~28 ℃范围内。
1.2.7 细胞对烟酰胺的耐受性
1 mol/L的烟酰胺经蠕动泵以1 mL/min的流速加入30 mL,OD600=4.0的菌液中并不断搅拌,定期取样并测定不同产物浓度下的细胞酶活,最终计算不同产物浓度下的相对酶活:
(1)
1.2.8 重组型基因工程菌的高密度发酵工艺
种子培养:从-8 ℃冰箱中取出保存的甘油管,吸取200 μL菌液接种于30 mL含有50 μg/mL卡那青霉素抗性的LB培养基中,置于37 ℃的恒温摇床中200 r/min培养7.5 h。
发酵罐培养:采用5 L Winpact全自动发酵罐,初始装液量为2 L。设置初始pH为7,培养温度为37 ℃,初始转速200 r/min,通气量为5 L/min。将上步所得的种子液以6%的接种量接入发酵罐中,同时加入卡那青霉素至终质量浓度为50 μg/mL,接入种子液后将发酵罐的溶氧与转速相偶联,控制罐内溶氧维持在30%,在接种5~6 h后出现溶氧反弹现象,此时以比生长速率μ=0.2/h的指数流加补料策略进行补料。每隔2~3 h取样1次并测定罐内细胞密度、葡萄糖浓度、乙酸浓度以及细胞酶活。整个发酵过程中通过流加25%的氨水调节pH,使罐内pH始终维持在7.0左右,当OD600达到60左右时,改变温度为30 ℃,以0.22 g/(L·h)的恒流速流加乳糖和钴离子进行诱导。
1.2.9 分析方法
菌体生物量:紫外可见分光光度计测定OD600值,根据OD600值和细胞干重关系进行换算:1 g DCW/L=0.368 3 OD600
葡萄糖含量:使用SBA-40E生物传感分析仪。
乙酸含量:采用高压液相法。
2 结果与分析
2.1 摇瓶培养基的正交优化
本研究选取甘油、酵母提取物、胰蛋白胨、Co2+这4个主要培养基组,分别采用4因素3水平正交试验L9(34),对高分子量腈水合酶大肠杆菌重组菌(BAG)在不同组合下的菌体生物量和细胞产酶情况进行了考察,并与优化前的培养基作对比。
依据正交试验设计表,各个因素分别选取3个水平。表1是正交试验设计表,表2是试验结果的直观分析表。实验后通过极差分析(表3)和方差分析(表4)可得出培养基中各组分对酶活影响大小的排序和影响酶活的显著性因素,最终得到优化组合。
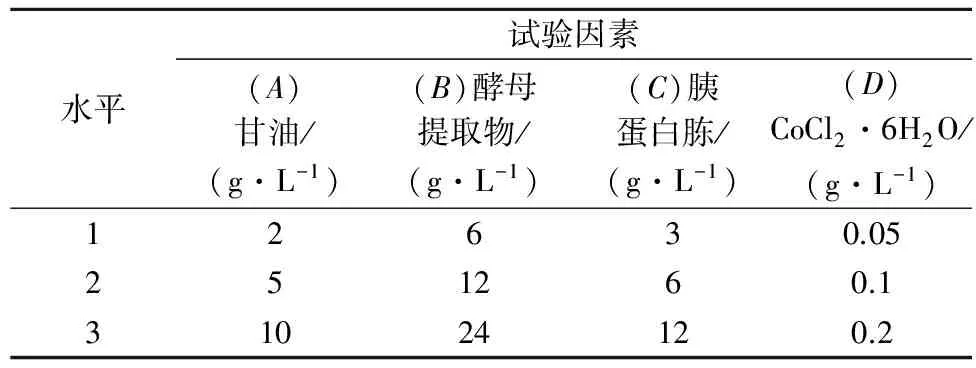
表1 正交试验设计表
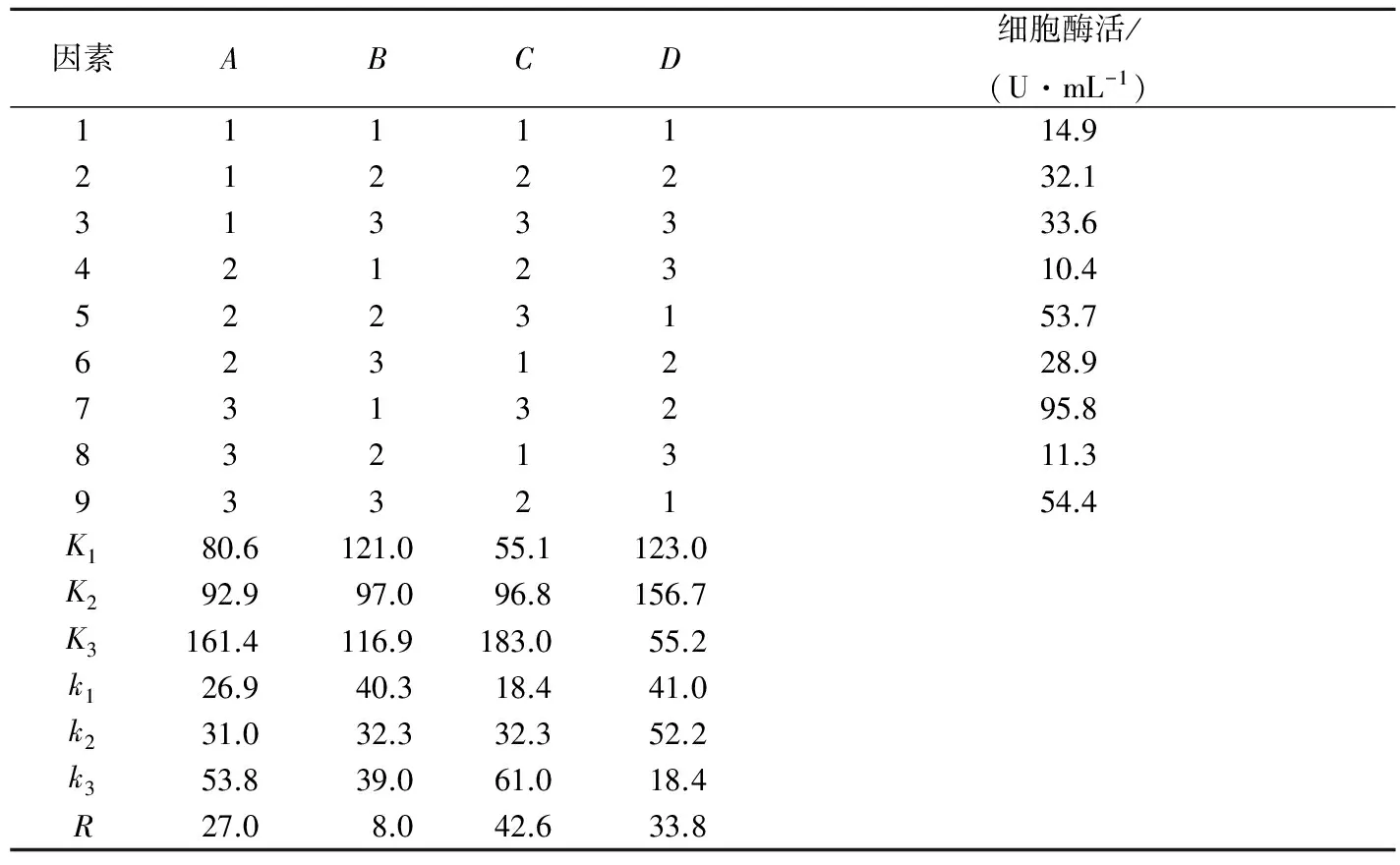
表2 L9(34)直观分析表Table 2 L9(34) visual analysis test

表3 培养基中各组分的重要性排序及分析Table 3 Significance analysis for factors of fermentationmedium
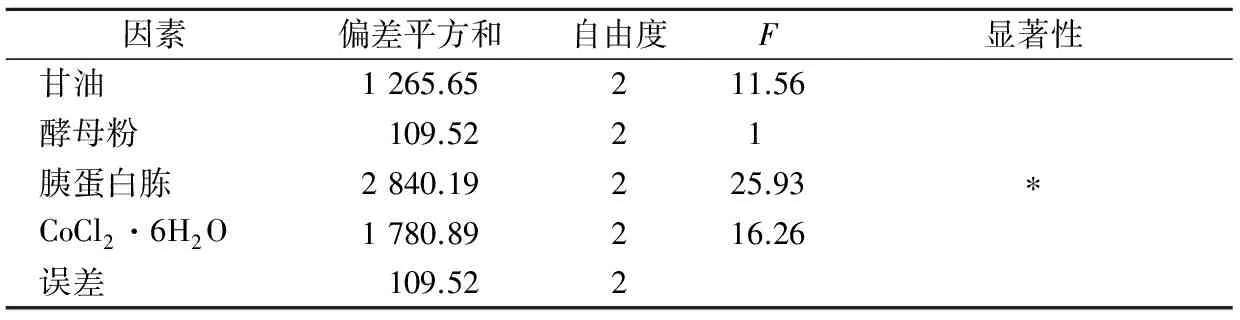
表4 培养基中各组分方差分析Table 4 Variance analysis for factors of fermentation medium
通过极差分析,得到了培养基中各组分对酶活影响大小的排序,而根据方差分析中的F值大小,发现其得到的重要性程度排序与极差分析得到的结果相同,其中影响最显著的因素是胰蛋白胨。根据以上实验结果,得到了最优培养基:胰蛋白胨12 g/L,酵母提取物6 g/L,甘油10 g/L,CoCl2·6H2O 0.1 g/L。
如表5所示,BAG优化后的细胞生物量是优化前的1.2倍,从3.68 gDCW/L提高到4.42 gDCW/L,细胞比酶活为优化前的3.8倍,从8.06 U/mgDCW提高到30.91 U/mgDCW。
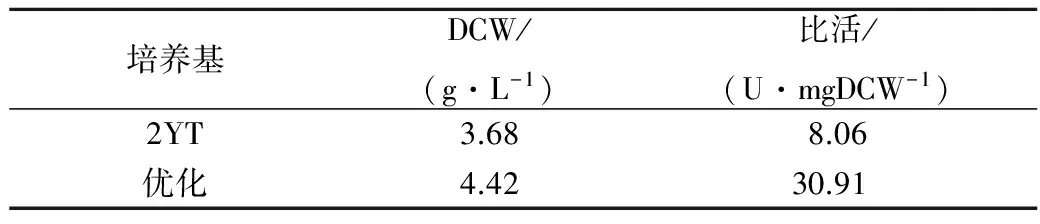
表5 培养基优化前后BAG菌体量和酶活变化Table 5 The change of biomass and enzyme activity ofBAG after the optimization of culture media
2.2 BAG烟腈底物流加工艺
参考丙烯酰胺生产中的丙烯腈流加工艺,对BAG进行烟腈流加实验,并测定生成的烟酰胺质量浓度。如图1,无论底物流加速率快慢(0.38 mL/min、1.5 mL/min),在反应开始都有底物积累现象,终产物质量浓度只到39.12 g/L。考虑到烟腈在水中溶解度较低,所以下步实验考虑采用固体底物分批补料的工艺来提高产物浓度。
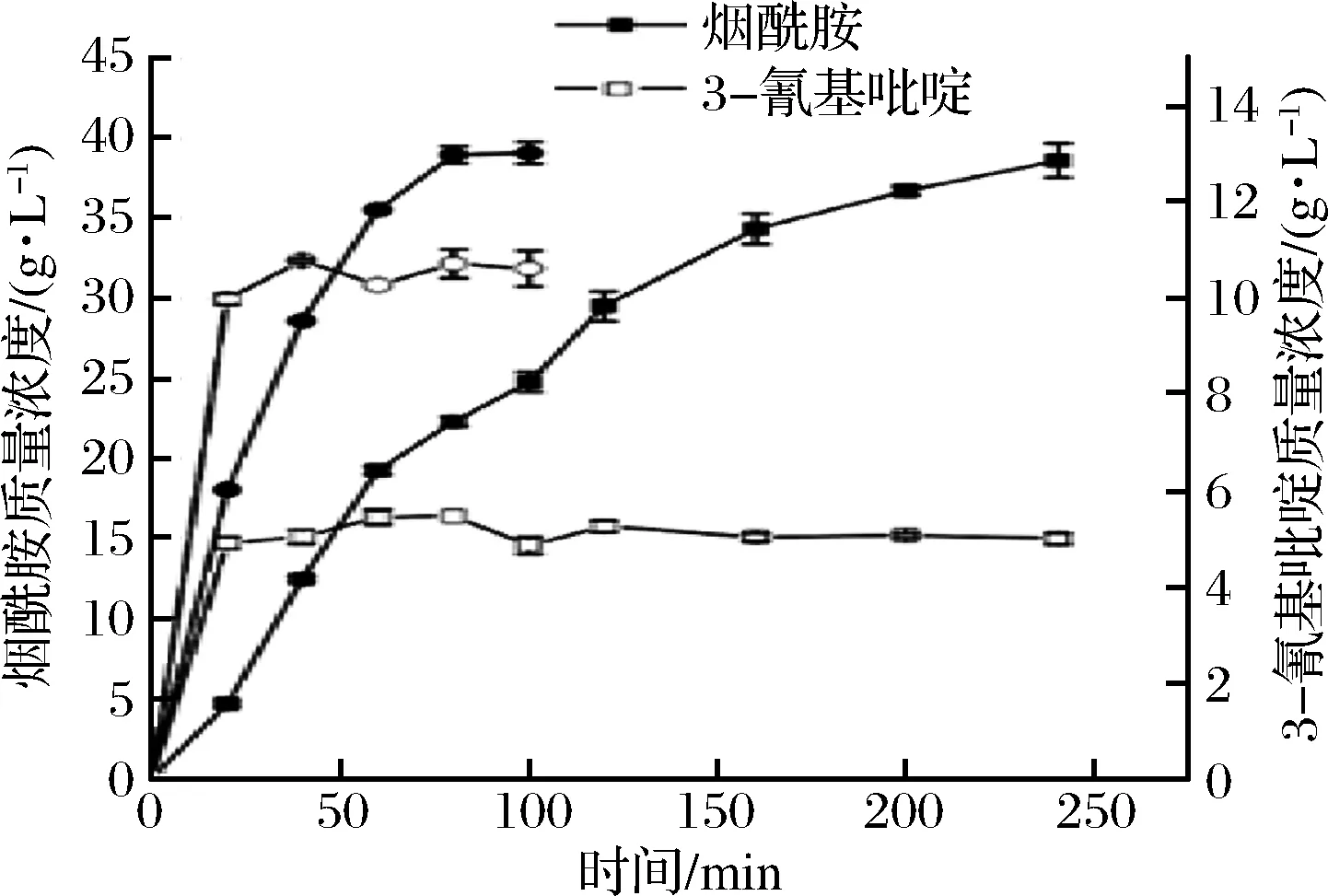
图1 BAG催化烟腈生成烟酰胺Fig.1 Hydration reaction of 3-cyanopyridine to nicotinamide catalyzed by BAG注:流加速率:—■—0.38mL/min、—●—1.5 mL/min,实心表示产物,空心表示底物,形状相同表示流速相同。
2.3 烟腈底物分批补料工艺
为了使反应结束后产物浓度更高,对基因工程重组菌进行烟腈底物分批补料实验,并对生成的烟酰胺浓度进行测定。因为反应体系基本不变而不断添加底物,所以这种催化方式会使产物大量积累,考虑到高浓度烟酰胺对酶和细胞可能会产生影响,首先考察了BAG对产物的耐受性,同时用低分子量腈水合酶大肠杆菌重组菌(BAE)作为对照。如图2,BAE对烟酰胺耐受能力较差,在烟酰胺浓度达到0.8 mol/L时相对酶活已经低于10%,而重组菌BAG对产物耐受能力较强,产物浓度到达0.8 mol/L时相对酶活依旧维持在95%以上。这就说明高分子量腈水合酶具有更优的产物耐受性,其反应持续性较强,在产物大量积累的情况下有能力维持反应初期的催化速率。
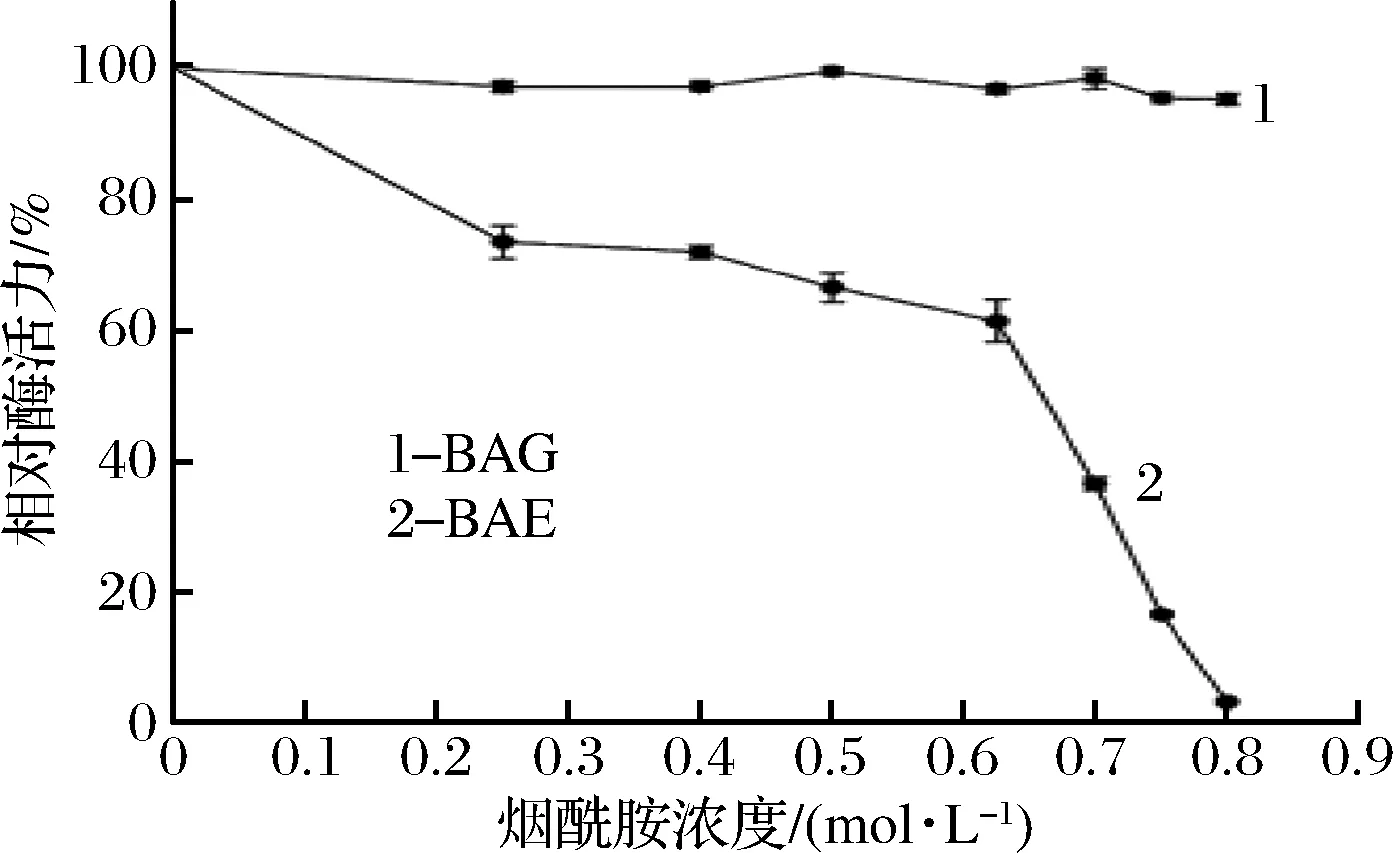
图2 BAG和BAE对烟酰胺的耐受性Fig.2 The tolerance to nicotinamide of BAG and BAE
向体积为30 mL,OD600=8.0 的重组菌BAG中分批加入终浓度为0.2 mol/L(20.82 g/L)的烟腈,待底物完全反应后再加入下一批,当反应速率下降后停止反应结果如图3所示。

图3 BAG分批补料催化烟腈生成烟酰胺(OD600=8.0)Fig.3 Hydration reaction of 3-Cyanopyridine to nicotinamide catalyzed by BAG (OD600=8.0) during the fed-batch reaction
可以看到在反应初期细胞大约10 min可反应完一批底物,且可以在比较长的时间维持这个速率,在产物质量浓度达到250 g/L左右时反应速率依旧不变,随后虽然有所下降,但下降幅度不大,在反应进行到220 min时停止了反应,产物终质量浓度达到了390 g/L,与之前文献报道[11]用重组大肠杆菌催化烟腈8 h产物质量浓度达到240 g/L相比有了很大的提高。
2.4 重组菌BAG高密度发酵工艺
要想使重组基因工程菌应用到工业生产当中就必须实现大肠杆菌的高密度发酵,通过菌体密度的提高实现重组蛋白的大量表达。因此我们以分批补料的策略实现重组大肠杆菌的扩大培养。如图4,经过50 h的发酵,菌体在48 h时生物量达到最大值,为 73.97 gDCW/L(OD600=200);细胞比酶活在44~48 h达到最高,为 42.7 U/mgDCW,与摇瓶发酵相比(30.91 U/mgDCW)有一定的提高;单位发酵液酶活在44 h左右最高,为 2 813 U/mL。与之前文献报道[24]的单位发酵液酶活在60 h达到2 170 U/mL相比有一定提高。整个补料过程中葡萄糖积累量不超过0.3 g/L,乙酸基本没有积累(图5),说明按照比生长速率μ=0.21 /h进行指数流加是合适的。由此,重组菌BAG的高密度发酵工艺初步建立。
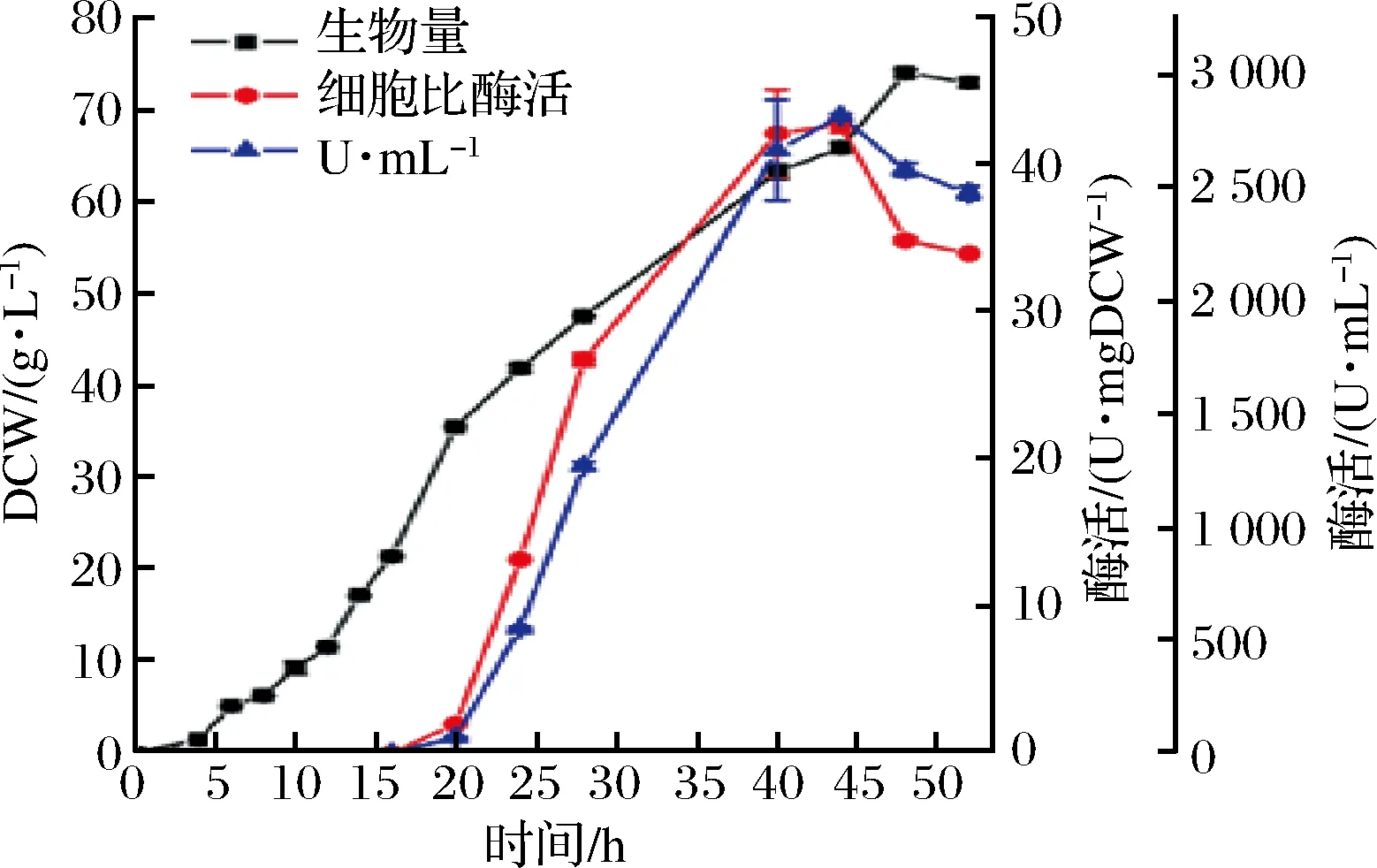
图4 BAG5 L发酵罐分批补料培养Fig.4 Fed-batch culture course in a 5 L fermenter of BAG
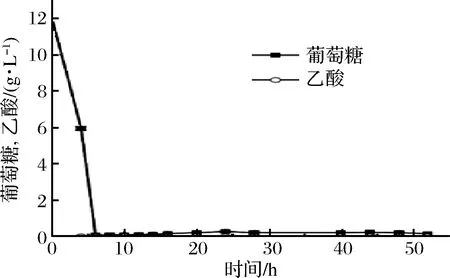
图5 葡萄糖和乙酸质量浓度Fig.5 Concentration of glucose and acetic acid
结合实际的生产情况,取发酵液并将其调整到 OD600=160.0,向总体积为 30 mL 的发酵液中间歇添加 0.4 mol/L(41.64 g/L)烟腈进行底物分批补料反应。如图6,因为菌体生物量较大,反应速率很快,产物质量浓度在1 h即达到537 g/L,随后反应速率因产物抑制已经不高,所以中止反应。
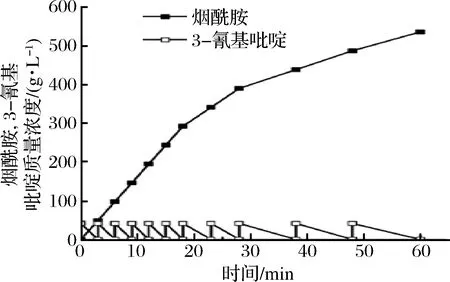
图6 BAG(OD600=160.0)分批补料催化烟腈生成烟酰胺Fig.6 Hydration reaction of 3-Cyanopyridine to nicotinamide catalyzed byBAG (OD600=160.0) during the fed-batch reaction
由图6可知,在高密度菌体的情况下,反应开始时只用较短时间进行1个反应周期,但是随着烟酰胺浓度增高,催化速率都开始下降。R.rhodochrousJ1在 5 h 时能生成 732 g/L 产物[25],与其相比,重组菌BAG还达不到这样高的产物浓度,这与红球菌和大肠杆菌本身对有机物的耐受性差异有关。
R.rhodochrousJ1的发酵周期需要100 h,并且细胞酶活(2 100 U/mL[26])比BAG要低很多。相反,BAG只有R.rhodochrousJ1一半的发酵周期就可以生产出浓度符合工业标准的产品。BAG的生产效率[537 g/(L·h)]是R.rhodochrousJ1[162.8 g/(L·h)][27]的212%。
J1发酵周期较长,反应速率也较慢,催化时间长,生产效率不高,而大肠杆菌与之相比则有自己的优势,在达到工业生产的标准下,其较短的发酵周期和较高的蛋白表达量可以大大提升生产效率,具有广阔的应用前景。
3 结论
本研究通过摇瓶培养基的优化使重组菌BAG的生物量提高到4.42 gDCW/L,细胞比酶活达到30.91 U/mgDCW。通过比较不同的烟腈催化工艺,发现底物分批补料的方法最适合烟酰胺的生产。烟腈经过摇瓶发酵的BAG催化后,最终生成390 g/L烟酰胺。对BAG进行高密度发酵培养,菌体生物量达到73.97 gDCW/L,细胞比酶活为42.70 U/mgDCW,单位发酵液为2 813 U/mL,用较高密度的发酵罐培养后重组菌进行烟腈催化反应,产物质量浓度能达到537 g/L。
参考文献
[1] KOBAYASHI M, SHIMIZU S. Cobalt proteins[J]. European Journal of Biochemistry, 1999, 261(1): 1-9.
[2] YAMADA H, KOBAYASHI M. Nitrile hydratase and its application to industrial production of acrylamide[J]. Bioscience, Biotechnology, and Biochemistry, 1996, 60(9): 1 391-1 400.
[3] PRASAD S, BHALLA TC. Nitrile hydratases (NHases): at the interface of academia and industry[J]. Biotechnology Advances, 2010, 28(6): 725-741.
[4] NOGUCHI T, NOJIRI M, TAKEI KI, et al. Protonation structures of Cys-Sulfinic and Cys-Sulfenic acids in the photosensitive nitrile hydratase revealed byfourier transform infrared spectroscopy[J]. Biochemistry, 2003, 42(40): 11 642-11 650.
[5] NAGASHIMA S, NAKASAKO M, DOHMAE N, et al. Novel non-heme iron center of nitrile hydratase with a claw setting of oxygen atoms[J]. Nature Structural &Molecular Biology, 1998, 5(5): 347-351.
[6] MURAKAMI T, NOJIRI M, NAKAYAMA H, et al. Post-translational modification is essential for catalytic activity of nitrile hydratase[J]. Protein Science, 2000,9(5): 1 024-1 030.
[7] KOBAYASHI M, FUJIWARA Y, GODA M, et al. Identification of active sites in amidase evolutionary relationship between amide bond-and peptidebond-cleaving enzymes[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(22): 11 986-11 991.
[8] DE CARVALHO C CC R. Enzymatic and whole cell catalysis: finding new strategies for old processes[J]. Biotechnology Advances, 2010, 29(1): 75-83.
[9] SCHMID A, DORDICK J S, HAUER B, et al. Industrial biocatalysis today and tomorrow[J]. Nature, 2001, 409(6 817): 258-268.
[10] RAVI S, MATHEW K M, SIVAPRASAD N. A rapid microwave induced synthesis of [carboxyl-14C]-nicotinic acid (vitamin B3) and [carbonyl-14C]-nicotinamide using K14CN[J]. Journal of Radio Analytical and Nuclear Chemistry, 2008, 275(2): 441-444.
[11] LI Bin-feng, SU Jin-huan, TAO Jun-hua. Enzyme and process development for production of nicotinamide[J]. Organic Process Research & Development, 2011, 15(1): 291-293.
[12] NAGASAWA T, TAKEUCHI K, YAMADA H. Occurrence of a cobalt-induced and cobalt-containing nitrile hydratase inRhodococcusrhodochrousJ1[J]. Biochemical and Biophysical Research Communications, 1988, 155(2): 1 008-1 016.
[13] NAGASAWA T, TAKEUCHI K, YAMADA H. Characterization of a new cobalt-containing nitrile hydratase purified from urea-induced cells ofRhodococcusrhodochrousJ1[J]. European Journal of Biochemistry, 1991, 196(3): 581-589.
[14] NAGASAWA T, TAKEUCHI K, NARDI-DEI V, et al. Optimum culture conditions for the production of cobalt-containing nitrile hydratase byRhodococcusrhodochrousJ1[J]. Applied Microbiology and Biotechnology, 1991, 34(6): 783-788.
[15] ZHOU Zhe-min, HASHIMOTO Y, SHIRAKI K, et al. Discovery of posttranslational maturation by self-subunit swapping[J]. Proceedings of the National Academy of Sciences, 2008, 105(39): 14 849-1 4854.
[16] 余越春, 崔文璟, 刘义, 等.RhodococcusrhodochrousJ1腈水合酶在大肠杆菌中的表达策略[J].工业微生物, 2014, 44(2): 14-19.
[17] 张晓欢,崔文璟, 周哲敏. 高分子量腈水合酶在大肠杆菌中的表达策略及重组菌的细胞催化[J]. 微生物学通报, 2016,43(10):2 121-2 128.
[18] LEE S Y. High cell-density culture ofEscherichiacoli[J]. Trends in Biotechnology, 1996, 14(3): 98-105.
[19] YEE L, BLANCH H W. Recombinant protein expression in high cell density fed-batch cultures ofEscherichiacoli[J]. Nature Biotechnology, 1992, 10(12): 1 550-1 556.
[20] SHILOACH J, FASS R. GrowingE.colito high cell density—a historical perspective on method development[J]. Biotechnology Advances, 2005, 23(5): 345-357.
[21] CHOI J H, KEUM K C, LEE S Y, et al. Production of recombinant proteins by high cell density culture ofEscherichiacoli[J]. Chemical Engineering Science, 2006, 61(3): 876-885.
[22] TIAN Yu-xuan, CHEN Jie, YU Hui-min, et al. Overproduction of theEscherichiacolichaperones GroEL-GroES inRhodococcusruberimproves the activity and stability of cell catalysts harboring a nitrile hydratase[J]. Journal of Microbiology and Biotechnology, 2016, 26(2): 337-346.
[23] SHARMA N N, SHARMA M, BHALLA T C. An improved nitrilase-mediated bioprocess for synthesis of nicotinic acid from 3-cyanopyridine with hyperinducedNocardiagloberulaNHB-2[J]. Journal of Industrial Microbiology & Biotechnology, 2010, 38(9): 1 235-1 243.
[24] PEI Xiao-lin, ZHANG Hong-yu, MENG Li-jun, et al. Efficient cloning and expression of a thermostable nitrile hydratase inEscherichiacoliusing an auto-induction fed-batch strategy[J]. Process Biochemistry, 2013, 48(12): 1 921-1 927.
[25] PEI Xiao-lin. Discovery of nitrile hydratase genes and their recombinant expression for the production of nicotinamide[D]. Zhejiang: Zhejiang University, 2013.
[26] NAGASAWA T, SHIMIZUH, YAMADA H. The superiority of the third-generation catalyst,RhodococcusrhodochrousJ1 nitrile hydratase,for industrial production of acrylamide[J]. Applied Microbiology and Biotechnology, 1993, 40(2-3): 189-195.
[27] NAGASAWA T, MATHEW C D, MAUGER J, et al. Nitrile hydratase-catalyzed production of nicotinamide from 3-cyanopyridine inRhodococcusrhodochrousJ1[J]. Applied and Environmental Microbiology, 1988, 54(7): 1 766-1 769.
