陆地棉钾转运体基因GhHAK5的序列特征及表达分析
2018-03-01晁毛妮温青玉张志勇胡根海张金宝王清连河南科技学院现代生物育种河南省协同创新中心河南省棉麦分子生态与种质创新重点实验室河南新乡453003河南省农业科学院河南郑州45000
晁毛妮 温青玉 张志勇 胡根海 张金宝 王 果 王清连,*河南科技学院 / 现代生物育种河南省协同创新中心 / 河南省棉麦分子生态与种质创新重点实验室, 河南新乡 453003; 河南省农业科学院, 河南郑州 45000
钾是植物生长发育所必需的大量元素之一, 它参与多种生理生化过程, 如调节细胞的膨压, 参与蛋白质合成及调节各种酶的活性等[1-2]。钾缺乏会影响作物的产量、品质和抗逆性[3-4]。近年来, 随着我国土壤缺钾面积日益扩大, 钾肥的供需矛盾较为突出[5]。棉花是喜钾作物, 对钾更敏感和更易缺钾[6],缺钾不仅易导致棉花早衰[7-8], 而且对其产量和品质有较大影响[9-11]。鉴定和克隆参与棉花钾高效吸收和利用的基因, 改良棉花的钾吸收和利用特性, 培育钾高效棉花品种已成为近年来研究热点和方向之一。
KUP/HAK/KT钾转运体是植物中最早发现的钾转运体基因家族, 在介导植物对 K+的高亲和吸收、转运与分配等方面都起着关键作用[12-13]。目前, 已从拟南芥、水稻、玉米和大豆等多种作物中对 KUP/HAK/KT家族基因进行了全基因组鉴定和克隆[14-17]。在拟南芥KUP/HAK/KT家族基因中, 以AtHAK5基因的功能研究最为深入, 其在拟南芥根部对 K+的高亲和吸收过程中发挥着重要作用, 低钾胁迫可诱导AtHAK5基因表达量显著升高, 恢复 K+供应时, 其表达量又恢复正常水平, 故 AtHAK5常常被作为拟南芥响应钾缺乏时的标记基因[18-20]。拟南芥AtHAK5的同源基因, 如大麦HvHAK1和番茄LeHAK5, 在低钾环境中表达量均显著上调, 表明这些基因可能参与低钾环境中植物根部对 K+的吸收[21-22]。在棉花KUP/HAK/KT家族基因中, 目前GhKT2基因已被克隆和研究[23-24]。研究发现, GhKT2基因是拟南芥AtKT2的同源基因, 其表达受低钾胁迫诱导, 将GhKT2在拟南芥中过表达, 可显著增加拟南芥根中K+的积累量[23]; 相反地, 通过 VIGS技术沉默GhKT2后, 在充分供 K+和低K+胁迫下棉花幼苗 K+积累量分别降低 15%左右和 25%以上, 此外, 还会导致棉花幼苗对K+的最大吸收速率和亲和能力出现不同程度地下降[24]。同样地, 在烟草中过表达拟南芥钾转运体基因 AtKUP1, 能提高转基因植株叶片中钾含量约 45%[25]。这些研究表明 KUP/HAK/KT钾转运体基因在植物对K+的吸收过程中发挥着重要作用, 鉴定和克隆植物的 KUP/HAK/KT基因, 在提高植物对环境中的K+的吸收方面具有很大潜力。
棉花是重要的经济作物, 也是喜钾作物, 其产量和品质受钾素影响较大[9-11]。克隆和分析陆地棉钾转运体基因, 不仅有助于提高棉花的钾吸收效率,对于提高棉花的产量和品质也具有重要意义。本研究基于已测序的陆地棉基因组序列[26], 通过同源克隆的方法鉴定到陆地棉钾转运体基因 GhHAK5, 并对其序列特征、进化关系、亚细胞定位和表达模式进行了分析。旨在为改良棉花的钾吸收特性, 培育钾高效棉花品种提供重要的基因信息。
1 材料与方法
1.1 田间试验材料
陆地棉品种百棉 1号, 由河南科技学院棉花研究所提供。于2016年4月25日种植于河南科技学院乔谢试验田, 田间管理同常规。取开花期植株的根、茎、叶和花(花瓣和花萼), 初铃期的纤维, 以液氮速冻后, 保存于–80ºC冰箱。
1.2 材料低钾处理
将百棉 1号种植于河南科技学院光照培养室。光照强度为 450 µmol m–2s–1, 光照/黑暗时间为 14 h/10 h, 昼/夜温度为(30~33) °C/(23~26) °C。
挑选整齐一致且饱满的棉花脱绒种子, 用 10%双氧水消毒30 min, 用自来水冲洗3次后, 在自来水中浸泡 12 h, 然后置湿沙中萌发和出苗。子叶展开后转移至1/2 Hoagland’s营养液[27]中培养, 3 d后选取长势一致的幼苗, 一部分在正常的营养液中生长作为对照, 另外一部分用于低钾处理。对照的钾离子浓度为2.5 mmol L–1KCl; 低钾处理的钾离子浓度为0.05 mmol L–1KCl, NaCl用于平衡钾离子, 其他营养液元素组成不变。培养盒尺寸为 20 cm×13 cm×15 cm, 含3.9 L培养液。每盒移入8株幼苗, 24 h通气, 每3 d换一次营养液。处理7 d后, 取对照(CK)和处理(LK)的根系, 以液氮速冻后, 保存于–80°C冰箱。
1.3 RNA的提取和cDNA的合成
使用RNA提取试剂盒(TIANGEN, DP441)提取植株样品的总RNA, 并通过1%琼脂糖凝胶电泳检测RNA质量。按照TaKaRa反转录试剂盒操作说明, 取约2 µg RNA反转录生成cDNA, 保存于–20ºC冰箱。
1.4 GhHAK5基因的鉴定及其CDS序列的扩增
从网站 http://mascotton.njau.edu.cn/下载陆地棉全基因组蛋白质序列数据[26], 建立本地 Blast数据库。以拟南芥的AtHAK5蛋白序列(GenBank登录号为 NP_567404.1)作为查询序列, 运行本地 Blastp检索程序, 获得 AtHAK5在陆地棉中的同源基因, 该基因的 ID号为 Gh_D01G1760。根据陆地棉基因组中该基因的序列信息, 使用 Premier5.0软件设计引物, 引物序列为 F: 5′-TCTCACCGTCATCGCAAC TT-3′; R: 5′-ACTCACTCAGCTTTTCATCCTCT-3′。以cDNA为模板进行PCR扩增, 反应体系为50 µL,包括 10 μL 5× PrimeSTAR GXL buffer (Mg2+plus),4.0 μL 2.5 mmol L–1dNTPs, 1.5 μL 10 mmol L–1正向引物, 1.5 μL 10 mmol L–1反向引物, 1 μL 1.2 U μL–1PrimeSTAR GXL DNA Polymerase (TaKaRa, R050A),5.0 μL cDNA模板和27.0 μL ddH2O。PCR程序为94°C 5 min; 94°C 30 s, 62°C 2.5 min, 32个循环; 72°C 10 min。PCR扩增产物经胶回收试剂盒(Axygen,AP-GX-4)回收后与pMD20-T载体连接, 4ºC连接过夜。连接产物转化到大肠杆菌 DH5α(TIANGEN)中,随机挑选阳性克隆, 经菌液PCR验证后送深圳华大公司测序。
1.5 GhHAK5蛋白的生物信息学分析
利用网站 http://web.expasy.org/protparam/在线分析 GhHAK5蛋白的等电点和分子量; 运用 NCBI的 CDD (Conserved Domain Database)在线工具(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cg i?)进行功能结构域分析; 使用 ClustalX软件进行多序列比对, 用 GeneDoc编辑比对后的序列; 采用TMHMM 在线软件(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构域分析; 使用 WoLF PSORT (http://www.genscript.com/wolf-psort.html)软件预测亚细胞定位。
1.6 进化树的构建
从NCBI网站下载拟南芥和水稻KUP/HAK/KT家族基因的蛋白序列。利用MEGA5.02软件中的邻接法(Neighbor-Joining method, NJ)构建系统进化树[28], 主要参数设置为基因树稳健性检测, bootstrap法, 1000次重复; 距离模型选择p-distance和空位缺失数据的处理选择pairwise deletion。
1.7 亚细胞定位
采用 Golden Gate克隆法[29]构建亚细胞定位用瞬时表达载体 pBWA(V)HS-GhHAK5-osgfp。首先,根据GhHAK5的CDS序列(不含终止密码子)设计特异性引物, GhHAK5-GFP-F: 5′-CAGTCACCTGCAA AACAAC ATGGAAGAGAACAAACCAGG-3′ (虚线和实线分别为IIS型内切酶Aar I的识别序列和切割位点); GhHAK5-GFP-R: 5′-CAGTCACCTGCAAAA TACA AATCTCGTACGCCATTCCAA-3′(虚 线 和 实线分别为 IIS型内切酶 Aar I的识别序列和切割位点)。接着, 通过PCR扩增将酶切位点引入GhHAK5基因完整CDS序列(不含终止密码子)的上下游。对载体pBWA(V)HS-osgfp和扩增的片段分别用IIS型内切酶Bsa I和Aar I酶切, 回收后进行连接。将连接产物转化大肠杆菌感受态细胞 DH5α, 涂布含有卡那霉素的LB抗性平板, 37ºC培养12 h后, 挑单克隆并进行菌液PCR。阳性菌液提取质粒后进行酶切验证, 将含有目的基因片段的菌液送深圳华大公司测序。将测序正确的菌液用质粒大提试剂盒(TIANGEN, DP117)进行质粒提取, 并对质粒进行浓缩, 以备用于水稻原生质体的转化。
参考 Chen等[30]的亚细胞定位实验方法。首先,在PEG介导下将空载体(pBWA(V)HS-osgfp)和瞬时表达载体(pBWA(V)HS-GhHAK5-osgfp)的质粒分别转入水稻原生质体。然后, 于光照培养箱中28°C条件下暗培养24~48 h。最后, 采用激光共聚焦显微镜(FV10-ASW, OLYMPUS), 于激发光波长480 nm和发射光波长510 nm条件下观察GFP信号, 确定蛋白的亚细胞定位。
1.8 GhHAK5基因的表达特性分析
采用qRT-PCR检测GhHAK5基因在棉花不同组织及低钾处理条件下的表达。棉花内参基因 Actin(GenBank登录号为AY907703.1)用于标准化每个样品的转录水平。GhHAK5和内参基因Actin的荧光定量 PCR引物为: GhHAK5-F: 5′-GTAAGGACGGGTG GATA-3′; GhHAK5-R: 5′-AGTAAAGCAGGCAAGG TA-3′; Actin-F: 5′-GACCGCATGAGCAAGGAGAT-3′;Actin-R: 5′-GCTGGAAGGTGCTGAGTGAT-3′。qRTPCR体系总体积 20 μL, 包括 10 μL 2× SYBR Premix Ex Taq (TaKaRa, RR820L), 0.8 μL 10 mmol L–1正向引物, 0.8 μL 10 mmol L–1反向引物, 1 μL cDNA 和7.4 μL ddH2O。每个样品设3次重复, 于BIOER荧光定量PCR仪上扩增, 反应程序为95°C 30 s; 95°C 5 s, 60°C 20 s, 40个循环后增加熔解曲线。采用 2–ΔΔCt法计算基因在不同样品中的相对表达量。
2 结果与分析
2.1 陆地棉GhHAK5的序列特征分析
通过同源克隆的方法鉴定到陆地棉基因组中[26]与拟南芥钾转运体基因 AtHAK5高度同源的基因,其 ID号为 Gh_D01G1760, 本研究将其命名为GhHAK5。基于该基因的CDS序列设计特异性引物,利用PCR的方法从陆地棉品种百棉1号的根cDNA中扩增得到与 GhHAK5基因大小一致的 cDNA序列。测序结果表明, 该序列长2495 bp, 其中CDS全长为2451 bp, 与陆地棉基因组中GhHAK5基因的相似性达99.70%。进一步对GhHAK5基因编码的蛋白分析发现, GhHAK5蛋白编码816个氨基酸, 分子量和等电点分别为91.23 kD和8.15。运用NCBI的CDD在线工具进行GhHAK5蛋白功能保守域的分析表明,GhHAK5是钾转运体超级家族(K-trans superfamily)的一员, 并具有KUP/HAK/KT钾转运体家族的保守结构域“K-trans” (Pfam02705)(图 1-A)。将 GhHAK5蛋白与拟南芥 KUP/HAK/KT家族成员进行氨基酸序列比对分析表明, GhHAK5与拟南芥 KUP/HAK/KT家族成员一样, 均具有该家族的标志性序列GXXXGDXXXSPLY (图1-B)。利用TMHMM工具进行跨膜区分析表明, GhHAK5含有11个跨膜区(图 1-C)。
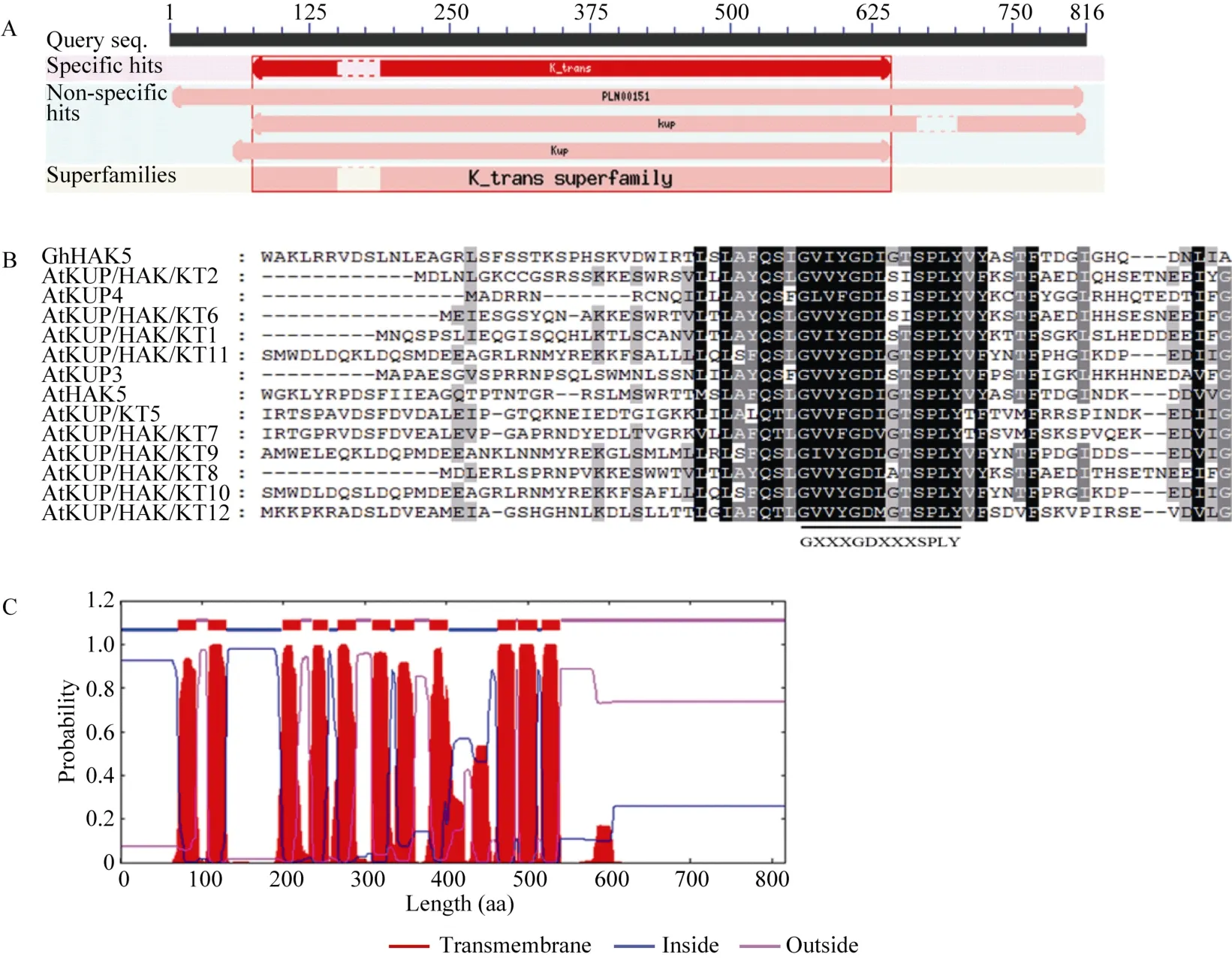
图1 陆地棉GhHAK5蛋白的序列分析Fig. 1 Sequence analysis of GhHAK5 protein in upland cotton (Gossypium hirsutum L.)
2.2 陆地棉GhHAK5蛋白的进化关系分析
系统进化树表明, GhHAK5蛋白在进化上与拟南芥AtHAK5亲缘关系最近, 同源性达58.40%, 其次是水稻的 OsHAK5, 同源性达 55.90% (图 2)。在植物中, KUP/HAK/KT家族基因大致可划分为4个进化簇 Cluster I~IV[31], GhHAK5在进化上与AtHAK5和OsHAK5同属Cluster I进化簇(图2)。
2.3 陆地棉GhHAK5蛋白的亚细胞定位
利用WoLF PSORT软件预测GhHAK5蛋白定位于质膜上。通过水稻原生质体瞬时表达系统进一步确定 GhHAK5的亚细胞定位情况, 对于空载体在质膜、细胞质和细胞核中均可检测到绿色荧光蛋白, 而对于含有 GhHAK5基因的表达载体只在质膜上检测到绿色荧光蛋白(图 3), 表明GhHAK5是一个定位于质膜的蛋白, 该结果与预测结果一致。
2.4 陆地棉GhHAK5基因的表达特性分析
qRT-PCR分析表明, GhHAK5在棉花的不同组织中表达量存在明显差异, 在根中表达量最高, 在茎、叶、花瓣、花萼和纤维中表达量很低(图4-A); 低钾处理后, GhHAK5在根中表达量显著上调(图4-B),表明它是一个响应低钾胁迫诱导表达的基因。
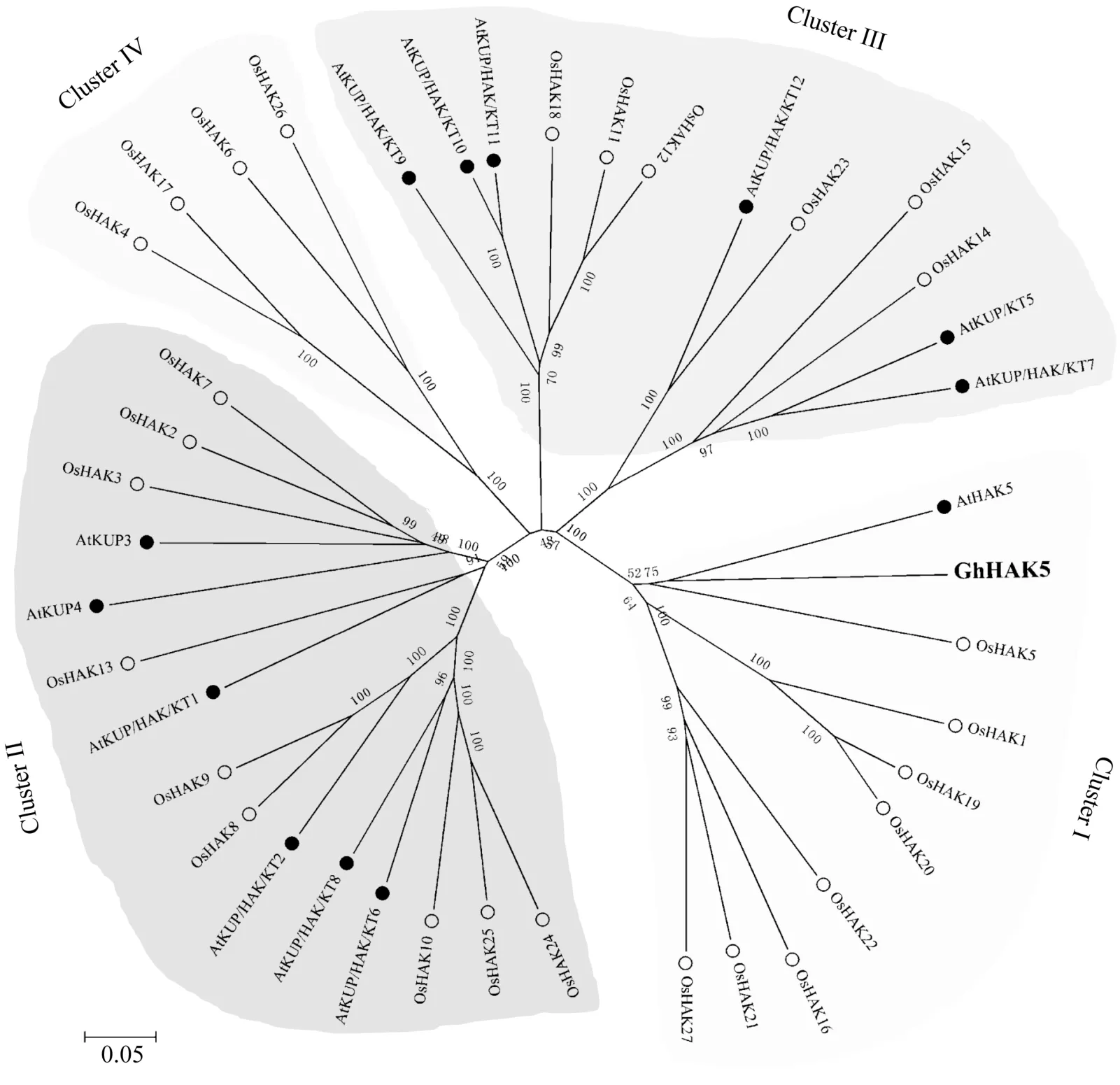
图2 陆地棉GhHAK5蛋白与拟南芥和水稻KUP/HAK/KT家族成员的进化关系分析Fig. 2 Phylogenetic relationships between GhHAK5 protein and KUP/HAK/KT family members in Arabidopsis and rice
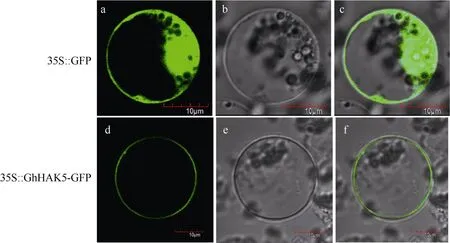
图3 陆地棉GhHAK5蛋白的亚细胞定位Fig. 3 Subcellular localization of GhHAK5 protein in upland cotton (Gossypium hirsutum L.)
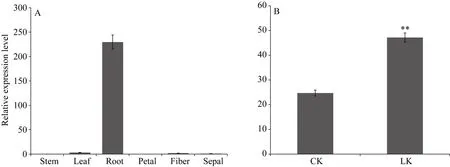
图4 陆地棉GhHAK5基因表达水平Fig. 4 Expression level of GhHAK5 gene in upland cotton (Gossypium hirsutum L.)
3 讨论
植物生长发育过程中, 其根际土壤经常处于缺钾状态[32]。低钾环境中, 常常需要高亲和性钾转运体基因的参与, 以完成植物根部对 K+的吸收[33]。AtHAK5是拟南芥KUP/HAK/KT钾转运体家族中的一个高亲和性钾转运体, 在根中大量表达, 也是唯一对低钾胁迫产生响应的基因[12,34]。通过同源克隆的方法, 本研究在陆地棉基因组中鉴定到与拟南芥钾转运体基因 AtHAK5高度同源的 GhHAK5基因,该基因CDS全长为2451 bp, 编码816个氨基酸, 分子量和等电点分别为91.23 kD和8.15。已有研究表明,植物的 KUP/HAK/KT家族基因在结构上一般都具有10~14个跨膜区和一些相对保守的氨基酸序列[33,35],如拟南芥 KUP/HAK/KT家族基因均有数目不等的跨膜区[36]。本研究发现, GhHAK5蛋白具有11个跨膜区, 且具有KUP/HAK/KT钾转运体家族的保守结构域“K-trans”(Pfam02705)和标志性序列 GXXXGD XXXSPLY (图1), 表明其可能是编码钾转运体蛋白的基因。
前人研究发现植物的 KUP/HAK/KT家族基因大致可划分为4个进化簇Cluster I~IV, 不同进化簇的基因在表达和功能上存在差异[31]。Cluster I成员多为典型的高亲和性钾转运体, 主要在植物的根中行使功能, 其表达受低钾胁迫的诱导[19,21-22]。Cluster II成员则在各个组织均有表达, 不仅参与了植物对K+的吸收, 也起着调节植物生长发育的作用[37]。对Cluster III成员研究较少, 已有研究表明其除了具有钾吸收功能外, 在表达上还受盐胁迫诱导[38]。Cluster IV成员是一种具有Na+转运功能的钾转运体[39]。本研究表明, GhHAK5蛋白在进化上与拟南芥 AtHAK5亲缘关系最近, 其次是与水稻的 OsHAK5, 且与AtHAK5和OsHAK5同属Cluster I进化簇(图2), 表明其可能具有与拟南芥AtHAK5基因相似功能, 并可能作为高亲和性 K+转运体参与棉花根部低钾环境中的 K+吸收。与此功能相一致的是, GhHAK5蛋白与AtHAK5蛋白一样, 是一个定位于质膜的蛋白(图3)。
植物主要通过根从土壤中获取营养元素, 因此,根中大量表达的 KUP/HAK/KT可能主要参与植物根对土壤中 K+的吸收。在拟南芥中, KUP/HAK/KT家族具有13个成员, 它们呈现出不同表达模式。AtHAK5在根中表达量最高, 其表达具有组织特异性[12,34]; AtKUP1、AtKUP2、AtKUP3和 AtKUP4基因在拟南芥的根、茎、叶和花中均有表达, 不存在表达的组织特异性[37]; AtHAK11在盐胁迫处理后的根中表达量显著上调, 可能参与植物响应高盐胁迫的应对机制[38]; 拟南芥KUP/HAK/KT家族成员不同的表达模式暗示着它们可能行驶着不同的功能。本研究发现, GhHAK5在根中表达量最高, 在茎、叶、花瓣、花萼和纤维中表达量很低(图4-A), 推测GhHAK5在棉花根部对环境中 K+的吸收起重要作用。GhHAK5在低钾处理的根中表达量显著上调(图4-B), 表明其表达受低钾胁迫诱导, 暗示着GhHAK5在陆地棉应对钾缺乏方面可能扮演着重要角色。
在植物中, 许多研究发现不同营养元素的信号途径之间可能存在交叉, 相关离子转运蛋白基因的表达可能参与促进植物体内营养元素平衡的过程[40]。例如, 拟南芥的 AAR6基因在氮、磷和钾缺乏条件下,表达量均显著上调[41]; 陆地棉的磷酸盐转运蛋白基因GhPT6和GhPT14同时被低磷和低钾胁迫诱导并上调表达[42], 表明磷和钾信号途径可能存在相互作用。由于植物在生长过程中往往不是受单一营养元素胁迫的影响, 而是同时受到多种营养元素胁迫的共同影响, 故鉴定那些参与多种元素共同营养信号转导途径的基因, 对于同步改良棉花的营养元素吸收效率可能具有重要意义。在棉花中, 响应低氮胁迫诱导的二硫键异构酶基因GhPDI[43], 响应低钾胁迫的钾转运体基因 GhKT2[23]等已被克隆, 不过研究者更侧重研究单一元素缺乏对植物生长状态影响,关于离子信号途径之间相互作用的研究很少。本研究克隆的钾转运体基因GhHAK5在根中表达量最高,且被低钾胁迫诱导上调表达(图4), 不过其表达是否受其他营养元素影响还不清楚。今后的研究将在陆地棉中过表达GhHAK5基因, 探讨转基因植株对氮、磷、钾和其他营养元素的吸收效率, 这有助于更好地理解棉花GhHAK5基因的功能及离子信号途径相互作用的分子机制。
4 结论
从陆地棉基因组中鉴定到一个钾转运体基因GhHAK5, 其 CDS全长为2451 bp, 编码816个氨基酸, 分子量和等电点分别为91.23 kD和8.15。GhHAK5具有 KUP/HAK/KT钾转运体家族的保守结 构 域 “K-trans” (Pfam02705) 和 标 志 性 序 列GXXXGDXXXSPLY, 并具有11个跨膜区, 是一个定位于质膜的蛋白。在进化上, GhHAK5与拟南芥的AtHAK5亲缘关系最近, 其次是与水稻的 OsHAK5,它们同属ClusterI进化簇。GhHAK5主要在根中表达,并受外界低钾环境的诱导。这些结果可为今后研究GhHAK5基因的功能及培育钾高效棉花品种奠定基础。
[1]Véry A A, Sentenac H. Molecular mechanisms and regulation of K+transport in higher plants. Annu Rev Plant Biol, 2003, 54:575–603
[2]Grabov A. Plant KT/KUP/HAK potassium transporters: single family-multiple functions. Ann Bot, 2007, 99: 1035–1041
[3]张炎, 李宁, 胡伟, 高媛, 姚银坤, 汤明尧. 钾肥用量对棉花蕾铃脱落及产量品质的影响. 新疆农业科学, 2012, 11:2088–2095 Zhang Y, Li N, Hu W, Gao Y, Yao Y K, Tang M Y. Bud & boll falling, yield and quality of cotton affected by rates of potassium.Xinjiang Agric Sci, 2012, 11: 2088–2095 (in Chinese with English abstract)
[4]Mian A, Oomen R J, Isayenkov S, Sentenac H, Maathuis F J,Véry A A. Over-expression of an Na+- and K+-permeable HKT transporter in barley improves salt tolerance. Plant J, 2011, 68:468–479
[5]高祥照, 马文奇. 我国耕地土壤养分变化与肥料投入状况. 植物营养与肥料学报, 2000, 6: 363–369 Gao X Z, Ma W Q. Changes of soil nutrient contents and input of nutrientsin arable of China. Plant Nutr Fert Sci, 2000, 6: 363–369(in Chinese with English abstract)
[6]张志勇, 王清连, 李召虎, 段留生, 田晓莉. 缺钾对棉花幼苗根系生长的影响及其生理机制. 作物学报, 2009, 35: 718–723 Zhang Z Y, Wang Q L, Li Z H, Duan L S, Tian X L. Effect of potassium deficiency on root growth of cotton (Gossypium hirsutum L.) seedlings and its physiological mechanisms involved. Acta Agron Sin, 2009, 35: 718–723 (in Chinese with English abstract)
[7]孔祥强, 罗振, 李存东, 董合忠. 棉花早衰的分子机理研究进展. 棉花学报, 2015, 27: 71–79 Kong X Q, Luo Z, Li C D, Dong H Z. Molecular mechanisms of premature senescence in cotton. Cott Sci, 2015, 27: 71–79 (in Chinese with English abstract)
[8]刘冬青, 刘锐. 转基因抗虫棉早衰与土壤肥力的相关性分析.土壤肥料, 2002, (6): 41–42 Liu D Q, Liu R. Correlation analysis between soil fertility and premature senescence of transgenic cotton. Soils & Fert, 2002,(6): 41–42 (in Chinese with English abstract)
[9]Pettigrew W, Meredith Jr W. Dry matter production, nutrient uptake, and growth of cotton as affected by potassium fertilization.J Plant Nutr, 1997, 20: 531–548
[10]李书田, 邢素丽, 张炎, 崔荣宗. 钾肥用量和施用时期对棉花产量品质和棉田钾素平衡的影响. 植物营养与肥料学报, 2016,22: 111–121 Li S T, Xing S L, Zhang Y, Cui R Z. Application rate and time of potash for high cotton yield, quality and balance of soil potassium.Plant Nutr Fert Sci, 2016, 22: 111–121 (in Chinese with English abstract)
[11]宋美珍, 毛树春, 邢金松, 杨惠元. 钾素对棉花光合产物的积累及产量形成的影响. 棉花学报, 1994, 6(增刊): 52–57 Song M Z, Mao S C, Xing J S, Yang H Y. Effects of potassium on photosynthetic matter accumulation and yield. Acta Gossipii Sin,1994, 6(suppl): 52–57 (in Chinese with English abstract)
[12]Ahn S J, Shin R, Schachtman D P. Expression of KT/KUP genes in Arabidopsis and the role of root hairs in K+uptake. Plant Physiol, 2004, 134: 1135–1145
[13]Véry A A, Nieves-Cordones M, Daly M, Khan I, Fizames C,Sentenac H. Molecular biology of K+transport across the plant cell membrane: what do we learn from comparison between plant species? J Plant Physiol, 2014, 171: 748–769
[14]Mäser P, Thomine S, Schroeder J I, Ward J M, Hirschi K, Sze H,Talke I N, Amtmann A, Maathuis F J, Sanders D, Harper J F,Tchieu J, Gribskov M, Persans M W, Salt D E, Kim S A, Guerinot M L. Phylogenetic relationships within cation transporter families of Arabidopsis. Plant Physiol, 2001, 126: 1646–1667
[15]Yang Z, Gao Q, Sun C, Li W, Gu S, Xu C. Molecular evolution and functional divergence of HAK potassium transporter gene family in rice (Oryza sativa L.). J Genet Genomics, 2009, 36:161–172
[16]Zhang Z, Zhang J, Chen Y, Li R, Wang H, Wei J. Genome-wide analysis and identification of HAK potassium transporter gene family in maize (Zea mays L.). Mol Biol Rep, 2012, 39:8465–8473
[17]晁毛妮, 温青玉, 张晋玉, 张志勇, 董洁, 于亚鑫. 大豆KUP/HAK/KT钾转运体基因家族的鉴定与表达分析. 西北植物学报, 2017, 37: 239–249 Chao M N, Wen Q Y, Zhang J Y, Zhang Z Y, Dong J, Yu Y X.Identification and expression analysis of KUP/HAK/KT potassium transporter gene family in soybean [Glycine max (L.)Merr.]. Acta Bot Boreal-Occident Sin, 2017, 37: 239–249 (in Chinese with Englishabstract)
[18]Shin R, Schachtman D P. Hydrogen peroxide mediates plant root cell response to nutrient deprivation. Proc Natl Acad Sci USA,2004, 101: 8827–8832
[19]Gierth M, Mäser P, Schroeder J I. The potassium transporter AtHAK5 functions in K+deprivation-induced high-affinity K+uptake and AKT1 K+channel contribution to K+uptake kinetics in Arabidopsis roots. Plant Physiol, 2005, 137: 1105–1114
[20]Zhao S, Zhang M L, Ma T L, Wang Y. Phosphorylation of ARF2 relieves its repression of transcription of the K+transporter gene HAK5 in response to low potassium stress. Plant Cell, 2016, 28:3005–3019
[21]Wang Y H, Garvin D F, Kochian L V. Rapid induction of regulatory and transporter genes in response to phosphorus,potassium, and iron deficiencies in tomato roots. Evidence for cross talk and root/rhizosphere-mediated signals. Plant Physiol,2002, 130: 1361–1370
[22]Santa-María G E, Rubio F, Dubcovsky J, Rodríguez-Navarro A.The HAK1 gene of barley is a member of a large gene family and encodes a high-affinity potassium transporter. Plant Cell, 1997, 9:2281–2289
[23]徐娟. 棉花钾离子通道基因GhAKT1和转运体基因GhKT2的克隆及功能分析. 中国农业大学博士学位论文, 北京, 2014 Xu J. Cloning and Functional Characterization of Potassium Channel Gene GhAKT1 and Potassium Transporter Gene GhKT2 from Cotton (Gossypium hirsutum L.). PhD Dissertation of China Agricultural University, Beijing, China, 2014 (in Chinese with English abstract)
[24]穆春, 安静, 李婷婷, 王逸茹, 张明才, 田晓莉. 应用病毒诱导基因沉默技术研究棉花GhAKT1和GhKT2基因的功能. 棉花学报, 2017, 29: 40–49 Mu C, An J, Li T T, Wang Y R, Zhang M C, Tian X L. Functional characterization of GhAKT1 and GhKT2 in cotton (Gossypium hirsutum L.) using virus-induced gene silencing. Cott Sci, 2017,29: 40–49 (in Chinese with English abstract)
[25]郭兆奎, 杨谦, 姚泉洪, 万秀清, 颜培强. 转拟南芥AtKup1基因高含钾量烟草获得. 中国生物工程杂志, 2005, 25: 24–28 Guo Z K, Yang Q, Yao Q H, Wan X Q, Yan P Q. Transgenic tobacco with Arabidopsis thaliana AtKup1 gene has high potassium content in leaves. J Chin Biotechnol, 2005, 25: 24–28(in Chinese with English abstract)
[26]Zhang T, Hu Y, Jiang W, Fang L, Guan X, Chen J, Zhang J, Saski C A, Scheffler B E, Stelly D M, Hulse-Kemp A M, Wan Q, Liu B,Liu C, Wang S, Pan M, Wang Y, Wang D, Ye W, Chang L, Zhang W, Song Q, Kirkbride R C, Chen X, Dennis E, Llewellyn D J,Peterson D G, Thaxton P, Jones D C, Wang Q, Xu X, Zhang H,Wu H, Zhou L, Mei G, Chen S, Tian Y, Xiang D, Li X, Ding J,Zuo Q, Tao L, Liu Y, Li J, Lin Y, Hui Y, Cao Z, Cai C, Zhu X,Jiang Z, Zhou B, Guo W, Li R, Chen Z J. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1)provides a resource for fiber improvement. Nat Biotechnol, 2015,33: 531–537
[27]Zhang Z, Chao M, Wang S, Bu J, Tang J, Li F, Wang Q, Zhang B.Proteome quantification of cotton xylem sap suggests the mechanisms of potassium-deficiency-induced changes in plant resistance to environmental stresses. Sci Rep, 2016, 6: 21060
[28]Kumar S, Nei M, Dudley J, Tamura K. MEGA: a biologistcentric software for evolutionary analysis of DNA and protein sequences. Brief Bioinform, 2008, 9: 299–306
[29]Engler C, Kandzia R, Marillonnet S. A one pot, one step,precision cloning method with high throughput capability. PLoS One, 2008, 3: e3647
[30]Chen S, Tao L, Zeng L, Vega-Sanchez M E, Umemura K, Wang G L. A highly efficient transient protoplast system for analyzing defence gene expression and protein-protein interactions in rice.Mol Plant Pathol, 2006, 7: 417–427
[31]宋毓峰, 张良, 董连红, 靳义荣, 史素娟, 白岩, 刘朝科, 冯广林, 冯祥国, 王倩, 刘好宝. 植物KUP/HAK/KT家族钾转运体研究进展. 中国农业科技导报, 2013, 15: 92–98 Song Y F, Zhang L, Dong L H, Jin Y R, Shi S J, Bai Y, Liu C K,Feng G L, Feng X G, Wang Q, Liu H B. Research progress on KUP/HAK/KT potassium transporter family in plant. J Agric Sci Tech China, 2013, 15: 92–98 (in Chinese with English abstract)
[32]Ashley M K, Grant M, Grabov A. Plant responses to potassium deficiencies: a role for potassium transport proteins. J Exp Bot,2006, 57: 425–436
[33]Rodrı́guez-Navarro A. Potassium transport in fungi and plants.Biochim Biophys Acta, 2000, 1469: 1–30
[34]Rubio F, Santamaría G E, Rodrígueznavarro A. Cloning of Arabidopsis and barley cDNAs encoding HAK potassium transporters in root and shoot cells. Physiol Plant, 2000, 109:34–43
[35]Véry A A, Sentenac H. Molecular mechanisms and regulation of K+transport in higher plants. Annu Rev Plant Biol, 2003, 54:575–603
[36]Sato Y, Nanatani K, Hamamoto S, Shimizu M, Takahashi M,Tabuchi-Kobayashi M, Mizutani A, Schroeder J I, Souma S,Uozumi N. Defining membrane spanning domains and crucial membrane-localized acidic amino acid residues for K+transport of a Kup/HAK/KT-type Escherichia coli potassium transporter. J Biochem, 2014, 155: 315–323
[37]Kim E J, Kwak J M, Uozumi N, Schroeder J I. AtKUP1: an Arabidopsis gene encoding high-affinity potassium transport activity. Plant Cell, 1998, 10:51-62
[38]Maathuis F J. The role of monovalent cation transporters in plant responses to salinity. J Exp Bot, 2006, 57: 1137–1147
[39]Takahashi R, Nishio T, Ichizen N, Takano T. High-affinity K+transporter PhaHAK5 is expressed only in salt-sensitive reed plants and shows Na+permeability under NaCl stress. Plant Cell Rep, 2007, 26: 1673–1679
[40]Grossman A, Takahashi H. Macronutrient utilization by photosynthetic eukaryotes and the fabric of interactions. Annu Rev Plant Physiol Plant Mol Biol, 2001, 52: 163–210
[41]Coello P, Polacco J C. ARR6, a response regulator from Arabidopsis, is differentially regulated by plant nutritional status.Plant Sci, 1999, 143: 211–220
[42]晁毛妮, 张志勇, 宋海娜, 李成奇, 张新, 胡根海, 张金宝, 王清连. 陆地棉 Pht1家族成员的全基因组鉴定及表达分析. 棉花学报, 2017, 29: 59–69 Chao M N, Zhang Z Y, Song H N, Li C Q, Zhang X, Hu G H,Zhang J B, Wang Q L. Genome-wide identification and expression analysis of Pht1 family genes in cotton (Gossypium hirsutum L.). Cott Sci, 2017, 29: 59–69 (in Chinese with English abstract)
[43]韩璐. 棉花氮高效基因型筛选及相关基因的克隆. 石河子大学硕士学位论文, 新疆石河子, 2011 Han L. Nitrogen Efficiency of Genotype Selection and Related Gene Cloning in Cotton. MS Thesis of Shihezi University, 2011,Shihezi, China (in Chinese with English abstract)
