水稻花器官数目突变体mf2的鉴定和基因定位
2018-03-01严贤诚陈立凯罗玉花罗文龙陈志强
严贤诚 陈立凯 罗玉花 罗文龙 王 慧 郭 涛 陈志强
华南农业大学国家植物航天育种工程技术研究中心, 广东广州 510642
花器官作为种子植物重要的繁殖器官, 一直是植物发育学研究中的热点之一。植物的花器官发育是一个十分复杂的过程, 包括花分生组织起始、花分生组织属性的特化与保持, 花器官原基的起始、花器官属性的特化、花分生细胞的终止以及花器官的最终成熟[1]。植物开花涉及大量的基因调控, 确定花器官的数目、属性等[2]。随着对双子叶模式植物拟南芥(Arabidopsis thaliana)、金鱼草(Antirrhinum majus)和矮牵牛(Petunia hybrida)花器官同源异型突变体的研究, 相继提出了“ABC”模型、“ABCD”模型和“ABCDE”模型[3]。水稻(Oryza sativa L.)作为单子叶植物的重要模式植物, 对于“ABCDE”模型也基本适用, 表现出一定的保守性和多样性[4]。
水稻花器官的基本结构单位是小穗, 1个小穗包含 1朵小花, 小花具四轮结构, 由外到内分别是内/外稃、浆片、雄蕊和雌蕊[5]。水稻花器官的变异不仅包括各轮结构的同源异型转化, 而且包括各轮结构数目的变化, 甚至产生新的轮结构。目前已报道的有关调控水稻花器官数目的基因主要有PAP1[6]、RFL[7]、LHS1/OsMADS1[8]、OsLRK1[9]、OsFOR1[10]、FON1[11]、FON2/FON4[12,13]、OsAP2-1[14]、SNB[15]、JMJ706[16]、EG1[17]、SL1[18]、MFS1[19]、NSG[20]、EG2/OsJAZ1[21]、OsIG1[22]、OsMADS32[23]和 YABBY4[24]。其中JMJ706通过编码H2K9去甲基酶调控花器官的发育; EG1-EG2和 YABBY4分别通过茉莉酸和赤霉素对花器官发育进行调控, 在表观遗传调控和激素调控上很好地丰富了水稻花器官发育调控路径。
虽然目前已找到大量的水稻花器官突变相关基因, 但是水稻花器官的调控机制(花分生组织确定性以及花器官属性的特化)仍然不够清晰。李云峰等[25]曾报道了一个多小花小穗突变体 mf1, 其一个小穗内出现两朵及以上小花。本研究通过重离子诱变获得的花器官数目突变体, 其四轮器官均有增多现象,个别器官还表现出同源异型转化, 暂命名为multi-floret 2 (mf2)。本研究对该突变体进行了表型鉴定、遗传分析和基因初步定位, 并借助定位区间的基因注释功能对候选基因进行筛选, 为候选基因的克隆及功能研究奠定基础。
1 材料与方法
1.1 供试材料
突变体 mf2是在籼稻航恢 7号重离子诱变 M2中筛选得到的一个花器官突变体, 经过多代的自交繁殖, 其性状稳定遗传。以 mf2为父本, 分别与02428 (粳稻)和 Francis (粳稻)配制杂交组合用于遗传分析。以02428×mf2的F2群体作为初步定位群体,以Francis×mf2的F2群体作为精细定位群体。
重离子诱变处理方法: 航恢7号干种子于2014年 4月在兰州重离子加速器(HIRFL)国家实验室进行辐照。重离子为12C6+, 80.55 MeV u–1, 束斑直径4 cm, 束斑均匀度>90%, 流强 5~20 nA。辐照剂量为80 Gy。
1.2 农艺性状调查
突变体材料mf2和野生型(WT)同期种植。全生育期观察 mf2和 WT的田间表型, 重点调查其抽穗期、株高、穗长、穗数、结实率、千粒重等主要农艺性状。
1.3 形态学和组织学观察
1.3.1 解剖观察 于抽穗期分别取突变体和野生型的未散粉颖花, 在 SZ780体视显微镜下解剖、观察并拍照。
1.3.2 石蜡切片观察 随机取突变体和野生型临近开放的颖花, 用FAA固定液(90 mL 70%酒精、5 mL福尔马林、5 mL冰醋酸)固定。经酒精梯度脱水、氯仿梯度透明后采用常规石蜡切片程序包埋。用石蜡切片机连续切片。用苯胺番红-固绿双重染色, 中性树胶封片保存。用Olympus SZX10型光学显微镜观察并拍照。
1.3.3 电镜观察 取突变体和野生型水稻幼穗用2.5%~5.0%的戊二醛固定。为了让固定液能够充分浸入材料, 将幼穗放入固定液时先抽气3~4 min, 在4℃条件下固定1~3 h后吸出固定液, 用0.1 mol L–1pH 6.8的PBS漂洗1 h, 期间换漂洗液3~4次。吸出PBS后用乙醇梯度脱水干燥。经无水乙醇干燥后使用导电双面胶黏贴在载物台上, 喷金后用XL-30-ESEM型号电镜扫描观察。
1.4 分子标记获取与开发
参照 gramene数据库(http://www.gramene.org/)的 SSR引物序列, 用 SSRHunter1.3软件搜索粳稻Nipponbare部分SSR引物。根据粳稻Nipponbare和籼稻 93-11的序列差异比对设计 InDel引物, 用Primer Premier 5.0软件设计引物, 引物均由上海生工生物工程有限公司合成。
1.5 DNA提取与PCR检测
采用 CTAB法[26]提取水稻基因组 DNA。PCR扩增体系含 5.0 µL 2×PCR Reaction Mix、0.1 µL 5 UµL–1Taq DNA 聚合酶、引物(10 µmol L–1)各 0.3 µL、1.0 µL模板DNA, ddH2O补至10 µL。PCR程序为94°C 预变性 5 min; 94°C 变性 30 s, 55°C 退火 30 s,72°C延伸 30 s, 35个循环; 72°C延伸7 min。扩增产物经 8.0%非变性聚丙烯酰胺凝胶电泳后银染观察。
1.6 基因定位
02428 ×mf2的F2群体用于基因的初定位。采用BSA 法定位目标基因[27]。即根据F2植株表型, 各取10株正常植株和突变体植株的叶片等量混合提取DNA构建近等基因池, 利用近等基因池筛选具有多态性的SSR标记。再用分离后代单株验证该多态性标记是否真正与目标基因连锁。
Francis×mf2的F2群体用于基因的精细定位。在初定位的基础上, 利用更大的定位群体, 通过高密度SSR标记与InDel标记对目的基因进行连锁分析,利用 gramene数据库确定与突变基因紧密连锁的标记在水稻基因组(Nipponbare)上的位置, 并利用水稻基因组注释(http://rice.plantbiology.msu.edu/)筛选候选基因。
2 结果与分析
2.1 表型观察
与野生型比较, 突变体 mf2的营养生长和生殖生长都受到了一定的影响, 主要表现在抽穗期推迟、株高降低、穗数增多, 其穗的穗重、结实率以及籽粒的千粒重都极显著降低(表1和图1-A)。

表1 野生型和突变体mf2主要农艺性状调查Table 1 Comparison of main agronomic traits between wild type and mf2 mutant
一个正常的小花具有确定数目的四轮结构, 包含1对闭合的内外稃、2个浆片、6枚雄蕊以及1个双柱头的雌蕊, 小花基部还有1对副护颖和护颖(图1-C, D)。突变体mf2的副护颖和护颖与野生型基本无异, 但是小花的四轮结构均异常, 表现为内外稃增多、开裂、雌雄蕊数目增多、浆片稃片化等(图1-E~J)。尤其是小花内三轮结构成倍增长, 形成类似2朵小花(图1-H)和3朵小花(图1-J), 且许多小花的雄蕊比正常的6枚都有不同程度的减少。对150个mf2颖花统计发现, 突变体小穗中包含1朵小花、2朵小花、3朵小花的颖花分别占 6.00%、75.33%和18.67%。对mf2成熟后的籽粒进行统计却发现小穗中包含1粒籽粒、2粒籽粒、3粒籽粒的小穗分别占81.56%、17.49%和 0.95%。表明多数突变体的小花并不能成功授粉、灌浆, 最终形成籽粒(图2)。
2.2 组织学观察
组织学观察(图 3)表明正常小花的内外稃分别有3条和5条维管束(图3, I-A)。突变体mf2具有多片稃状结构, 其内外稃大多不能很好地勾合, 而且形状和维管束都产生了一定程度的变化(图 3, I-B,C)。从维管束的数量和内外稃形状上看, 其外稃具有一定程度的内稃化。

图1 突变体mf2及其野生型的表型Fig. 1 Phenotypes of mf2 and the wild type
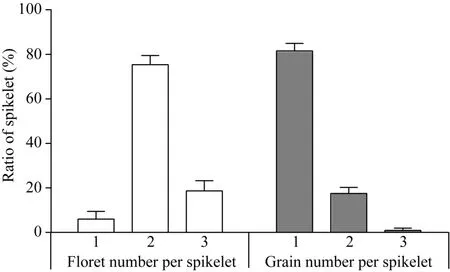
图2 mf2突变体小穗小花数和籽粒数所占比例Fig. 2 Ratio of the floret and grain number per spikelet of mf2 mutant
电镜观察(图3-II)发现, 突变体花器官的异常在幼穗的分化期, 各花器官在原基分化时就已经产生。一般正常的小花原基先后分化出外稃原基、内稃原基、浆片原基、雄蕊原基、雌蕊原基, 6个雌蕊原基以同心圆的方式围绕着雄蕊原基(图3, II-A, B)。突变体小花在雌雄蕊原基分化时期表现为雌雄蕊原基排列散乱, 数目和大小不一, 还有额外的稃片原基产生(图 3, II-C, D), 这是产生多个雌雄蕊和类稃器官的基础。
2.3 遗传分析
突变体mf2分别与02428和Francis杂交, F1所有植株表型均正常, 与野生型相同, 说明 mf2为隐性突变。F2群体抽穗期花器官正常的称为野生型, 异常的称为突变型, 两者的分离比符合3∶1 (表2), 说明mf2突变性状受1对隐性核基因控制, 暂命名为MF2。

图3 突变体mf2及其野生型小穗显微结构Fig. 3 Micrographs of wild-type and mf2 spikelets

表2 mf2突变体遗传分析Table 2 Genetic analysis of mf2 mutant
2.4 基因定位
选用均匀分布在水稻 12条染色体上的 302对SSR标记对两亲本02428、mf2进行多态性分析, 筛选出107对具多态性SSR标记, 多态率为34.97%。进一步利用亲本间具多态性的标记扩增F2近等基因池筛选多态性标记, 并分别随机选取野生型和突变型分离单株各10株进行验证, 最终发现标记RM315与MF2连锁(图4-A)。
为进一步确定基因所在的标记区间, 利用 Francis mf2的F2群体的24株突变表型单株在RM315附近筛选更高密度的多态性SSR标记。结果表明RM472、RM104与目标基因连锁, 其重组个体分别为5个和3个, 且2个标记的重组个体不重叠。因此, MF2基因初步被定位于RM472–RM104区间(图4-A)。
在 RM472和 RM104区间内设计多个 InDel、SSR标记(具多态性的标记列于表3), 利用全部隐性单株(321个)进一步缩小定位区间。在标记SSR39108和 InD39210各有 1个重组子, 且互不重叠。比对Nipponbare的序列(http://ensembl.gramene.org/Tools/Blast), 标记SSR39108–InD39210对应于日本晴第1染色体 39 198 782~39 210 592 bp, 物理距离约为102 kb (图 4-B)。

图4 MF2基因的精细定位Fig. 4 Fine mapping of MF2

表3 定位MF2所设计的引物序列Table 3 Primer sequence used for MF2 mapping
根据Rice Genome Annotation Project (http://rice.plantbiology.msu.edu/index.shtml)提供的基因注释信息, 在目的基因所在的102 kb定位区间内共有10个预测基因(表4)。其中Os01g67430为脂肪酶基因, 该基因目前已被克隆, 即EG1 (EXTRA GLUME 1), 参与花器官的调控[17]。Os01g67410是AP2/EREBP转录因子, 目前已发现多个 AP2亚家族基因, 在调控花分生组织确定性、花器官属性和种子发育上发挥着重要的作用[28]。因此本研究将EG1 (Os01g67430)和Os01g67410作为初步的候选基因。
3 讨论
植物的形态建成取决于植物分生组织的活动[29]。在生殖生长阶段, 花器官分生组织的异常导致花器官形态、数目的变异。在部分报道的调控花器官数目的基因中, FON1调控花分生组织大小。FON1基因突变导致花分生组织增大, 其突变体所有花器官的数目增多, 更有异位的花器官产生以及新的轮结构形成[11]; FON1还能调控干旱胁迫和种子萌发[30]。FON2控制花分生组织的保守性及分化, 其突变也导致花分生组织增大, 使花器官数目增多[13]。FON4与 FON2是同一基因[12]。FON3突变体花分生组织在外稃原基分化后就明显增大, 导致异常的小花内轮发育[31]。Fon(t)在雄蕊原基分化时也观察到了分生组织的异常[32]。本研究中所获得的突变体mf2雌雄蕊原基排列散乱, 数目和大小不一, 还有额外的稃片原基产生, 暗示花器官分生组织的异常致使其花器官变异。
本研究将 MF2定位在第 1染色体的标记SSR39108和InD39210之间, 与已报道的MF1基因在不同染色体上[25]。定位区间内具多个可能的基因,其中Os01g67410为AP2/EREBP转录因子基因, 在植物发育中起重要调控作用, 比如调控花器官的发育, 影响株高、育性, 对生物胁迫和非生物胁迫的应答反应等[28]。拟南芥的 AP2基因是 AP2亚族成员,属于“ABCDE”花器官模型中的A类基因, 在调控花分生组织确定性、花器官属性和种子发育上具有重要作用[28]。该类基因在单子叶植物中具有一定的保守性。玉米IDS1基因是拟南芥AP2的同源基因, 调控小穗分生组织特性。ids1突变体小穗分生组织的确定性丢失, 每个小穗产生了多个小花[33]。水稻SNB基因同样属于AP2亚族, 调控小穗分生组织向花分生组织转化以及花器官的发育[15]。SNB与另一个AP2-LIKE基因OsIDS1协同控制花序结构和花分生组织建成[34]。OsAP2-1基因属于AP2亚族, 该基因下调导致雄蕊减少, 心皮或柱头增加, 也会产生额外的内外稃器官, 表明该基因也参与花器官数目的调控[14]。此外, Os01g67430基因即EG1已被定位克隆。EG1涉及花分生组织的确定性, 其突变导致空颖的属性以及数目变化, 产生异位的花器官[17]。该脂肪酶基因与已知的其他植物的脂肪酶基因不同,拟南芥中与之高度同源的基因(At2g31690)并不影响花器官的发育[35], 表明其功能在水稻中存在分化。EG1作为质体定位脂肪酶, 参与JA的生物合成, 揭示了禾本科植物中一个新的小穗发育调控机制[21]。Zhang等[36]的实验证明, EG1可以通过一个高度依赖温度的线粒体脂肪酶途径使确定花器官属性的基因安全表达, 进而提高植株花器官的稳定性以应对温度的波动, 这又对该基因调控花器官发育机制有了新的理解。

表4 MF2定位区间内的候选基因Table 4 Candidate genes of MF2 mapped region
突变体 mf2的花器官发育受到严重影响, 其各轮结构的数目异常、形状畸变, 浆片稃片化以及部分外稃内稃化, 表明其花分生组织的确定性以及花器官属性的调控受到影响。虽然其表型与EG1突变体的花器官表型相似但也存在差异。mf2表现出生育期推迟、植株变矮、分蘖增多, 表明其营养生长也受到严重影响, 而EG1突变体eg1-1和eg1-2的营养生长和圆锥花序的数目并不受影响[17]。初步的测序结果表明, 突变体 mf2与野生型的 Os01g67430/EG1基因和 Os01g67410基因的编码区以及 5′UTR都未发现碱基序列改变, 需进一步对这 2个候选基因及定位区间内的其他基因进行表达分析以及测序验证。
4 结论
水稻突变体 mf2的营养生长和生殖生长都受到影响。其花器官表现出多向性缺陷, 主要体现在各轮花器官数目的变化, 包括稃片、浆片、雄蕊、雌蕊, 多数小穗内具 2~3朵类似小花。花器官的变异在幼穗分化期的各花器官原基分化时就已形成。mf2突变体表型受单隐性核基因控制, 该基因被定位于第1染色体的标记SSR39108和InD39210之间, 区间大小约为102 kb。
[1]Guo S, Sun B, Looi L S, Xu Y, Gan E S, Huang J, Ito T.Co-ordination of flower development through epigenetic regulation in two model species: rice and Arabidopsis. Plant Cell Physiol, 2015, 56: 830–842
[2]Yanofsky M F. Floral meristems to floral organs: genes controlling early events in Arabidopsis flower development. Annu Rev Plant Physiol Plant Mol Biol, 1995, 46: 167–188
[3]Fornara F, Marziani G, Mizzi L, Kater M, Colombo L.MADS-box genes controlling flower development in rice. Plant Biol, 2003, 5: 16–22
[4]田大刚, 刘华清, 苏军, 张礼华, 王锋. 水稻与拟南芥中控制花器官发育MADS-box基因的比较研究进展. 福建农业学报,2011, 26: 309–320 Tian D G, Liu H Q, Su J, Zhang L H, Wang F. Flower-Development-Controlling MADS-box genes in rice and Arabidopsis thaliana. Fujian J Agric Sci, 2011, 36: 309–320 (in Chinese with English abstract)
[5]Yoshida H, Nagato Y. Flower development in rice. J Exp Bot,2011, 62: 4719–4730
[6]Takahashi M, Nagasawa N, Kitano H, Nagato Y. Panicle phytomer 1 mutations affect the panicle architecture of rice. Theor Appl Genet, 1998, 96: 1050–1056
[7]Kyozuka J, Konishi S, Nemoto K, Izawa T, Shimamoto K.Down-regulation of RFL, the FLO/LFY homolog of rice, accompanied with panicle branch initiation. Proc Natl Acad Sci USA,1998, 95: 1979–1982
[8]Jeon J S, Jang S, Lee S, Nam J, Kim C, Lee S H, Chung Y Y,Kim S R, Lee Y H, Cho Y G. Leafy hull sterile 1 is a homeotic mutation in a rice MADS box gene affecting rice flower development. Plant Cell, 2000, 12: 871–884
[9]Chanhong K, Donghoon J, An G H. Molecular cloning and characterization of OsLRK1 encoding a putative receptor-like protein kinase from Oryza sativa. Plant Sci, 2000, 152: 17–26
[10]Jang S, Lee B, Kim C, Kim S J, Yim J, Han J J, Lee S, Kim S R,An G. The OsFOR1 gene encodes a polygalacturonase-inhibiting protein (PGIP) that regulates floral organ number in rice. Plant Mol Biol, 2003, 53: 357–369
[11]Suzaki T, Sato M, Ashikari M, Miyoshi M, Nagato Y, Hirano H Y. The gene FLORAL ORGAN NUMBER1 regulates floral meristern size in rice and encodes a leucine-rich repeat receptor kinase orthologous to Arabidopsis CLAVATA1. Development,2004, 131: 5649–5657
[12]Chu H, Qian Q, Liang W, Yin C, Tan H, Yao X, Yuan Z, Yang J,Huang H, Luo D. The FLORAL ORGAN NUMBER4 gene encoding a putative ortholog of Arabidopsis CLAVATA3 regulates apical meristem size in rice. Plant Physiol, 2006, 142: 1039–1052[13]Suzaki T, Toriba T, Fujimoto M, Tsutsumi N, Kitano H, Hirano H Y. Conservation and diversification of meristem maintenance mechanism in Oryza sativa: function of the FLORAL ORGAN NUMBER2 gene. Plant Cell Physiol, 2006, 47: 1591–1602
[14]Zhao L, Xu S, Chai T, Tai W. OsAP2-1, an AP2-like gene from Oryza sativa, is required for flower development and male fertility. Plant Reprod, 2006, 19: 197–206
[15]Lee D Y, Lee J, Moon S, Park S Y, An G. The rice heterochronic gene SUPERNUMERARY BRACT regulates the transition from spikelet meristem to floral meristem. Plant J, 2007, 49: 64–78
[16]Sun Q, Zhou D X. Rice jmjC domain-containing gene JMJ706 encodes H3K9 demethylase required for floral organ development. Proc Natl Acad Sci USA, 2008, 105: 13679–13684
[17]Li H, Xue D, Gao Z, Yan M, Xu W, Xing Z, Huang D, Qian Q,Xue Y. A putative lipase gene EXTRA GLUME1 regulates both empty-glume fate and spikelet development in rice. Plant J Cell Mol Biol, 2009, 57: 593–605
[18]Xiao H, Tang J, Li Y, Wang W, Li X, Jin L, Xie R, Luo H, Zhao X, Meng Z. STAMENLESS 1, encoding a single C2H2 zinc finger protein, regulates floral organ identity in rice. Plant J, 2009, 59:789–801
[19]Ren D Y, Li Y F, Wang Z, Xu F F, Guo S, Zhao F M, Sang X C,Ling Y H, He G H.. Identification and gene mapping of a multi-floret spikelet 1 (mfsl) mutant associated with spikelet development in rice. J Integr Agric, 2012, 11: 1574–1579
[20]Wang N, Li Y F, Sang X C, Ling Y H, Zhao F M, Yang Z L, He G H. Nonstop glumes (nsg), a novel mutant affects spikelet development in rice. Genes & Genomics, 2013, 35: 149–157
[21]Cai Q, Yuan Z, Chen M, Yin C, Luo Z, Zhao X, Liang W, Hu J,Zhang D. Jasmonic acid regulates spikelet development in rice.Nat Commun, 2014, 5: 3476
[22]Zhang J, Tang W, Huang Y, Niu X, Zhao Y, Han Y, Liu Y.Down-regulation of a LBD-like gene, OsIG1, leads to occurrence of unusual double ovules and developmental abnormalities of various floral organs and megagametophyte in rice. J Exp Bot,2015, 66: 99–112
[23]Wang H H, Zhang L, Cai Q, Jin Z M, Zhao X X, Huang Q M,Luo Z J, Chen M J, Zhang D B, Yuan Z. OsMADS32 interacts with PI-like proteins and regulates rice flower development. J Integr Plant Biol, 2015, 57: 504–513
[24]Yang C, Ma Y, Li J. The rice YABBY4 gene regulates plant growth and development through modulating the gibberellin pathway. J Exp Bot, 2016, 67: 5545–5556
[25]李云峰, 杨正林, 凌英华, 王楠, 任德勇, 王增, 何光华. 水稻多小花小穗突变体 mf1的鉴定与基因定位. 作物学报, 2011,37: 280–285 Li Y F, Yang Z L, Ling Y H, Wang N, Ren D Y, Wang Z, He G H. Characterization and gene mapping of a spikelet mutant mf1 in rice. Acta Agron Sin, 2011, 37: 280–285 (in Chinese with English abstract)
[26]Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA. Nucl Acids Res, 1980, 8: 4321–4326
[27]Michelmore R W, Paran I, Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations. Proc Natl Acad Sci USA, 1991, 88:9828–9832
[28]赵利峰, 柴团耀. AP2/EREBP转录因子在植物发育和胁迫应答中的作用. 植物学通报, 2008, 25: 89–101 Zhao L F, Chai T Y. Roles of AP2/EREBP family of transcription factors in development and stress response of plants, Chin Bull Bot, 2008, 25: 89–101 (in Chinese with English abstract)
[29]Poethig R S. Phase change and the regulation of developmental timing in plants. Science, 2003, 301: 334–336
[30]Feng L, Gao Z, Xiao G, Huang R, Zhang H. Leucine-rich repeat receptor-like kinase FON1 regulates drought stress and seed germination by activating the expression of ABA-responsive fenes in rice. Plant Mol Biol Rep, 2014, 32: 1158–1168
[31]Jiang L, Qian Q, Mao L, Zhou Q Y, Zhai W X. Characterization of the rice floral organ number mutant fon3. J Integr Plant Biol,2005, 47: 100–106
[32]Li Y, Xu P, Zhang H, Peng H, Zhang Q, Wang X, Wu X. Characterization and identification of a novel mutant fon(t) on floral organ number and floral organ identity in rice. J Genet Genomics,2007, 34: 730–737
[33]Kaplinsky N J, Freeling M. Combinatorial control of meristem identity in maize inflorescences. Development, 2003, 130:1149–1158
[34]Lee D Y, An G. Two AP2 family genes, SUPERNUMERARY BRACT (SNB) and OsINDETERMINATE SPIKELET 1 (OsIDS1),synergistically control inflorescence architecture and floral meristem establishment in rice. Plant J, 2012, 69: 445–461
[35]Padham A K, Hopkins M T, Wang T W, Mcnamara L M, Lo M,Richardson L G, Smith M D, Taylor C A, Thompson J E. Characterization of a plastid triacylglycerol lipase from Arabidopsis.Plant Physiol, 2007, 143: 1372–1384
[36]Zhang B, Wu S, Zhang Y, Xu T, Guo F, Tang H, Li X, Wang P,Qian W, Xue Y. A high temperature-dependent mitochondrial lipase EXTRA GLUME1 promotes floral phenotypic robustness against temperature fluctuation in rice (Oryza sativa L.). PLoS Genet, 2016, 12: e1006152
