R2R3-MYB转录因子GmMYB184调节大豆异黄酮合成
2018-03-01褚姗姗张培培喻德跃南京农业大学大豆研究所作物遗传育种与种质创新国家重点实验室江苏省现代作物生产协同创新中心国家大豆改良中心江苏南京0095河南农业大学农学院河南省粮食作物协同创新中心河南郑州45000
朱 莹 褚姗姗 张培培 程 浩 喻德跃 王 娇,*南京农业大学大豆研究所 / 作物遗传育种与种质创新国家重点实验室 / 江苏省现代作物生产协同创新中心 / 国家大豆改良中心,江苏南京0095; 河南农业大学农学院 / 河南省粮食作物协同创新中心, 河南郑州 45000
异黄酮是苯丙烷类次生代谢物, 主要含在豆科植物中, 尤其是大豆中[1]。它具有雌激素、抗癌、抗氧化、治疗心血管病和预防骨质疏松等多种生理活性[2-3], 且在植物与微生物的相互作用中发挥重要作用, 如在大豆中, 异黄酮一方面可以作为信号物,参与大豆和根瘤菌的共生; 另一方面, 可以抵制病原菌的侵害, 保护植株正常生长[4]。大豆异黄酮除本身具有抗病原菌活性外[5], 还参与大豆抗毒素的合成。大豆抗毒素是大豆特异的植物抗毒素, 具有广谱抗性, 可以保护大豆免受病原菌、食草动物等的侵害[6]。
大豆异黄酮通过植物共有的苯丙氨酸代谢途径和豆科特异的异黄酮分支途径被合成, 涉及苯丙氨酸解氨酶(PAL)、肉桂酸 4-羟化酶(C4H)、4-香豆酸:CoA连接酶(4CL)、查尔酮合成酶(CHS)、查尔酮还原酶(CHR)、异黄酮合酶(IFS)等多种酶。此外, 苯丙氨酸代谢途径还可以通过其他分支途径合成木质素、花青素、黄酮等物质[1]。
随着对大豆异黄酮合成分子机制理解的深入,代谢工程成为大豆异黄酮品质改良的重要手段。调节与异黄酮合成相关基因的表达是研究大豆异黄酮代谢工程的主要策略之一。但是, 对大豆异黄酮遗传控制的研究还存在很多问题。首先, 大豆异黄酮的合成受多基因控制, 这些基因间存在相互作用,形成一个代谢复合体, 调控大豆异黄酮含量[7-8]。另外, 苯丙氨酸代谢途径还涉及许多其他物质的合成,与异黄酮代谢途径存在微妙的互作关系。过表达大豆异黄酮代谢途径中的单个或多个基因, 如 PAL、CHS和 IFS, 通常对提高大豆异黄酮的含量影响不显著[9-10]。因此若要将代谢前体和中间产物有效导入异黄酮分支途径, 调控途径基因可能不是高效的方法。
除了途径基因, 转录因子也受到关注。转录因子可以作为分子开关与结构基因启动子特异结合,通过调控结构基因的表达, 可高效诱导或抑制目标代谢产物的积累[11-12]。因此, 可以寻找与异黄酮合成关键酶相互作用的转录因子, 通过转录因子调节异黄酮的合成。近年来鉴定了许多与黄酮、异黄酮代谢有关的不同类型的转录因子, 包括MYB、bHLH(碱性螺旋-环-螺旋)、bZIP (碱性亮氨酸拉链)等[13]。其中, MYB转录因子是高等植物最大的转录因子家族之一[14]。在拟南芥中报道了近 200个MYB转录因子[15], 其中绝大多数是 R2R3类型[16-17]; 大豆中大约有700个MYB转录因子, 其中244个是R2R3类型的 MYB转录因子[18]。目前已鉴定 3个R2R3-MYB转录因子与大豆异黄酮合成相关, 如GmMYB29能够激活CHS8和IFS2的表达, 过表达和干扰该基因能够分别提高和抑制异黄酮的合成[19]。GmMYB39能够抑制CHS的表达活性, 过表达该转录因子可以降低PAL、C4H、CHS、4CL和CHR的转录水平, 进而抑制异黄酮的生物合成[20]。GmMYB100在异黄酮的积累中显示了负调控的功能, 过表达该基因能够降低大豆转基因株系的异黄酮含量[21]。当然很可能还存在更多的R2R3-MYB转录因子参与异黄酮的调控, 而如何有效地搜寻并鉴定影响异黄酮积累的调控因子成为科学家们努力攻克的难点和方向。
谷胱甘肽(glutathione, GSH)可以影响生物胁迫相关基因的表达, 并诱导异黄酮的产生[22-23]。前人系统分析了百脉根中 39个主要的转录因子家族在GSH诱导后的表达情况, 发现R2R3-MYB转录因子亚家族 20在 GSH诱导下表达量显著提高, 如LjMYB62在GSH胁迫处理后3 h上调表达86倍, 处理6 h后上调表达106倍。推测此亚家族可能存在与抗病抗逆和异黄酮调控相关的转录因子[23-24]。本研究利用生物信息学方法, 分析预测了一个与大豆异黄酮合成相关的转录因子GmMYB184, 并克隆该转录因子进行调控机制研究。首先, 分析该转录因子组织表达和诱导表达模式, 以探索它与大豆异黄酮合成关键基因之间的关系。其次, 采用双荧光素酶报告系统分析其对异黄酮合成途径关键基因转录激活活性。最后通过发根农杆菌介导的转化系统, 找到该转录因子在异黄酮合成调控中的直接作用证据。
1 材料与方法
1.1 试验材料
用于基因克隆的大豆品种为已完成基因组测序的Williams 82。大肠杆菌菌株DH5α购自天根生化科技有限公司。组织表达和诱导表达分析使用的大豆材料是监利牛毛黄品种。遗传转化分析使用的是大豆 Jack品种。原生质体的提取使用的是拟南芥Columbia-0生态型的叶片。以上材料均保存于南京农业大学国家大豆改良中心。
菌株载体包括发根农杆菌(Agrobacterium rhizogenes) K599、植物原生质体表达载体pAN580、入门载体pDONR221、表达载体pBA002、干扰载体pB7GWIWG2(II), 均由本实验室保存提供。
KOD plus neo高保真聚合酶购自TOYOBO。限制性内切酶购自 New England Biolabs (NEB)。pMD19-Tsimple载体、T4连接酶购自TaKaRa公司。DNA标准分子量购自北京全式金生物技术有限公司。还原型谷胱甘肽(GSH)、MES和 Silwet购自Sigma公司。氨苄青霉素Amp、卡那霉素Kana、利福平Rif等抗生素试剂购自Roche公司。MS、White培养基购自南京寿德生物科技有限公司。
质粒小提试剂盒、DNA凝胶回收试剂盒购自Axygen公司。RNA提取试剂盒购自北京天根公司。cDNA合成试剂盒购自TaKaRa公司。无内毒素质粒大提试剂盒购自北京天根公司。双荧光素酶报告基因检测试剂盒购自北京普洛麦格(Promega)生物技术有限公司。
PCR引物合成由英潍捷基(上海)贸易有限公司完成。DNA测序由北京六合华大基因科技股份有限公司完成。
1.2 大豆GmMYB184基因的克隆
PCR扩增模板为大豆Williams 82品种的cDNA序列。分别使用RNA试剂盒和cDNA合成试剂盒提取 RNA序列和合成 cDNA序列。为了获取GmMYB184的全长 cDNA 序列, 设计大豆GmMYB184的特异 PCR扩增引物, 上游引物GmMYB184-F: 5′-GTTTCAGTGAGTGAGAATAGC-3′; 下游引物 GmMYB184-R: 5′-AGAGTTTTGGAC TTTTGGT-3′。PCR 扩增体系(50 μL)含: cDNA 模板4 μL, dNTP 10 μL, 2×buffer 25 μL, KOD 酶 1 μL, 上游引物 1.5 μL, 下游引物 1.5 μL, ddH2O 7 μL。PCR条件为: 94°C 预变性 2 min; 98°C 变性 10 s, 51°C 退火30 s, 68°C延伸40 s, 33个循环; 68°C延伸7 min。
PCR反应结束后, 将反应产物与Loading buffer混合, 用1%琼脂糖凝胶电泳检测条带大小。PCR产物经胶回收试剂盒回收后, 取适量进行加尾反应,随后按照 pGM-T载体连接试剂盒说明, pMD19-Tsimple载体与目的基因连接反应, 连接产物转化大肠杆菌 DH5α感受态细胞。挑取大而圆的单菌落进行菌液PCR。反应结束后将菌液PCR产物进行1%琼脂糖凝胶电泳检测。挑选电泳结果中与目的片段大小一致的产物结果送测序, 测序结果正确的重新接菌提取质粒。将质粒保存用于后续实验及载体构建的模板(以下称该质粒为8g-T)。
1.3 目的基因序列分析
使用 CLUSTALW 程序比对蛋白序列[25], 默认参数设置。使用 MEGA[26]绘制邻接树(neighborjoining tree), 每个节点的 bootstrap值重复次数为1000次。使用NCBI分析目标基因序列。
1.4 实时定量荧光PCR分析
1.4.1 组织表达样品 分别取播种于南京农业大学江浦试验站的大豆品种监利牛毛黄的根、茎、叶、花、开花10 d后的荚和种子, 开花20、25、30、40、50 d后的种子和成熟种子, 经液氮速冻并于–80°C保存。提取样品的RNA, 反转录成cDNA。
1.4.2 谷胱甘肽诱导表达样品 在光照培养箱中培养大豆监利牛毛黄材料4~7周。设置光照培养箱16 h光照, 8 h黑暗; 温度为光照28°C, 黑暗23°C。培养过程中需避免其他生物胁迫。摘取健康的完全展开叶片,用10 mmol L–1GSH溶液(含0.005% Silwet植物表面活性剂)黑暗处理8 h。处理条件为室温下(23~25°C),80 r min–1振荡。对照仅用0.005% Silwet植物表面活性剂处理, 其他条件均相同。分别在0、3、6、7 h时取样[22-23]。液氮速冻, 保存于–80°C冰箱备用。
1.4.3 实时荧光定量PCR 以大豆组成型表达的tubulin基因为内参, 设计基因特异引物, 引物序列如表1, 采用ABI 7500 system v1.4.0数据分析实验获得的数据。每个样品重复测定3次。
目标基因表达水平进行相对定量采用2–ΔΔCT法[27]。设诱导表达0 h Δ值为1, 计算出3、6、7 h相较于0 h的表达倍数。
1.5 亚细胞定位
首先构建亚细胞定位所需的表达载体。以8g-T载体为模板, 选择Xba I和Xma I酶切位点设计引物,PCR扩增得到 GmMYB184的完整开放阅读框(open reading frame, ORF)序列。引物MYB184SUB正向序列为 5′-TGCTCTAGAATGTCTACTTCAAAGAGC-3′,反向序列为 5′-TCCCCCCGGGTTTTTGTAACTTGC TAAA-3′。获得带酶切位点的GmMYB184完整ORF序列的PCR产物, 对该产物进行酶切、回收酶切产物, 用T4连接酶连入pAN580载体。由此方法获得表达 MYB184::GFP融合蛋白的重组质粒 pAN580-MYB184-GFP。重组质粒 pAN580-MYB184-GFP测序正确后, 与对照 pAN580-GFP质粒通过聚乙二醇(polyethylene glycol, PEG)法分别转入拟南芥原生质体。

表1 引物序列Table 1 Primer sequences
1.6 构建双荧光素酶报告系统
GmMYB184的表达载体是由亚细胞定位中所用的 pAN580表达载体改造的。首先用限制性内切酶BamH I和Not I对亚细胞定位中使用的pAN580表达载体进行双酶切, 切除 GFP片段。用 Klenow酶平末端处理后用T4连接酶连接, 完成中间载体的构建。随后将GmMYB184序列连入中间载体的多克隆位点区段, 构成 CaMV35S::MYB184重组质粒, 经测序正确即完成效应载体Effector的构建。
双荧光素酶报告系统报告载体Reporter的构建,首先设计带 Xba I和 Xma I酶切位点的引物, 以pGL3载体为模板通过 PCR扩增带有酶切位点的LUC基因的完整ORF。对PCR产物进行酶切、回收后用T4连接酶将LUC序列连入中间载体的多克隆位点区段, 构成CaMV35S::LUC重组质粒。构建完成的CaMV35S::LUC重组质粒中的CaMV35S片段需用Sac I和Nhe I限制性内切酶双酶切去除, 去除后的质粒片段用于构建最终报告载体。设计特异引物从大豆 Williams 82 DNA中扩增带酶切位点的IFS2和CHS8启动子片段, 引物序列如表1。对扩增而得的IFS2和CHS8启动子片段PCR产物酶切、回收, 分别连入去除 CaMV35S片段后的质粒片段从而构成IFS2pro::LUC和CHS8pro::LUC载体, 经测序正确即完成报告载体Reporter的构建。
使用海肾荧光素酶载体为内参载体。双荧光素酶报告系统的报告载体、效应载体和内参载体示意图如图1。
将报告载体、内参载体和效应载体, 或报告载体和内参载体(作为对照)分别共转染拟南芥原生质体, 每个转染实验重复 6次。参照荧光素酶双报告基因检测试剂盒使用手册(Promega)的荧光素酶报告基因检测步骤。
1.7 农杆菌介导的大豆毛状根转化系统的构建及遗传转化
RNA干涉载体是应用Gateway技术构建的。构建过程只需 BP和 LR两个反应。选择GmMYB184基因编码区域321 bp的特异片段作为干涉区段。设计带有 attB1和 attB2接头序列的正向引物MYB184Ri-F和反向引物MYB184Ri-R进行PCR扩增, 在 5′端接入 attB1位点, 3′端接入 attB2位点。MYB184Ri-F 序列为 5′-GGGGACAAGTTTGTACAA AAAAGCAGGCTTCAGACATTTGAAAATTTACA C-3′; MYB184Ri-R 序列为 5′-GGGGACCACTTTGT ACAAGAAAGCTGGGTCAAAATGCTGAGGCACT TTTG-3′。
PCR产物切胶回收后进行BP反应和LR反应,将目的片段插入pB7GWIWG2(II)载体, 最终得到重组RNA干扰载体pBI-MYB184Ri。
过表达载体的构建, 设计带有酶切位点Xho I和Mlu I的上下游引物, PCR扩增GmMYB184的全长序列。上游引物 MYB184OE-F序列为 5′-ATCCGCTC GAGATGTCTACTTCAAAGAGC-3′; 下 游 引 物MYB184 OE-R 序列为 5′-ATCGACGCGTTTATTTTT GTAACTTGCT-3′。
随后经酶切, 回收, 连接, 将 GmMYB184全长序列正向插入 pBA002载体的CaMV35S启动子后,构建成过表达载体pBA002-MYB184。
农杆菌介导的大豆毛状根转化, 用浓 HCl和NaClO溶液反应产生的氯气对成熟饱满的大豆Jack品种的种子灭菌6~8 h, 放入灭菌的容器备用。用灭菌水浸泡灭菌的大豆种子, 于黑暗环境浸泡 10~12 h。接着在超净工作台中去除种皮后将种子接种于萌发培养基, 每皿约10粒豆子, 放置培养箱培养(25°C,光照16 h, 黑暗 8 h)至刚长出真叶。
将干扰载体 pBI-MYB184Ri、过表达载体pBA002-MYB184以及对应的空载分别转化发根农杆菌 K599, 挑取阳性单克隆至 YEB液体培养基中(含有 Kana 50 µg L–1), 28°C 振荡培养 48 h, 3000 r min–1离心 10 min, 收集菌体。用 10 mmol L–1MgCl2溶液重悬菌体至终浓度OD600= 0.5。
在超净台剪下培养好的大豆的子叶并在每个子叶的背面挖一个凹槽。将处理后的大豆子叶摆放在White培养基上(含有头孢50 µg mL–1和羧苄霉素50µg mL–1)。将OD600为0.5的菌液滴在子叶的凹槽里,置于培养箱中生长18~20 d (25°C黑暗培养)。毛状根4~8 cm长时取样, 进行后续实验。
1.8 不同组织大豆异黄酮提取及含量检测
称取大豆不同组织粉末状样品0.015 g, 于2 mL离心管中。加入80%色谱级甲醇溶液1.5 mL (料液比 1∶100), 涡旋混匀 30s, 50°C超声(频率 40 kHz,功率300 W)辅助提取1 h, 超声期间每隔10 min取出样品上下颠倒混匀。12 000×g离心10 min。小心吸取上清液, 过 0.22 μm 有机相针式滤器, 注入Agilent 2 mL自动进样专用瓶, –20°C保存待上机检测。12种异黄酮标样(Daidzein、Genistein、Glycitein、Daidzin、Genistin、Glycitin、6"-O-acetyldaidzin、6"-O-acetylgenistin、6"-O-acetylglycitin、6"-O-malonyldaidzin、6"-O-malonylgenistinhe和 6"-O-malonylglycitin)购自 Sigma-Aldrich公司。用 80%甲醇溶液将这12种标样分别配制成0、5、10、20、50、100、500、1000 ng/进样针, 制作 12条标准曲线用于12种异黄酮单体含量的计算。
使用 HPLC法检测不同组织异黄酮含量, 采用ZorbaxSB-C18 柱(5 μm, 4.6 mm×150.0 mm), 柱温保持在36°C, 检测波长为254 nm (DAD), 进样量为10 μL。流动相为0.1% (v/v)乙酸水溶液(A)和100%的甲醇(B), 梯度洗脱为 27% B (v/v), 0~2 min; 27%~38%B, 2~3 min; 38% B, 3~10 min; 38%~39% B, 10~12 min; 39% B, 12~14 min; 39%~27% B, 14~15 min, 柱后3 min, 流速为2 mL min–1。本研究异黄酮总含量指大豆12种异黄酮组分的总和, 单位为每克大豆组织干物质所含异黄酮的量(μg g–1)[28]。
对于大豆转基因毛状根系的异黄酮测定, 取过表达和沉默GmMYB184转基因独立株系各4个, 截取部分根毛, 置烘箱105°C杀青0.5 h, 70°C烘至恒重, 称取干重 0.015 g, 测定根系中的异黄酮含量,每个转基因株系重复3次。
2 结果与分析
2.1 GmMYB184基因的克隆及序列分析
前人研究发现LjMYB62可能与生物、非生物胁迫相关, 通过序列比对发现 GmMYB184 (Glyma.08G042100)是 LjMYB62的同源基因(氨基酸序列相似度74%), 同属于R2R3-MYB转录因子亚家族20,该家族中可能存在与抗病抗逆和异黄酮调控相关的转录因子[23-24]。根据以上结果以及前人对于大豆GmMYB184基因响应干旱、盐害、冷害和 ABA(abscisic acid)处理的研究结果[29]推断, GmMYB184可能参与植物非生物胁迫响应及异黄酮生物合成途径调控。因此选择GmMYB184作为调控异黄酮合成的候选基因, 进行进一步功能分析。
为了解转录因子 GmMYB184在大豆异黄酮合成中的分子机制, 根据Phytozome (https://phytozome.jgi.doe.gov/)上大豆GmMYB184基因序列, 以大豆叶片 cDNA序列为模板, 使用设计的特异扩增引物GmMYB184-F和GmMYB184-R进行PCR扩增, 得到包含部分 5′-UTR 和部分 3′-UTR 在内的GmMYB184全长 CDS (coding sequence)序列(图 2),与数据库中目的片段大小(1009 bp)基本一致。经比对, 测序结果与大豆基因组数据库的序列一致。非翻译区, 3′-非翻译区, 3个外显子及2个内含子区域(图2)。ORF (open reading frame)序列长度为939 bp,编码蛋白分子量为31.15 kDa的312个氨基酸, 等电点(pI)为 7.15。经 Pfam 网站(http://pfam.xfam.org/)预测, GmMYB184属于R2R3-MYB亚家族。
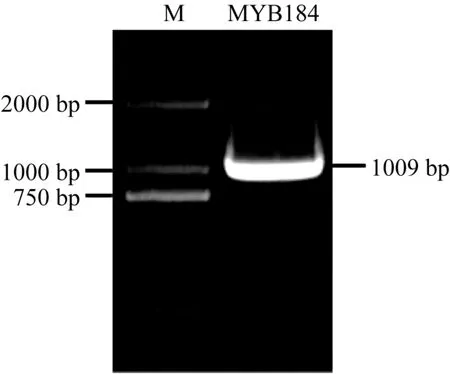
图2 GmMYB184扩增结果Fig. 2 Amplification result for GmMYB184
将GmMYB184和其他物种如拟南芥、百脉根、苜蓿中已报道的胁迫响应相关的R2R3-MYB转录因子蛋白序列比对表明, 这些 MYB序列在 N端区域有着非常高的保守性, 正是R2R3型DNA结合结构域(图3)。大豆GmMYB84与GmMYB184的氨基酸序列有着 92%的相似性, 表明 GmMYB84是GmMYB184的同源基因。

图3 胁迫响应相关R2R3-MYB转录因子序列比对Fig. 3 Sequence alignment for stress responsive R2R3-MYB transcription factors
通过邻接法构建这些序列的进化树, 进行分子发育分析发现, GmMYB184与已报道的拟南芥、百脉根和苜蓿中与苯丙烷代谢途径相关的 MYB转录因子关系密切(图4)。
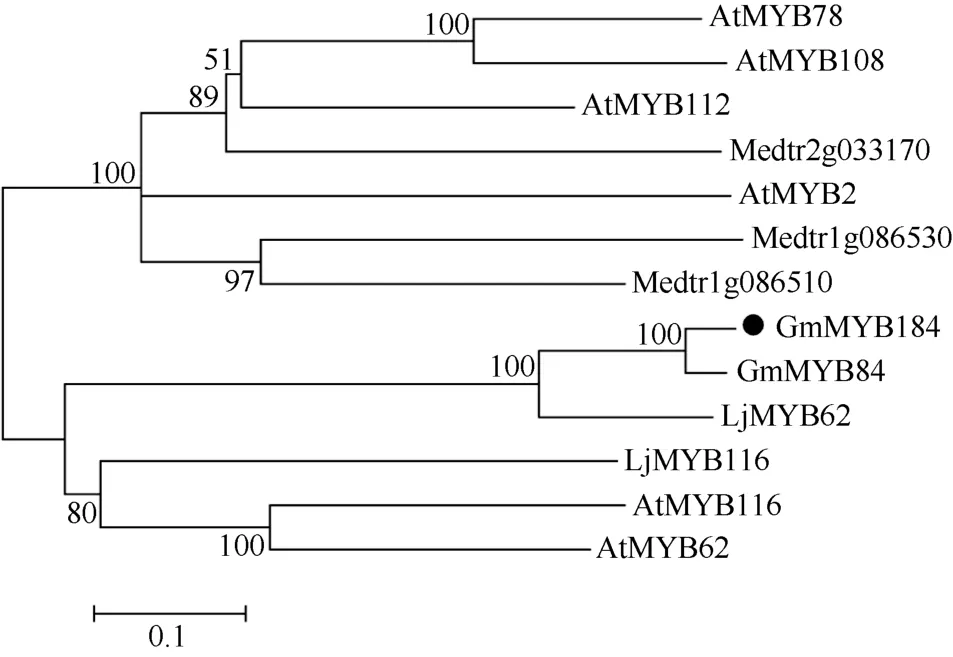
图4 胁迫响应相关R2R3-MYB转录因子系统发育分析Fig. 4 Phylogenetic analysis for stress responsive R2R3-MYB transcription factors
2.2 表达分析
实时荧光定量 PCR分析结果发现 GmMYB184在不同组织中的表达量均较低, 主要在根和种子中表达(图5-A)。在大豆种子的不同发育时期中, 随着籽粒成熟, GmMYB184的表达量也逐渐增加(图5-D)。
比较GmMYB184和IFS2的转录水平发现, IFS2基因的表达与GmMYB184的表达模式相似, IFS2在根和种子中大量表达(图 5-B), 并且随着籽粒成熟,IFS2的表达量也逐渐增加(图5-E)。又取大豆不同组织的材料提取异黄酮, 发现大豆不同组织和发育时期的异黄酮含量积累模式(图 5-C, F)也与GmMYB184和IFS2的表达模式相同。因此, 异黄酮的积累与 GmMYB184和IFS2在大豆不同组织和种子发育时期的表达模式密切相关。
GSH处理3 h后, GmMYB184与对照相比增加了12倍, IFS2基因与对照相比增加4倍。相似地, 7 h后, GmMYB184比对照提高了64倍, IFS2的表达量也提高了 10倍(图 6)。这些结果证实了 IFS2和GmMYB184在受到胁迫的大豆叶片中的强烈的共表达。此外, 处理期间的表达模式显示, 大豆GmMYB184转录因子有很大的可能性与IFS2共同参与大豆应对胁迫的诱导表达活动, 以及其他相似的生物过程。
2.3 亚细胞定位
单独的 GFP蛋白分布于整个细胞(图 7-A), 而GmMYB184-GFP融合蛋白定位于细胞核(图 7-B)。这与转录因子一般在核内进行转录调控活动相吻合。
2.4 GmMYB184对CHS8和IFS2的启动子激活活性分析
运用拟南芥原生质体和双荧光素酶报告系统瞬时表达分析发现, GmMYB184能够使IFS2和CHS8的启动子活性分别提高5倍(图8-A)和7倍(图8-B),这表明 GmMYB184能够显著增强异黄酮合成途径关键结构基因IFS2和CHS8的启动子活性, 并在转录水平调控异黄酮的合成。
2.5 在大豆毛状根中过表达和干扰 GmMYB184对异黄酮积累的影响
使用大豆毛状根转化系统, RNAi沉默和过表达GmMYB184基因, 沉默株系GmMYB184的表达水平与对照相比显著降低 2~6倍; 过表达株系GmMYB184的表达与对照相比显著上升 2~4倍(图9)。并且, GmMYB184沉默株系的异黄酮含量与对照相比显著降低, 为对照的 39%至 59%。但是,GmMYB184过表达株系的异黄酮含量与对照相比并没有显著提高(图10)。
3 讨论
大豆异黄酮的积累受多基因和复杂代谢网络控制[7]。对代谢途径中一个或两个酶基因的修饰或改造不能显著改变异黄酮含量[1]。转录因子可以同时调节多个基因共同表达, 通过转录调控因子的引入有望对整个代谢途径协调控制。本研究鉴定了一个与大豆异黄酮合成相关的转录因子GmMYB184。通过亚细胞定位、表达分析、转录激活活性分析以及大豆根毛转化等实验, 验证了该转录因子对异黄酮合成途径关键基因的转录激活活性及其在异黄酮合成中的正向调控作用。本研究为大豆异黄酮合成调控的分子机制提供了理论依据, 并为大豆异黄酮品质改良提供了新的靶标位点。
3.1 MYB转录因子对异黄酮合成的调控作用
高等植物中 MYB转录因子是最大的转录因子家族之一, 大豆中已鉴定了约700个MYB转录因子[18]。不同 MYB蛋白在不同植物中的功能已得到广泛研究。它们的功能多种多样, 比如参与细胞形态与模式建成[30-31], 植物生长发育[32-34], 生物和非生物胁迫的响应[35-36]以及苯丙烷代谢途径的调控[37-39]。但是 MYB家族的大部分成员的功能仍不清楚。因此从这个大家族中鉴定出异黄酮合成的候选调控子,将使我们更好地剖析异黄酮合成的潜在机制, 从而最终为更有效地操控异黄酮的生物合成以满足不同植物的特异需求奠定坚实的基础。
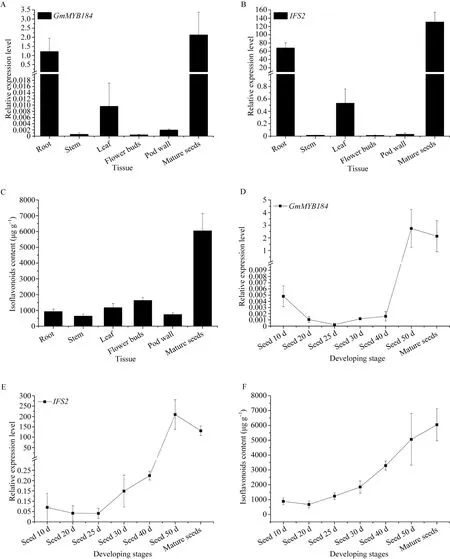
图5 大豆不同组织和不同发育时期的种子中GmMYB184、IFS2的相对表达量及异黄酮含量Fig. 5 Relative expression levels of GmMYB184 and IFS2, and isoflavone contents in different tissues at different developing stages of soybean seed
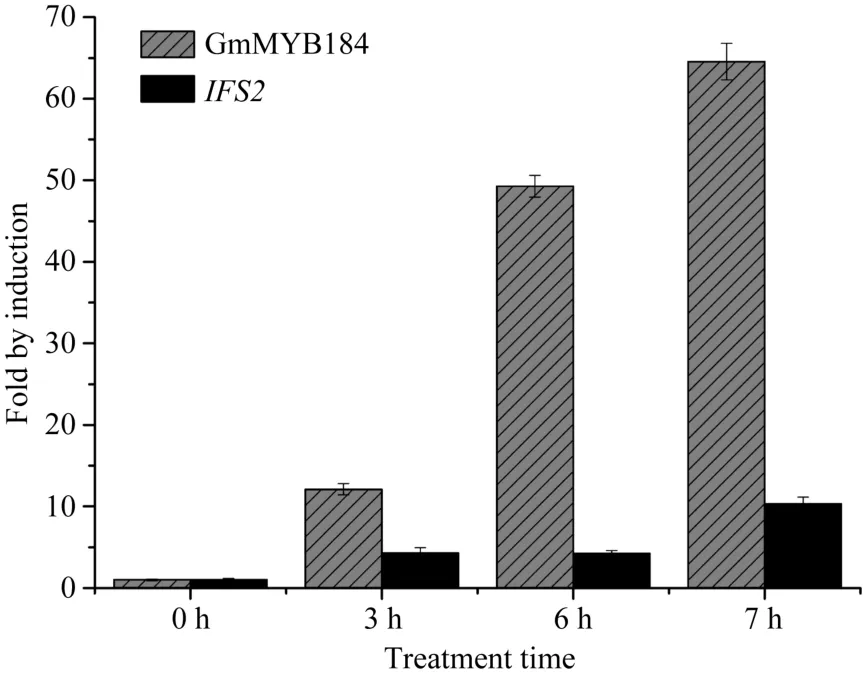
图6 GmMYB184和IFS2的谷胱甘肽诱导表达模式Fig. 6 GSH induced expression pattern of GmMYB184 and IFS2
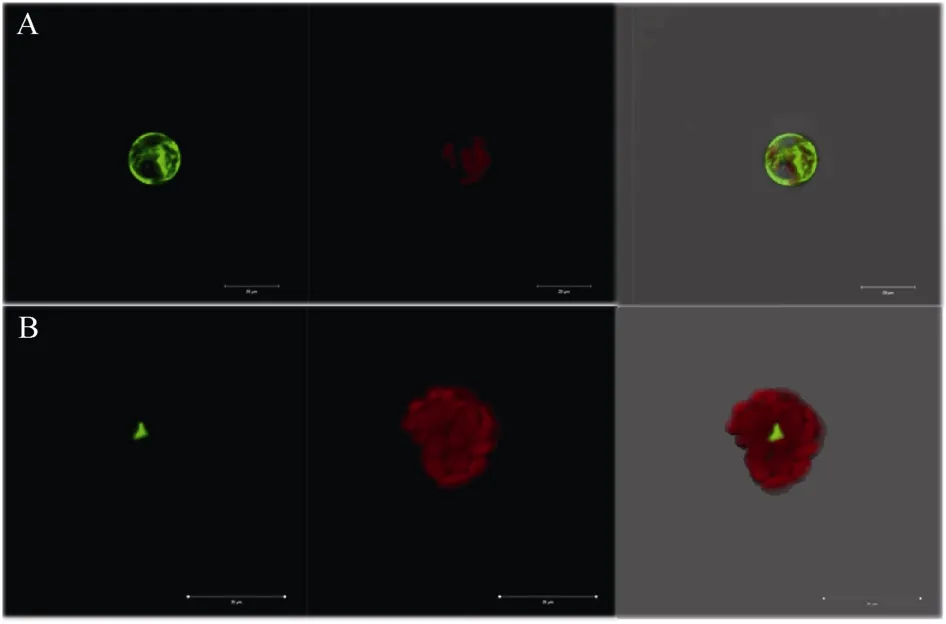
图7 GmMYB184的亚细胞定位Fig. 7 Subcellular localization analysis of GmMYB184

图8 GmMYB184对IFS2和CHS8的启动子激活活性Fig. 8 GmMYB184 induce the promoter activity of IFS2 and CHS8

图9 毛状根中沉默和过表达GmMYB184的相对表达量Fig. 9 Relative expression levels of RNAi and over expression of GmMYB184 in hairy roots
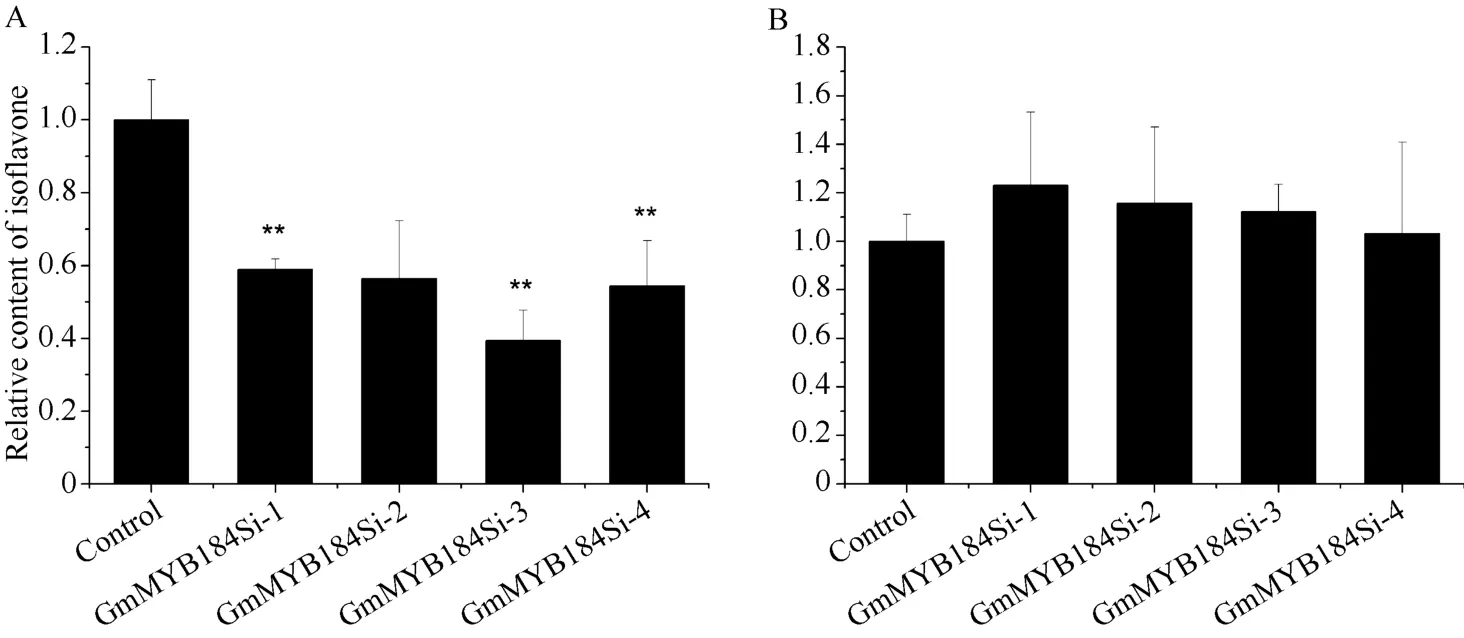
图10 沉默和过表达GmMYB184毛状根相对异黄酮含量Fig. 10 Relative isoflavone contents of RNAi and over expression of GmMYB184 in hairy roots
在大豆内源调控异黄酮合成的转录因子发现之前, 人们通过引入外源转录因子调控大豆异黄酮合成, 如引入玉米的C1和R转录因子, 用于提高大豆籽粒中的异黄酮含量[40]。C1是一个 R2R3-MYB转录因子, 参与玉米花青素积累, 行使功能时需要一个含有R-MYC的bHLH辅助因子[41]。R插入C1的DNA结合和激活结构域, 形成一种嵌合蛋白(CRC),可以使异黄酮含量比野生型上升4倍。与此同时, 黄酮合成途径的基因表达被抑制了, 代谢流和底物转向了异黄酮途径[40], 这也暗示了苯丙烷代谢途径各分支之间对于代谢前体的竞争。
大豆内源转录因子的发现为利用大豆自身转录因子调控异黄酮合成提供了新的契机。目前已发现3个 R2R3-MYB转录因子与大豆异黄酮合成相关,其中GmMYB39和GmMYB100在异黄酮的积累中表现负调控的功能, 过表达目的基因抑制异黄酮的积累[20-21]。最近报道的GmMYB29转录因子在异黄酮合成中起正调控作用, 该转录因子能够激活CHS8和 IFS2的表达, 过表达和干扰该基因能够分别提高和抑制异黄酮的合成[19]。与GmMYB29类似,本研究鉴定的 GmMYB184也对异黄酮的生物合成起正调控作用。这些大豆内源转录因子的发现为调控异黄酮合成提供了新的靶向基因位点。
3.2 大豆GmMYB184对异黄酮合成的调控作用
GmMYB184的表达受干旱、盐害、冷害和ABA胁迫响应, 暗示它可能在植物非生物胁迫中发挥重要作用[29]。同时, 此基因与豆科植物百脉根中的LjMYB62同源, 后者受GSH诱导, 因为GSH可以诱导异黄酮产生[22-23], 因此推测GmMYB184可能与异黄酮合成相关。表达分析发现, GmMYB184的组织表达和诱导表达与异黄酮合成关键基因IFS2的表达模式一致, 暗示二者可能参与相同的代谢途径。在GSH诱导表达中, 我们注意到6 h后, GmMYB184的诱导表达早于IFS2基因, 这与一般认为转录因子先于靶标基因的表达一致[42]。启动子激活活性分析表明, GmMYB184与GmMYB29都可以激活异黄酮代谢途径的 IFS2和 CHS8基因, 但激活活性GmMYB184低于 GmMYB29, 可能 GmMYB184还作用于其他靶标基因。另外, 尽管过表达GmMYB184能够使自身转录水平提高, 但是, 异黄酮的含量并没有显著提高。对于这样的结果, 可能的原因是 GmMYB184能够在拟南芥原生质体中与CHS8和 IFS2的启动子结合, 并激活基因的表达;但是毛状根中GmMYB184的过表达是否造成异黄酮合成相关的结构基因的大量表达还有待检测。即使这些异黄酮合成相关的结构基因表达量上升, 其中CHS8可能还会限制代谢流进入异黄酮合成途径,导致异黄酮含量没有显著变化, 甚至降低[43]。此外,由于某一组织中的异黄酮是由该组织合成的或者由其他母体组织转运而来的, 因此, 异黄酮合成相关
4 结论
基因以及其调控子的转录水平可能反映的是它们合成异黄酮的能力, 而不是在这个组织中积累异黄酮的能力。
这些研究表明转录因子对大豆异黄酮的积累存在复杂的调控机制。随着基因组学和分子生物学技术的发展, 越来越多的大豆异黄酮合成相关的转录因子将被鉴定出来, 今后需要研究这些转录因子调控大豆异黄酮合成的分子机制, 并评估对大豆异黄酮含量的影响, 从而可以有效地利用转录因子操控异黄酮的生物合成。
克隆了一个与大豆异黄酮合成相关的 R2R3类型MYB转录因子GmMYB184。验证了该转录因子对异黄酮合成途径关键基因的转录激活活性及其在异黄酮合成中的正向调控作用。
[1]Yu O, McGonigle B. Metabolic engineering of isoflavone biosynthesis. Adv Agron 2005, 86: 147–190
[2]Cederroth C R, Nef S. Soy, phytoestrogens and metabolism: a review. Mol Cell Endocrinol 2009, 304: 30–42
[3]Rochfort S, Panozzo J. Phytochemicals for health, the role of pulses. J Agric Food Chem 2007, 55: 7981–7994
[4]Dixon R A. Natural products and plant disease resistance. Nature 2001, 411: 843–847
[5]Graham T L, Graham M Y. Signaling in soybean phenylpropanoid responses (dissection of primary, secondary, and conditioning effects of light, wounding, and elicitor treatments). Plant Physiol, 1996, 110: 1123–1133
[6]Hammerschmidt R. PHYTOALEXINS: what have we learned after 60 years? Annu Rev Phytopathol, 1999, 37: 285–306
[7]Gutierrez-Gonzalez J J, Wu X, Gillman J D, Lee J D, Zhong R,Yu O, Shannon G, Ellersieck M, Nguyen H T, Sleper D A. Intricate environment-modulated genetic networks control isoflavone accumulation in soybean seeds. BMC Plant Biol, 2010, 10: 105
[8]Dixon R A, Steele C L. Flavonoids and isoflavonoids: a gold mine for metabolic engineering. Trends Plant Sci, 1999, 4:394–400
[9]Zernova O V, Lygin A V, Widholm J M, Lozovaya V V. Modification of isoflavones in soybean seeds via expression of multiple phenolic biosynthetic genes. Plant Physiol Biochem, 2009, 47:769–777
[10]易金鑫, 徐照龙, 王峻峰, 张大勇, 何晓兰, 朱虹润, 马鸿翔.GmCHS8和GmIFS2基因共同决定大豆中异黄酮的积累. 作物学报, 2011, 37: 571–578 Yi J X, Xu Z L, Wang J F, Zhang D Y, He X L, Zhu H R, Ma H X.The GmCHS8 and GmIFS2 genes together determine the accumulation of isoflavones in soybeans. Acta Agron Sin, 2011, 37:571–578 (in Chinese with English abstract)
[11]Butelli E, Titta L, Giorgio M, Mock H P, Matros A, Peterek S,Schijlen E G, Hall R D, Bovy A G, Luo J, Martin, C. Enrichment of tomato fruit with health-promoting anthocyanins by expression of select transcription factors. Nat Biotechnol, 2008, 26:1301–1308
[12]Cutanda-Perez M C, Ageorges A, Gomez C, Vialet S, Terrier N,Romieu C, Torregrosa L. Ectopic expression of VlmybA1 in grapevine activates a narrow set of genes involved in anthocyanin synthesis and transport. Plant Mol Biol, 2009, 69: 633–648
[13]Hichri I, Barrieu F, Bogs J, Kappel C, Delrot S, Lauvergeat V.Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway. J Exp Bot, 2011, 62: 2465–2483
[14]Zhou M L, Hou H L, Zhu X M, Shao J R, Wu Y M, Tang Y X.Soybean transcription factor GmMYBZ2 represses catharanthine biosynthesis in hairy roots of Catharanthus roseus. Appl Microbiol Biotechnol, 2011, 91: 1095–1105
[15]Chen Y H, Yang X Y, He K, Liu M H, Li J G, Gao Z F, Lin Z Q,Zhang Y F, Wang X X, Qiu X M, Shen Y P, Zhang L, Deng X H,Luo J C, Deng X W, Chen Z L, Gu H Y, Qu L J. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family. Plant Mol Biol, 2006, 60: 107–124
[16]Jin H, Martin C. Multifunctionality and diversity within the plant MYB-gene family. Plant Mol Biol, 1999, 41: 577–585
[17]Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana. Curr Opin Plant Biol, 2001, 4: 447–456
[18]Du H, Yang S S, Liang Z, Feng B R, Liu L, Huang Y B, Tang Y X.Genome-wide analysis of the MYB transcription factor superfamily in soybean. BMC Plant Biol, 2012, 12: 106
[19]Chu S, Wang J, Zhu Y, Liu S, Zhou X, Zhang H, Wang C E, Yang W, Tian Z, Cheng H, Yu D Y. An R2R3-type MYB transcription factor, GmMYB29, regulates isoflavone biosynthesis in soybean.PLoS Genet, 2017, 13: e1006770
[20]Liu X, Yuan L, Xu L, Xu Z, Huang Y, He X, Ma H, Yi J, Zhang D.Over-expression of GmMYB39 leads to an inhibition of the isoflavonoid biosynthesis in soybean (Glycine max L.). Plant Biotechnol Rep, 2013, 7: 445–455
[21]Yan J, Wang B, Zhong Y, Yao L, Cheng L, Wu T. The soybean R2R3 MYB transcription factor GmMYB100 negatively regulates plant flavonoid biosynthesis. Plant Mol Biol, 2015, 89: 35–48
[22]Robbins M P, Hartnoll J, Morris P. Phenylpropanoid defence responses in transgenic Lotus corniculatus 1. Glutathione elicitation of isoflavan phytoalexins in transformed root cultures. Plant Cell Rep, 1991, 10: 59–62
[23]Shelton D, Stranne M, Mikkelsen L, Pakseresht N, Welham T,Hiraka H, Tabata S, Sato S, Paquette S, Wang T L, Martin C,Bailey P. Transcription factors of Lotus: regulation of isoflavonoid biosynthesis requires coordinated changes in transcription factor activity. Plant Physiol, 2012, 159: 531–547
[24]Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15: 573–581
[25]Higgins D G, Thompson J D, Gibson T J. Using CLUSTAL for multiple sequence alignments. Method Enzymol, 1996, 266:383–402
[26]Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:Molecular Evolutionary Genetics Analysis version 6.0. Mol Biol Evol, 2013, 30: 2725–2729
[27]Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T))method. Methods, 2001, 25: 402–408
[28]Sun J M, Sun B L, Han F X, Yan S R, Yang H, Kikuchi A. Rapid HPLC method for determination of 12 isoflavone components in soybean seeds. Agric Sci China, 2011, 10: 70–77
[29]Chung E S, Kim K M, Lee J H. Molecular cloning and characterization of a soybean GmMBY184 induced by abiotic stresses. J Plant Biotechnol, 2012, 39: 175–181
[30]Ishida T, Kurata T, Okada K, Wada T. A genetic regulatory network in the development of trichomes and root hairs. Annu Review Plant Biol, 2008, 59: 365–386
[31]Nadeau J A. Stomatal development: new signals and fate determinants. Curr Opin Plant Biol, 2009, 12: 29–35
[32]Wang X, Niu Q W, Teng C, Li C, Mu J, Chua N H, Zuo J.Overexpression of PGA37/MYB118 and MYB115 promotes vegetative-to-embryonic transition in Arabidopsis. Cell Res, 2009,19: 224–235
[33]Muller D, Schmitz G, Theres K. Blind homologous R2R3 Myb genes control the pattern of lateral meristem initiation in Arabidopsis. Plant Cell, 2006, 18: 586–597
[34]Mu R L, Cao Y R, Liu Y F, Lei G, Zou H F, Liao Y, Wang H W,Zhang W K, Ma B, Du J Z, Yuan M, Zhang J S, Chen S Y. An R2R3-type transcription factor gene AtMYB59 regulates root growth and cell cycle progression in Arabidopsis. Cell Res, 2009,19: 1291–1304
[35]Abe H, Yamaguchi-Shinozaki K, Urao T, Iwasaki T, Hosokawa D,Shinozaki K. Role of Arabidopsis MYC and MYB homologs in drought- and abscisic acid-regulated gene expression. Plant Cell,1997, 9: 1859–1868
[36]Mengiste T, Chen X, Salmeron J, Dietrich R. The BOTRYTIS SUSCEPTIBLE1 gene encodes an R2R3MYB transcription factor protein that is required for biotic and abiotic stress responses in Arabidopsis. Plant Cell, 2003, 15: 2551–2565
[37]Borevitz J O, Xia Y, Blount J, Dixon R A, Lamb C. Activation tagging identifies a conserved MYB regulator of phenylpropanoid biosynthesis. Plant Cell, 2000, 12: 2383–2394
[38]Sablowski R W, Moyano E, Culianez-Macia F A, Schuch W,Martin C, Bevan M. A flower-specific Myb protein activates transcription of phenylpropanoid biosynthetic genes. EMBO J,1994, 13: 128–137
[39]Solano R, Nieto C, Avila J, Canas L, Diaz I, Paz-Ares J. Dual DNA binding specificity of a petal epidermis-specific MYB transcription factor (MYB.Ph3) from Petunia hybrida. EMBO J,1995, 14: 1773–1784
[40]Yu O, Shi J, Hession A O, Maxwell C A, McGonigle B, Odell J T.Metabolic engineering to increase isoflavone biosynthesis in soybean seed. Phytochemistry, 2003, 63: 753–763
[41]Grotewold E, Chamberlin M, Snook M, Siame B, Butler L,Swenson J, Maddock S, St Clair G, Bowen B. Engineering secondary metabolism in maize cells by ectopic expression of transcription factors. Plant Cell, 1998, 10: 721–740
[42]Holl J, Vannozzi A, Czemmel S, D′Onofrio C, Walker A R,Rausch T, Lucchin M, Boss P K, Dry I B, Bogs J. The R2R3-MYB transcription factors MYB14 and MYB15 regulate stilbene biosynthesis in Vitis vinifera. Plant Cell, 2013, 25:4135–4149
[43]Dhaubhadel S, Gijzen M, Moy P, Farhangkhoee M. Transcriptome analysis reveals a critical role of CHS7 and CHS8 genes for isoflavonoid synthesis in soybean seeds. Plant Physiol, 2007, 143:326–338
