甘蓝型油菜蔗糖磷酸合酶(SPS)基因家族成员鉴定及表达分析
2018-03-01荐红举张翱翔张立源刘列钊徐新福李加纳
张 莉 荐红举 杨 博 张翱翔 张 超 杨 鸿 张立源刘列钊 徐新福 卢 坤 李加纳
西南大学农学与生物科技学院, 重庆 400715
蔗糖是高等植物光合作用的主要产物, 也是碳从源器官到库器官的移动形式, 蔗糖在作物生长和产量形成过程中起着重要作用[1]。蔗糖磷酸合酶(sucrose phosphate synthase, SPS)催化 UDPG (尿苷二磷酸葡糖)和F6P (果糖-6-磷酸)反应生成S6P (蔗糖-6-磷酸), S6P再进一步被SPP (磷酸蔗糖磷酸化酶)催化水解产生蔗糖[2], 而SPS和SPP以复合体形式存在于植物中, 因此生成蔗糖这一过程不可逆[3]。一般认为, SPS-Spase (蔗糖磷酸合酶-蔗糖磷酸化酶)系统是蔗糖合成主要途径, 在此途径中 SPS是关键限速酶[4]。SPS作为糖基转移酶超家族成员, 是由2个或4个亚基构成的二聚体或者四聚体[5]。目前, 在植物中共发现了4个SPS基因亚家族, 双子叶植物有A、B、C共3个基因亚家族[6-7], D家族为单子叶植物独有[8-9], 每个基因家族中至少有1个基因。在拟南芥中有 4个 SPS基因[10], 分别是 SPSA1(AT5G20280)、SPSA2 (AT5G11110)、SPSB(AT1G04920)和 SPSC (AT4G10120)。SPSA1在拟南芥光合作用合成蔗糖中起主要作用[10], SPSA2参与拟南芥花粉发育后期蔗糖代谢[11], SPSA1和 SPSA2在拟南芥成熟蜜腺中高度表达, 是花蜜分泌必不可少的基因[12]; SPSB在烟草花药和子房中大量表达,为花粉粒萌发提供碳源[13]; SPSC是参与拟南芥和烟草夜间蔗糖合成的主要基因, 同时能够维持淀粉正常运输[14]。
SPS作为蔗糖合成催化剂, 其活力大小与蔗糖积累量成正比[3,15], SPS在蔗糖代谢中主要影响源强和库强, 调节光合产物在蔗糖和淀粉的分配, 参与细胞分化与纤维细胞壁合成[16]。Park等[14]在烟草中转入 AtSPSA1基因, 导致转基因烟草库器官蔗糖浓度提高, 而源器官蔗糖浓度不变, 所有转基因植株高度增加、茎秆增粗、纤维增长、总干重增加。Baxter等[17]在烟草中转入玉米 SPS基因, 导致转基因烟草开花时间提前, 生物量有增加趋势, 花的数量、蔗糖积累量和碳同化量都大幅提高。Ishimaru等[18]在土豆中转入玉米SPS基因, 导致转基因土豆中SPS活性增加, 光合产物从源到库的转移增加, 光合作用时间延长, 产量提高。
在许多作物中, SPS活性受昼夜循环调控[19], 受低温[20]、渗透胁迫[21]和高CO2[22]的诱导。作为控制CO2同化形成蔗糖的一个关键点, SPS蛋白合成不仅受 SPS基因的转录调控[23-24], 同时还受包括氧化磷酸化蛋白翻译后的修饰作用、G6P (葡萄糖-6-磷酸)的活化作用和无机正磷酸盐的抑制作用在内的多个机能多个层次结构控制[3,25]。
油菜是我国播种面积最大, 地区分布最广的油料作物。油菜中蔗糖主要在光合器官细胞质基质中合成, 通过韧皮组织向其他器官和组织运输, 为油菜生长发育提供碳源和能量。SPS作为油菜蔗糖合成过程关键调控酶, 主要促进蔗糖合成。SPS的活性体现了籽粒对同化物的需求程度, 高活性 SPS有利于油菜籽油分累积[26]。本研究利用生物信息学分析方法, 基于已经公布的油菜基因组全序列信息, 对油菜 SPS基因全部成员进行鉴定, 从基因组水平上分析了油菜SPS基因数目、基因进化和结构、在染色体上的分布、启动子区域顺式作用元件以及在不同组织器官中的表达情况, 为进一步研究该家族成员的生物学功能奠定理论基础。
1 材料与方法
1.1 试验材料
2015年9月至2016年5月在重庆市油菜工程技术研究中心歇马实验基地种植各甘蓝型油菜品种。田间试验按 3个重复随机区组设计, 每个小区种植2行, 行距40 cm, 株距20 cm。分别于盛花期取甘蓝型油菜“中双 11”的根、茎、叶、蕾和花, 花后30 d种子和角果皮, 在液氮中速冻并存于–80℃,用于分析SPS基因在甘蓝型油菜不同器官的表达水平。为分析SPS基因在高、低生物产量甘蓝型油菜组织器官中表达差异情况, 从33个国内甘蓝型油菜自交系(见附表 1)中筛选出 3份高生物产量(生物产量分别为155、171和172 g 株-1)和2份低生物产量材料(生物产量分别为105 g 株-1和111 g 株-1)。这33份材料组成的群体生物产量性状值符合正态分布(见附图1)。分别于盛花期取其茎, 叶, 花后30 d种子和角果皮, 在液氮中速冻并存于–80℃待用。
1.2 甘蓝型油菜、白菜和甘蓝中SPS基因的鉴定
拟南芥SPS基因序列来源于TAIR (http://www.arabidopsis.org/), 利用拟南芥 SPS蛋白序列分别在甘蓝型油菜基因组数据库(http://www.genoscope.cns.fr/brassicanapus/)和BRAD (http://brassicadb.org/brad/index.php)进行 BLASTP分析, 获取甘蓝型油菜、白菜及甘蓝的SPS蛋白序列。
1.3 甘蓝型油菜SPS基因的系统进化分析
利用ClustalW默认设置对SPS氨基酸序列进行多重序列比对, 并在 MEGA 6软件中使用邻接法(neighbour-joining, NJ)构建进化树[27], 校验参数bootstrap为重复1000次。
1.4 蛋白特性和序列分析
利用ExPASy Proteomics Sever (http://expasy.org/)预测甘蓝型油菜SPS蛋白氨基酸序列分子量和等电点[28]。利用GSDS2.0 (http://gsds.cbi.pku.edu.cn/index.php)绘出甘蓝型油菜 SPS基因结构[29]。通过MEME 网站(http://meme-suite.org/tools/meme)分析甘蓝型油菜SPS蛋白的保守元件[30], 设置最大元件数量为 25个, 元件的最佳宽度 6~50。利用InterProScan网站(http://www.ebi.ac.uk/interpro/search/sequence-search)对元件进行注释[31]。
1.5 甘蓝型油菜 SPS基因启动子顺式作用元件分析
从甘蓝型油菜基因组网站(http://www.genoscope.cns.fr/brassicanapus/)获得SPS基因启动子序列(转录起始位点上游 1.5 kb), 并利用 PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测甘蓝型油菜SPS基因启动子顺式作用元件[32-33]。
1.6 RNA提取和实时荧光定量PCR
使用 RNA提取试剂盒(Invitrogen)提取各组织样品总RNA。首先利用DNase I去除受污染基因组DNA, 根据试剂盒说明, 利用反转录酶(TaKaRa)将1 μg RNA反转录形成cDNA。根据Wei等[34]描述的实时荧光定量PCR方法确定SPS基因在油菜不同组织的表达水平, 各样品3个重复。引物序列见附表2。
2 结果与分析
2.1 甘蓝型油菜SPS基因鉴定和系统发生分析
通过BLASTP分析, 获得11个甘蓝型油菜、6个白菜和5个甘蓝的SPS基因家族成员。这11个油菜SPS蛋白的氨基酸长度从473 (BnSPSC-1)到1065(BnSPSB-2), 平均 953; 相对分子质量从 53.8 kD(BnSPSC-1)到119.6 kD (BnSPSB-2); 等电点从5.52(BnSPSA1-3)到8.06 (BnSPSC-1), 除1个成员外, 其余SPS蛋白成员的等电点均在7以下, 即呈酸性形式存在(表1)。

表1 甘蓝型油菜SPS预测编码蛋白信息Table 1 Information of predicted BnSPS proteins
对拟南芥、甘蓝型油菜、白菜和甘蓝的SPS氨基酸序列系统进化树分析表明, SPS基因群被分成A、B和C 3个亚家族(图1), 其中, A家族包括7个甘蓝型油菜、2个拟南芥、4个白菜和3个甘蓝SPS基因, B家族包括2个甘蓝型油菜、1个拟南芥、1个白菜和1个甘蓝SPS基因, C家族则由2个甘蓝型油菜、1个拟南芥、1个白菜和1个甘蓝SPS基因构成。
2.2 甘蓝型油菜中SPS基因的染色体位置, 基因结构和保守结构域
6个白菜SPS基因分布在4条染色体上(图2-a),5个甘蓝SPS基因分布在4条染色体上(图2-b), 11个甘蓝型油菜SPS基因分布在9条染色体上(图2-c),每条染色体上有 1~2个基因。根据染色体定位分布信息和序列比对分析, 发现该家族成员没有串联重复, BnSPSA1-1/A1-2/A1-3、BnSPSA2-1/A2-2/A2-4、BnSPSA2-3/A2-4、BnSPSB-1/B-2和 BnSPSC-1/C-2参与基因的片段复制。

图1 拟南芥、甘蓝、白菜和甘蓝型油菜中SPS家族进化树(邻接法)Fig. 1 Phylogenetic tree of SPS proteins in A. thaliana, B. oleracea, B. rape, and B. napus (neighbor-joining method)

图2 白菜(a)、甘蓝(b)和甘蓝型油菜(c)的SPS基因染色体分布Fig. 2 Chromosomal mapping of BrSPS (a), BoSPS (b), and BnSPS (c)
在同一类型基因中, 可以通过分析它们内含子长度或者外显子数目来判断它们之间的紧密程度。例如, A家族基因有13~15个外显子, 有3个基因含有 14个外显子, 3个基因含有 15个外显子, 只有SPSA2-1含有13个外显子; B家族的基因都含有11个外显子; C家族中 SPSC-1只有 5个外显子, 而SPSC-2含有13个外显子, 而且SPSC-2的长度明显大于 SPSC-1 (图 3-A)。
利用MEME软件, 在甘蓝型油菜SPS蛋白中预测出 25个保守元件。元件 1~4、8、11、12、16和18在所有SPS蛋白中都存在; 除SPSC-1之外, 其他10个 SPS蛋白均含有元件 5~7、9、10、13、15、17、19、20和23。元件24和25只在A家族中存在, 元件14和21同时在A、B家族中存在, 元件22在除SPSA2-4和 SPSC-1外的其他 SPS蛋白中存在(图3-B)。根据InterProScan注释, 元件1~8都是糖基转移酶蛋白, 元件2~4、6和8是SPS相关蛋白。SPSC-1在基因结构和保守元件中具有较大特异性。
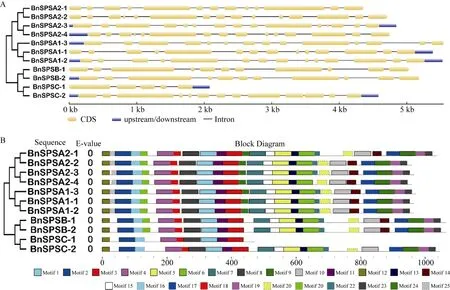
图3 油菜SPS基因家族的结构(A)和保守元件(B)Fig. 3 Structures (A) and identified motifs (B) of BnSPS gene family
2.3 油菜SPS基因启动子的顺式调控元件
在油菜 SPS上除了有 TATA-box和 CAAT-box等一些基本的启动子保守区以外, 还有许多与逆境和激素反应相关的重要顺式作用元件(表 2), 如 23个抗氧化反应元件(ARE), 18个CGTCA-motif 和18个TGACG-motif茉莉酸甲酯响应元件, 17个赤霉素响应元件(GARE-motif), 17个脱落酸响应元件(ABRE), 11个干旱响应元件(MBS), 以及11个水杨酸响应元件(TCA-element)。
2.4 SPS基因在甘蓝型油菜不同组织器官中的表达分析
BnSPSA1-1、BnSPSA1-2和 BnSPSA1-3均在花中表达量最高, BnSPSA1-2和BnSPSA1-3在蕾和角果皮中也有较高表达; BnSPSA2-1、BnSPSA2-2、BnSPSA2-3和BnSPSA2-4在各部位均有不同程度表达, BnSPSA2-1和 BnSPSA2-2在花中表达量最高, BnSPSA2-3和BnSPSA2-4在蕾中表达量最高; BnSPSB-1和BnSPSB-2只在叶、蕾和花中表达, 且蕾中表达量最高; BnSPSC-1和 BnSPSC-2在叶中表达量最高, 在蕾和花中也有不同程度的表达, 在其他组织器官中基本不表达(图4)。
2.5 SPS基因在高、低生物产量油菜不同组织器官中的表达分析
BnSPSA-1、BnSPSA-2和BnSPSA-3在叶片中的表达量基本上是高生物产量油菜高于低生物产量油菜; BnSPSB-1和BnSPSB-2只在叶片中表达, 且高生物产量油菜中的表达量极显著低于低生物产量油菜;BnSPSC-1和BnSPSC-2主要在叶片中表达, 且低生物产量油菜高于高生物产量油菜(图5)。
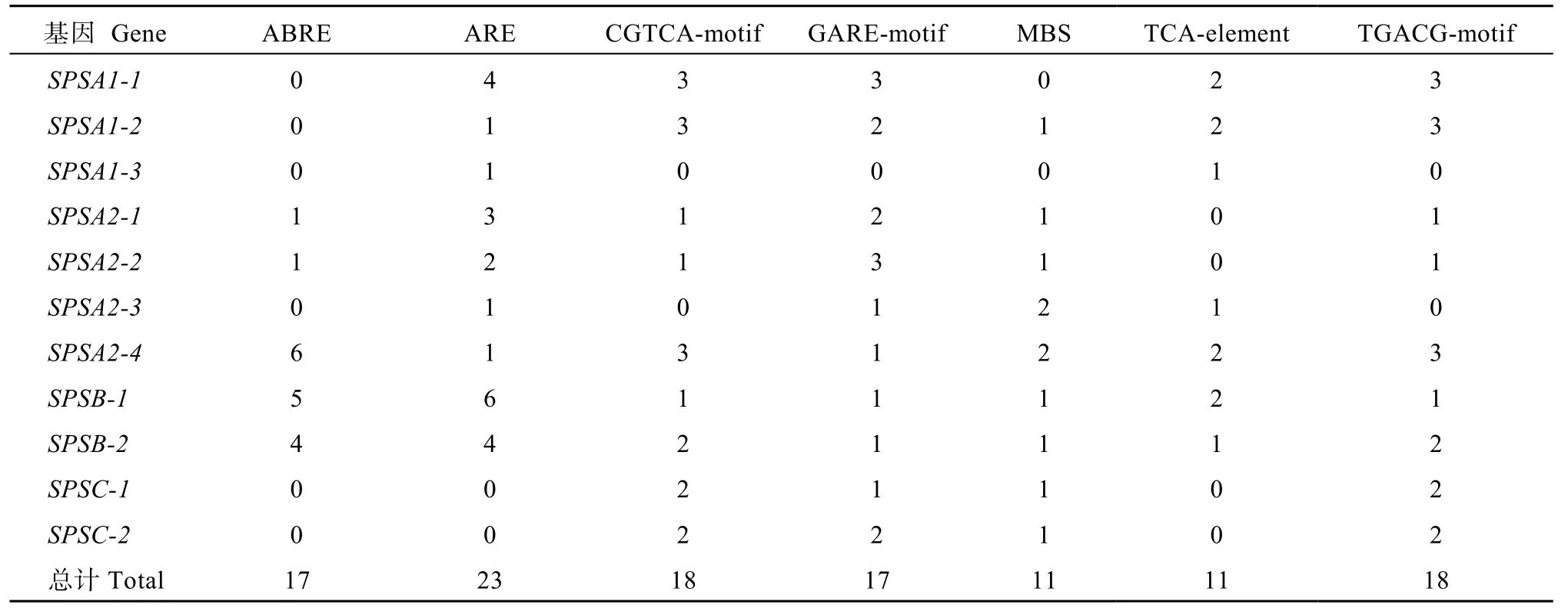
表2 甘蓝型油菜SPS基因启动子中顺式作用元件的种类与数量Table 2 Types and quantities of cis-acting elements in BnSPS promoters

图4 BnSPS基因在中双11中的组织特异表达分析Fig. 4 Tissue-specific expression patterns of BnSPS genes in Zhongshuang 11

图5 高、低生物产量甘蓝型油菜SPS基因在不同器官中的差异表达Fig. 5 Differential expression of BnSPS genes in different organs between high-biomass and low-biomass rapeseed varieties
3 讨论
随着大规模基因组测序的发展, 比较基因组学在基因组分析和注释方面的功能逐渐展露, 利用比较基因组学进行基因家族分析已经成为现今基因功能研究得热点。目前已经在多种粮食作物和水果中进行了类似研究, 包括大豆 LEA基因家族[35], 番茄LBD基因家族[36]以及梨CBL基因家族[37]等, 为各基因家族功能研究奠定了理论基础。甘蓝型油菜作为重要的油料作物, 基因组测序完成后, 对 SUC和SWEET等[38], SnRK2等[39]及WRKY等[40]家族都进行了广泛的生物信息学分析。SPS在作物生长和产量形成中起着重要的作用, 但是在油菜中有关 SPS基因的研究未见报道。本文分析了甘蓝型油菜SPS基因的系统发育关系, 内含子和外显子结构, 染色体定位, 保守元件, 不同组织器官中的表达模式和在高、低生物产量油菜中的差异表达。
3.1 甘蓝型油菜SPS基因的进化
多倍体化是真核生物进化的主要推动者[41]。芸薹属植物由拟南芥进化之后, 经历全基因组三倍化事件, 基因数目增多[42]。白菜和甘蓝作为二倍体芸薹属植物, 理论上拟南芥的单个基因在其染色体上应该有3个拷贝[43-44]。甘蓝型油菜(Brassica napus L.,2n = 4x = 38, AACC)是由白菜(B. rapa, 2n = 2x = 20,AA)和甘蓝(B. oleracea, 2n = 2x = 18, CC)自然杂交形成的异源四倍体植物, 因此拟南芥的单个基因在其染色体上应该有 6个拷贝[45]。然而, 由于基因组的坍塌和丢失, 拟南芥的单个基因在白菜[43]和甘蓝型油菜[45]中分别只有1.5~2.0和2.0~6.0个拷贝。本研究共鉴定出11个甘蓝型油菜SPS基因, 分布在9条染色体上, 其中BnSPSA2-2定位在C03染色体上,结合白菜和甘蓝中 SPS基因在染色体上的分布, 发现并没有BoSPS基因定位在C03染色体上, 猜测这可能是甘蓝型油菜在进化过程中染色体片段交换的结果。本研究还发现各BnSPS基因之间没有串联重复, 有 4组片段重复, 说明片段复制事件是 BnSPS在基因组内扩增的主要方式。
油菜各 SPS分子特征有较大差异, 其分子量和等电点是根据油菜SPS基因家族的分类而变化的。外显子结构差异对基因家族的进化起重要作用[46-47],本研究对SPS基因家族结构分析也解释了基因结构的多样性。A家族中外显子数目略多于B、C家族, 同时 C家族中两个基因的外显子数目差异也较大, 这暗示各基因功能的差异。例如, 11个 SPS基因中,SPSC-1外显子数目明显低于其他基因, 且由该基因合成的蛋白质其氨基酸平均长度最短, 等电点相对最高, 这可能是在进化过程中基因片段丢失、N端缩短等原因造成的[48]。外显子结构和保守元件分析,也揭示了在不同物种中的SPS基因具有相同的起源和进化模式, 从整体上看, 具有相同保守元件组成的基因都聚集在同一家族中, 这暗示了同一家族内部各基因的功能有相似性。
3.2 甘蓝型油菜SPS基因的差异表达
11个SPS基因在甘蓝型油菜不同组织器官中有差异表达, BnSPSA1-2、BnSPSA1-3、BnSPSC-1和BnSPSC-2在叶片中表达量较高, 这与拟南芥叶片中主要是SPSA1和SPSC表达的结果[49]一致。水稻中OsSPS1 (B家族)是花粉发育必不可少的基因[50], 烟草中 SPSB基因在花药和子房中大量表达[51]。与此一致, 本研究中BnSPSB-1和BnSPSB-2均在油菜蕾中表达量最高。由此推测, BnSPSB在油菜花粉发育中可能起重要作用。据报道, SPSB基因在种子中的表达量较高[2,10,49]。然而, 本研究中 BnSPSB-1和BnSPSB-2在种子中基本不表达, 这可能是由于甘蓝型油菜在进化过程中SPS基因功能发生分化。
以叶、茎和角果皮等光合器官作为研究材料,BnSPSA1和BnSPSC作为在叶片中主要表达的基因,其在高生物产量油菜叶片中的表达量高于低生物产量油菜, 这与前人报道一致。Park等[14]在烟草中转入AtSPSA1基因, Baxter等[17]在烟草中转入ZmSPS基因, Ishimaru等[18]在马铃薯中转入ZmSPS基因, 均发现转基因作物的产量或干重提高。由此推测,BnSPSA1和BnSPSC与甘蓝型油菜生物产量的积累相关。BnSPSB在低生物产量油菜叶片中的表达量高于高生物产量油菜, 可能是由于低生物产量油菜的蔗糖合成不足, BnSPSB作为蔗糖合成候补基因发挥功能, 而高生物产量油菜蔗糖合成充足抑制了 BnSPSB在叶片中的表达。Lu等[52]通过全基因组关联分析发现SPSA2-3是与甘蓝型油菜生物产量相关的候选基因,而在本研究中SPSA2-3在高、低生物产量油菜光合器官中的表达没有明显规律, 需要进一步研究论证。
4 结论
通过对油菜 SPS基因家族全基因组鉴定, 共获得11个基因, 分属3个不同家族, 分布在9条染色体上, 具有较高的保守性, 大部分基因都含有与逆境和激素相关的顺式作用元件, 基因的表达具有明显的组织器官特异性, 与油菜生物产量密切相关。
[1]Farrar J, Pollock C, Gallagher J. Sucrose and the integration of metabolism in vascular plants. Plant Sci, 2000, 154: 1–11
[2]Bahaji A, Baroja F E, Ricarte B A, Sánchez L Á M, Muñoz F J,Romero J M, Ruiz M T, Baslam M, Almagro G, Sesma M T, Pozueta R J. Characterization of multiple SPS knockout mutants reveals redundant functions of the four Arabidopsis sucrose phosphate synthase isoforms in plant viability, and strongly indicates that enhanced respiration and accelerated starch turnover can alleviate the blockage of sucrose biosynthesis. Plant Sci,2015, 238: 135–147
[3]Huber S C, Huber J L. Role and regulation of sucrose-phosphate synthase in higher plants. Annu Rev Plant Biol, 1996, 47:199–222
[4]李永庚, 于振文, 姜东, 余松烈. 冬小麦旗叶蔗糖和籽粒淀粉合成动态及与其有关的酶活性的研究. 作物学报, 2001, 27:658–664 Li Y G, Yu Z W, Jiang D, Yu S L. Studies on the dynamic changes of the synthesis of sucrose in the flag leaf and starch in the grain and related enzymes of high yielding wheat. Acta Agron Sin,2001, 27: 658–664 (in Chinese with English abstract)
[5]Sawitri W D, Narita H, Ishizaka I E, Sugiharto B, Hase T, Nakagawa A. Purification and characterization of recombinant sugarcane sucrose phosphate synthase expressed in E. coli and insect Sf9 cells: an importance of the N-terminal domain for an allosteric regulatory property. J Biochem, 2016, 159: 599–607
[6]Langenkämper G, Fung R W M, Newcomb R D, Atkinson R G.Gardner R C, MacRae E A. Sucrose phosphate synthase genes in plants belong to three different families. J Mol Evol, 2002, 54:322–332
[7]Lunn J E, Macrae E. New complexities in the synthesis of sucrose. Curr Opin Plant Biol, 2003, 6: 208–214
[8]Castleden C K, Aoki N, Gillespie V J, MacRae E A, Quick W P,Buchner P, Foyer C H, Furbank R T, Lunn J E. Evolution and function of the sucrose-phosphate synthase gene families in wheat and other grasses. Plant Physiol, 2004, 135: 1753–1764
[9]Grof C P L, So C T E, Perroux J M, Bonnett G, Forrester R I. The five families of sucrose-phosphate synthase genes in Saccharum spp. are differentially expressed in leaves and stem. Funct Plant Biol, 2006, 33: 605–610
[10]Sun J D, Zhang J S, Larue C T, Larue C T, Huber S C. Decrease in leaf sucrose synthesis leads to increased leaf starch turnover and decreased RuBP regeneration-limited photosynthesis but not Rubisco-limited photosynthesis in Arabidopsis null mutants of SPSA1. Plant, Cell & Environ, 2011, 34: 592–604
[11]Jiang J, Zhang Z, Cao J. Pollen wall development: the associated enzymes and metabolic pathways. Plant Biol, 2013, 15: 249–263
[12]Lin I W, Sosso D, Chen L Q, Gase K, Kim S G, Kessler D,Klinkenberg P M, Gorder M K, Hou B H, Qu X Q, Carter C J,Baldwin I T, Frommer W B. Nectar secretion requires sucrose phosphate synthases and the sugar transporter SWEET9. Nature,2014, 508 : 546–549
[13]Chen S, HajirezaeI M, Börnke F. Differential expression of sucrosephosphate synthase isoenzymes in tobacco reflects their functional specialization during dark-governed starch mobilization in source leaves. Plant Physiol, 2005, 139: 1163–1174
[14]Park J Y, Canam T, Kang K Y, Ellis D D, Mansfild S D.Over-expression of an Arabidopsis family A sucrose phosphate synthase (SPS) gene alters plant growth and fibre development.Transgenic Res, 2008, 17: 181–192
[15]Huber S C. Role of sucrose-phosphate synthase in partitioning of carbon in leaves. Plant Physiol, 1983, 71: 818–821
[16]刘凌霄, 沈法富, 卢合全, 韩庆点, 刘云国. 蔗糖代谢中蔗糖磷酸合成酶(SPS)的研究进展. 分子植物育种, 2005, 3:275–281 Liu L X, Shen F F, Lu H Q, Han Q D, Liu Y G. Research Advance on sucrose phosphate synthase in sucrose metabolism. Mol Plant Breed, 2005, 3: 275–281 (in Chinese with English abstract)
[17]Baxter C J, Foyer C H, Turner J, Rolfe S A, Quick W P. Elevated sucrose phosphate synthase activity in transgenic tobacco sustains photosynthesis in old leaves and alters development. J Exp Bot,2003, 54: 1813–1820
[18]Ishimaru K, Hirotsu N, Kashiwagi T, Madoka Y, Nagasuga K,Ono K, Ohsugi R. Over-expression of a maize SPS gene improves yield characters of potato under field conditions. Plant Prod Sci, 2008, 11: 104–107
[19]Ohsugi R, Huber S C. Light modulation and localization of sucrose phosphate synthase activity between mesophyll cells and bundle sheath cells in C4species. Plant Physiol, 1987, 84: 1096–1101
[20]Reimholz R, Geiger M, Haake V, Deiting U, Krause K P, Sonnewald U, Stitt M. Potato plants contain multiple forms of sucrose phosphate synthase, which differ in their tissue distributions, their levels during development, and their responses to low temperature. Plant, Cell & Environ, 1997, 20: 291–305
[21]Quick P, Siegl G, Neuhaus E, Feil R, Stitt M. Shortterm water stress leads to a stimulation of sucrose synthesis by activating sucrose-phosphate synthase. Planta, 1989, 177: 535–546
[22]Seneweera S P, Basra A S, Barlow E W, Conroy J P. Diurnal regulation of leaf blade elongation in rice by CO2(Is it related to sucrose-phosphate synthase activity?). Plant Physiol, 1995, 108:1471–1477
[23]Gibon Y, Bläsing O E, Palacios R N, Pankovic D, Hendriks J H M, Fisahn J, Höhne M, Gunther M, Stitt M. Adjustment of diurnal starch turnover to short days: depletion of sugar during the night leads to a temporary inhibition of carbohydrate utilization,accumulation of sugars and post-translational activation of ADP-glucose pyrophosphorylase in the following light period.Plant J, 2004, 39: 847–862
[24]Okamura M, Aoki N, Hirose T, Yonekura M, Ohto C, Ohsugi R.Tissue specificity and diurnal change in gene expression of the sucrose phosphate synthase gene family in rice. Plant Sci, 2011,181: 159–166
[25]Winter H, Huber S C. Regulation of sucrose metabolism in higher plants: localization and regulation of activity of key enzymes.Crit Rev Biochem Mol Biol, 2000, 35: 253–289
[26]唐湘如, 官春云. 施氮对油菜几种酶活性的影响及其与产量和品质的关系. 中国油料作物学报, 2001, 23(4): 32–37 Tang X R, Guan C Y. Chinese journal of oil crop sciences, Effect of N application on activities of several enzymes and trait of yield and quality in rapeseed cultivar Xiangyou No.13. Chin J Oil Crop Sci, 2001, 23(4): 32–37 (in Chinese)
[27]Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:Molecular evolutionary genetics analysis version 6.0. Mol Biol Evol, 2013, 30: 2725–2729
[28]Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E, Grosdidier A, Hernandez C, Ioannidis V, Kuznetsov D, Liechti R, Moretti S,Mostaguir K, Redaschi N, Rossier G, Xenarios I, Stockinger H.ExPASy: SIB bioinformatics resource portal. Nucl Acids Res,2012, 40: W597–W603
[29]Hu B, Jin J P, Guo A Y, Zhang H, Luo J H, Gao G. GSDS 2.0: an upgraded gene feature visualization server. Bioinformatics, 2015,31: 1296–1297
[30]Bailey T L, Boden M, Buske F A, Frith M, Grant C E, Clementi L, Ren J Y, Li W W, Noble W S. MEME SUITE: tools for motif discovery and searching. Nucl Acids Res, 2009, 37: W202–W208
[31]Jones P, Binns D, Chang H Y, Fraser M, Li W Z, McAnulla C,McWilliam H, Maslen J, Mitchell A, Nuka G, Pesseat S, Quinn A F, Sangrador V A, Scheremetjew M, Yong S Y, Lopez R, Hunter S. InterProScan 5: genome-scale protein function classification.Bioinformatics, 2014, 30: 1236–1240
[32]Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Peer Y V D,Rouzé P, Rombauts S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucl Acids Res, 2002, 30: 325–327
[33]Rombauts S, Déhais P, Van M M, Rouzé P. PlantCARE, a plant cis-acting regulatory element database. Nucl Acids Res, 1999, 27:295–296
[34]Wei L J, Jian H J, Lu K, Filardo F, Yin N W, Liu L Z, Qu C M, Li W, Du H, Li J N. Genome-wide association analysis and differential expression analysis of resistance to Sclerotinia stem rot in Brassica napus. Plant Biotechnol J, 2016, 14: 1368–1380
[35]李乐, 许红亮, 杨兴露, 李雅轩, 胡英考. 大豆 LEA基因家族全基因组鉴定、分类和表达. 中国农业科学, 2011, 44:3945–3954 Li L, Xu H L, Yang X L, Li Y X, Hu Y K, Genome-wide identification, classification and expression analysis of LEA gene family in soybean. Sci Agric Sin, 2011, 44: 3945–3954 (in Chinese with English abstract)
[36]王小非, 刘鑫, 苏玲, 孙永江, 张世忠, 郝玉金, 由春香. 番茄LBD基因家族的全基因组序列鉴定及其进化和表达分析. 中国农业科学, 2013, 46: 2501–2513 Wang X F, Liu X, Su L, Sun Y J, Zhang S Z, Hao Y J, You C X.Identification, evolution and expression analysis of the LBD gene family in tomato. Sci Agric Sin, 2013, 46: 2501–2513 (in Chinese with English abstract)
[37]许园园, 蔺经, 李晓刚, 常有宏. 梨CBL基因家族全基因组序列的鉴定及非生物胁迫下的表达分析. 中国农业科学, 2015,48: 735–747 Xu Y Y, Lin J, Li X G, Chang Y H. Identification and expression analysis under abiotic stresses of the CBL gene family in pear. Sci Agric Sin, 2015, 48: 735–747 (in Chinese with English abstract)
[38]Jian H J, Lu K, Yang B, Wang T Y, Zhang L, Zhang A X, Wang J,Liu L Z, Qu C M, Li J N. Genome-wide analysis and expression profiling of the SUC and SWEET gene families of sucrose transporters in oilseed rape (Brassica napus L.). Front Plant Sci, 2016,7: 1464–1480
[39]Yoo M J, Ma T Y, Zhu N, Liu L H, Harmon A C, Wang Q M,Chen S X. Genome-wide identification and homeolog-specific expression analysis of the SnRK2 genes in Brassica napus guard cells. Plant Mol Biol, 2016, 91: 211–227
[40]He Y J, Mao S S, Gao Y L, Wu D M, Cui Y X, Li J N, Qian W.Genome-wide identification and expression analysis of WRKY transcription factors under multiple stresses in Brassica napus.PLoS One, 2016, 11: e0157558
[41]Dun X L, Shen W H, Hu K N, Zhou Z F, Xia S Q, Wen J, Yi B,Shen J X, Ma C Z, Tu J X, Fu T D, Lagercrantz U. Neofunctionalization of duplicated Tic40 genes caused a gain-of-function variation related to male fertility in Brassica oleracea lineages.Plant Physiol, 2014, 166: 1403–1419
[42]Lysak M A, Cheung K, Kitschke M, Bureš P. Ancestral chromosomal blocks are triplicated in Brassiceae species with varying chromosome number and genome size. Plant Physiol, 2007, 145:402–410
[43]Wang X W, Wang H Z, Wang J, Sun R F, Wu J, Liu S Y, Bai Y Q,Mun J H, Bancroft I, Cheng F, Huang S W, Li X X, Hua W, Wang J Y, Wang X Y, Freeling M, Pires J C, Paterson A H, Chalhoub B,Wang B, Hayward A, Sharpe A G, Park B S, Wesshaar B, Liu B H,Li B, Liu B, Tong C B, Song C, Duran C, Peng C F, Geng C Y,Koh C, Lin C Y, Edwards D, Mu D S, Shen D, Soumpourou E, Li F, Fraser F, Conant G, Lassalle G, King G J, Bommema G, Tang H B, Wang H P, Belcram H, Zhou H L, Hirakawa H, Abe H, Guo H,Wang H, Jin H Z, Parkin I A P, Batley J, Kim J S, Just J, Li J W,Xun J H, Deng J, Kim J A, Li J P, Yu J Y, Meng J L, Wang J P,Min J M, Poulain J, Wang J, Hatakeyama K, Wu K, Wang L,Fang L, Trick M, Links M G, Zhao M X, Jin M N, Ramchiary N,Drou N, Berkman P J, Cai Q L, Huang Q F, Li R Q, Tabata S,Cheng S F, Zhang S, Zhang S J, Huang S M, Sato S, Sun S L,Kwon S J, Choi S R, Lee T H, Fan W, Zhao X, Tan X, Xu X,Wang Y, Qiu Y, Yin Y, Li Y R, Du Y C, Liao Y C, Lim Y, Narusaka Y, Wang Y P, Wang Z Y, Li Z Y, Wang Z W, Xiong Z Y,Zhang Z H. The genome of the mesopolyploid crop species Brassica rapa. Nat Genet, 2011, 43: 1035–1039
[44]Liu S Y, Liu Y M, Yang X H, Tong C B, Edwards D, Parkin I A.P., Zhao M X, Ma J X, Yu J Y, Huang S M, Wang X Y, Wang J Y,Lu K, Fang Z Y, Bancroft I, Yang T, Hu Q, Wang X F, Yue Z, Li H J, Yang L F, Wu J, Zhou Q, Wang W X, King G J, Pires J C, Lu C X, Wu Z Y, Sampath P, Wang Z, Guo H, Pan S K, Yang L M,Zhang D, Jin D C, Li W S, Belcram H, Tu J X, Guan M, Qi C K,Du D Z, Li J N, Jiang L C, Batley J, Sharpe A G, Park B, Ruperao P, Cheng F, Waminal N E, Huang Y, Dong C H, Wang L, Li J P,Hu Z Y, Zhuang M, Huang Y, Huang J Y, Shi J Q, Mei D S, Liu J,Lee T, Wang J P, Jin H Z, Li Z Y, Li X, Zhang J F, Xiao L, Zhou Y M, Liu Z S, Liu X Q, Qin R, Tang X, Liu W B, Wang Y P,Zhang Y Y, Lee J , Kim H H, Denoeud F, Xu X, Liang X M, Hua W, Wang X W, Wang J, Chalhoub B, Paterson A H. The Brassica oleracea genome reveals the asymmetrical evolution of polyploidy genomes. Nat Commun, 2014, 5: 3930
[45]Chalhoub B, Denoeud F, Liu S, Parkin I A P, Tang H B, Wang X Y, Chiquet J, Belcram H, Tong C B, Samans B, Corréa, Silva C D,Just J, Falentin C, Koh C S, Clainche I L, Bernard M, Bento P,Noel B, Labadie K, Alberti A, Charles M, Arnaud D, Guo H, Daviaud C, Alamery S, Jabbari K, Zhao M X, Edger P P, Chelaifa H,Tack D, Lassalle G, Mestiri I, Schnel N, Paslier M L, Fan G Y,Renault V, Bayer P E, Golicz A A, Manoli S, Lee T, Thi V H D,Chalabi S, Hu Q, Fan C C, Tollenaere R, Lu Y H, Battail C, Shen J X, Sidebottom C H. D, Wang X F, Canaguier A, Chauveau A,Bérard A, Deniot G, Guan M, Liu Z S, Sun F M, Lim Y P, Lyons E, Town C D, Bancroft I, Wang X W, Meng J L, Ma J X, Pires J C, King G J, Brunel D, Delourme R, Renard M, Aury J, Adams K L, Batley J, Snowdon R J, Tost J, Ewards D, Zhou Y M, Hua W,Sharpe A G, Paterson A H, Guan C Y, Wincker P. Early allopolyploid evolution in the post-Neolithic Brassica napus oilseed genome. Science, 2014, 345: 950–953
[46]Xu G X, Guo C, Shan H Y, Kong H Z. Divergence of duplicate genes in exon-intron structure. Proc Natl Acad Sci USA, 2012,109: 1187–1192
[47]Rogozin I B, Sverdlov A V, Babenko V N, Koonin E V. Analysis of evolution of exon-intron structure of eukaryotic genes. Briefings Bioinform, 2005, 6: 118–134
[48]Johansson I, Karlsson M, Johanson U, Larsson C, Kjellbom P.The role of aquaporins in cellular and whole plant water balance.Biochim Biophys Acta, 2000, 1465: 324–342
[49]Volkert K, Debast S, Voll L M, Voll H, Schießl I, Hofmann J,Schneider S, Börnke F. Loss of the two major leaf isoforms of sucrose-phosphate synthase in Arabidopsis thaliana limits sucrose synthesis and nocturnal starch degradation but does not alter carbon partitioning during photosynthesis. J Exp Bot, 2014,65: 5217–5229
[50]Hirose T, Hashida Y, Aoki N, Okamura M, Yonekura M, Ohto C,Terao T, Ohsugi R. Analysis of gene-disruption mutants of a sucrose phosphate synthase gene in rise, OsSPS1, shows the importance of sucrose synthesis in pollen germination. Plant Sci,2014, 225: 102–106
[51]Chen S, Hajirezaei M, Börnke F. Differential expression of sucrose-phosphate synthase isoenzymes in tobacco reflects their functional specialization during dark-governed starch mobilization in source leaves. Plant Physiol, 2005, 139: 1163–1174
[52]Lu K, Xiao Z C, Jian H J, Peng L, Qu C M, Fu M L, He B, Tie L M, Liang Y, Xu X F, Li J N. A combination of genome-wide association and transcriptome analysis reveals candidate genes controlling harvest index-related traits in Brassica napus. Sci Rep,2016, 6: 36452
