基于玛氏骨条藻转录组的脂肪酸合成途径分析❋
2018-02-28米铁柱王华龙
张 梅, 米铁柱❋❋, 甄 毓, 王华龙
(1. 中国海洋大学环境科学与工程学院,山东 青岛 266100; 2. 海洋环境与生态教育部重点实验室,山东 青岛 266100;3. 青岛海洋科学与技术国家实验室海洋生态与环境科学功能实验室,山东 青岛 266071;4.中国海洋大学海洋生命学院,山东 青岛 266003 )
化石燃料的大量燃烧是导致全球变暖的主要原因,生物燃料是可替代化石燃料的可再生能源,脂肪族生物燃料由于其热值高、性能好而受到广泛重视。其中,微藻脂肪酸的高生产量使其成为生物燃料研究的热点之一[1]。近年来随着微藻脂肪酸生物功能研究的不断深入,科学界发现微藻生产油脂具有其他生物无法替代的优点:微藻是光合效率最高的原始植物之一,周期短,生产量大;很多微藻可以在海水中生长,节省了淡水资源和土地资源;微藻的油脂含量较高,一般可以达到藻体干重的20%,最高甚至可达60%[2-3]。尽管产油微藻具有代替化石燃料的潜力[4],其商业化应用仍存在巨大的经济挑战,需要从分子生物学和基因工程等方面加强对微藻脂肪酸生物合成的研究,以达到降低生产成本的目的。因此,研究微藻的脂肪酸合成途径不仅对其生理过程有重要意义,而且还有较为广泛的应用前景。
从生态学角度来看,微藻是海洋初级生产力的主要贡献者,其生产力的高低直接影响着整个海洋生态系统[5]。从分子水平解释微藻的生理功能、合成和代谢途径,对研究微藻的生长、发育和繁殖具有重要的意义。在实际应用方面,转录组学研究有助于解释赤潮发生、发展以及消亡的内在分子机制,能够为赤潮的预防和治理提供有效的理论依据。
骨条藻(Skeletonemaspp.)是一类广温广盐型海洋微型浮游硅藻,有时会引发赤潮[6]。由于骨条藻种类之间的形态学差异非常细微,鉴定难度大,长期以来一直以中肋骨条藻(S.costatum)作为该属的模式种和代表进行研究,在涉及到骨条藻的生理生化研究中,并未对研究对象进行严格的区分鉴定,一般都被当作是中肋骨条藻。玛氏骨条藻(S.marinoi)是骨条藻属5种今生种之一,于2005年由Sarno等人分离确认[7]。S.marinoi分布广泛,在我国的长江口及香港海域均有发现,其从3月份开始进入快速繁殖期,是长江口赤潮高发区的优势种。本文以S.marinoi为研究对象,确定了其脂肪酸生物合成途径中的相关基因,并在此基础上分析了S.marinoi的脂肪酸生物合成途径及其在不同生长时期的基因表达差异,以期为阐明微藻脂肪酸生物合成的内在机制提供理论基础。
1 材料与方法
1.1 S. marinoi藻种的培养
玛氏骨条藻(Skeletonemamarinoi)自青岛近海海域分离得到,保存于中国海洋大学海洋环境与生态教育部重点实验室藻种室,培养基为常规f/2培养基,培养温度为(20±1)℃,培养过程光暗比控制为12 h/12 h,光照强度为80~100 μmol·m-2·s-1。实验所用海水取自青岛近海,盐度为33,pH为7.8~8.0。
1.2 测序样品准备
将分离得到的S.marinoi在实验室进行培养,基于S.marinoi的生长趋势,收集指数期(Exponential Phase, EP)、稳定期(Stationary Phase, SP)以及衰亡期(Decline Phase, DP)的藻细胞。对不同生长时期的S.marinoi分别进行离心收集,将离心收集到的藻细胞快速放入液氮中速冻保存。每个样品设置3个平行样进行生物学重复,样品由北京诺禾致源生物信息科技有限公司进行总RNA的提取及Illumina Hiseq2000双端测序。
1.3数据预处理及从头拼接
将获得的核苷酸原始序列(Raw Reads)进行可信度分析和质量评估,去除核苷酸序列中带接头的、低质量序列得到有效序列(Clean Reads)。用拼接软件Trinity[8](版本:r2012-10-05)将有效序列进行从头拼接,得到的转录本序列信息的储存方式为FASTA格式,以此形成一个转录组,作为分析的参考序列。
1.4 功能注释及代谢途径分析
功能注释和代谢途径分析使用的数据库有7个,分别为Nr、Nt、Pfam、KOG、Swiss-Prot、KEGG以及GO数据库。利用NCBI blast 2.2.28+[9]对拼接形成的转录本与Nr、Nt、Swiss-Prot以及KOG数据库进行比对,其中,Nr、Nt和Swiss-Pro数据库比对evalue阈值为1e-5,对于每条unigene展示top10比对结果(如果符合条件),KOG比对evalue阈值为1e-3。通过HMMER 3.0程序,搜索已建好的蛋白结构域的HMM模型,对Pfam蛋白结构域进行预测。GO功能注释利用Blast2GO v2.5[10]和自写脚本来完成。利用KAAS服务器进行KEGG代谢途径分析,KAAS对转录本序列进行逐一注释,对成功注释的转录本分配到相应的EC编号和其所参与的代谢途径[11-12]。最后,将Trinity拼接得到的转录组作为参考序列(ref),利用RSEM[13]软件将每个样品上的clean reads往ref上做mapping,对bowtie的比对结果进行统计,得到每个样品比对到每一个基因上的read count数目。
2 结果与讨论
2.1 转录组测序信息及拼接
通过Illumina Hiseq2000测序平台进行双端测序,总共获得60 936 371条原始数据,经过测序数据过滤后得到的有效序列为58 592 306条,占原始序列的96.15%。利用从头拼接得到的转录本数为39 058个,非重复序列基因(unigene)为20 139个,长度范围为200 ~ 20 000 bp。
2.2 功能注释及代谢分析
通过与上述Nr、Nt等7个数据库进行比对,发现注释成功的unigene个数为14 693个,占总unigene数的71.3%。其中,在Nr数据库中注释的成功率最高,为65.25%,在Nt数据库中的注释率最低,仅为8.52%。GO 和Pfam数据库注释的成功率较高,分别为53.28%和51.44%,而Swiss-Prot、KOG和KO数据库注释的成功率较低,分别为33.46%、20.07%和16.73%。
对拼接得到的所有转录本进行GO注释,将注释成功的转录本按照GO三个大类(生物学过程、细胞成分、分子功能)的下一层级进行分类,划分为46个类别,包括核糖体结构组分、大分子生物合成、碳利用等。与KOG数据库进行比对,将注释成功的转录本按照功能分成26个子家族,包括RNA加工与修饰、能量产生与转化、核染色质结构与变化等。对转录本进行KO注释后,将它们参与的KEGG代谢通路进行分类,得到了31个类群,可以匹配到EC编号的转录本为3 816个,并注释为225条不同的代谢途径,包括氮代谢途径、脂肪酸生物合成途径以及氨基酸代谢途径等。
2.3 脂肪酸合成途径分析
基于S.marinoi转录组的GO功能注释和KEGG代谢途径分析,本文已经对S.marinoi的氮代谢途径[14]、碳固定途径[15]以及硫代谢途径(未发表数据)进行了分析。为了全面构建S.marinoi的代谢网络,计划对脂肪酸、氨基酸以及淀粉的生物合成及降解等多个代谢途径进行解析。在本研究中,主要对S.marinoi的脂肪酸生物合成途径进行了解析。
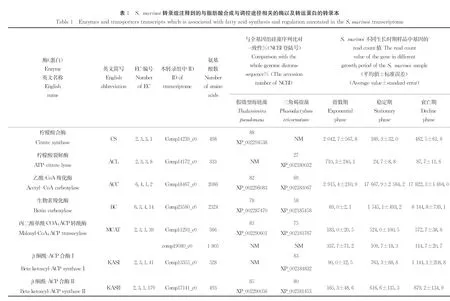
通过分析,发现了与脂肪酸生物合成途径相关的26种酶以及33个编码基因的转录本。由于微型海链藻(Thalassiosirapseudonana)和三角褐指藻(Phaeodactylumtricornutum)已进行了全基因组测序,基因注释较为完整,且与S.marinoi同属硅藻,为了分析3 种硅藻脂肪酸合成相关编码基因的同源性,利用BLASTx比较之后发现:S.marinoi与同属中心纲的假微型海链藻的基因序列一致性相对较高,而与羽纹纲的三角褐指藻的序列一致性相对较低(见表1)。
结合KEGG的比对结果构建了S.marinoi的脂肪酸生物合成途径,脂肪酸生物合成包括三个方面:脂肪酸的从头合成(见图1)、脂肪酸碳链的延长以及不饱和脂肪酸的生成。脂肪酸的从头合成起始于线粒体中乙酰-CoA的生成,其中,糖酵解过程是产生乙酰-CoA的主要方式,β-氧化和氨基酸代谢也产生乙酰-CoA。真核藻类的脂肪酸从头合成是在叶绿体中进行的,由于乙酰-CoA是在线粒体中产生,而线粒体内膜不允许乙酰-CoA穿透,乙酰-CoA从线粒体到叶绿体中的输送需要以柠檬酸为载体来完成。在线粒体中,乙酰-CoA和草酰乙酸在柠檬酸合酶(citrate synthase, CS)的作用下生成柠檬酸,随后,柠檬酸跨越线粒体内膜进入到细胞溶液中与柠檬酸裂解酶(ATP-citrate lyase, ACL)作用生成乙酰-CoA和草酰乙酸,之后乙酰-CoA进入到叶绿体中进行脂肪酸的从头合成。

S.marinoi脂肪酸碳链的延长分为两个阶段,分别在叶绿体和内质网上进行。第一阶段是发生在叶绿体中的脂肪酸从头合成(见图1),合成过程中以不断延长的酰基-ACP为底物,直至C16或C18脂肪酸的生成。第二阶段是内质网中的脂酰-CoA延长酶催化C18脂肪酸延长并产生更长碳链的脂肪酸(见图2),该合成过程中以酰基-CoA为底物,以丙二酸单酰-CoA作为碳链延伸的供体,其延长机制与脂肪酸的从头合成相似。脂酰-CoA延长酶属于膜绑定的多酶复合体,由β-酮酰-CoA合酶(beta-ketoacyl-CoA synthase, KCS)、β-酮酰-CoA还原酶(beta-ketoacyl-CoA reductase, KCR)、β-羟酰-CoA脱水酶(beta-hydroxyacyl-CoA dehydratase, HCD)以及烯酰- CoA还原酶(enoyl-CoA reductase, ECR)组成,其相继催化缩合、还原、脱水、还原反应,每一循环使碳链增加2个碳子长度。
S.marinoi单不饱和脂肪酸的合成主要发生在叶绿体中,其主要形式为叶绿体中的C16:0-ACP或C18:0-ACP在ADD的作用下生成单不饱和脂肪酸C16:1(9)或C18:1(9)。S.marinoi多不饱和脂肪酸的合成是通过原核途径和真核途径来配合完成的,原核途径发生在叶绿体中,真核途径发生在内质网上。真核途径与原核途径的主要区别是真核途径可以在脂酰-CoA延长酶的作用下使脂酰基的碳链延伸。植物中的去饱和酶不能直接作用于游离脂肪酸,但可作用于甘油脂质。甘油-3-磷酸与酰基-CoA或酰基-ACP经过两次酰化反应生成甘油脂质。合成甘油脂质后,脂肪酸仍可进一步加工。基于转录组的信息,推测S.marinoi多不饱和脂肪酸形成的途径主要有两种:一是在叶绿体中形成的C18:1-ACP被转移到sn-1或sn-2位形成磷酸酰甘油,其与半乳糖残基作用生成单或二半乳糖二酰基甘油,进一步在Δ12-去饱和酶(delta-12 fatty acid desaturase, Δ12D)的作用下生成亚油酸,亚油酸在Δ15-去饱和酶(delta-15 fatty acid desaturase, Δ15D)的作用下生成α-亚麻酸;二是发生在内质网上的多不饱和脂肪酸的生成,在叶绿体中生成的C18:0或C18:1(9)游离脂肪酸自由扩散到叶绿体外膜上,被长链酰基-CoA合成酶(long-chain acyl-CoA synthetase, LACS)催化为C18:0-CoA或C18:1-CoA,进一步被催化形成磷酸脂,内质网定位的延长酶和去饱和酶可分别催化磷脂酸的脂酰基进行碳链延伸和去饱和,其合成途径主要为ω3和ω6途径。除了脂酰-CoA延长酶外,还有Δ4D、Δ5D、Δ6D、Δ9D、Δ12D以及Δ15D参与多不饱和脂肪酸的真核合成途径。多不饱和脂肪酸具体的合成途径如图3所示。




(酶由长方形标出,代谢通路由椭圆标出,图中X6和X7表示循环次数。Enzymes or protein are shown in solid boxes and metabolism pathways are shown in cycles. In the Figure, X6 and X7 is the cycle times.)
图1 基于S.marinoi转录组数据的从头拼接及注释结果构建的脂肪酸从头合成途径
Fig.1 Denovo fatty acid synthesis pathway reconstructed based on the de novo assembly and annotation ofS.marinoitranscriptome

(酶由长方形标出。Enzymes or protein are shown in solid boxes.)
图2 基于S.marinoi转录组数据的从头拼接及注释结果构建的脂肪酸碳链延长途径
Fig.2 Elongation fatty acid synthesis pathway reconstructed based on the de novo assembly and annotation ofS.marinoitranscriptome
2.4 脂肪酸合成途径中基因差异表达的分析
差异基因表达的输入数据为基因表达水平分析中得到的read count数据,采用DESeq对其进行分析[16-18],筛选阈值为padj<0.05[19],该分析方法基于的模型是负二项分布,第i个基因在第j个样本中的read count为Kij,则有
以第i个基因的指数期和稳定期的差异表达计算为例,A为指数期j个样本中第i个基因read count的平均值,B为稳定期j个样本中第i个基因read count的平均值。设定f=Log2(B/A),若f的绝对值大于2则视为差异显著。正值视为上调表达,负值视为下调表达。
通过分析S.marinoi脂肪酸生物合成途径中相关基因的表达情况发现:在稳定期和衰亡期,该通路中相关酶基因的表达量均无显著差异,而与指数期相比有着较显著的差异。在与脂肪酸合成途径相关的26种酶对应的33个基因中,存在差异表达的基因有15个。图4中列出了这些差异表达基因在以指数期的表达水平为基准的情况下的差异表达倍数。
CS和ACL是植物脂肪酸合成途径中的关键调控酶。童晋[20]通过基因工程的方法在油菜中过量表达CS基因发现该酶表达量的上升会造成脂肪酸合成量的增加,反义表达则会造成脂肪酸合成量的降低。ACL催化柠檬酸与辅酶A产生乙酰-CoA和草酰乙酸,这是细胞质中产生乙酰-CoA的唯一途径,除此之外再无其他途径可补充植物生长发育所需的乙酰-CoA[21]。在拟南芥中,反义表达ACL基因发现造成植物矮化、果荚缩短、叶片变小等现象,同时也造成了植物体内脂肪酸合成量的降低[20]。如图4a,b所示,在S.marinoi脂肪酸合成途径中,CS的编码基因在稳定期出现了显著的下调,而ACL的编码基因在稳定期和衰亡期均出现了显著的下调,这表明在S.marinoi生长的指数期,由这两种酶参与的代谢活动可能更为旺盛。CS和ACL作为脂肪酸合成代谢调控途径中的关键酶,影响着细胞内乙酰-CoA的含量,而乙酰-CoA是脂肪酸合成的主要原料,其含量的变化会直接影响脂肪酸的合成。

(酶由长方形标出。Enzymes or protein are shown in solid boxes.)
图3 基于S.marinoi转录组数据的从头拼接及注释结果构建的多不饱和脂肪酸生成途径
Fig.3 Polyunsaturated fatty acid synthesis pathway reconstructed based on thedenovoassembly and annotation ofS.marinoitranscriptome
ACC严密控制着脂肪酸从头合成的速度,是脂肪酸合成过程中的关键限速酶[22]。ACC催化生成的一部分丙二酸单酰-CoA存在于质体中用于脂肪酸的从头合成;另一部分存在于细胞溶胶中用于脂肪酸链的延伸及其他次生代谢产物的合成[23]。Roesler等[24]利用基因工程的方法将拟南芥的乙酰-CoA羧化酶基因导入到甘蓝型油菜中发现,转基因油菜第一代种子中ACC的活性提高了1.7~1.9倍,种子含油量提高了5%左右。Roessler[25]和Dunahay[26]等利用克隆的方法将硅藻的ACC基因经修饰后转化到硅藻自身中后发现,硅藻ACC的活性提高了2~3倍,脂质的含量小幅度增加。如图4c所示,ACC的编码基因在稳定期和衰亡期出现显著上调,S.marinoi生长进入稳定期后,细胞生长缓慢,碳代谢流主要转向脂肪酸的生物合成,细胞开始进行油脂积累。作为脂肪酸合成途径中的限速酶,ACC编码基因的上调表达促使更多的乙酰-CoA转化为丙二酸单酰-CoA而加速脂肪酸生物合成途径,从而为脂肪酸的合成提供一定的物质保证。本文在S.marinoi转录组中还发现了生物素羧化酶(Biotin Carboxylase, BC)的编码基因,BC是ACC组成的亚基单元之一,BC的编码基因在不同生长时期的差异表达情况与ACC的编码基因基本一致,具体如图4d所示。
MCAT是肪酸生物合成途径中的关键酶,MCAT的活性与脂肪酸含量的高低密切相关。Cheng等[27]将Schizochytriumsp. TIO1101的MCAT基因在酿酒酵母中过量表达,发现其生物量和脂肪酸总量分别提高了16.8%和62%。研究者利用基因技术在拟南芥、油菜等植物中过量表达MCAT基因,发现对C16和C18类脂肪酸的积累有很好的促进作用[28-30]。如图4e所示,MCAT的编码基因在稳定期和衰亡期均出现显著上调,间接说明S.marinoi在稳定期和衰亡期的脂肪酸合成能力可能要高于指数期。
脂肪酸从头合成过程中的另一个关键酶是脂肪酸合成酶(FAS),它催化脂肪酸碳链的延长。植物FAS为原核形式的多酶复合体(FASII),由ACP、KAS(KASI, KASII, KASIII)、KAR、HAD、EAR和OAT构成[31]。
在FAS中,KASIII是当前脂肪酸生物合成积累研究的主要热点。研究者通过过量表达KASIII基因来调控脂肪酸的生物合成,Dehesh等[32]将菠菜的KASIII基因在烟草、拟南芥、油菜中三种植物中超量表达后发现,植物体内C16:0脂肪酸的含量增加但脂肪酸合成速率降低。Verwoert等[33]将E.coli的KASIII基因在油菜籽中超量表达后也得到了类似的结果:脂肪酸的组成虽然发生了变化,但脂肪酸的含量没有提高且细胞生长严重受阻。上述现象的产生可能与植物FAS是多酶复合体有关,每个亚基的表达活性与其他亚基相互依存,相互制约[34]。在脂肪酸的生物合成中FAS基因的调控机制十分复杂,但可以明确的是FAS活性提高,有助于促进脂肪酸的生物合成。目前,在国内外的研究中鲜有发现微藻FAS超量表达的相关报告。
较指数期相比,FAS中3种酶的5个编码基因在稳定期和衰亡期均出现显著上调,分别为KASI、KASII、KASIII、KAR以及EARI的编码基因,差异表达情况具体如图4f,j所示。S.marinoi在进入稳定期后,KASI等5个编码基因的显著上调为细胞的油脂积累提供了有利的物质条件,同时,KASI等编码基因表达的变化体现了S.marinoi能量固定以及储能代谢途径的变化,这与藻类在不同生长阶段的分子响应有着密切的关系。

(各柱状图横坐标表示不同生长时期:EP,指数期;SP,稳定期;DP,衰亡期。纵坐标表示在以指数期的表达水平为基准的情况下的f数值。In each bar chart, the horizontal ordinate represents different growth stages: EP, exponential phase; SP, stationary phase; DP, decline phase. The numbers of vertucal ordinate are thefvalue as a benchmark of EP.)
图4S.marinoi脂肪酸生物合成途径中差异表达的基因
Fig.4 Different gene expression involving in fatty acid synthesis
在脂肪酸的碳链延长途径中,发现有3种酶的4个编码基因的表达在S.marinoi的不同生长时期表现出显著差异,其中,HCD、ECR和LACS(Comp17540_c0)的编码基因在稳定期和衰亡期均出现显著上调,而LACS(Comp14198_c0)的编码基因在稳定和衰亡期则出现显著下调。HCD等3个编码基因的上调意味着S.marinoi在进入稳定期后可能生成了更多的长链脂肪酸,以满足细胞正常生命活动的需求,而Comp14198_c0编码基因显著下调意味着一部分编码基因表达量的降低,可能会造成相关代谢的减缓。
在脂肪酸的去饱和途径中,发现Δ12D的编码基因在稳定期和衰亡期均出现显著上调(见图4o),Δ12D能够催化油酸形成亚油酸。Δ12D编码基因的表达量的提高有助于亚油酸的生物合成,而亚油酸是多不饱和脂肪酸合成的重要前体物质,Δ12D编码基因的上调表达为藻类多不饱和脂肪酸的合成提供了一定的物质保证。
通过对S.marinoi脂肪酸生物合成相关编码基因在不同生长时期表达量的分析,初步推测在稳定期S.marinoi的脂肪酸的生物合成更为旺盛,造成上述结果的原因可能与碳代谢流的竞争有关。在指数期,培养基中N、P等营养元素较为充足,在营养盐充足的情况下,微藻细胞生长繁殖快速,致使主要的碳代谢流向了细胞生长;而当微藻的生长进入稳定期后,培养基中的营养元素逐渐被耗尽,使微藻的生长处于营养限制的条件下,致使细胞的生长繁殖变缓,促使主要的碳代谢流转向细胞内物质的积累,例如,部分碳代谢流以乙酰-CoA的形式转向脂肪酸的生物合成。在S.marinoi的脂肪酸生物合成途径中,发现以ACC编码基因为代表的12个编码基因在稳定期和衰亡期均出现了显著上调,而CS、ACL和LACS(Comp14198_c0)的编码基因在不同生长时期出现了显著下调。在后续的研究中,将着重加强对这15个编码基因的研究,计划利用荧光定量检测的方法来验证转录组的测序结果,从而分析S.marinoi的脂肪酸生物合成在转录水平的调控途径。通过监测S.marinoi脂肪酸生物合成途径中相关酶的基因表达情况,有助于阐述脂肪酸生物合成的内在分子机制,为微藻的脂肪酸生物合成机制提供一定的理论依据。
研究发现,S.marinoi是中国海域的广布种,同时,也是一种常见的赤潮引发种。在对赤潮的研究中,可以将S.marinoi作为典型,对其的生理生化机理进行研究,从而阐明该藻引发赤潮的发生、发展以及消亡的内在分子机制,为赤潮的预警和有效防治提供一定的理论基础。利用转录组数据对S.marinoi的代谢网络进行构建,不但有助于从分子水平了解微藻的生理代谢机制,而且对赤潮的预警和防治提供了新的思路。
3 结语
本研究中对不同生长时期的S.marinoi进行了转录组测序,通过分析发现存在26个与脂肪酸合成相关的酶及33个对应的编码基因,经过序列对比分析发现,这些编码基因与假微型海链藻有较高的序列一致性,而与三角褐指藻的序列一致性较低。通过比较S.marinoi不同生长时期的转录组以及KEGG差异的分析结果,确定了脂肪酸合成途径中基因的表达差异,发现在该代谢通路中存在显著差异表达的基因有15个。与指数期相比, ACL和LACS(Comp14198_c0)的编码基因在稳定期和衰亡期出现显著下调表达,而CS的编码基因仅在稳定期出现显著下调表达。与之相反的是,以ACC编码基因为代表的12个编码基因在稳定期和衰亡期均出现了显著上调。基于上述研究结果,可以初步推测出S.marinoi在稳定期的脂肪酸合成能力高于指数期。这些信息为海洋微藻脂肪酸生物合成途径的研究提供了理论基础,有助于对S.marinoi进行基因改造,提高脂肪酸的合成量。同时,又能为赤潮的防治提供新的思路。
[1] Williams P J L B, Laurens L M L. Microalgae as biodiesel & biomass feedstocks: Review & analysis of the biochemistry, energetics & economics[J]. Energy & Environmental Science, 2010, 3(5): 554-590.
[2] Gouveia L, Oliveira A C. Microalgae as a raw material for biofuels production[J]. Journal of Industrial Microbiology & Biotechnology, 2009, 36(2): 269-74.
[3] Das P, Obbard J P. Incremental energy supply for microalgae culture in a photobioreactor[J]. Bioresource Technology, 2011, 102(3): 2973-8.
[4] Chisti Y. Biodiesel from microalgae[J]. Biotechnol Adv, 2007, 25(3): 294-306.
[5] 高亚辉. 海洋微藻分类生态及生物活性物质研究[J]. 厦门大学学报(自然科学版), 2001, 40(2): 566-573. Gao Y H. Studies on taxonomy, ecology and bioactive products of marine microalgae[J]. Journal of Xiamen University (Natural Science) , 2001, 40(2): 566-573.
[6] 程金凤, 高亚辉, 梁君荣, 等. 骨条藻的种类与基因多样性研究进展[J]. 自然科学进展, 2007, 17(5): 586-594. Cheng J X, Gao Y H, Liang J R, et al. Progress ofSkeletonemaspecies and genetic diversity[J]. Progress in Natural Science, 2007, 17(5): 586-594.
[7] Diana Sarno, Kooistra W H C F, Medlin L K, et al. Diversity in the genus skeletonema, (bacillariophyceae). Ii. An assessment of the taxonomy of s. Costatum -like species with the description of four new species 1[J]. Journal of Phycology, 2005, 41(1): 151-176.
[8] Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652.
[9] Camacho C, Coulouris G, Avagyan V, et al. BLAST+: architecture and applications[J]. BMC Bioinformatics, 2009, 10(1): 1-9.
[10] Götz S, Garcíagómez J M, Terol J, et al. High-throughput functional annotation and data mining with the Blast2GO suite[J]. Nucleic Acids Research, 2008, 36(10): 3420-3435.
[11] Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30.
[12] Moriya Y, Itoh M, Okuda S, et al. KAAS: an automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research, 2007, 35(suppl 2): 182-185.
[13] Li B, Dewey C N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1): 93-99.
[14] 荆晓丽, 米铁柱, 甄毓, 等. 基于玛氏骨条藻(Skeletonemamarinoi)转录组的氮代谢途径解析[J]. 海洋环境科学, 2016, 35(5): 703-711. Jing X L, Mi T Z, Zhen Y, et al. Description of nitrogen metabolism pathway based onSkeletonemamarinoitranscriptome[J]. Marine Environmental Science , 2016, 35(5): 703-711.
[15] 刘乾, 米铁柱, 甄毓, 等. 基于玛氏骨条藻(Skeletonemamarinoi)转录组的碳固定代谢途径分析[J]. 科学通报, 2016(22): 2483-2493. Liu Q, Mi T Z, Zhen Y, et al. Description of carbon fixation pathway based onSkeletonemamarinoitranscriptome[J]. Chinese Science Bulletin, 2016(22): 2483-2493.
[16] Anders S, Huber W. Differential expression analysis for sequence count data[J]. Genome Biol, 2010, 11(10): R106.
[17] Trapnell C, Williams B A, Pertea G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5): 511-515.
[18] Dillies M A, Rau A, Aubert J, et al. A comprehensive evaluation of normalization methods for Illumina high-throughput RNA sequencing data analysis[J]. Briefings in Bioinformatics, 2013, 14(6): 671-683.
[19] Storey J D, Tibshirani R. Statistical significance for genomewide studies[J]. Proceedings of the National Academy of Sciences, 2003, 100(16): 9440-9445.
[20] 童晋. 油菜柠檬酸合酶与柠檬酸裂解酶基因克隆及功能研究[D]. 北京: 中国农业科学院, 2009. Tong J. Cloning and Functional Research of Citrate Synthase and ATP-Citrate Lyase of Brassica Napus[D]. Beijing: Chinese Academy of Agricultural Sciences, 2009.
[21] Fatland B L, Nikolau B J, Wurtele E S. Reverse genetic characterization of cytosolic acetyl-CoA generation by ATP-citrate lyase in Arabidopsis[J]. Plant Cell, 2005, 17(1): 182-203.
[22] Ohlrogge J B, Jaworski J G. Regulation of fatty acid synthesis[J]. Plant Biology, 1997, 48(48): 109-136.
[23] Nikolau B J, Ohlrogge J B, Wurtele E S. Plant biotin-containing carboxylases [J]. Archives of Biochemistry & Biophysics, 2003, 414(2): 211-22.
[24] Roesler K, Shintani D, Savage L, et al. Targeting of the Arabidopsis homomeric acetyl-coenzyme A carboxylase to plastids of rapeseeds[J]. Plant Physiology, 1997, 113(1): 75-81.
[25] Roessler P G, Ohlrogge J B. Cloning and characterization of the gene that encodes acetyl-coenzyme A carboxylase in the alga Cyclotella cryptica[J]. Journal of Biological Chemistry, 1993, 268(26): 19254-19259.
[26] Dunahay T G, Jarvis E E, Roessler P G. Genetic transformation of the diatoms cyclotella cryptica, and navicula saprophila[J]. Journal of Phycology, 1995, 31(6): 1004-1012.
[27] Cheng R, Ge Y, Yang B, et al. Cloning and functional analysis of putative malonyl-CoA: acyl-carrier protein transacylase gene from the docosahexaenoic acid-producer Schizochytrium sp. TIO1101[J]. World Journal of Microbiology and Biotechnology, 2013, 29(6): 959-967.
[28] Eccleston V S, Ohlrogge J B. Expression of lauroyl-acyl carrier protein thioesterase in Brassica napus seeds induces pathways for both fatty acid oxidation and biosynthesis and implies a set point for triacylglycerol accumulation[J]. Plant Cell, 1998, 10(10): 613-22.
[29] Bonaventure G, Ohlrogge J B. Differential regulation of mRNA levels of acyl carrier protein isoforms in Arabidopsis[J]. Plant Physiology, 2002, 128(1): 223-35.
[30] Jha J K, Sinha S, Maiti M K, et al. Functional expression of an acyl carrier protein (ACP) from Azospirillum brasilense alters fatty acid profiles in Escherichia coli and Brassica juncea[J]. Plant Physiology & Biochemistry Ppb, 2007, 45(6-7): 490-500.
[31] 卢善发. 植物脂肪酸的生物合成与基因工程[J]. 植物学报, 2000, 17(6): 481-491. Lu S F. Fatty acid biosynthesis and genetic engineering of plants[J]. Bulletin of Botany, 2000, 17(6): 481-491.
[32] Dehesh K, Tai H, Edwards P, et al. Overexpression of 3-ketoacyl-acyl-carrier protein synthase IIIs in plants reduces the rate of lipid synthesis[J]. Plant Physiology, 2001, 125(2): 1103-14.
[33] Verwoert I I G S, Linden K H V D, Walsh M C, et al. Modification of Brassica napus, seed oil by expression of the Escherichia coli fabH, gene, encoding 3-ketoacyl-acyl carrier protein synthase III[J]. Plant Molecular Biology, 1995, 27(5): 875-86.
[34] 朱顺妮, 王忠铭, 尚常花, 等. 微藻脂肪合成与代谢调控[J]. 化学进展, 2011, 23(10): 2169-2176. Zhu S N, Wang Z M, Shang C H, et al. Lipid biosynthesis and metabolic regulation in microalgae[J]. Progress in Chemistry, 2011, 23(10): 2169-2176.
