转基因大豆MON89788双重数字PCR通用定量检测方法的建立
2018-02-09冼钰茵吴希阳高东微
刘 津,李 婷,冼钰茵,吴希阳,凌 莉,高东微,*
(1.广东检验检疫技术中心,广东省动植物与食品进出口技术措施研究重点实验室,广东 广州 510623;2.暨南大学理工学院,广东 广州 510632)
全球转基因作物种植已逾20 年,种植面积从1996年的170万 hm2增加了100 倍,达到了2015年的1.797亿 hm2,20 年来累计种植面积达到空前的20亿 hm2,相当于中国大陆总面积的2 倍[1]。随着转基因作物在全球范围内的大规模种植和消费,越来越多的国家和地区开始实施转基因产品标识管理制度。鉴于转基因作物在种植收割、生产加工和运输消费等过程中会出现不可避免的混杂,目前有60多个国家和地区对转基因产品标识设置了阈值,如欧盟规定合法转基因成分标识阈值为0.9%、非法转基因成分标识阈值为0.5%,巴西、澳大利亚、新西兰、沙特阿拉伯、以色列设定的标识阈值为1%,韩国设定的标识阈值为3%,日本和俄罗斯设定的标识阈值为5%[2]。这些国家和地区规定,产品中转基因成分含量低于阈值可以免于标识。转基因标识阈值逐渐成为大势所趋,我国相关部门也正在研究将其引入转基因安全管理体系当中。标识阈值的采用,为转基因安全管理带来更加科学合理的技术措施,同时也对转基因成分的精准定量提出了迫切而具体的需求。
在数字聚合酶链式反应(digital polymerase chain reaction,dPCR)技术出现以前,转基因成分的定量检测基本采用实时荧光定量PCR(real-time fluorescence quantitative PCR,qPCR)技术[3-5],通过绘制不同浓度梯度标准物质与循环阈值之间的线性标准曲线,基于标准物质和待测样品的DNA提取与PCR扩增效率一致的假设,用待测样品qPCR的循环阈值对照标准曲线计算得到其中转基因成分的含量。这种定量检测技术在实际应用中局限性极大:梯度标准物质不易获得,标准物质与待测样品存在天然的本质差异,实验步骤和计算过程繁琐导致测量误差在多个环节累积放大,加之痕量转基因成分的测定结果重复性差等,因此定量结果相对粗略,不能达到阈值管理所需要的痕量、精准的定量技术水平。
dPCR基于单分子目标基因PCR扩增,PCR反应液被分配到大量独立的反应室中进行单拷贝核酸独立荧光PCR扩增,根据泊松分布原理进行矫正并计算出初始样品中目标核酸的精确拷贝数浓度[6-8]。dPCR主要有微滴式数字PCR(droplet digital PCR,ddPCR)和芯片式数字PCR(chip digital PCR,cdPCR)[9],目前这两种dPCR在研究和应用领域可谓平分秋色。由于无需依赖梯度浓度标准物质绘制标准曲线即可对目标核酸进行准确的拷贝数定量,利用外源基因与内源基因拷贝数比例即可计算出转基因成分相对含量,避免了qPCR技术在实际应用中的各种局限,因此dPCR技术已被数次报道用于转基因成分定量检测的研究中[10-16]。
MON89788品系是美国孟山都公司研发的抗草甘膦除草剂转基因大豆,最早于2007年被美国、菲律宾和加拿大批准种植,允许作为食品或饲料直接食用或加工。2008年8月28日MON89788品系首次获得我国农业部颁发的进口安全证书,仅可用于原料加工[17]。目前,除我国以外,澳大利亚、哥伦比亚、欧盟、新西兰等22 个国家或地区批准该转基因大豆品系用于食品和饲料中。目前转基因大豆MON89788品系的官方检测方法包括农业部1485号公告—6—2010《转基因植物及其产品成分检测 耐除草剂大豆MON89788及其衍生品种定性PCR方法》[18]和欧盟官方方法CRLVL05/06VR《大豆MON89788品系特异性定量实时PCR检测方法》[3],农业部标准方法采用普通PCR进行定性检测,欧盟官方方法采用实时荧光PCR进行相对定量检测。国内外鲜有采用dPCR进行精准定量检测的报道。尽快建立转基因大豆MON89788品系的dPCR定量检测方法,使之通用于主流的ddPCR和cdPCR平台,对构建我国转基因定量检测技术体系,为日后阈值管理做好技术储备,具有重要的实用价值和示范作用。
1 材料与方法
1.1 材料与试剂
本实验以转基因大豆MON89788品系为研究对象,25 份其他种类转基因标准物质和4 份本实验室留存的非转基因植物材料为特异性实验材料,1 份来自食品分析能力评价体系(food analysis performance assessment scheme,FAPAS)国际能力验证项目的动物饲料样品为盲样检测实验材料,具体情况见表1。
ddPCR:ddPCRTM预混液、微滴生成油、微滴分析油、微滴生成卡槽、微滴生成卡槽胶垫和96 孔板,购自美国Bio-Rad公司;cdPCR:QuantStudio®预混液(3D Digital PCR Master Mix v2)、芯片试剂盒(3D Digital PCR 20K Chip Kit v2,包含芯片、芯片盖、刷头、封油注射器),购自美国Applied Biosystems by Life Technologies公司;TIANGEN®植物基因组DNA提取试剂盒(CAT:#DP30502)购自天根生化科技(北京)有限公司;引物和探针均由上海闪晶分子生物科技有限公司合成。
1.2 仪器与设备
QX200TMDroplet Digital PCR系统(包括C1000 TouchTM热循环仪、微滴生成仪、微滴分析仪和封膜仪4 个部分) 美国Bio-Rad公司;QuantStudioTM3D Digital PCR系统(包括Dual Flat Block GeneAmp® PCR System 9700、芯片装载仪和芯片分析仪3 个部分)美国Applied Biosystems by Life Technologies公司;Nanodrop 1000核酸蛋白分析仪 美国Thermo Scientific公司;Rainin®E4-200XLS+单通道电动移液枪 美国瑞宁公司。
1.3 方法
1.3.1 DNA的提取
实验材料基因组DNA的提取参照TIANGEN®植物基因组DNA提取试剂盒说明书。
1.3.2 引物和探针及特异性实验
建立MON89788品系双重dPCR定量检测方法,一个反应体系中同时进行品系特异性外源基因和内源基因扩增,对拷贝数定量检测。品系特异性序列MON89788为外源基因插入序列与大豆基因组序列之间5′边界的跨界序列,参考欧盟方法CRLVL05/06VR《大豆MON89788品系特异性定量实时PCR检测方法》[3]的序列信息。大豆内源基因序列Lectin为植物凝集素基因lectin,参考欧盟方法CRLVL01/08VP《大豆A5547-127品系特异性定量实时PCR检测方法》[19]的序列信息。MON89788探针采用5′-FAM—3′-BHQ1荧光标记,Lectin探针在欧盟方法的序列5′端增加A、G两个碱基,并采用5′-VIC—3′-BHQ1荧光标记。两个目标序列在大豆基因组中均为单拷贝,具体见表2。

表2 引物和探针序列Table2 Sequences of primers and probes used in this study
采用表1中序号1~30的实验材料,对植物组织类型的实验材料按照1.3.1节进行DNA提取,对30 个实验材料的基因组DNA按照欧盟方法CRLVL01/08VP的反应体系和反应条件进行实时荧光PCR扩增,每个样品设置2 个PCR平行,考察引物探针的特异性。
1.3.3 ddPCR体系和反应条件
ddPCR体系为20μL:ddPCRTM预混液 10 μL;MON89788和Lectin正反向引物各0.8 μL(10 pmol/μL)、MON89788和Lectin探针各0.4 μL(10 pmol/μL),DNA模板2 μL,补水至20 μL。分别将20 μL的反应体系和70 μL微滴生成油加入微滴生成卡槽中,盖上胶垫后放入微滴生成仪中进行微滴生成,待微滴生成结束后用单通道电动移液枪将生成的微滴(大约40 μL)全部转移至96 孔板中,用封膜仪进行封膜后置于热循环仪中进行PCR。
反应条件:95 ℃ 5min(1 ℃/s);94 ℃ 15 s(1 ℃/s);60 ℃ 1 min(1 ℃/s),共49 个循环;98 ℃ 10 min(1 ℃/s),12 ℃保存反应产物。
扩增结束后将96 孔板置于微滴分析仪中读取荧光信号,并用QuantaSoft V1.3.2软件分析实验数据。
1.3.4 cdPCR体系和反应条件
cdPCR体系为15 μL:QuantStudio®预混液7.5 μL;MON89788和Lectin正反向引物各0.6 μL(10 pmol/μL)、MON89788和Lectin探针各0.3 μL(10 pmol/μL),DNA模板1.5 μL,补水至15 μL。将配制好的15 μL反应体系通过芯片装载仪自动加载到芯片上的微孔中,体系加载完成后立即使用封油注射器将封油覆盖于芯片表面并将芯片密封。密封后的芯片放置于PCR系统上进行扩增。
反应条件:96 ℃ 10 min;60 ℃ 2 min,98 ℃ 30 s,49 个循环;60 ℃ 2 min;10 ℃保存反应产物。
扩增结束后,待芯片恢复至室温,将芯片置于芯片分析仪中读取并初步分析芯片结果,再经由QuantStudioTM3D AnalysisSuiteTM Cloud Software二次分析实验数据。
1.3.5 重复性实验
以50 ng/μL的转基因大豆MON89788标准物质基因组DNA为模板,分别按照ddPCR和cdPCR的反应体系和反应条件进行PCR扩增。每种dPCR设置3 个平行,软件分析后记录有效反应数和拷贝数浓度,分别计算平均值和相对标准偏差(relative standard deviation,RSD),以RSD不大于25%作为有效定量数据的判断依据,评价方法的可重复性。
1.3.6 定量线性范围的测定
将150 ng/μL的转基因大豆MON89788标准物质基因组DNA用Tris-EDTA缓冲液稀释,得到150、50、10、2、0.4、0.08、0.04 ng/μL共7 个工作质量浓度的DNA溶液。分别取7 个工作质量浓度的DNA溶液为模板,按照ddPCR和cdPCR的反应体系和反应条件进行PCR扩增,每个质量浓度设置3 个平行。计算各质量浓度DNA检测结果的平均值和RSD值。以RSD不大于25%作为有效定量数据的判断依据,并定义绝对定量限为检测结果RSD不大于25%时最低拷贝数浓度。以DNA工作质量浓度为横坐标,以拷贝数浓度的有效定量数据为纵坐标,分别绘制MON89788和Lectin检测的ddPCR和cdPCR线性相关性曲线。
1.3.7 相对定量检测低限的测定
将150 ng/μL的转基因大豆MON89788标准物质基因组DNA用含150 ng/μL的非转基因大豆基因组DNA的Tris-EDTA缓冲液进行10 倍梯度稀释,得到100%、10%、1%、0.1%和0.01%共5 个梯度相对含量的转基因大豆MON89788 DNA溶液。分别取5 个梯度相对含量的DNA溶液为模板,按照ddPCR和cdPCR的反应体系和反应条件进行PCR扩增,每个梯度设置3 个平行。根据测得的MON89788和Lectin基因拷贝数浓度,按照下式求得转基因大豆MON89788品系的相对含量,并计算测得的相对含量与理论相对含量的偏差、3 个平行相对含量的RSD。以RSD不大于25%作为有效定量数据的判断依据,并定义相对定量限为检测结果RSD不大于25%时最低相对含量。同时,利用上述偏差评价定量检测方法的准确度和精密度。
转基因品系MON89788的相对含量/%=100×MON89788拷贝数浓度/Lectin拷贝数浓度
1.3.8 盲样检测
为了评估本方法的适用性,应用建立的转基因大豆MON89788双重dPCR检测方法完成FAPAS国际能力验证项目“GeMMP26动物饲料中转基因品系检测”中的MON89788含量测定,按照1.3.1节进行基因组DNA提取和适当稀释,按照ddPCR和cdPCR的反应体系和反应条件进行PCR扩增,每个浓度设置3 个平行。以FAPAS能力验证评价结果作为本方法适用性的评判依据,并比较ddPCR和cdPCR的实际检测效果。
2 结果与分析
2.1 引物探针特异性实验结果
由表3可知,进行Lectin检测时,序号1~13和序号27的实验材料基因组DNA均够检出阳性信号,其他序号检测结果阴性,说明Lectin引物探针在大豆检测中具有良好的特异性。进行MON89788检测时,只有序号1的实验材料基因组DNA能够检出阳性信号,其他序号检测结果阴性,说明MON89788引物探针对转基因大豆MON89788品系检测特异性好,不会对转基因玉米、转基因马铃薯、转基因苜蓿、转基因油菜、转基因水稻和各种非转基因植物发生交叉反应。特异性实验结果表明,Lectin和MON89788引物探针适于进行后续dPCR定量检测技术研究。

表3 引物和探针特异性实验结果Table3 Specificity evaluation of primers and probes
2.2 dPCR定量检测结果
对序号1的转基因大豆MON89788基因组DNA进行双重dPCR定量检测,在ddPCR与cdPCR平台的二维实验结果图分别见图1和图2。由图1可知,ddPCR中,MON89788的本底荧光强度介于0~350之间,阳性扩增荧光强度介于4 500~7 800之间;Lectin的本底荧光强度介于1 500~1 900之间,阳性扩增荧光强度介于3 000~4 200之间,2 个基因的阳性微滴与阴性微滴之间界限分明。由图2可知,cdPCR中,MON89788本底荧光强度介于0~3 200之间,阳性扩增荧光强度介于5 500~15 000之间;Lectin的本底荧光强度介于0~2 300之间,阳性扩增荧光强度介于3 200~7 000之间,2 个基因阳性微孔与阴性微孔之间界限分明。根据dPCR技术要求,上述结果表明本方法在ddPCR与cdPCR平台都表现良好。相比较而言,ddPCR平台的阳性扩增热点比cdPCR平台分布更为集中,拖尾情况更弱,阴阳性热点之间的间隔更为分明,说明ddPCR的各个反应室间扩增效率更为均一,室间差异更小。
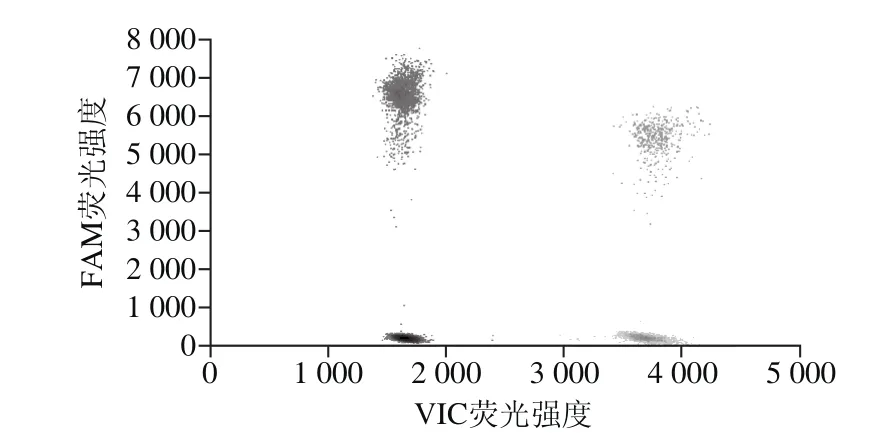
图1 转基因大豆MON89788双重ddPCR 2D图Fig. 1 2D amplitude map of duplex ddPCR for GM soybean event MON89788

图2 转基因大豆MON89788双重cdPCR 2D图Fig. 2 2D amplitude map of duplex cdPCR for GM soybean event MON89788
2.3 重复性实验结果
由表4可知,2 个平台产生的有效反应数平均值均在16 000以上,满足泊松分布统计学原理对有效反应数大于10 000的要求。ddPCR和cdPCR平台有效反应数的RSD值分别为5.49%和1.46%,均远小于25%的评价标准。cdPCR有效反应数RSD值比ddPCR的小,考虑到cdPCR采用芯片装载仪而ddPCR采用微滴生成仪进行反应体系的分散,相较之下ddPCR存在更多的人工操作步骤所致。2 个平台对MON89788和Lectin的拷贝数浓度检测,3 个平行结果RSD值在0.84%~1.99%之间,远小于25%的评价标准。以上数据说明,2 个平台均能得到重复性良好的检测结果。
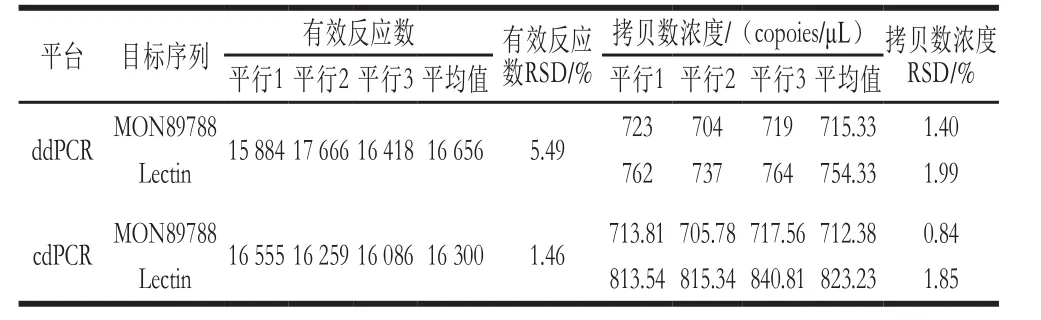
表4 转基因大豆MON89788双重dPCR重复性实验结果Table4 Repeatability of duplex dPCR for detection of GM soybean event MON89788
2.4 定量线性范围实验结果
由表5可知,根据1.3.6节的判断依据,ddPCR检测MON89788的绝对定量限为8.0 copies/μL,RSD为15.66%;检测Lectin的绝对定量限为8.2 copies/μL,RSD为19.22%。cdPCR检测MON89788的绝对定量限为7.443 copies/μL,RSD为7.79%;检测Lectin的绝对定量限为7.646 copies/μL,RSD为7.24%。
以反应体系中DNA模板添加质量浓度0.4~150 ng/μL为横坐标,分别以ddPCR和cdPCR平台的3 个平行检测结果为纵坐标绘制线性范围拟合曲线,由图3和图4可知,所得的4 条曲线中MON89788和Lectin的拷贝数浓度均与DNA模板添加浓度呈现高度的线性相关,相关系数达到0.99以上。说明建立的dPCR方法在2 个平台均具有良好的定量线性相关性,适用于MON89788和Lectin的拷贝数浓度定量检测。
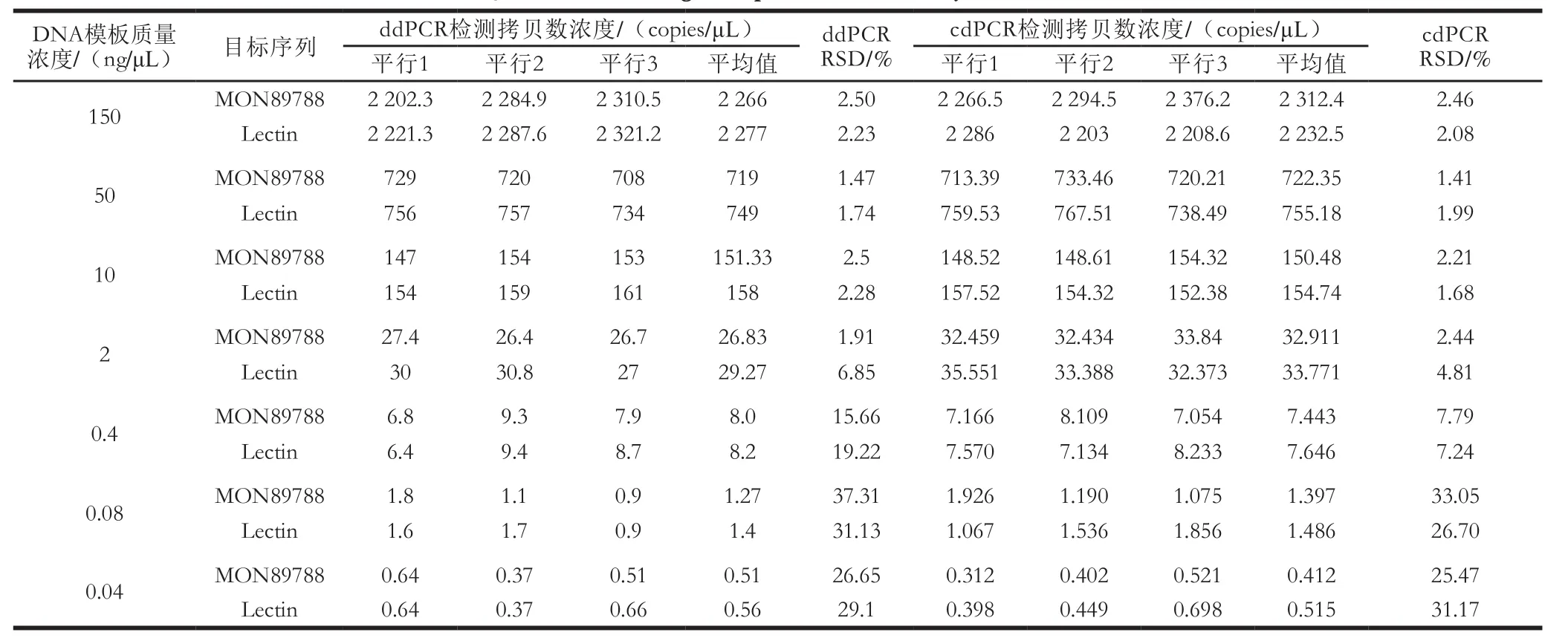
表5 转基因大豆MON89788双重dPCR定量线性范围实验结果Table5 Quantitative linear range of duplex dPCR for GM soybean event MON89788

图3 转基因大豆MON89788双重ddPCR定量线性范围拟合曲线Fig. 3 Quantitative linear range fitting curves of duplex ddPCR for GM soybean event MON89788
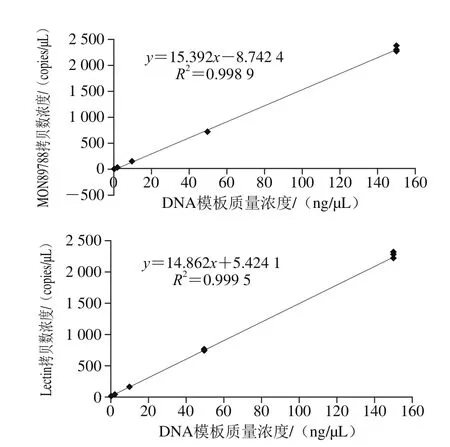
图4 转基因大豆MON89788双重cdPCR定量线性范围拟合曲线Fig. 4 Quantitative linear range fitting curves of duplex cdPCR for GM soybean event MON89788
2.5 相对定量限结果
由表6可知,对于100%、10%、1%、0.1%和0.01%共5 个梯度相对含量的转基因大豆MON89788 DNA溶液,ddPCR测得值分别为98.1%、10.2%、1.05%、0.17%和0.03%,偏差分别为-1.91%、0.20%、0.05%、0.07%和0.02%,RSD分别为3.24%、4.20%、5.99%、16.67%和48.49%;cdPCR测得值分别为99.4%、9.66%、1.2%、0.17%和0.03%,偏差分别为-3.63%、-0.34%、1.12%、0.07%和0.02%,RSD分别为2.71%、2.83%、6.97%、19.88%和40.05%。根据1.3.7节的判断依据,ddPCR和cdPCR的相对定量限均为0.1%,且当转基因大豆MON89788相对含量低至0.01%时2 个平台仍能够稳定的检出阳性结果。
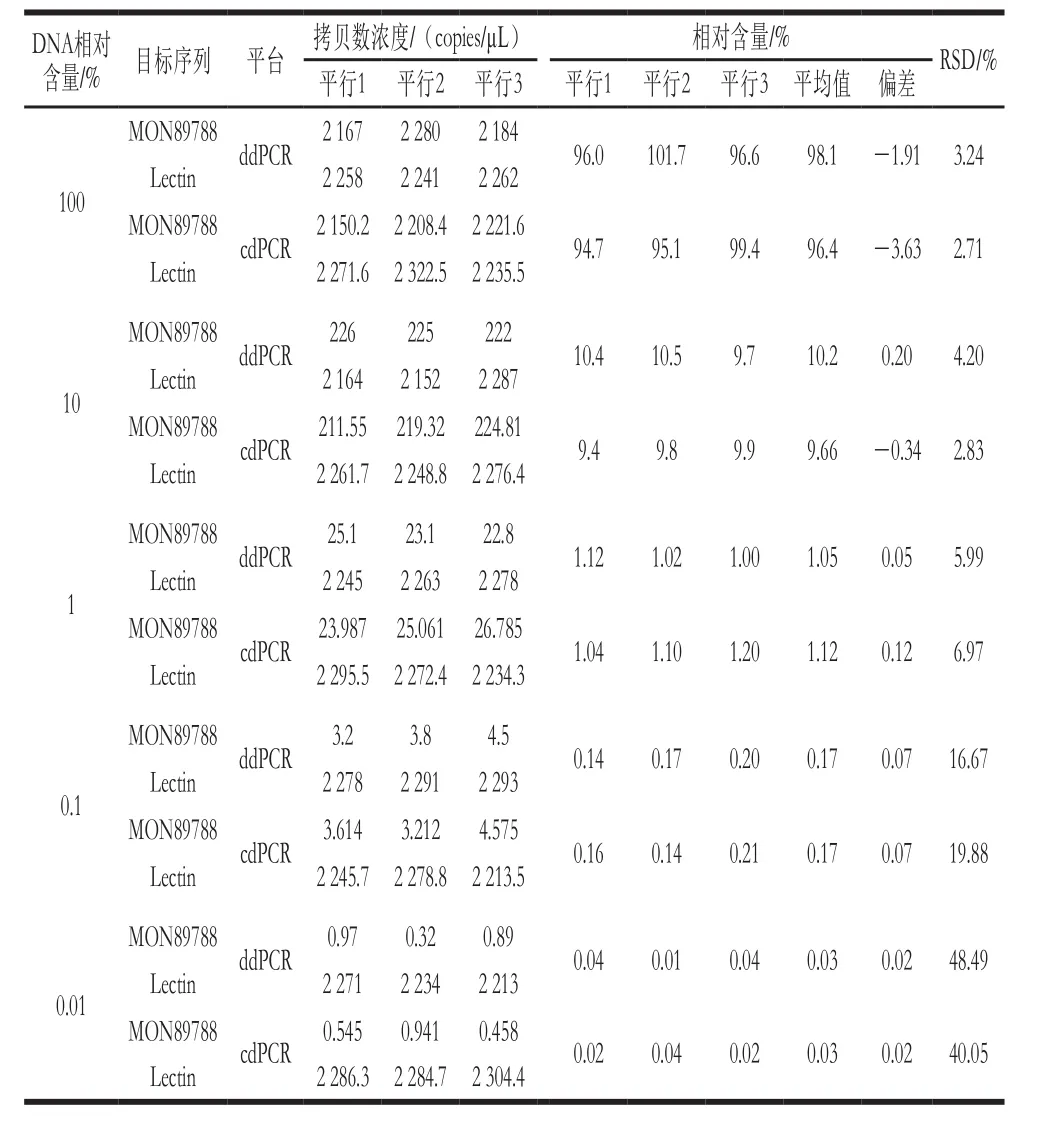
表6 转基因大豆MON89788双重dPCR相对定量限实验结果Table6 Relative LOQs of duplex dPCR for GM soybean event MON89788
2.6 盲样检测结果

表7 盲样检测实验结果Table7 Results of detection of blind samples
盲样提取基因组DNA并适当稀释,得到质量浓度为1 235、400、100 ng/μL的DNA溶液,分别进行MON89788定量检测。由表7可知,当DNA模板质量浓度为1 235 ng/μL时,cdPCR检测MON89788的RSD值最小为3.21%,相对含量平均值为0.887%。因此,本实验室以MON89788相对含量为0.887%报告检测结果。FAPAS对全部18 家参试实验室的定量结果进行统计,盲样中MON89788含量中位值为1%,Z值±2以内评价为“满意”。实验建立的检测方法对盲样的检测结果获得了Z值-0.3的结果,表现优异。根据FAPAS评价标准,在DNA模板质量浓度为1 235 ng/μL和400 ng/μL时,所建立的检测方法在ddPCR和cdPCR 2 个平台均能获得“满意”的检测结果。
3 讨 论
国内外开展dPCR转基因成分定量检测技术研究虽然刚刚起步,但是发展极快。Corbisier[10]、潘广[20-21]、Demeke[22]、于晓帆[23]和任怡菲[24]等应用ddPCR分别建立了转基因玉米MON810、玉米T25、玉米GA21、油菜OXY235、大豆DP305423、大豆DAS-44406-6和水稻LL62的品系特异性定量检测方法;胡佳莹等[25]应用cdPCR建立了转基因玉米MON863的品系特异性定量检测方法;Fu Wei等[26]应用ddPCR和cdPCR建立了CaMV35s启动子和NOS终止子筛选基因定量检测方法;Dobnik等[27]应用ddPCR技术建立了欧盟批准的12 种玉米品系的多重定量检测方法。目前国内ddPCR和cdPCR两种平台几乎各占一半,显然绝大多数研究仅仅基于一种dPCR平台,不能满足现实需要,导致很多研究成果在很多实验室无法应用。虽然Fu Wei等[26]的研究同时在两种平台上开展,但是局限于两种筛选基因的定量检测,对于转基因检测中最大量、最迫切的品系检测尚缺乏通用dPCR定量检测技术开发和比较研究的报道。
ddPCR与cdPCR虽然均基于泊松分布原理进行目标核酸的拷贝数定量,但是由于反应体系分散系统和荧光信号检测系统的差异,实验设计、实验操作和技术性能等方面存在明显区别。总体上,ddPCR的技术要求更高,主要表现在:ddPCR推荐采用本底信号值更低的BHQ1或MGB作为淬灭基团,以便于荧光信号检测系统更加有效的区分阴阳性信号;ddPCR体系分散为油包水的微滴,移液操作不慎易造成微滴破裂并导致有效反应数下降,移液设备优先选择电动移液枪和细长枪头。与此同时,cdPCR采用芯片装载仪进行反应体系的分散,实验数据平行间的重复性较好。不过,由于cdPCR一次最多只能进行24 个反应且芯片装载仪每次仅制作1 张芯片,而ddPCR最多能容纳96 个反应并一次可制备8 个反应的微滴,所以一般情况下超过24 个样品时采用ddPCR在操作上的耗时更短。但是,在进行少量样品的检测实验时,ddPCR要求微滴生成操作数量必须是8的倍数,显然在试剂损耗和时间消耗上cdPCR更为经济和便利。
在检测灵敏度方面,由于无需梯度浓度标准物质制作标准曲线,有效避免了标准物质与待测样品因状态差异导致的DNA提取效率和PCR扩增效率不一致,从而极大降低了因此引起的测量误差,dPCR与qPCR相比对目标核酸的检测更加灵敏准确。根据我国已经颁布的各种标准检测方法[28],转基因成分qPCR的定性限为0.1%。本实验相对定量限实验结果表明,采用dPCR可稳定检出相对含量0.01%的转基因大豆MON89788,与qPCR检测技术相比定性检测限降低10 倍。
目前全球转基因标识管理最为谨慎的欧盟地区规定,转基因成分标识阈值为0.9%[29],转基因成分定量检测平行数据RSD可接受阈值为25%[30],本实验建立的转基因大豆MON89788双重dPCR通用定量检测方法各项技术指标均可满足最为严苛的管理规定,为我国相关标识阈值管理的技术储备提供一种可靠的检测手段。
[1] JAMES C. 2015年全球生物技术/转基因作物商业化发展态势[J].中国生物工程杂志, 2016, 36(4): 1-11. DOI:10.13523/j.cb.20160401.
[2] Global Legal Research Center. Restrictions on genetically modified organisms[EB/OL]. The Law Library of Congress, 2014 (2014-03-01) [2017-02-01]. http://www.loc.gov/law/help/restrictions-on-gmos/restrictions-on-gmos.pdf.
[3] CHRYSTÈLE C D, ALESSIA B, GREGOR P, et al. Event-specific method for the quantification of soybean line MON 89788 using realtime PCR v1.01[J]. Luxembourg: Publications Office of the European Union, 2013: 1-32. DOI:10.2788/20464.
[4] JACCHIA S, NARDINI E, SAVINI C, et al. Development, optimization,and single laboratory validation of an event-specific real-time PCR method for the detection and quantification of Golden Rice 2 using a novel taxon-specific assay[J]. Journal of Agricultural and Food Chemistry,2015, 63(6): 1711-1721. DOI:10.1021/jf505516y.
[5] TSUKAHARA K, TAKABATAKE R, MASUBUCHI T, et al.Development and evaluation of event-specific quantitative PCR method for genetically modified soybean MON87701[J]. Shokuhin Eiseigaku Zasshi, 2016, 57(6): 187-192. DOI:10.3358/shokueishi.57.187.
[6] SYKES P J, NEOH S H, BRISCO M J, et al. Quantitation of targets for PCR by use of limiting dilution[J]. Biotechniques, 1992, 13(3): 444-449.
[7] VOGELSTEIN B, KINZLER K W. Digital PCR[J]. The Proceedings of the National Academy of Sciences of the United States of America, 1999,96(16): 9236-9241. DOI:10.1073/pnas.96.16.9236.
[8] PINHEIRO L B, COLEMAN V A, HINDSON C M, et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J]. Analytical Chemistry, 2012, 84(2): 1003-1011.DOI:10.1021/ac202578x.
[9] 刘津, 刘二龙, 谢力, 等. 数字聚合酶链式反应技术在食品安全检测领域的研究应用进展[J]. 食品科学, 2016, 37(17): 275-280.DOI:10.7506/spkx1002-6630-201617046.
[10] CORBISIER P, BHAT S, PARTIS L, et al. Absolute quantification of genetically modified MON810 maize (Zea mays L.) by digital polymerase chain reaction[J]. Analytical and Bioanalytical Chemistry, 2010, 396(6):2143-2150. DOI:10.1007/s00216-009-3200-3.
[11] BURNS M J, BURRELL A M, FOY C A. The applicability of digital PCR for the assessment of detection limits in GMO analysis[J]. European Food Research and Technology, 2010, 231(3): 353-362. DOI:10.1007/s00217-010-1277-8.
[12] MILAVEC M, DOBNIK D, YANG L, et al. GMO quantification:valuable experience and insights for the future[J]. Analytical and Bioanalytical Chemistry, 2014, 406(26): 6485-6497. DOI:10.1007/s00216-014-8077-0.
[13] PÉREZ U M, AI S A. Copy number ratios determined by two digital polymerase chain reaction systems in genetically modified grains[J].Metrologia, 2014, 51: 61-66. DOI:10.1088/0026-1394/51/1/61.
[14] 中国检验检疫科学研究院. 转基因大米的定量检测[EB/OL]. (2015-10-20) [2015-10-20]. http://sn2.cnca.cn/standard/loginInform/zqyj_lojin_other.jsp.
[15] 姜羽, 胡佳莹, 杨立桃. 利用微滴数字PCR分析转基因生物外源基因拷贝数[J]. 农业生物技术学报, 2014, 22(10): 1298-1305. DOI:10.3969/j.issn.1674-7968.2014.10.013.
[16] 姜志军, 江颖, 徐摇光, 等. 利用微滴数字PCR方法快速分析转基因玉米中外源基因的拷贝数[J]. 生物技术进展, 2016, 6(4): 288-294.DOI:10.3969 /j.issn.2095-2341.2016.04.12.
[17] 农业部农业转基因生物安全管理办公室. 2004-2011年进口用作加工原料的农业转基因生物审批情况[EB/OL]. http://www.moa.gov.cn/ztzl/zjyqwgz/spxx/201202/t20120203_2474426.htm,2012.
[18] 农业部. 转基因植物及其产品成分检测 耐除草剂大豆MON89788及其衍生品种定性PCR方法: 农业部1485号公告-6-2010[S]. 北京: 中国农业出版社, 2010: 1-5.
[19] CHRYSTÈLE C D, ALESSIA B, MAZZARA M, et al. Event-specific qethod for the quantification of soybean Line A5547-127 using real-time PCR-validation report and protocol[J]. Luxembourg: Publications Office of the European Union, 2009: 1-10. DOI:10.2788/60083.
[20] 潘广, 章桂明, 黄新, 等. 应用双重数字PCR对转基因玉米成分进行定量方法研究[J]. 植物检疫, 2016, 30(3): 65-71.
[21] 潘广, 程颖慧, 黄新. 转基因玉米品系GA21拷贝数百分含量与质量百分含量关系研究[J]. 植物检疫, 2016, 30(5): 58-62.
[22] DEMEKE T, GRÄFENHAN T, HOLIGROSKI M, et al. Assessment of droplet digital PCR for absolute quantification of genetically engineered OXY235 canola and DP305423 soybean samples[J]. Food Control, 2014, 46: 470-474. DOI:10.1016/j.foodcont.2014.06.018.
[23] 于晓帆, 高宏伟, 孙敏, 等. 荧光PCR和数字PCR法检测转基因DAS-44406-6品系大豆[J]. 食品科学, 2016, 37(16): 235-241. DOI:10.7506/spkx1002-6630-201616038.
[24] 任怡菲, 高琴, 邓婷婷, 等. 基于数字PCR的转基因水稻LL62品系精准定量检测方法建立[J]. 生物技术通报, 2016, 32(8): 69-76.DOI:10.13560/j.cnki.biotech.bull.1985.2016.08.011.
[25] 胡佳莹, 姜羽, 杨立桃. 利用QuantStudioTM 3D数字PCR分析转基因玉米MON863含量[J]. 农业生物技术学报, 2016, 24(8): 1216-1224.DOI:10.3969/j.issn.1674-7968.2016.08.013.
[26] FU W, ZHU P Y, WANG C G, et al. A highly sensitive and specific method for the screening detection of genetically modified organisms based on digital PCR without pretreatment[J]. Scientific Reports, 2015, 5:1-10. DOI:10.1038/srep12715.
[27] DOBNIK D, SPILSBERG B, BOGOZALEC KOSIR A, et al.Multiplex quantification of 12 European Union authorized genetically modified maize lines with droplet digital polymerase chain reaction[J].Analytical Chemistry, 2015, 87(16): 8218-8226. DOI:10.1021/acs.analchem.5b01208.
[28] 国家质量监督检验检疫总局. 食品中转基因植物成分定性PCR检测方法: SN/T 1202—2010[S]. 北京: 中国标准出版社, 2010.
[29] The European Parliament and The Council of The European Union.Regulation (EC) No 1829/2003 of the European Parliament and of the Council of 22 September 2003 on genetically modified food and feed[J].Official Journal of the European Communities, 2003, L268: 1-23.
[30] European Network of GMO Laboratories. Definition of minimum performance requirements for analytical methods of GMO testing[EB/OL]. (2015-10-20) [2017-02-01]. http://gmocrl.jrc.ec.europa.eu/doc/Min_Perf_Requirements_Analytical_methods.pdf, 2015: 1-24.
