嗜酸乳杆菌ATCC4356分选酶A基因克隆、表达及功能分析
2018-02-09王文文熊蓉露潘道东曾小群郭宇星
吴 京,王文文,熊蓉露,吴 振,*,潘道东,,*,曾小群,郭宇星
(1.宁波大学 浙江省动物蛋白食品精深加工技术重点实验室,浙江 宁波 315211;2.南京师范大学金陵女子学院,江苏 南京 210097)
革兰氏阳性细菌的表面蛋白可以使细菌黏附于一些细胞和组织表面,这种机制可以促使细菌成功定殖于机体内并逃避宿主的免疫监视[1]。部分表面蛋白发挥作用之前需要分选酶A(sortase A,SrtA)的参与,它可以识别表面蛋白特定的LPXTG序列(X表示任意一种氨基酸),并将表面蛋白在苏氨酸(T)和甘氨酸(G)之间切开,使表面蛋白锚定在细胞壁上[2-3]。
分选酶是一种膜结合类的转肽酶,很多表面蛋白发挥功能之前都需要分选酶的参与。目前为止发现的6 种分选酶中,SrtA可以识别LPXTG等序列,使表面蛋白正常发挥功能[4];可催化细菌的多种黏附素或毒力因子锚定在细胞壁表面,在致病菌入侵宿主细胞的过程中发挥着重要作用[5-6]。在对多种革兰氏阳性细菌的SrtA基因进行敲除后发现,SrtA缺陷菌株生理状况没有明显变化,但对宿主细胞的黏附作用大幅下降,从而很大程度上降低了病原菌的致病性[7-8]。
相较于其他革兰氏阳性细菌,乳酸菌的分选酶及表面蛋白没有毒性,因此其分选酶锚定机制被认为是比较安全的。研究发现,乳酸菌SrtA在细菌黏附过程中同样扮演着重要角色。Malik等[9]研究了植物乳杆菌CMPG5300对阴道上皮细胞的黏附,结果发现,CMPG5300 SrtA基因被敲除后,自身的聚集效应明显降低,同时对阴道上皮细胞的黏附作用也会减弱,而对SrtA缺陷菌株进行基因回补后,CMPG5300对细胞的黏附能力又有所回升,表明乳酸菌SrtA同样会影响它对宿主细胞的黏附;SrtA基因的缺失也会影响其他基因的表达,并影响乳酸菌细胞膜的形成。
乳酸菌SrtA可以识别多种表面蛋白,并介导乳酸菌对肠道细胞的黏附,抑制病原菌的入侵[10-11]。虽然这些表面蛋白与肠道细胞的结合位点尚不明确,但可以肯定的是,乳酸菌的表面蛋白可以抑制病原菌对肠道细胞的黏附,并提高乳酸菌在肠胃中的存活率;表面蛋白与肠道细胞受体结合还可以刺激免疫细胞的增殖,增强人体免疫力[12-13];乳酸菌SrtA也有望被开发为一种蛋白质连接工具[14]。因此,对乳酸菌SrtA的研究有助于进一步探究表面蛋白的具体功能,揭示乳酸菌的益生机理。
目前为止,对SrtA的研究主要集中在致病菌及其抑制物的开发上[15-17],虽然获得了诸多成果,但它们对分选酶家族的广谱抑制效应尚少见研究。乳酸菌作为人体肠道中的优势菌群,鲜有文献报道这些抑制剂对乳酸菌SrtA的影响。本研究拟利用pET28a(+)载体对Lactobacillus acidophilus ATCC4356的SrtA进行异源表达,并分析其酶学特性,期望获得具有活性的SrtA,验证分选酶抑制剂对乳酸菌SrtA的抑制作用,为提高乳酸菌分选酶的催化效率以及为后续分选酶抑制剂的筛选奠定基础。
1 材料与方法
1.1 材料
L. acidophilus ATCC4356、pET28a(+)为本实验室保存;荧光多肽Dabcyl-QALPTTGEE(Edans)由上海强耀生物科技有限公司合成;大肠杆菌感受态细胞Trans1-T1、Transetta(DE3)、克隆载体pEASY-Blunt Zero、亲和层析柱Ni-NTA Resin 北京全式金生物技术有限公司;高保真DNA聚合酶、限制性内切酶、T4 DNA连接酶 日本TaKaRa公司;查尔酮 天津一方科技有限公司。
1.2 仪器与设备
INFINITE M200 PRO酶标仪 瑞士TECAN公司;H2500R-2、H-2050R低温离心机 长沙湘仪离心机仪器有限公司;聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Labnet公司;超声波细胞粉碎机 宁波新芝生物科技股份有限公司;Universal HoodⅡ凝胶成像分析系统 美国Bio-Rad公司;Mini Drop超微量分光光度计 依科赛生物科技有限公司;MC99-3自动液相分离层析仪 上海沪西分析仪器厂有限公司。
1.3 方法
1.3.1 嗜酸乳杆菌SrtA基因的克隆及鉴定
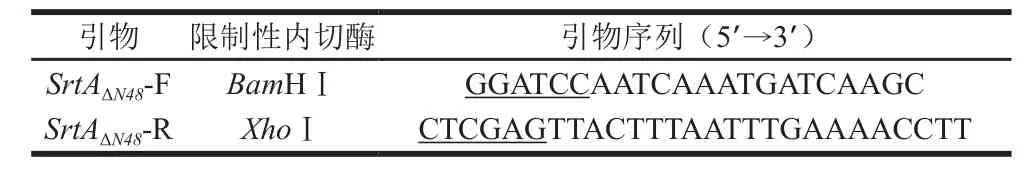
表1 PCR引物序列Table1 Sequences of primers used for PCR
使用信号肽分析软件SignaIP分析SrtA的氨基酸序列,发现L. acidophilus ATCC4356 SrtA(Lap-SrtA)存在一个信号肽剪切位点,位于第48个氨基酸处。根据分析结果,如表1所示,设计引物SrtAΔN48-F、SrtAΔN48-R去除Lap-SrtA N端48 个氨基酸,以XhoⅠ和BamHⅠ作为酶切位点,通过PCR克隆得到目的基因SrtAΔN48,用琼脂糖凝胶电泳检测PCR结果。PCR程序:98 ℃预变性4 min;98 ℃ 10 s,58 ℃ 5 s,72 ℃ 1 min,30 个循环;72 ℃延伸10 min。
PCR产物割胶回收后与Blunt Zero克隆载体连接,转入大肠杆菌Trans1-T1感受态细胞,卡纳霉素筛选转化子,双酶切验证,并送至上海桑尼生物技术有限公司测序。
1.3.2 重组原核表达载体的构建
用BamH I和Xho I分别双酶切Blunt Zero-SrtAΔN48和完整质粒pET28a(+),琼脂糖凝胶电泳检测酶切效果,然后割胶回收目的基因SrtAΔN48和被切开的载体pET28a(+)。用T4 DNA连接酶连接pET28a(+)与SrtAΔN48,连接产物转入大肠杆菌Transetta(DE3),卡那霉素筛选转化子。并通过双酶切、测序等获取重组表达载体pET28a-SrtAΔN48。
1.3.3 重组载体在大肠杆菌中的诱导表达
将含有重组表达载体pET28a-SrtAΔN48的大肠杆菌接种于卡那霉素质量浓度为100 mg/mL的LB肉汤培养基中,37 ℃ 180 r/min 摇床培养至600 nm波长吸光度约为0.6,加入异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG)至终浓度为0.4 mmol/L,25 ℃继续培养16 h,同时以未加IPTG的培养物作为对照。诱导结束后,5 000 r/min离心收集菌体,用0.01 mol/L PBS缓冲液重悬,超声波破碎细菌,10 000 r/min离心30 min,收集上清液,利用Ni-NTA亲和层析柱纯化目的蛋白,然后进行十二烷基硫酸钠-聚丙烯酰胺凝胶蛋白电泳分析,确定蛋白表达情况。
1.3.4 重组蛋白的活性测定
分选酶活性测定选用Dabcyl-QALPTTGEE(Edans)荧光多肽,分选酶可以识别LPTTG序列,并在T与G之间切割肽键,使荧光基团Edans脱落并发出荧光,通过测定反应体系中的荧光强度来测定分选酶的活性。荧光多肽Dabcyl-QALPTTGEE(Edans)用pH 7.5的Tris-HCl缓冲液稀释至5 mmol/L作为储备液。
纯化的蛋白用蛋白活性缓冲液(50 mmol/L Tris-HCl缓冲液,150 mmol/L NaCl溶液,pH 8.0)稀释至0.5 mg/mL,加入96 孔板黑板中,并加入分选酶底物肽至终浓度为10 μmol/L(终反应体系为200 μL),在37 ℃培养箱中孵育2 h,使用酶标仪记录孵育前后体系中的荧光强度(激发波长350 nm,发射波长520 nm),以蛋白活性缓冲液作为阴性对照。
为研究金属离子对Lap-SrtA酶活性的影响,反应体系中分别加入5 mmol/L的CaCl2、MgCl2、ZnCl2及MnCl2溶液(终反应体系为200 μL),以不加任何金属离子的反应体系作为对照,37 ℃培养箱中孵育2 h,测定反应体系中荧光强度,计算相对酶活力。
1.3.5 分选酶抑制剂查尔酮对重组蛋白活性的影响
查尔酮用二甲基亚砜溶解,加入反应体系中,使其终浓度分别为20、40、80、120、160、200 μmol/L,在37 ℃培养箱中孵育30 min后,加入荧光肽底物至终浓度为10 μmol/L(终反应体系为200 μL),继续孵育2 h,按之前的方法读取荧光强度,计算相对酶活力。
1.3.6 嗜酸乳杆菌SrtA生物信息学分析
分别用ExPASY、TMHMM、PSIPRED、SWISS MODEL[18-19]等蛋白质在线分析软件分析分选酶的结构特点并与其他分选酶进行比对。
1.4 数据处理
所有数据用Origin 8.5软件绘制趋势曲线图,采用一维方差分析比较数据平均值之间的差异显著性。
2 结果与分析
2.1 SrtA基因的PCR扩增和重组表达载体的构建
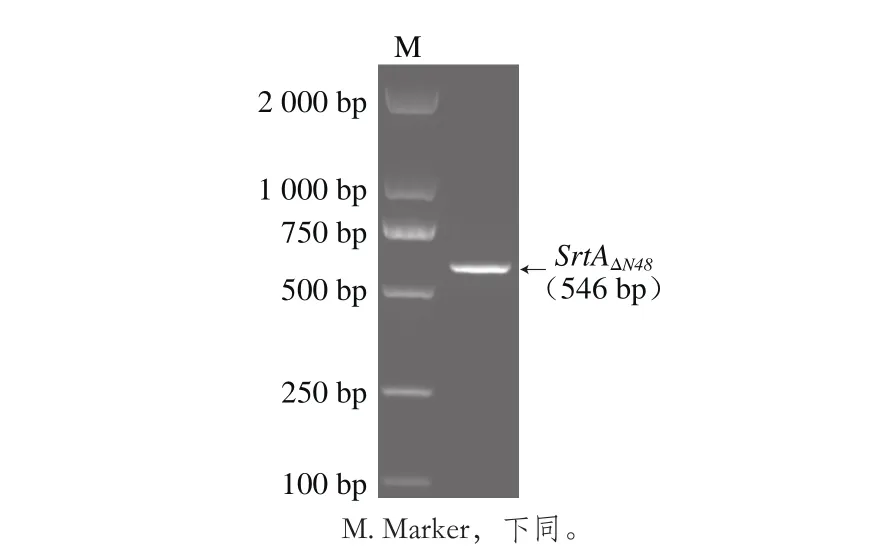
图1 SrtAΔN48基因PCR产物电泳图Fig. 1 Agarose gel electrophoresis of PCR products of SrtAΔN48 gene
由图1可知,以L. acidophilus ATCC4356基因组DNA为模板扩增产物长度为546 bp,符合预计长度。测序结果经过NCBI对比,与L. acidophilus NCFM序列相似性为99%,氨基酸序列完全相同。将克隆载体Blunt Zero-SrtAΔN48双酶切后得到的SrtAΔN48与pET28a(+)连接,卡那霉素筛选阳性菌,并送至上海桑尼生物技术有限公司测序,测序结果显示整个克隆过程中没有引入突变碱基。
2.2 SrtA基因在大肠杆菌中的表达与纯化
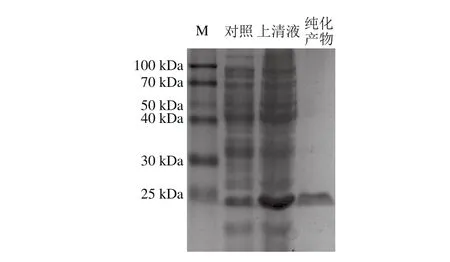
图2 Transetta(DE3)/pET28a-SrtAΔN48的表达产物十二烷基硫酸钠-聚丙烯酰胺凝胶电泳分析Fig. 2 SDS-PAGE analysis of expression product from Transetta (ED3)/pET28a-SrtAΔN48
将重组载体pET28a-SrtAΔN48转入表达菌株Transetta(DE3),在25 ℃、0.4 mmol/L IPTG条件下表达及纯化结果如图2所示。SrtA作为一种膜蛋白,其氨基酸序列的N端含有一段强疏水性序列,包括跨膜区域和信号肽,这段序列可以使膜蛋白成功锚定在细胞壁上;但在基因异源表达过程中,疏水性序列会严重影响蛋白质的正确折叠以及可溶性表达[7,20]。为获得具有活性的SrtA蛋白,本研究设计引物去除了SrtA酶N端的48 个氨基酸,然后使用pET系列载体在大肠杆菌内表达目的基因Lap-SrtAΔN48。L. acidophilus ATCC4356 SrtA基因原本编码229 个氨基酸,切除N端48 个氨基酸后,相对分子质量会有所减小。由图2可知,SrtAΔN48在约20 kDa的位置出现了明显的条带,同时,利用Ni-NTA亲和层析柱,获得了高纯度的SrtAΔN48蛋白。
2.3 金属离子及查尔酮对嗜酸乳杆菌SrtA酶活力的影响
分选酶进行转肽作用时不需要ATP的参与,因此在体外可以较为方便地测定Lap-SrtAΔN48的活性。利用带有荧光基团的底物肽Dabcyl-QALPTTGEE(Edans)测定SrtA的活性,Lap-SrtAΔN48切割荧光基团结果如图3所示。由于对照组没有分选酶活性,在孵育过程中,荧光强度会衰减,因此相对酶活力呈负值,SrtA在37 ℃时可以成功切断肽段LPTTG,使荧光基团Edans发出荧光。
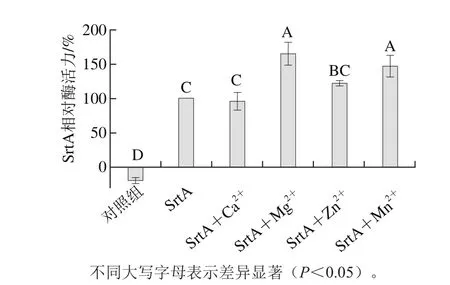
图3 不同金属离子对Lap-SrtAΔN48蛋白活性的影响Fig. 3 Effect of different metal ions on Lap-SrtAΔN48 activity
金属离子是生命体不可缺少的微量元素,对酶的活力和机体生长发育有着重要的影响。由图3可知,Mg2+、Mn2+可显著提高Lap-SrtAΔN48的活力(P<0.05);Zn2+也可以提高Lap-SrtAΔN48的活力,但效果并不显著(P>0.05);Ca2+不会影响Lap-SrtAΔN48的活力(P>0.05),明显区别于其他SrtA酶。Ilangovan[3]、Li Hong’en[21]等在对金黄色葡萄球菌OS2分选酶(Sa-SrtA)及单核细胞性李斯特菌BUG1600分选酶(Lm-SrtA)的研究过程中发现,Ca2+可以提高Sa-SrtA及Lm-SrtA的活力至原来的4 倍以上。Naik等[22]研究了Sa-SrtAΔN59与Ca2+的关系,结果表明,Sa-SrtA β6与β7之间的谷氨酸(Glu171)可以使Ca2+结合到酶活中心附近,改变Sa-SrtA的动力学结构,提高Sa-SrtA的催化效率,而Lap-SrtAΔN48在这一区域并未发现功能相似的氨基酸(图6)。
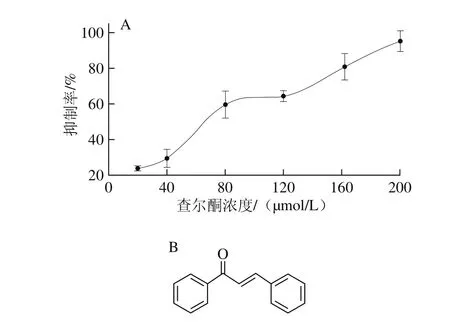
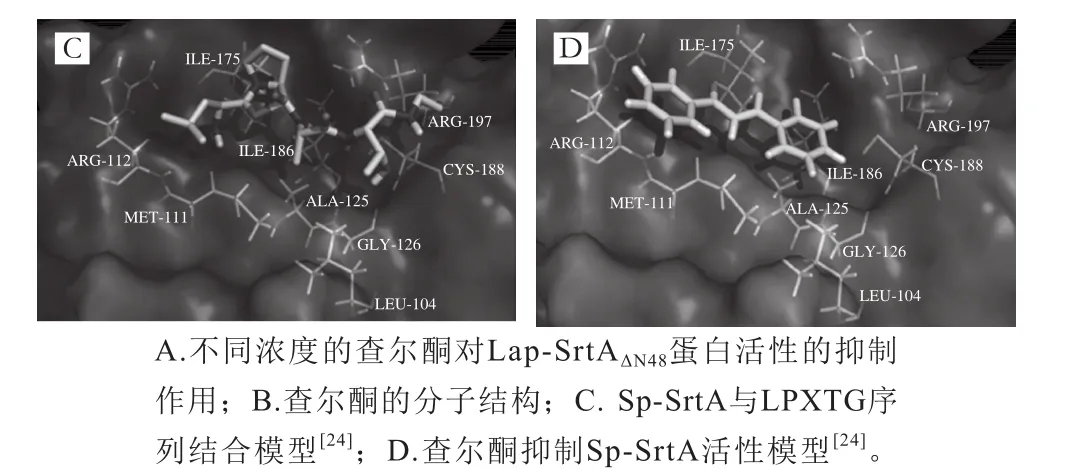
图4 查尔酮对SrtA蛋白活性的抑制作用Fig. 4 Inhibitory activity of chalcone on SrtA
由图4可知,查尔酮在体外可以显著抑制Lap-SrtAΔN48的活性,抑制效果与查尔酮的浓度有关,浓度越高,查尔酮的抑制作用越强。结合以往的研究结果,查尔酮可以与荧光肽底物竞争酶活位点,使Lap-SrtAΔN48丧失转肽能力。
查尔酮是一种黄酮类化合物,普遍存在于植物体内[23]。研究发现,查尔酮可以直接封闭蛋白酶的活性位点,阻止SrtA的转肽作用,抑制Listeria monocytogenes、Streptococcus mutans、S. pyogenes[21,24]等多种致病菌对宿主细胞的黏附,具有良好的应用前景。由于查尔酮有较高的广谱抑制效果,本实验选择它作为研究Lap-SrtAΔN48特性的工具。结果显示,查尔酮同样会抑制Lap-SrtAΔN48的活性,查尔酮特殊的分子结构具有较大的柔性,如果人体肠道中其他益生菌的SrtA结构与L. acidophilus ATCC4356相似,查尔酮就会干扰正常的肠道微生物环境,影响人体健康。查尔酮对多种SrtA的抑制效应也表明,依靠特殊的分子结构抑制SrtA酶活力的方法没有特异性,因而在通过查尔酮阻止肠道致病菌对肠道的吸附作用进行治疗时,可能会影响人体内原有的微生物,引发一定的副作用。
2.4 Lap-SrtAΔN48重组蛋白生物信息学分析
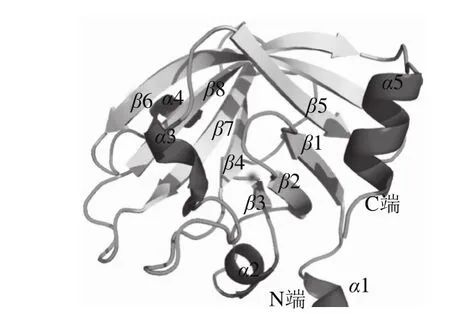
图5 预测的Lap-SrtA三维结构Fig. 5 Structure of Lap-SrtA predicted by SWISS MODEL
使用ExPASY在线检测工具对Lap-SrtAΔN48分析后发现,Lap-SrtAΔN48总氨基酸数为181,分子质量约为20 kDa,理论等电点为9.95,分子式为C891H1466N244O255S11,脂肪指数为85.14,不稳定指数为23.95,属于稳定蛋白质(不稳定指数大于40属于不稳定蛋白质)。SrtA属于Ⅱ类膜拓扑结构(N端位于细胞膜内侧,C端位于细胞膜外侧),蛋白质在细胞壁外侧发挥作用。蛋白质二级结构预测显示,Lap-SrtAΔN48中含有14.92%的α螺旋结构和26.52%的β折叠结构;可能存在11 个蛋白质识别位点和1 个蛋白质-核酸结合位点。SrtA可以催化多种表面蛋白,这些蛋白质识别位点可能对应的是不同的表面蛋白。
由图5可知,采用SWISS MODEL构建的蛋白质三维结构显示,Lap-SrtAΔN48由5 段α螺旋和8 段β折叠结构组成,其中,8 条反向平行的β折叠形成了一个β中心桶状结构,与单核细胞性李斯特菌、金黄色葡萄球菌、酿脓链球菌的基本结构相同。β中心桶状结构是SrtA的经典结构,是分选酶进行转肽作用的基础,研究表明,位于β4、β7和β8上活性位点会形成一个口袋结构,这种结构可以识别表面蛋白的LPXTG序列,并攻击T与G之间的肽键,催化转肽过程(图4)[25]。
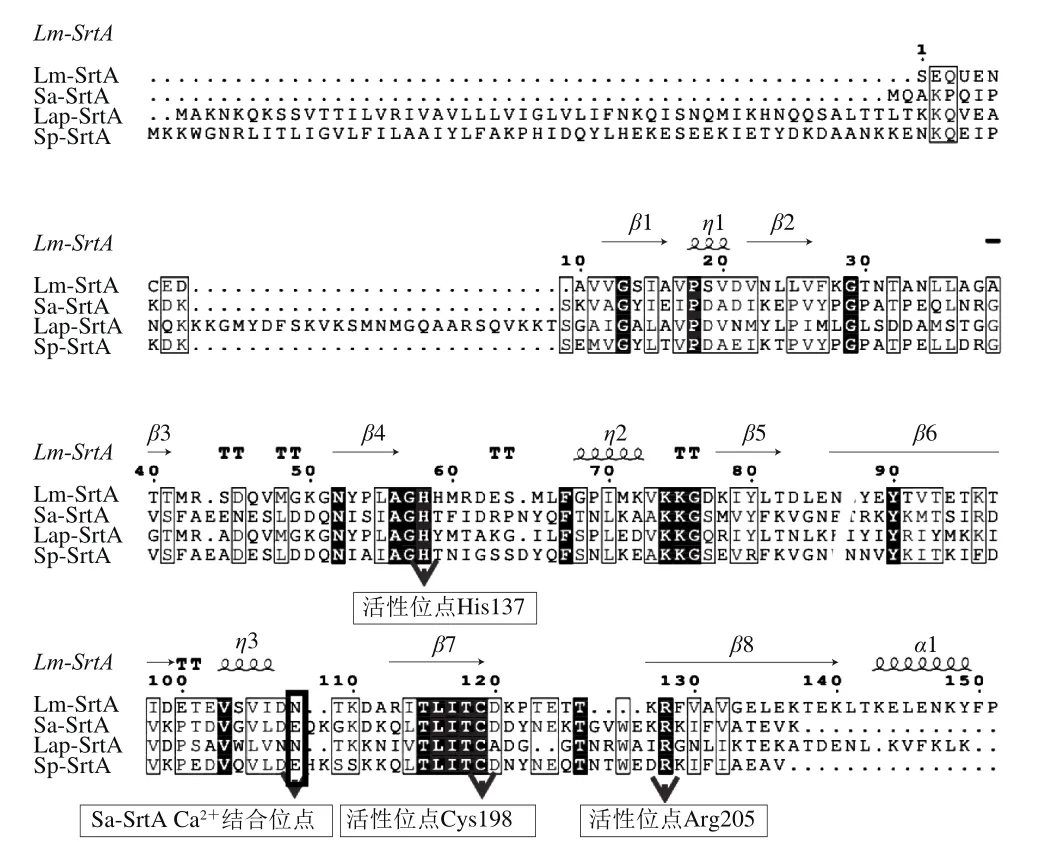
图6 Lap-SrtA蛋白与同源蛋白序列比对结果Fig. 6 Results of multiple sequence alignment of Lap-SrtA protein and homologous proteins
由图6可知,以Lm-SrtA为模板,将Lap-SrtAΔN48与其他革兰氏阳性细菌分选酶序列比对后发现,多种革兰氏阳性细菌SrtA之间有很高的同源性,并且均存在组氨酸(H)-半胱氨酸(C)-精氨酸(R)三联体活性中心,这是SrtA的主要活性氨基酸,此外,组氨酸附近存在LAGH等保守序列,半胱氨酸附近也存在TLITC保守序列,可能与分选酶活性有关。结合序列比对结果,本研究推测Lap-SrtA活性氨基酸为His137、Cys198、Arg205。组氨酸和精氨酸是SrtA酶抓取LPXTG序列的必需氨基酸,而半胱氨酸可以将底物从TG之间切开[26]。
3 讨 论
分选酶锚定机制是细菌外蛋白的5 种锚定方式之一[27],本研究利用大肠杆菌表达载体获得了具有活性的Lap-SrtA蛋白,并对其分子生物学特性作了初步分析。
相较于Sa-SrtA、Lm-SrtA,Ca2+不会影响Lap-SrtA的活性,氨基酸序列比对发现,Lap-SrtA在β6与β7之间没有谷氨酸(Glu),这可能是Lap-SrtA对Ca2+不敏感的原因之一。谷氨酸是Sa-SrtA结合Ca2+的关键氨基酸 ,在Sa-SrtA催化转肽作用时,Ca2+可以同时结合β6/β7之间的谷氨酸和β3/β4之间天冬氨酸、天冬酰胺、谷氨酸等氨基酸,拉近两段肽链之间的距离,使Sa-SrtA的活性位点发生形变,提高催化效率。另外,金黄色葡萄球菌,单核细胞性李斯特菌均为致病菌,其分选酶及分选酶依赖蛋白多数为毒性蛋白,Sa-SrtA及Lm-SrtA对Ca2+的敏感性很可能是为了适应毒性蛋白的锚定而进化的特殊性质[22]。
生物信息学分析结果显示,Lap-SrtA的结构和活性位点与Sa-SrtA、Sp-SrtA和Lm-SrtA相似,均由8条β折叠和若干α螺旋及部分β转角组成;其中,位于β4、β7、β8上的组氨酸-半胱氨酸-精氨酸构成了SrtA的三联体活性中心。由于具有相似的结构和相同的活性位点,查尔酮同样可以抑制Lap-SrtA的活性。通过与Lap-SrtA的活性位点相结合,查尔酮可以阻止Lap-SrtA与底物蛋白间的相互作用,抑制Lap-SrtA的催化作用[24]。因此,查尔酮虽然对多种分选酶有抑制效果,但它也有一定的副作用,如果长期作用于生物体,很可能会影响机体内的正常菌群。鉴于查尔酮的广谱抑制效应,本研究认为,以竞争性抑制为原理筛得的SrtA抑制剂有一定的缺陷,针对致病菌分选酶抑制剂的开发可以从它们对Ca2+的敏感性入手,如果可以进一步确定Ca2+的作用机制,设计相关实验,降低SrtA与Ca2+的相互作用,同样可以有效抑制致病菌SrtA的活性,同时不影响嗜酸乳杆菌ATCC4356分选酶的正常功能。
乳酸菌必须定殖于人体肠道中才能发挥益生作用,因此,乳酸菌对肠道细胞的黏附被认为是定殖的首要条件[28]。但是近年来不断有研究显示,目前的益生菌产品中的乳酸菌在肠道中的滞留能力并不理想。虽然乳酸菌可以耐受人体消化道严苛的环境,成功到达肠道,但它们大部分并不能成功定殖,多数外源乳酸菌在肠道内存在时间很短,一旦停止从外界摄入,它们的数目会在2 周内迅速衰减[29-30]。因此,提高乳酸菌的定殖能力一直是乳品研究者们努力的方向。本实验研究发现,Lap-SrtA对Ca2+不敏感,催化效率较低,会导致细菌表面黏附蛋白含量减少,对宿主细胞的黏附能力也会降低,因此,嗜酸乳杆菌在肠道中定殖成功率不高,很容易被其他肠道菌群排斥。目前,可以通过改变乳酸菌的培养条件,筛选黏附性较强的乳酸菌菌种,改善乳酸菌在肠道中的定殖率;另一方面,通过分子生物学的方法增强乳酸菌分选酶的含量,进而提高乳酸菌的黏附能力也是一个可行的方法。
分选酶虽然在多种乳酸菌中被发现,但它识别表面蛋白的种类及底物识别机制尚不明确,SrtA在肠道环境中的催化效率、转运方式、相应的激活剂及抑制剂等仍需要进一步阐明。只有获得这些酶更为详细的信息,才能使乳酸菌及其制品获得更好的应用。
[1] 石洁, 王长军. 病原菌逃避宿主细胞防御的策略[J]. 微生物学通报,2012, 39(1): 90-98.
[2] MARRAFFINI L A, DEDENT A C, SCHNEEWIND O. Sortases and the art of anchoring proteins to the envelopes of Gram-positive bacteria[J]. Microbiology & Molecular Biology Reviews Mmbr, 2006,70(1): 192-221. DOI:10.1128/MMBR.70.1.192-221.2006.
[3] ILANGOVAN U, TONTHAT H, IWAHARA J, et al. Structure of sortase, the transpeptidase that anchors proteins to the cell wall of Staphylococcus aureus[J]. Proceedings of the National Academy of Sciences, 2001, 98(11): 6056-6061. DOI:10.1073/pnas.101064198.
[4] 罗立新, 施周铭. 一种抗革兰氏阳性致病菌新型靶酶: 分选酶[J]. 生物化学与生物物理进展, 2006, 33(9): 828-833.
[5] CHEN J, DONG H, MURFIN K E, et al. Active site analysis of sortase A from Staphylococcus simulans, indicates function in cleavage of putative cell wall proteins[J]. Biochemical & Biophysical Research Communications, 2016, 478(4): 1653-1659. DOI:10.1016/j.bbrc.2016.08.175.
[6] 李森, 热依汉古丽·麦麦提力. 单增李斯特菌srtA基因敲除菌株的构建及其生物学特性[J]. 食品科学, 2017, 38(4): 113-117. DOI:10.7506/spkx1002-6630-201704019.
[7] MAZMANIAN S K, LIU G, TONTHAT H, et al. Staphylococcus aureus sortase, an enzyme that anchors surface proteins to the cell wall[J]. Science, 1999, 285(5428): 760-763.
[8] PATERSON G K, MITCHELL T J. The role of Streptococcus pneumoniae, sortase A in colonisation and pathogenesis[J]. Microbes &Infection, 2006, 8(1): 145-153. DOI:10.1016/j.micinf.2005.06.009.
[9] MALIK S, PETROVA M I, CLAES I J, et al. The highly autoaggregative and adhesive phenotype of the vaginal Lactobacillus plantarum strain CMPG5300 is sortase dependent[J]. Applied & Environmental Microbiology, 2013, 79(15): 4576-4585. DOI:10.1128/AEM.00926-13.
[10] LEBEER S. Functional analysis of Lactobacillus rhamnosus GG pili in relation to adhesion and immunomodulatory interactions with intestinal epithelial cells[J]. Applied & Environmental Microbiology, 2012, 78(1):185-193. DOI:10.1128/AEM.06192-11.
[11] WU Z, GUO X, WANG Q, et al. Sortase A-catalyzed transpeptidation of glycosylphosphatidylinositol derivatives for chemoenzymatic synthesis of GPI-anchored proteins[J]. Journal of the American Chemical Society,2010, 132(5): 1567-1571. DOI:10.1021/ja906611x.
[12] KONSTANTINOV S R, SMIDT H, DE VOS W M, et al. S layer protein A of Lactobacillus acidophilus NCFM regulates immature dendritic cell and T cell functions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(49): 19474-19479.DOI:10.1073/pnas.0810305105.
[13] RASK C, ADLERBERTH I, BERGGREN A, et al. Differential effect on cell-mediated immunity in human volunteers after intake of different lactobacilli[J]. Clinical & Experimental Immunology, 2013, 172(2): 321-332. DOI:10.1111/cei.12055.
[14] CALL E K, KLAENHAMMER T R. Relevance and application of sortase and sortase-dependent proteins in lactic acid bacteria[J]. Frontiers in Microbiology, 2013, 4(3): 73. DOI:10.3389/fmicb.2013.00073.
[15] ZHULENKOVS D, RUDEVICA Z, JAUDZEMS K, et al.Discovery and structure-activity relationship studies of irreversible benzisothiazolinone-based inhibitors against Staphylococcus aureus sortase A transpeptidase[J]. Bioorganic & Medicinal Chemistry, 2014,22(21): 5988-6003. DOI:10.1016/j.bmc.2014.09.011.
[16] BI C, WANG L, NIU X, et al. The use of chlorogenic acid and its analogues as inhibitors: an investigation of the inhibition of sortase A of Staphylococcus aureus, using molecular docking and dynamic simulation[J]. Biotechnology Letters, 2016, 38(8): 1-7. DOI:10.1007/s10529-016-2112-5.
[17] LIU B, CHEN F, BI C, et al. Quercitrin, an inhibitor of sortase A,interferes with the adhesion of Staphylococcal aureus[J]. Molecules,2015, 20(4): 6533-6543. DOI:10.3390/molecules20046533.
[18] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.DOI:10.1093/bioinformatics/btm404.
[19] GOUET P, COURCELLE E, STUART D I. ESPript: analysis of multiple sequence alignments in PostScript[J]. Bioinformatics, 1999,15(4): 305-308.
[20] 罗立新, 江彬强, 陈谋通. 新抗菌靶点分选酶基因(srtA)在两种原核载体中的克隆表达[J]. 微生物学报, 2009, 49(2): 186-190.
[21] LI H, CHEN Y, ZHANG B, et al. Inhibition of sortase A by chalcone prevents Listeria monocytogenes, infection[J]. Biochemical Pharmacology, 2016, 106: 19-29. DOI:10.1016/j.bcp.2016.01.018.
[22] NAIK M T, SUREE N, ILANGOVAN U, et al. Staphylococcus aureus sortase A transpeptidase. calcium promotes sorting signal binding by altering the mobility and structure of an active site loop[J]. Journal of Biological Chemistry, 2005, 281(3): 1817-1826. DOI:10.1074/jbc.M506123200.
[23] KUMAR D, KUMAR M, KUMAR A, et al. Chalcone and curcumin derivatives: a way ahead for malarial treatment[J]. Mini Reviews in Medicinal Chemistry, 2013, 13(14): 2116.
[24] 李洪恩. 查尔酮对单增李斯特杆菌以及肺炎链球菌D39分选酶A抑制活性的分子机制研究[D]. 长春: 吉林大学, 2016: 51-70.
[25] WALLOCK-RICHARDS D J, MARLES-WRIGHT J, CLARKE D J,et al. Molecular basis of Streptococcus mutans sortase A inhibition by the flavonoid natural product trans-chalcone[J]. Chemical Communications, 2015, 51(52): 10483-10485. DOI:10.1039/c5cc01816a.
[26] ZONG Y, BICE T T H, SCHNEEWIND O, et al. Crystal structures of Staphylococcus aureus sortase A and its substrate complex[J]. Journal of Biological Chemistry, 2004, 279(30): 31383-31389. DOI:10.1074/jbc.M401374200.
[27] COSSART P, JONQUIÈRES R. Sortase, a universal target for therapeutic agents against Gram-positive bacteria?[J]. Proceedings of the National Academy of Sciences, 2000, 97(10): 5013-5015.
[28] 周方方, 郭本恒, 吴正钧, 等. 干酪乳杆菌LC2W对两种不同胃癌细胞的黏附作用[J]. 中国乳品工业, 2007, 35(8): 15-18.
[29] CHARBONNEAU D, GIBB R D, QUIGLEY E M M. Fecal excretion of Bifidobacterium infantis 35624 and changes in fecal microbiota after eight weeks of oral supplementation with encapsulated probiotic[J].Gut Microbes, 2013, 4(3): 201-211. DOI:10.4161/gmic.24196.
[30] FRESE S A, HUTKINS R W, WALTER J. Comparison of the colonization ability of autochthonous and allochthonous strains of lactobacilli in the human gastrointestinal tract[J]. Advances in Microbiology, 2012, 2(3): 399-409. DOI:10.4236/aim.2012.23051.
