犏牛体外受精胚胎的发育转录组分析
2018-01-26字向东熊显荣张正帆李志雄钟金城朱江江
字向东,刘 霜,夏 威,熊显荣,黄 林,张正帆,李志雄,李 键,钟金城,王 利,朱江江
(1.西南民族大学生命科学与技术学院,成都 610041;2.西南民族大学青藏高原研究院,成都 610041)
牦牛(Bosgrunniens)是分布在海拔2 000~5 000 m以青藏高原为中心,及其毗邻高山、亚高山地区的特有牛种。对高寒低氧的独特适应能力决定了它在青藏高原畜牧业中不可替代的地位[1]。但是,相对于普通牛品种而言,牦牛生产生长速度慢,产肉性能和产奶性能都很低。利用普通牛(Bostaurus)优良奶牛品种杂交改良牦牛繁殖的后代(犏牛)产奶性能成倍提高,深受牧民欢迎。但是,牦牛与普通牛种间杂交受胎率低[1-2],体外受精(Invitrofertilization, IVF)的杂种胚胎发育率也不高[3-4],其原因还不清楚。因此,研究犏牛胚胎发育调控机制,对提高牦牛种间杂交效率,完善犏牛胚胎体外生产技术具有十分重要的意义。
胚胎发育是众多基因表达在时间和空间上的联系及配合共同作用的结果[5-8],逐一研究每个候选基因的表达模式与胚胎发育的关系从效率上和可行性上都受到限制。目前,犏牛胚胎发育调控机制方面的研究尚属空白。随着RNA高通量测序技术的发展[9-10]和牦牛基因组测序的完成[11],为从组学水平研究犏牛胚胎发育的分子机制和保存提供了快速、有效的方法。因此,本研究拟用娟珊牛精子体外受精牦牛卵母细胞,通过体外培养获得杂种胚胎,然后利用微量RNA高通量测序技术分析不同发育阶段的犏牛胚胎转录组,揭示犏牛早期胚胎发育机制, 为提高犏牛胚胎体外生产效率提供理论基础。
1 材料与方法
1.1 犏牛胚胎的生产
在屠宰场采集的牦牛卵巢放入盛有DPBS液(29~33 ℃)的保温瓶中, 2 h内运回到实验室。参照X. Xiao等[4]的方法开展牦牛卵母细胞体外成熟(Invitromaturation, IVM):将从直径为2~8 mm的卵泡中抽取的卵丘-卵母细胞复合体(COC)放入含10% FCS、5 μg·mL-1FSH、50 IU·mL-1LH和1 μg·mL-117β-E2的TCM199液中,在38.5 ℃、5%CO2、饱和湿度的CO2培养箱中成熟培养24 h。采用娟珊牛冷冻精液进行体外受精(IVF)。参照石仙等[12]的方法:用Vitrolife(瑞典)体外胚胎生产系列试剂在38.5 ℃、5% CO2、5% O2、90% N2、饱和湿度的三气培养箱中进行精子体外获能、IVF和胚胎的体外培养(Invitroculture, IVC)。在受精48 h的2-细胞、68~72 h的4-细胞、88~96 h的8-细胞,每个阶段收集10枚发育良好的胚胎;受精第6 天的桑椹胚和第7天囊胚各收集1枚,提取RNA。
1.2 胚胎总RNA提取、cDNA文库构建及测序
采用RNeasy Micro Kit分别提取胚胎总RNA后,用Smart-Seq2方法[9]进行扩增:加入反应buffer、反转录酶、含公共序列的Oligo-dT引物和TSO引物,反应得到第一条cDNA链;不转管,直接加入含共同序列的ISPCR引物和PCR扩增试剂,反应得到长度1~2 kb的第二条cDNA链。对富集扩增的cDNA采用Agilent 2100 High Sensitivity DNA Assay Kit检测扩增产物cDNA样品片段分布情况。峰图显示在1~2 kb片段长度存在明显的目的产物主峰,表明原始样本完整性较好,可以进行文库构建。每个样本各选取20 ng扩增产物cDNA作为起始样本构建文库。使用Bioruptor®Sonication System(Diagenode Inc.)进行样本cDNA片段化,使之断裂为200 bp左右的小片段。经超声打断后,进行样本cDNA末端修复、加 poly(A)并连接测序接头,每步反应后使用Beckman Ampure XP磁珠进行纯化。取接头产物进行PCR扩增,样本分别引入不同的Index标签。最后用2%(w/v)琼脂糖凝胶电泳检测PCR扩增产物后,切取DNA片段的凝胶块,使用CWBIO Gel Extraction Kit快速琼脂糖凝胶DNA回收试剂盒回收DNA,再溶于EB缓冲液中,获得测序文库。用HiSeqTM2500进行高通量测序。
1.3 转录组数据分析
llumina HisSeqTM2500测序产生的原始图像数据经Base calling转化为序列数据(Raw Reads),结果以FASTQ文件格式存储,包含Reads的序列以及碱基的测序质量。Raw Reads经公共序列污染及酶切位点、Adapter污染、含N过多(>5%)、低质量(Q≤19)以及比对到rRNA库上的Reads过滤后,获得纯净 Reads(Clean Reads)。采用TopHat v2.0.12软件[13]将测序过滤后所得的Clean Reads与牦牛基因组(http://me.lzu.edu.cn/yak)[11]进行比对分析,获得唯一比对上参考基因的 Reads(Unique reads)。采用RPKM法(每百万 Reads 中来自于某基因每千碱基长度的 Reads 数,Reads per kilobase transcriptome per million mapped Reads)计算基因表达量[14],参照无生物学重复样品的DEGseq法基因差异表达分析[15],以|log2Ratio|≥1和Q<0.05为阈值, 筛选出不同发育阶段间的差异表达基因(Differentially expressed genes, DEGs)。将DEGs与参考基因比较,得出DEGs显著富集的Gene Ontology(GO)功能条目,并进一步挖掘出与DEGs显著相关的生物学功能。然后将DEGs向GO数据库(http://www.geneontology.org/)各个条目进行映射,计算其数目,用Y. Benjamini和Y. Hochberg方法[16]对P-value进行校正后,以Q-value<0.05为阈值定义DEGs中显著富集的GO条目。通过与京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)数据库(http://wego.genomics.org.cn)进行比对,对基因涉及的信号通路或代谢途径(Pathway)进行分析。
1.4 差异表达基因荧光定量 PCR验证
随机选择与发育相关的SKP1、CD63、ZAR1和H3等4个DEGs,采用Primer5.0软件进行基因的定量引物设计(表1),以1.2中反转录获得的2-、4-、8-细胞、桑椹胚和囊胚的cDNA等体积混合,然后倍比稀释,将稀释的样本作为模板,构建其荧光定量标准曲线。qRT-PCR反应体系为10 μL:上下游引物各0.8 μL,Sso AdvancedTMSYBR®Green Super mix 5 μL,ddH2O 2.9 μL,cDNA模板0.5 μL。扩增条件:95 ℃预变性3 min,95 ℃ 10 s、59.5 ℃ 20 s,35个循环。以H2A为内参,采用2-ΔΔCt计算基因的差异倍数。每个样品设3次重复。
表1验证RNA-Seq的qRT-PCR基因及其引物
Table1GenesandqPCRprimersusedforRNA-Seqvalidation
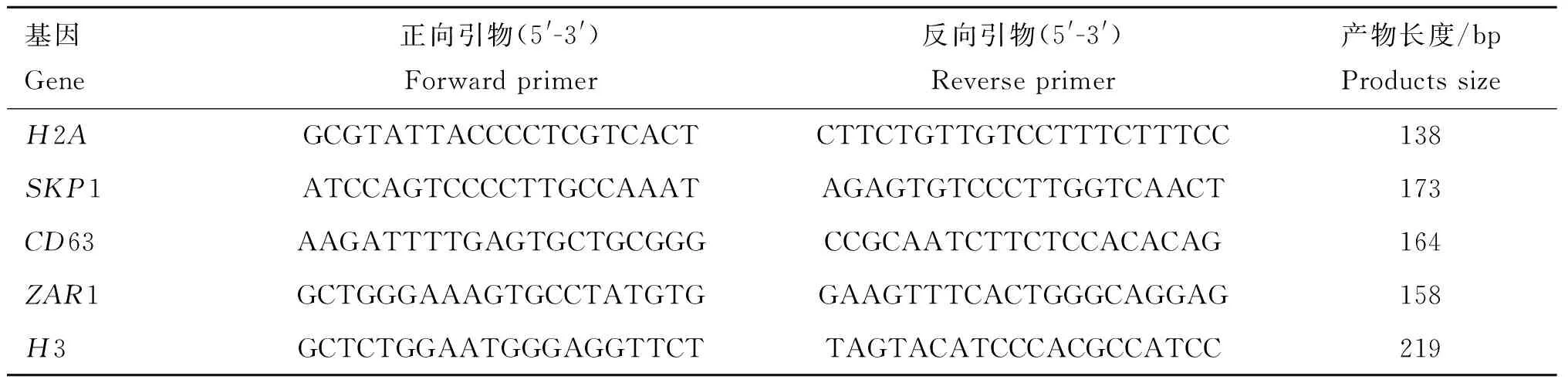
基因Gene正向引物(5'-3')Forwardprimer反向引物(5'-3')Reverseprimer产物长度/bpProductssizeH2AGCGTATTACCCCTCGTCACTCTTCTGTTGTCCTTTCTTTCC138SKP1ATCCAGTCCCCTTGCCAAATAGAGTGTCCCTTGGTCAACT173CD63AAGATTTTGAGTGCTGCGGGCCGCAATCTTCTCCACACAG164ZAR1GCTGGGAAAGTGCCTATGTGGAAGTTTCACTGGGCAGGAG158H3GCTCTGGAATGGGAGGTTCTTAGTACATCCCACGCCATCC219
2 结 果
2.1 体外受精效果与测序质量分析
通过3个批次的重复试验,IVF/IVC后杂种胚胎的平均卵裂率为78.4%,囊胚率为36.3%,说明采用的IVF/IVC体系效果好,测序结果能代表正常胚胎转录组水平。通过分析碱基的组成和质量值分布可以控制原始数据的质量。本研究中2-、4-、8-细胞、桑椹胚和囊胚 5 个发育时期的犏牛胚胎 RNA 样本测序所得原始数据的碱基质量分布和碱基含量分布良好,且Q30>85%,说明测序质量较好。原始数据经过滤后获得47 791 850(8-细胞)~63 332 216(4-细胞)条 Clean Reads。将 Clean Reads与牦牛基因组比对,有80.00%(囊胚)~91.13%(2-细胞)的序列可被牦牛基因组注释,比对到基因组多位的Reads比例为3.86%~5.24%(表2)。
表2测序和比对结果概述
Table2TheresultsofRNA-seqandmappingtothereferencegenome

比对数据Mappeddata2-细胞2-cell4-细胞4-cell8-细胞8-cell桑椹胚Morula囊胚Blastocyst干净数据序列数CleanReads6088044663332216477918505492401849296682比对上基因组的序列数MappedReads5547920556339924430873724899907139435615比对上基因组的Reads比例/%Mappingrate91.1388.9690.1689.2180.00未比对到基因组的Reads数UnmappedReads54012416992292470447859249479861067比对到基因组多位点的Reads数Multi-mapReads25187072643767221959321191722583620比对到基因组多位点的Reads比例/%Multi-maprate4.144.174.643.865.24
2.2 犏牛胚胎的基因表达的分析
采用RPKM法[14]计算基因表达量的结果表明,犏牛胚胎从2-细胞期发育到囊胚期每个阶段有9 604(桑椹胚)~15 400(4-细胞)个表达基因;共有19 072个基因表达,其中7 785个为共表达基因,11 287个为阶段特异表达基因。4-细胞开始表达的基因数目最多(1 606个),而桑椹胚开始表达的基因数目最少(148个)。随着胚胎发育的进行,BMP15、GDF9和ZP4等母源基因的表达量降低,而ATP5B、UBEA3和SNURF等胚胎基因的开始表达且表达量不断增加(图1)。以|log2Ratio|≥1和Q<0.05为筛选条件筛选不同发育阶段间的DEGs,结果表明,桑椹胚~囊胚的DEGs最多(10 298个),而2-~4-细胞胚的DEGs最少(3 690个)(表3)。

图1 母源基因(A)和胚胎基因(B)的表达变化趋势Fig.1 The expression tendency of maternal genes (A) and embryonic genes (B)
表3犏牛胚胎在不同发育阶段的基因表达分析统计表
Table3Geneexpressionatthedifferentdevelopmentalstagesofcrossbredembryosoftheyak

项目Item2-细胞2-cell4-细胞4-cell8-细胞8-cell桑椹胚Morula囊胚Blastocyst表达的基因数Expressedgenes144911540013792960411860新转录本Noveltranscripts496564747441519首次表达的基因数1sttimeexpressedgenes-1606390148535差异表达基因数Diferetiallyexpressedgenes36906332896510298上调基因数Up-regulatedgenes2861205829085747下调基因数Down-regulatedgenes829427460574551
2.3 差异表达基因的 qRT-PCR验证
为了进一步验证转录组分析结果的可靠性,选出4个基因,采用qRT-PCR 技术进行表达验证,结果表明,RNA-seq测序结果与qRT-PCR的结果基本一致(图 2)。说明本研究利用RNA-seq技术分析基因的结果是可靠的。

图2 差异表达基因的qRT-PCR与RNA-seq结果的对比Fig.2 Comparison of the differentially expressed genes between qRT-PCR and RNA-seq
2.4 差异表达基因的 GO 分类和显著性富集分析
GO分析显示(图3),从2-~4-细胞、4-~8-细胞、8-细胞~桑椹胚及桑椹胚~囊胚4个发育阶段分别有3 461(占93.8%)、5 972(占94.3%)、8 438(占94.1%)和9 749(占94.7%)个DEGs得到归类注释,都涉及生物过程(Biological process,BP)、细胞组分(Cellular component,CC)和分子功能(Molecular function,MF)3大类67个二级条目。在BP分类中有23个二级条目,其中占比例最大的4个二级条目在4个发育阶段相同,从高到低依次为细胞过程(Cellular process)、单有机体过程(Single-organism process)、生物调节(Biological regulation)和代谢过程(Metabolic process)。但是,各发育阶段第5个二级条目开始出现差异。在CC分类中有22个二级条目,二级条目中细胞部分(Cell part)占比例最多,其次为细胞器 (Organelle)及细胞器部件(Organelle part),前10个二级条目的排序在各发育阶段相同,但从第11个二级条目开始排序在不同发育阶段出现差异。在MF分类中有22个二级条目,占比例最大的2个二级条目在4个发育阶段完全相同,依次是绑定分子(Binding)所占比例最多和催化活性(Catalytic activity),但从第3个二级条目开始排序在不同发育阶段出现差异。
2.5 差异表达基因的代谢路径分析
犏牛胚胎发育过程中差异表达基因KEGG分析表明,2-~4-细胞涉及到300条通路(Pathways),4-~8-细胞有302条通路,8-细胞~桑椹胚有314条通路,桑椹胚~囊胚有314条通路,各发育阶段的富集前5条通路(Top 5 pathways)如表4所示,每个阶段的通路种类及富集性都有差异。2-~4-细胞、4-~8-细胞、8-细胞~桑椹胚和桑椹胚~囊胚4个阶段分别有15、0、18和1条通路显著富集(Q<0.05)。
3 讨 论
本研究利用RNA-seq技术从转录组学的角度揭示了犏牛早期胚胎发育机制, 为完善犏牛胚胎体外生产,提高犏牛胚胎体外生产效率具有重要价值。普通牛从2-细胞~囊胚,每个胚胎的总RNA只有200~2 000 pg[17]。犏牛胚胎应该与此相近,这样微量RNA不能满足转录组测序文库的构建及高通量测序的基本要求,因此,分别提取每个发育阶段的犏牛早期胚胎总RNA后,使用Smart-Seq2扩增技术对样本进行富集并构建测序文库[9],再应用RNA高通量测序技术对其进行高通量测序分析。通过从扩增产物cDNA样品片段分布、碱基的组成和质量值分布、Q30、饱和量、qRT-PCR验证等全面分析的结果表明,以犏牛胚胎微量RNA作为模板,采用Smart-Seq2扩增技术与RNA-seq技术相结合开展犏牛胚胎转录组高通量测序分析,结果可靠。
犏牛胚胎从2-细胞发育到囊胚每个阶段有9 604(桑椹胚)~15 400(4-细胞胚)个表达基因。与普通牛[18-19]和牦牛的胚胎[20]比较,犏牛桑椹胚的转录本少。这可能是由于犏牛胚胎为种间杂种胚胎,胚胎的遗传信息决定了在这一阶段不可避免地出现“发育阻滞”现象,也有可能是胚胎体外培养条件的不完善引起“体外发育阻滞”[21]。可以通过对体内发育的犏牛胚胎的转录组进一步深入研究,探明原因。如果确认为“体外发育阻滞”,则可以通过优化胚胎培养条件得到改善。犏牛胚胎从2-细胞发育到囊胚共有19 072个基因表达,其中7 785个为共表达基因,11 287个为阶段特异表达基因。4-细胞开始表达的基因数目最多(1 606个),而桑椹胚开始表达的基因数目最少(148个),显示胚胎发育是众多基因表达在时间和空间上的联系和配合共同作用的结果,不是单个基因控制的。但是,有些基因在胚胎发育的特异阶段发挥关键的调控作用,例如,NANOG能调控合子基因组的激活, 缺乏NANOG的胚胎合子基因组激活率低,发育受阻[22]。本研究发现,与普通牛[7,18-19]和牦牛胚胎[20]一样,犏牛胚胎NANOG基因在8-细胞开始表达。CLDN4是Claudin蛋白家族的成员之一,妇女黄体期子宫内膜CLDN4 mRNA的表达量与妊娠相关[23],囊胚中基因的表达与囊胚的发育与附植相关[24]。推测CLDN4基因在犏牛囊胚中开始表达,可能在囊胚发育和妊娠的建立中发挥重要作用。早期胚胎的发育受到由来自卵母细胞发生及成熟期间合成的大量转录本和蛋白质的调控[7,18-19,25],但随着发育的进行,母源转录本和蛋白质降解,而胚胎基因组激活(Embryonic genome activation, EGA),发育从母型调控向胚胎型调控的过渡(Maternal-to-embryonic transition, MET)。本研究发现,随着胚胎发育的进行,犏牛胚胎的BMP15、GDF9、ZP4和ZP3等母源基因表达量逐渐减少,而ATP5B、UBEA3和SNURF等胚胎基因的开始表达且表达量不断增加(图2),这与在其它物种的研究结果一致[7,18-19,26]。


A.2-~4-细胞;B.4-~8-细胞;C.8-细胞~桑椹胚;D.桑椹胚~囊胚A.2-cell-4-cell; B.4-cell-8-cell; C.8-cell-morula; D.Morula-blastocyst图3 犏牛早期胚胎发育过程中的差异表达基因GO分类注释图Fig.3 Gene Ontology classification of the DEGs of 4 developmental stages of crossbred embryos of the yak
表4牦牛胚胎发育过程中差异表达基因富集前5(Top5)KEGG通路
Table4Top5ofenrichedKEGGpathwaysofDEGsduringdevelopmentofcrossbredembryosoftheyak

发育阶段DevelopmentstageKEGG通路KEGGpathway上调基因Up-regulatedgenes下调基因Down-regulatedgenesQ-值Q-value2-细胞~4-细胞2-cell-4-cell泛素介导调节Ubiquitinmediatedproteolysis1020.0005内质网加工蛋白Proteinprocessinginendoplasmicreticulum1920.0081细胞周期Cellcycle1410.0152剪切体Spliceosome1410.0176RNA转运RNAtransport1630.02364-细胞~8-细胞4-cell-8-cell基底细胞癌Basalcellcarcinoma7190.0572RNA转运RNAtransport10300.0572系统性红斑狼疮Systemiclupuserythematosus33220.0798神经活性配体-受体互作Neuroactiveligand-receptorinteraction20480.0798剪切体Spliceosome15190.07988-细胞~桑椹胚8-cell-morula核糖体Ribosome13561.63E-13氧化磷酸化机制Oxidativephosphorylation21311.82E-06亨廷顿氏病Huntington'sdisease31531.88E-06RNA转运RNAtransport18461.52E-05神经活性配体-受体互作Neuroactiveligand-receptorinteraction18538.03E-05桑椹胚~囊胚Morula-blastocystRNA转运RNAtransport61340.0387核糖体Ribosome17340.1013内质网加工蛋白Proteinprocessinginendoplasmicreticulum72390.1799核苷酸切除修复Nucleotideexcisionrepair17100.1799氨基酸合成Biosynthesisofaminoacids29100.1799
以|log2Ratio|≥1和Q<0.05为筛选条件筛选出桑椹胚~囊胚的DEGs最多(10 298个),而2-~4-细胞胚的DEGs最少(3 690个)(表3)。GO分析显示,从2-~4-细胞、4-~8-细胞、8-细胞~桑椹胚及桑椹胚~囊胚4个发育阶段分别有3 461、5 972、8 438和9 749个DEGs(占DEGs数目的94%左右)得到归类注释,都涉及生物过程(BP)、细胞组分(CC)和分子功能(MF)3大类67个二级条目(图3)。在BP分类中,比例最大的4个二级条目在4个发育阶段都相同,但各发育阶段第5个二级条目开始出现差异;在CC分类中前10个二级条目的排序在4个发育阶段都相同,但从第11个二级条目开始在不同发育阶段出现差异;在MF分类中,只有占比例最大的2个二级条目在4个发育阶段都相同,但从第3个二级条目开始排序在不同发育阶段出现差异。说明在犏牛胚胎发育过程中,胚胎细胞组分相对稳定,但细胞内分子过程和分子功能在不断地发生变化。例如,分子功能调节器(Molecular function regulator)具有调节基因产物活性的功能,它从2-~4-细胞和4-~8-细胞位列BF分类中的第5位上调到8-细胞~桑椹胚中的第4位和桑椹胚~囊胚中的第3位。
本研究发现, 犏牛早期胚胎发育过程中DEGs富集的主要通路有剪接体(Spliceosome)、神经活性配体-受体互作(Neuroactive ligand-receptor interaction)、细胞因子及其受体相互作用(Cytokine-cytokine receptor interactions)、泛素介导的蛋白水解(Ubiquitin mediated proteolysis)、RNA转运(RNA transport)、核糖体(Ribosome)等(表4),这与在其它动物的发现基本一致[22,27-28],说明这些通路在犏牛胚胎发育过程中发挥重要调控作用。
4 结 论
本研究首次利用RNA-Seq技术从转录组学揭示犏牛早期胚胎发育机制, 获得了众多差异基因和有关通路的富集,为完善犏牛胚胎体外生产技术提供理论基础。
[1] WIENER G, HAN J L, LONG R J. The yak[M]. 2nd ed. Bangkok: Regional Office for Asia and the Pacific of the Food and Agriculture Organization of the United Nations, 2003: 1-13.
[2] ZI X D, HE S M, LU H, et al. Induction of estrus in suckled female yaks (Bosgrunniens) and synchronization of ovulation in the non-sucklers for timed artificial insemination using progesterone treatments and Co-Synch regimens[J].AnimReprodSci, 2006, 92(1-2): 183-192.
[3] ZI X D, YIN R H, CHEN S W, et al. Developmental competence of embryos derived from reciprocalinvitrofertilization between yak (Bosgrunniens) and cattle (Bostaurus)[J].JReprodDev, 2009, 55(5): 480-483.
[4] XIAO X, ZI X D, NIU H R, et al. Effect of addition of FSH, LH and proteasome inhibitor MG132 toinvitromaturation medium on the developmental competence of yak (Bosgrunniens) oocytes[J].ReprodBiolEndocrinol, 2014, 12: 30.
[5] YAN L Y, YANG M Y, GUO H S, et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells[J].NatStructMolBiol, 2013, 20(9): 1131-1139.
[6] CAO S Y, HAN J Y, WU J, et al. Specific gene-regulation networks during the pre-implantation development of the pig embryo as revealed by deep sequencing[J].BMCGenomics, 2014, 15: 4.
[7] GRAF A, KREBS S, HEININEN-BROWN M, et al. Genome activation in bovine embryos: review of the literature and new insights from RNA sequencing experiments[J].AnimReprodSci, 2014, 149(1-2): 46-58.
[8] JIANG Z L, SUN J W, DONG H, et al. Transcriptional profiles of bovineinvivopre-implantation development[J].BMCGenomics, 2014, 15: 756.
[9] PICELLI S, BJÖRKLUND A K, FARIDANI O R, et al. Smart-Seq2 for sensitive full-length transcriptome profiling in single cells[J].NatMethods, 2013, 10(11): 1096-1098.
[10] 郑 杰, 蒲思颖, 杨远潇,等.基于高通量测序的犏牛囊胚玻璃化冷冻损伤机制研究[J].畜牧兽医学报,2017,48(10):1871-1881.
ZHENG Jie, PU Si-ying, YANG Yuan-xiao, et al.Exploring Mechanism for Vitrification Damage of the Cross-bred Blastocysts of the Yak via High-throughput Sequencing[J].ActaVeterinariaetZootechnicaSinica, 2017, 48(10): 1871-1881. (in Chinese)
[11] QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J].NatGenet, 2012, 44(8): 946-949.
[12] 石 仙, 熊显荣, 兰道亮, 等. 糖对牦牛卵母细胞体外成熟及其发育能力的影响[J]. 中国畜牧兽医, 2017, 44(1): 155-160.
SHI X, XIONG X R, LAN D L, et al. Effect of sugar oninvitromaturation and developmental competence of yak oocytes[J].ChinaAnimalHusbandry&VeterinaryMedicine, 2017, 44(1): 155-160. (in Chinese)
[13] TRAPNELL C, PACHTER L, SALZBERG S L. TopHat: discovering splice junctions with RNA-seq[J].Bioinformatics, 2009, 25(9): 1105-1111.
[14] WANGER G P, KIN K, LYNCH V J. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples[J].TheoryBiosci, 2012, 131(4): 281-285.
[15] WANG L K, FENG Z X, WANG X, et al. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data[J].Bioinformatics, 2010, 26(1): 136-138.
[16] BENJAMINI Y, HOCHBERG Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing[J].JRStatistSocB, 1995, 57(1): 289-300.
[17] GILBERT I, SCANTLAND S, SYLVESTRE E L, et al. The dynamics of gene products fluctuation during bovine pre-hatching development[J].MolReprodDev, 2009, 76(8): 762-772.
[18] KUES W A, SUDHEER S, HERRMANN D, et al. Genome-wide expression profiling reveals distinct clusters of transcriptional regulation during bovine preimplantation developmentinvivo[J].ProcNatlAcadSciUSA, 2008, 105(50): 19768-19773.
[19] GRAF A, KREBS S, ZAKHARTCHENKO V, et al. Fine mapping of genome activation in bovine embryos by RNA sequencing[J].ProcNatlAcadSciUSA, 2014, 111(11): 4139-4144.
[20] 罗 斌. 牦牛体外受精胚胎的发育转录组研究[D]. 成都: 西南民族大学, 2016.
LUO B. Transcriptomic analysis of the regulation of yak embryonic developmentinvitro[D]. Chengdu: Southwest University for Nationalities, 2016. (in Chinese)
[21] GARCIA S M, MARINHO L S R, LUNARDELLI P A, et al. Developmental block and programmed cell death inBosindicusembryos: effects of protein supplementation source and developmental kinetics[J].PLoSOne, 2015, 10(3): e0119463.
[22] LEE M T, BONNEAU A R, TAKACS C M, et al. Nanog, Pou5f1 and SoxB1 activate zygotic gene expression during the maternal-to-zygotic transition[J].Nature, 2013, 503(7476): 360-364.
[23] SERAFINI P C, SILVA I D, SMITH G D, et al. Endometrial claudin-4 and leukemia inhibitory factor are associated with assisted reproduction outcome[J].ReprodBiolEndocrinol, 2009, 7: 30.
[24] MUNCH E M, SPARKS A E, GONZALEZ BOSQUET J, et al. Differentially expressed genes in preimplantation human embryos: potential candidate genes for blastocyst formation and implantation[J].JAssistReprodGenet, 2016, 33(8): 1017-1025.
[25] TADROS W, LIPSHITZ H D. The maternal-to-zygotic transition: a play in two acts[J].Development, 2009, 136(18): 3033-3042.
[26] BRAUDE P, BOLTON V, MOORE S. Human gene expression first occurs between the four- and eight-cell stages of preimplantation development[J].Nature, 1988, 332(6163): 459-461.
[27] ZUO Y C, SU G H, WANG S S, et al. Exploring timing activation of functional pathway based on differential co-expression analysis in preimplantation embryogenesis[J].Oncotarget, 2016, 7(45): 74120-74131.
[28] RAUWERDA H, PAGANO J F B, DE LEEUW W C, et al. Transcriptome dynamics in early zebrafish embryogenesis determined by high-resolution time course analysis of 180 successive, individual zebrafish embryos[J].BMCGenomics, 2017, 18: 287.
