利用MALDI-TOF MS鉴定芽苗菜中解鸟氨酸拉乌尔菌和肺炎克雷伯菌的研究
2018-01-25代晓航郭灵安
魏 超,代晓航,郭灵安,刘 炜
(四川省农科院分析测试中心,四川 成都 610066)
基质解析电离飞行时间质谱(MALDI-TOF MS )是一种软电离有机质谱,通过基质吸收激光的能量,并将部分能量传递给样品,帮助样品离子化,根据其质量与电荷的比(质核比m/z)进行检测[1]。MALDI-TOF MS通过基质辅助电离微生物蛋白质,使其片段化成大分子多肽,而获得肽指纹图谱进行检测,因为微生物蛋白质具有特异性和保守性,不受培养基和培养条件影响,所以其多肽指纹图谱也具有特异性和保守性。根据得到的不同微生物的特征性谱峰,通过检索特征性质谱峰数据库或与已知微生物的质谱峰比较,达到对微生物进行快速检测、鉴定和分型的目的[2]。从分子生物学的角度看,核酸决定蛋白的表达,但蛋白质的丰度受其稳定性、翻译效率和其他蛋白质的丰度调控以及后修饰等影响,因此,基于蛋白质水平的微生物分析相比核酸水平进行的分析可获得更多的微生物信息[1,3-4]。解鸟氨酸拉乌尔菌(Raoultellaornithinolytica)因与克雷伯菌属在基因水平的差异被从克雷伯菌属中的解鸟氨酸克雷伯菌分离出来[5-6],此菌可引起溶血症,在患者的痰液、尿液、胆汁中均有检出[7-9],其毒力基因blaKPC-2与肺炎克雷伯菌相同[10],此外解鸟氨酸拉乌尔菌的一些分离菌次生代谢产物具有抗肿瘤活性,常被用作工程菌株[11]。肺炎克雷伯菌(Klebsiellapneumoniae)在自然界中广泛分布于土壤、水、果蔬,也见于人和动物的肠道中,该菌常被分离于医学临床标本中,在适宜或特定的条件下,能引起人感染发病,致病作用较广泛[5],此外由于拉乌尔菌属与克雷伯菌属特征生化反应比较接近,所以在商业化鉴定系统中常发生误判的情况[12-13]。
本实验采用MALDI-TOF MS方法对芽苗菜中解鸟氨酸拉乌尔菌和肺炎克雷伯菌进行鉴定和蛋白质特征性分析,目的在于优化数据库使其更适合农产品非常见微生物的检测与危害性评估。
1 实验部分
1.1 仪器、试剂与材料
MALDI-TOF-MS(岛津,AXIMA Confidence),微生物鉴定仪(生物梅里埃公司,ATB),生化培养箱(宁波东南生物仪器有限公司,DHP-9082),PCR扩增仪(赛默飞世尔科技(中国)有限公司,SimpliAmp)。
蛋白质缓冲水(BPW)、麦康凯琼脂(MAC)、伊红美蓝琼脂(EMB)(广东环凯微生物科技有限公司);乙醇、乙腈(色谱纯,Fisher Scientific);甲酸(色谱纯,Tedia公司);α-氰基-4-羟基肉桂酸(CHCA)(Sigma公司 );微生物鉴定生化试剂条(生物梅里埃公司,ID32E),实验用水为去离子水。
芽苗菜(金丝柳苗,松柳苗)30份样品来自成都大型超市,超市分布覆盖大成都范围。
1.2 实验方法
1.2.1微生物的盲筛称取25 g样品,225 mL BPW均质后36 ℃培养16 h,增菌液划线于EMB和MAC培养基36 ℃培养24 h,每份样品每种培养条件分别挑取6个单菌落用于进一步纯化,36 ℃培养36 h后进行质谱鉴定。
1.2.2分离微生物的质谱鉴定样品前处理:在1.0 mL乙醇水溶液(体积比70∶30)中加入10 μL接种环移取适当菌量,振荡30 s后离心1 min,弃溶液,加入0.75 mL乙酸-水溶液(体积比5∶25)振荡20 s,于25 ℃、40 kHz超声10 min,加入0.75 mL乙腈振荡均匀,4 000 r/min离心1 min取上清液1 μL点至靶板,挥干加入1 μL CHCA,挥干。
质谱条件:电喷雾离子源(ESI),氮气激光器,固定聚焦337 nm,检测器线性模式-电子倍增管(Multiple dynode),激光束能频75~80 Hz,收集范围2 000~20 000(m/z),每个样品200次收集峰叠加,校准品ATCC 8739大肠杆菌,每16个样品校正2次,分离微生物获得的质谱图数据导入MALDI-TOF/SARAMIS微生物鉴定系统与其中的超级谱图进行多重比较分析,获得微生物的质谱鉴定结果。
1.2.3微生物标识峰优化及相关性分析收集质谱图谱导入MALDI-TOF/SARAMIS微生物鉴定系统,采用SARAMIS Premium软件中的Superpectrum和Taxonomy进行标记峰足迹图绘制、标识峰整理与微生物同源性分析。
1.2.4微生物的生化鉴定“1.2.1”中EMB和MAC纯化单菌落划线到营养琼脂培养基上,36 ℃培养24 h,挑取营养琼脂培养菌落至0.85%生理盐水比浊管,振荡分散,将菌液调至0.5麦氏单位,每孔55 μL分装于ID32E 试剂条试剂杯中,36 ℃培养24 h,ATB系统自动读取试剂条信息并给出最终结果。
1.2.5微生物16srDNA的相关分析25 μL PCR反应体系,扩增引物27f:5′-AGAGTTTGATCCTGGCTCAG-3′和1492r:5′-TACGGCTACCTTGTTACGACTT-3′,PCR反应条件为预变性95 ℃ 5 min;94 ℃ 30 s、60 ℃ 30 s、72 ℃ 30 s,共进行35个循环;延伸72 ℃ 7 min。PCR产物进行双向测序并拼接,获得序列结果并提交NCBI比对,采用MAGA7.0软件最大简约法(Maximum parsimony)进行序列分析构建发育树。
2 结果与讨论
2.1 解鸟氨酸拉乌尔菌及肺炎克雷伯菌的分离与鉴定
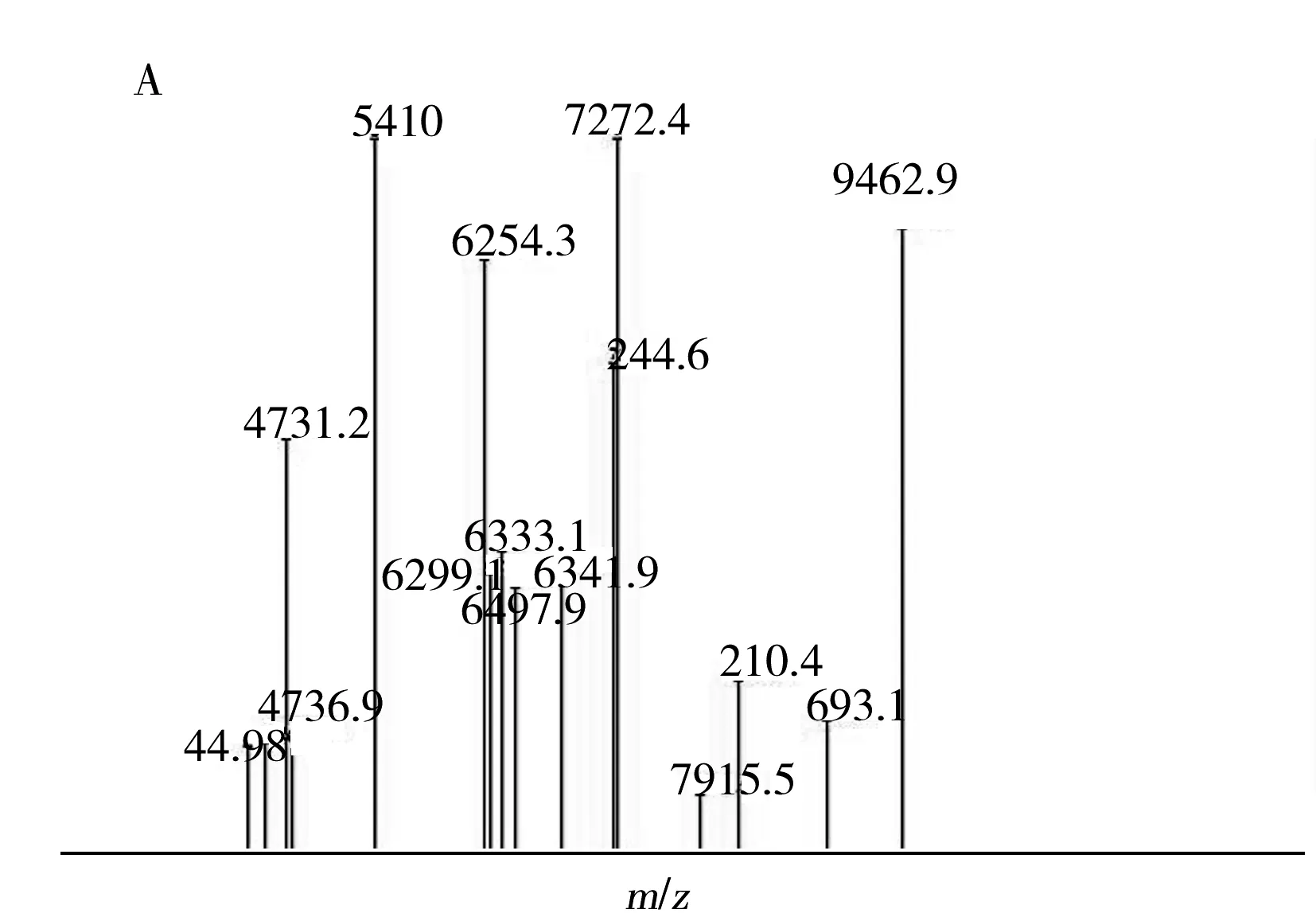
采用两种分离培养基在30份样品中分离得到解鸟氨酸拉乌尔菌12株(其中EMB培养基2株,MAC培养基10株,来自10份样品),解鸟氨酸拉乌尔菌的检出率为33.3%。肺炎克雷伯菌分离得到15株(其中MAC分离得到10株,EMB分离5株,来自13份样品),分离率43.3%,两种微生物的质谱图见图1,质谱对两种微生物获得很好的鉴定结果。取得有效结果的两种微生物的生化图谱见表1,部分质谱鉴定为解鸟氨酸拉乌尔菌的微生物生化结果为克雷伯菌(Klebsiellaspp)。
因生化反应的相似拉乌尔菌属经常被误认为克雷伯菌属细菌[5,13]。已有研究中芽苗菜中解鸟氨酸拉乌尔菌和肺炎克雷伯菌检出率较高,由生化图谱可以看出两种微生物在32个生化反应中大部分表现一致(表1),仅鸟氨酸脱羧酶和天门冬素芳胺酶生化反应不一致,其中鸟氨酸脱羧酶阳性为鉴定拉乌尔菌属内解鸟氨酸种的特征性反应,天门冬素芳胺酶实验在拉乌尔菌属其余种中也可能表现为阴性。虽然本实验对两种微生物的生化鉴定取得了有效的结果,但部分质谱鉴定解鸟氨酸拉乌尔菌的微生物生化鉴定结果有一定的差异,获得结果为克雷伯菌。实验结果表明两种微生物较难区分,与已有文献一致。
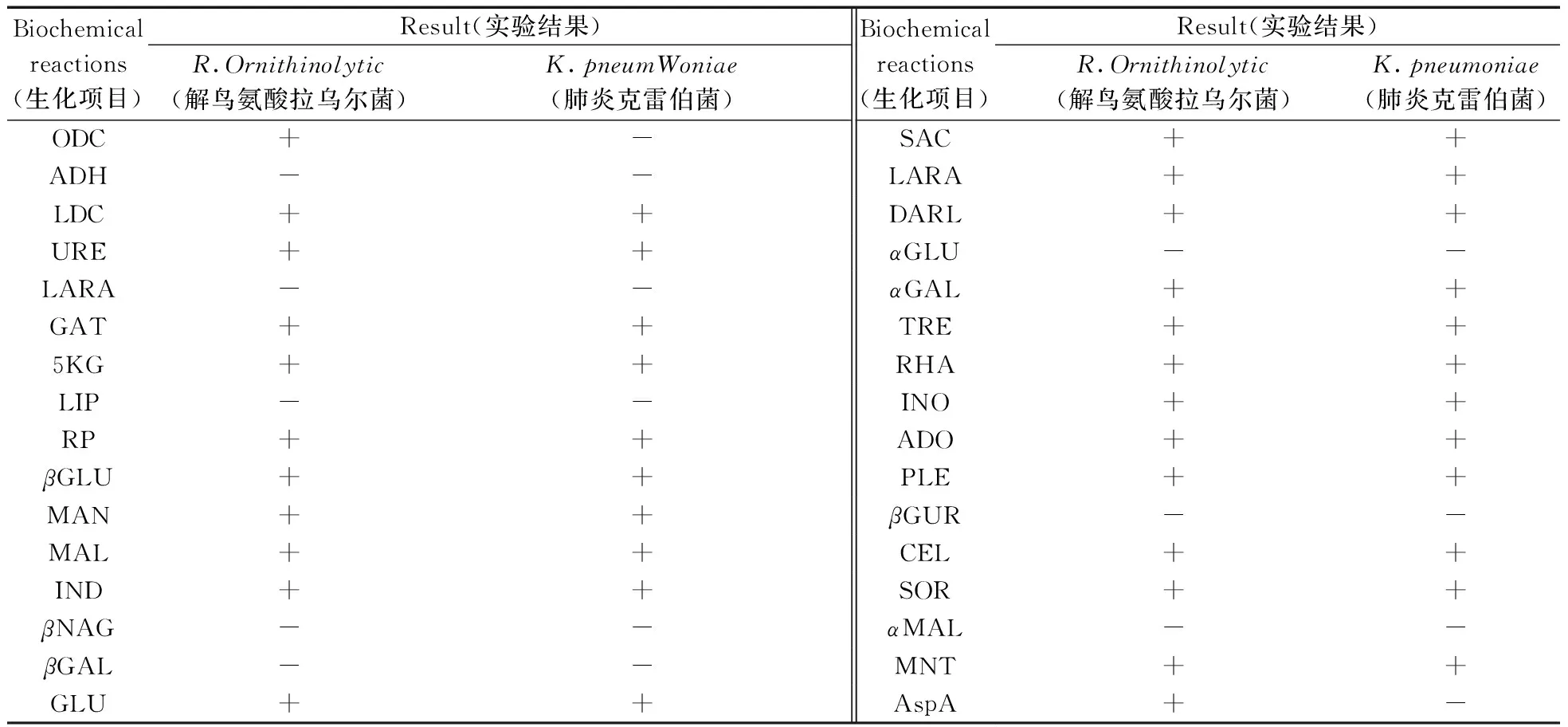
表1 解鸟氨酸拉乌尔菌和肺炎克雷伯菌的生化图谱Table 1 Biochemistry map of K.pneumoniae and R.ornithinolytica
ODC(鸟氨酸脱羧酶),AHD( 精氨酸双水解酶),LDC(赖氨酸脱羧酶),URE(尿素酶),LARL(L-阿拉伯醇),GAT(d-半乳糖酸盐同化),5KG(5酮基·葡萄糖酸钠),LIP(酯酶),RP(酚红),βGLU(β-葡萄糖苷酶),MAL(d-麦芽糖),IND(吲哚),βNAG(β-氮乙酰葡萄糖胺),βGAL(半乳糖苷酶),GLU(d-葡萄糖),SAC(蔗糖),LARA(L-阿拉伯糖),DARL(D-阿拉伯醇),αGLU(α-葡萄糖苷酶),αGAL(α-半乳糖苷酶),TRE(d-海藻糖),RHA(L-鼠李糖),INO(肌醇),ADO(侧金盏花醇),PLE(古老糖),βGUR(β-葡萄糖醛缩酶),CEL(d-纤维二糖),SOR(d-山梨醇),αMAL(α-麦芽糖苷酶),MAN(d-甘露醇),MNT(丙二酸盐),AspA(L-天门冬素芳胺酶)
2.2 解鸟氨酸拉乌尔菌和肺炎克雷伯菌质谱特征峰的研究
足迹图可直观表现微生物质谱标识峰的位置(包括<0.08的相对偏差),芽苗菜中分离的12株解鸟氨酸拉乌尔菌以及肺炎克雷伯菌的标识峰足迹图显示,在小质核比(m/z<5 000)区域内标识峰比较密集,随着质核比增大,标识峰逐渐减少,但尽管鉴定为同种微生物,其标识峰的数目、质核比轨迹仍相差较多,即便数据库中的超级谱图间差距也比较大。



图1 微生物鉴定质谱图谱Fig 1 Microorganism mass spectra of MALDI-TOF MS A:R.ornithinolytica(解鸟氨酸拉乌尔菌),B:K.pneumoniae(肺炎克雷伯菌),C:Calibration E.coli ATCC 8739(标准大肠杆菌 ATCC8739)
对芽苗菜中分离的解鸟氨酸拉乌尔菌的全部标识峰进行拟合(包含<0.08的相对偏差),12株微生物共有18组质核比标识峰完全拟合(图2A),再添加4组超级谱图数据后,16组解鸟氨酸拉乌尔菌完全拟合的标识峰为16组(图2B),减少为质核比(m/z)5 599.6和5 618.3(包含<0.08的绝对偏差)的两组标识峰,这两组峰响应值较高可认为是芽苗菜分离菌种的特有谱峰,完全拟合的16组标识峰可认定为解鸟氨酸拉乌尔菌的特征峰,将16组峰上传至SARAMIS Premium数据系统,对其加权以提高系统对解鸟氨酸拉乌尔菌的分辨能力。
对分离的肺炎克雷伯菌的全部标识峰进行拟合(包含<0.08的相对偏差),15株微生物共有16组标识峰完全拟合(图3A),再添加4组超级谱图数据后,19组肺炎克雷伯菌完全拟合的标识峰有10组(图3B),减少的5组标识峰中有3组峰的响应值较低,剩余2组峰的响应值较高可认为是芽苗菜分离菌种特有谱峰,完全拟合的10组标识峰可认定为解鸟氨酸拉乌尔菌的特征峰,将其上传至SARAMIS Premium数据系统,对其加权以提高系统对解鸟氨酸拉乌尔菌的分辨能力。
质谱鉴定微生物是根据其特征峰拟合进行数据计算获得鉴定结果,数据库的容量和特征峰的权重决定鉴定结果的准确性,SARAMIS Premium是一个开放平台,本实验对生化相似的两种微生物进行特征峰整理和加权,还可针对更多样的农产品特征微生物进行研究。


图4 解鸟氨酸拉乌尔菌聚类分析树Fig.4 R.ornithinolytica cluster dendrogram by MALDI-TOF MSA:cluster dendrogram of 12 strains of R.ornithinolytica;B:cluster dendrogram of R.ornithinolytica with weighted feature peak(A:12株解鸟氨酸拉乌尔菌进行聚类分析;B:解鸟氨酸拉乌尔菌征峰加权后聚类分析图)
2.3 解鸟氨酸拉乌尔菌及肺炎克雷伯菌系统发育学的研究
对芽苗菜中分离的12株解鸟氨酸拉乌尔菌进行聚类分析(图4A),对其特征峰加权后的聚类分析结果见图4B,对比两张分析图,在特征峰加权后微生物的相关系数有所提高,两张图中一级分支基本一致,但更高级的分支和相对距离发生了变化。在图4B中,13和14号图谱为超级谱图库中同源样品,聚类分析结果也显示其相关系数>100%;1和6号为芽苗菜分离解鸟氨酸拉乌尔菌,聚类分析结果显示其相关系数>100%,经溯源来源于同份芽苗菜样品的不同培养基的分离。肺炎克雷伯菌的聚类分析树与特征峰加权后分析树与图4的分析结果相近,加权特征峰后相关系数也有所提高,一级分支基本不变,但高级分支和相关位置发生了一定变化,聚类结果显示一级分支的5组微生物,除2组数据库中同源微生物,另外3组6株肺炎克雷伯菌为芽苗菜中分离,经溯源来自于3份芽苗菜样品的不同培养基。可见MALDI-TOF MS方法可以用于微生物的溯源,但其溯源的准确性还需进一步优化SARAMIS Premium系统。
2.4 解鸟氨酸拉乌尔菌与肺炎克雷伯菌相似性的研究
由生化鉴定结果可以看出,两种微生物的生化反应非常接近,容易误判,利用SARAMIS Premium将两种微生物的质谱图谱进行相关分析(图5),两种微生物虽有部分相交但在三四级分支中仍可分辨。图5显示两种微生物的相关性仍较高,最后一级分支系数仍大于50%,与“2.3”聚类分析树中种内最后一级分支相关系数接近,可见两种微生物在质谱图中较接近但可分辨。

对两种微生物进行16SrDNA测序并建立相关分析系统发育树(图6),系统发育树可将两种微生物分于两个分支,虽然遗传距离很接近但仍有一定差异,两种微生物在基因水平上区分明显。

图6 解鸟氨酸拉乌尔菌和肺炎克雷伯菌的16srDNA相关分析树Fig.6 K.pneumoniae and R.ornithinolytica related analysis tree by 16srDNA1-10:R.ornithinolytica;11-23:K.pneumoniae
3 结 论
本文针对芽苗菜中分离率最高且相似程度较高的两种微生物解鸟氨酸拉乌尔菌和肺炎克雷伯菌进行了MALDI-TOF MS鉴定相关分析并优化了标识峰,实验结果表明,MALDI-TOF MS在农产品相似微生物鉴定上准确度较高,但其溯源和相关分析能力比传统16SrDNA 方法还有一定差距,对开放数据库系统ARAMIS Premium进行添加和优化是提高MALDI-TOF MS对农产品微生物检测和溯源准确性的主要方法。
[1] Richard J S.ProteinsandProteomics:ALaboraryManual.New York:Cold Spring Harbor Laboratory Press,2003:397-412.
[2] Smith H I,Hecker M.MicobialProteomics:FunctionalBiologyofWholeOrganism.John Wiley & Sons,2006:5-7,311-315.
[3] Tekippe M E,Burnham C A.EuropeanJournalofClinicalMicrobiology&InfectiousDiseases,2014,33(12):2163-2171.
[4] Luo Y,Siu G K,Yeung A S,Chen J H,Ho P L,Leung K W.JournalofMedicalMicrobiology,2015,64(1):18-24.
[5] Fang H,Chen C Z,Zhang X J.PathogenceBacteriaofEnterobacteriaceae.Beijing:China Agriculture Press(房海,陈翠珍,张晓军.肠杆菌科病源细菌.北京:中国农业科学技术出版社),2011.
[6] Eefje de J,Arjan S J,Nathalie S B,Rob J R.DiagnosticMicrobiology&InfectiousDisease,2013,75(4):431-433.
[7] García-Lozano T,Pascual Plá F J,Aznar O E.MedicinaClinica,2013,141(3):138-147.
[9] Seng P,Boushab B M,Romain F,Gouriet F,Bruder N,Martin C.InternationalJournalofInfectiousDiseases,2016,45:65-71.
[11] Fioka M J,Lewtak K,Rzymowska J,Grzywnowicz K,Huasstasiak M,Sofińskachmiel W.Pathogens&Disease,2013,69(1):49-61.
[12] Al-Hulu S M,Al-Charrakh A H,Al-Saadi M A K.Med.J.Babylon.,2009,7(4):42-47.
[13] Kanki M,Yoda T,Tsukamoto T,Shibata T.Applied&EnvironmentalMicrobiology,2002,68(7):3462-3468.
