分化抑制因子1基因在急性髓系白血病患者骨髓单个核细胞中的表达
2018-01-18姚欣妤周静东张婷娟林江张巍钱军
姚欣妤,周静东,张婷娟,林江,张巍,钱军
(1.江苏大学附属人民医院血液内科,江苏镇江212002;2.江苏大学医学院,江苏镇江212013;3.江苏大学附属人民医院中心实验室,江苏镇江212002)
急性髓系白血病(acutemyeloid leukemia,AML)是一种细胞遗传学和分子生物学异质性较大的恶性克隆性疾病,其特征在于造血干细胞分化阻滞和成熟障碍,导致大量异常细胞即白血病细胞积累继而导致造血功能衰竭[1]。目前已经证实细胞遗传异常和基因突变不仅与疾病发生有关,而且对于疾病预后判断也很重要[2-3]。除遗传学改变之外,基因表达异常也是AML中常见的分子事件,并且可能与疾病的预后密切相关[3]。
分化抑制因子(inhibitor of differentiation,ID)家族基因编码螺旋—环—螺旋(helix-loop-helix,HLH)蛋白,对促进细胞分化的碱性HLH转录因子起负调控作用[4]。ID1是ID基因的家族一员,已经被鉴定为潜在的原癌基因,在细胞增殖以及侵袭中起作用,并保护细胞免受药物诱导的凋亡[4]。多种实体瘤中ID1呈现过表达,并且与预后相关[4]。有少量研究涉及AML患者中ID1呈现过表达趋势,但其临床意义不尽相同[5-6],其过表达对AML患者预后的影响仍然存在争议。因此,本研究旨在探讨初发AML患者骨髓单个核细胞(bone marrow mononuclear cells,BMMNCs)中ID1表达的临床意义。
1 对象与方法
1.1 研究对象
153例初诊 AML患者均为2005年10月至2016年7月在江苏大学附属人民医院血液科治疗案例。AML患者的具体临床参数见表1。诊断分型按FAB和2008版WHO标准[7-8]。治疗方案参照本课题组前期报道[9]。
1.2 BMMNCs分离、RNA提取和逆转录
所有AML患者行骨髓穿刺时抽取10 mL血,使用Ficoll密度梯度离心法分离 BMMNCs。总 RNA应用TRIzol(美国Invitrogen公司)提取,并逆转录为cDNA[10]。逆转录总反应体系为 40μL,含总 RNA(2μg)、随机引物(4μL×10μmol/L,日本 TaKaRa公司)、M-MLV逆转录酶(200 U,美国Thermo Fisher Scientific公司)、dNTPs(4μL×10 mmol/L)和 RNA酶抑制剂(25 U,美国 Thermo Fisher Scientific公司)。逆转录反应条件为25℃10 min、42℃60 min。
1.3 实时荧光定量PCR检测ID1表达
目的基因ID1引物序列为5′-CTCAGCACCCTCAACGG-3′(上游)、5′-GATCGGTCTTGTTCTCCCTC-3′(下游);选取ABL为内参基因,引物序列为5′-TCCTCCAGCTGTTATCTGGAAGA-3′(上 游)、5′-TCCAACGAGCGGCTTCAC-3′(下游),引物由上海华大基因科技有限公司合成。PCR反应体系共20μL,含10μL预染液(AceQ qPCR SYBR Green Master Mix,美国 Vazyme公司)、0.4μL ROX参比染料 1(美国Vazyme公司)、0.8μL上下游引物及20 ng cDNA。PCR反应在ABI 7300扩增仪(美国ABI公司)上进行,反应条件为95℃ 5 min(预变性)、95℃ 10 s(变性)、62℃ 30 s(退火)、72℃ 30 s(延伸)、80℃30 s(收集荧光),共35个循环;熔解曲线程序为95℃ 15 s、60℃ 60 s、95℃ 15 s、60℃ 15 s。每次扩增反应均含阳性对照(重组ID1质粒)和阴性对照(双蒸水)。ID1基因表达相对定量采用2-△△CT计算。
1.4 统计学分析
应用SPSS 20.0软件和GraphPad Prism 5软件进行统计学处理。两组连续性变量间的差异分析使用Mann-WhitneyU检验,分类变量之间的差异比较采用卡方检验或Fisher确切概率法,生存分析采用Kaplan-Meier法;以P<0.05为差异具有统计学意义。
2 结果
2.1 ID1表达与AML患者临床参数的关系
ID1基因在AML患者中的表达范围为0.000~3.536(中位为0.030)。以中位数值为界将AML患者分为高表达组和低表达组,两组常见的临床参数比较见表1。两组患者性别、外周血小板计数以及常见基因突变间无明显差异(P>0.05),外周血白细胞计数、血红蛋白含量、FAB分型、WHO分型、核型分组及核型危险程度分组趋近统计学差异(P<0.10)。然而,ID1高表达患者年龄以及骨髓原始细胞比例显著高于ID1低表达患者(P=0.033和0.035)。在 FAB、WHO分型及核型分组中,ID1高表达在 FAB-M3/WHO-t(15;17)中发生频率较低(29%,10/34),而在剩余其他类型中发生频率为56%(67/119),两者之间差异有统计学意义(χ2=7.649,P=0.007)。
2.2 ID1表达与AML患者预后的关系
2.2.1ID1表达与患者化疗反应之间的关系 经1~2个疗程诱导化疗后,有随访资料患者131例,ID1高表达患者完全缓解率显著低于ID1低表达患者(P<0.01,表1)。同时,定量分析发现经1~2个疗程诱导化疗获得完全缓解的AML患者初诊时ID1表达水平显著低于未获得完全缓解的AML患者(U=1408.0,P=0.001,图1)。
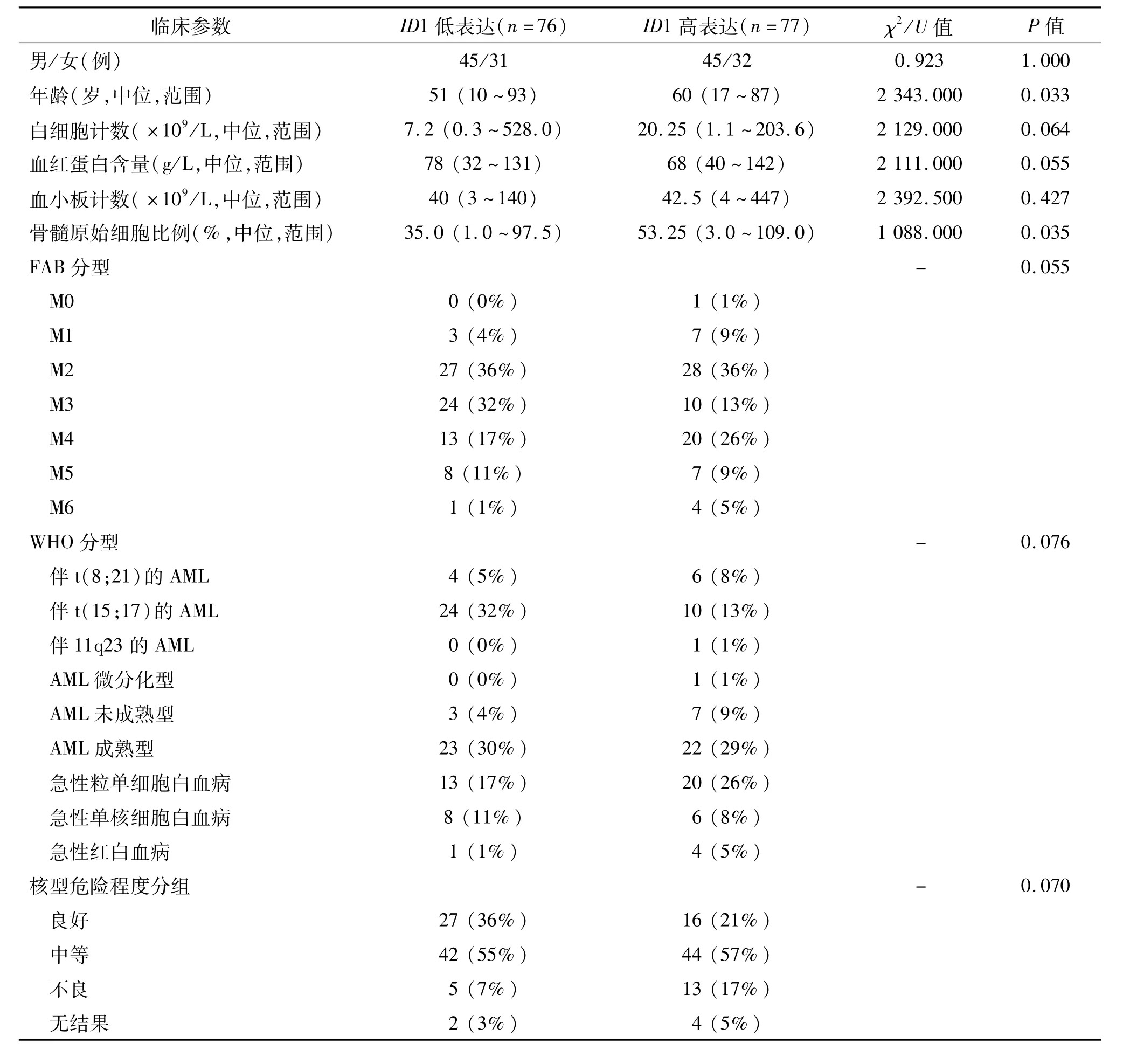
表1 ID1表达与AM L患者临床参数的关系
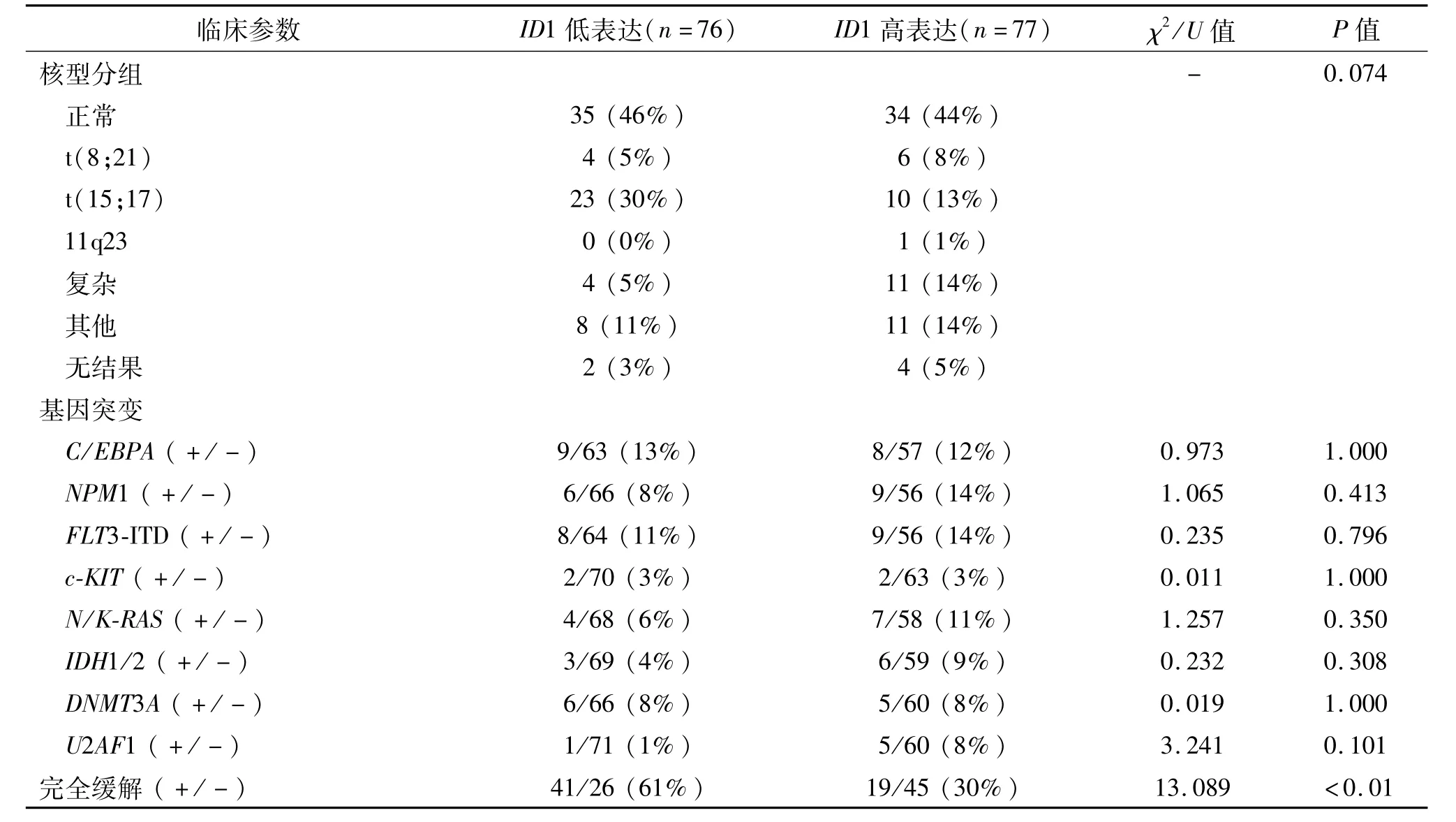
续表1
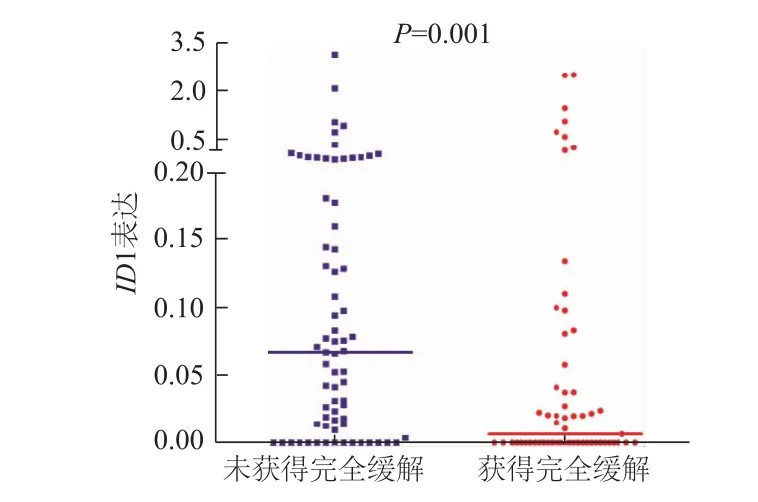
图1 获得完全缓解以及未获得完全缓解的AML患者初诊时ID1表达
2.2.2ID1表达与患者总体生存时间的关系ID1高表达患者的总体生存时间明显低于ID1低表达患者(中位生存时间分别为5个月和14个月,P=0.002)。在非M3患者中,ID1高表达患者的总体生存时间也明显低于ID1低表达患者(中位生存时间分别为4个月和8个月,P=0.008)。在正常核型患者中,ID1高表达患者的总体生存时间亦有明显的趋势低于ID1低表达患者(中位生存时间分别为6个月和12个月,P=0.050)。见图2。
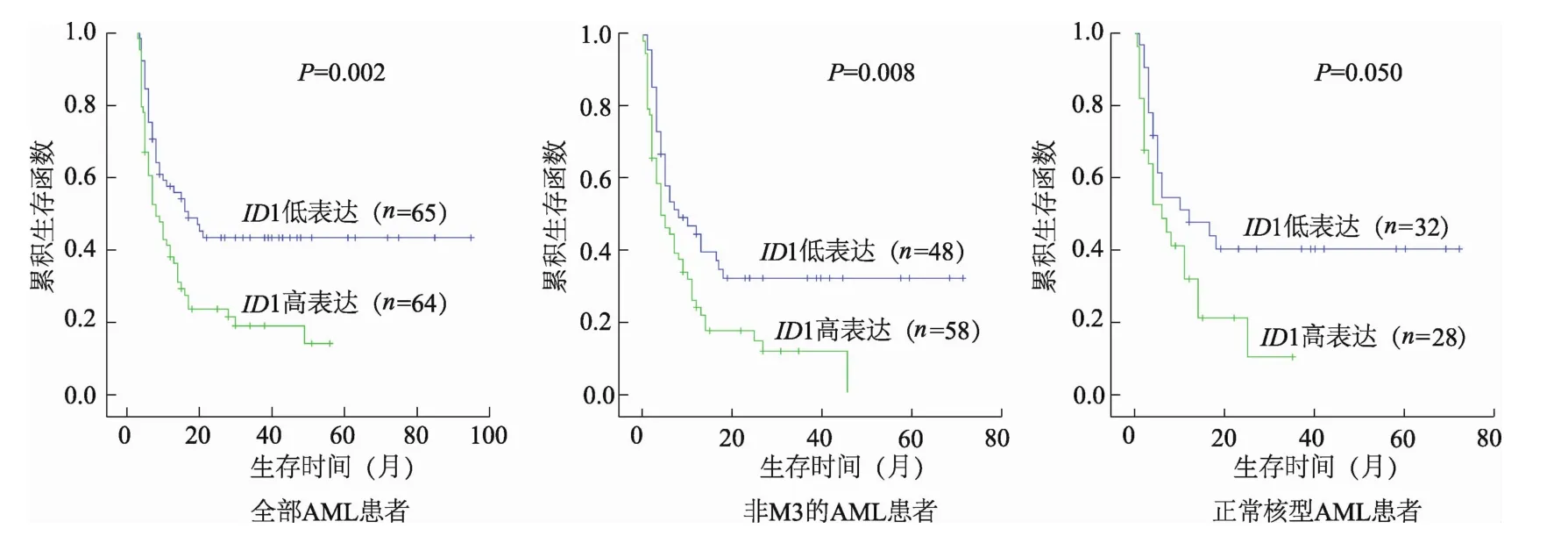
图2 AM L患者ID1表达对总体生存时间的影响
3 讨论
ID1过表达与大多数实体瘤的预后不良相关[4],但是ID1过表达对AML患者预后的影响仍然存在争议。Tang等[5]报道ID1高表达预示年龄<60岁的非M3或者正常核型AML患者完全缓解率降低、总体生存时间和无白血病生存时间缩短。然而,Damm等[6]揭示ID1过表达不是正常核型AML患者的独立预后因素,可能是与CEBPA野生型和(或)FLT3-ITD突变相关。我们前期的研究结果显示,ID1过表达与CEBPA及FLT3-ITD等基因突变并无明显相关,可能是由于这些基因突变的频率在中国人群中较低[11-13]。这种差异最可能归因于种族和AML亚型分布的差异[16]。本组资料的预后相关分析显示,ID1高表达与AML患者较低的完全缓解率相关,提示ID1可能参与耐药,进一步的生存分析证实了ID1高表达无论是在全部、非M3还是正常核型AML中都与预后不良密切相关。
此外,本研究还显示ID1表达尤其是在t(15;17)/M3中的表达,与核型显著相关。然而,如果M3患者被排除在分析之外,我们没有观察到ID1表达与核型分型之间的显著相关性,这与国外的研究结果一致[5]。既往研究发现急性早幼粒细胞白血病(即M3)患者原代白血病细胞以及NB4细胞系(M3细胞株)在全反式维甲酸治疗诱导分化后,ID1表达上调,提示ID1是全反式维甲酸的反应基因[14]。此外,ID1过表达抑制增殖并导致NB4细胞中的G0/G1阻滞[15]。然而,后来的研究显示,ID1过表达促进了原始骨髓祖细胞的增殖,ID1沉默抑制了白血病细胞株生长[16]。这些结果表明ID1在白血病发生过程中的作用可能取决于不同细胞遗传学背景。
综上所述,ID1基因表达水平可作为AML患者化疗反应以及预后判断的分子标志。
[1]Estey E,Döhner H.Acutemyeloid leukemia[J].Lancet,2006,368(9550):1894-1907.
[2]Grimwade D,Mrózek K.Diagnostic and prognostic value of cytogenetics in acute myeloid leukemia[J].HematolOncol Clin North Am,2011,25(6):1135-1161.
[3]Mrózek K,MarcucciG,Paschka P,etal.Clinical relevance ofmutations and gene-expression changes in adult acutemyeloid leukemiawith normal cytogenetics:arewe ready for a prognostically prioritizedmolecular classification?[J].Blood,2007,109(2):431-448.
[4]Lasorella A,Benezra R,Iavarone A.The ID proteins:master regulators of cancer stem cells and tumour aggressiveness[J].Nat Rev Cancer,2014,14(2):77-91.
[5]Tang R,Hirsch P,Fava F,et al.High Id1 expression is associated with poor prognosis in 237 patients with acute myeloid leukemia[J].Blood,2009,114(14):2993-3000.
[6]Damm F,Wagner K,G rlich K,et al.ID1 expression associates with othermolecularmarkers and is notan independent prognostic factor in cytogenetically normal acutemyeloid leukemia[J].Br JHaematol,2012,158(2):208-215.
[7]Bennett JM,Catovsky D,Daniel MT,et al.Proposed revised criteria for the classification of acute myeloid leukemia.A report of the French-American-British Cooperative Group[J].Ann Intern Med,1985,103(4):620-625.
[8]Vardiman JW,Thiele J,Arber DA,et al.The 2008 revision of the World Health Organization(WHO)classification ofmyeloid neoplasms and acute leukemia:rationale and important changes[J].Blood,2009,114(5):937-951.
[9]Zhou JD,Zhang TJ,Li XX,et al.Epigenetic dysregulation of ID4 predicts disease progression and treatment outcome in myeloid malignancies[J].JCell Mol Med,2017,21(8):1468-1481.
[10]杨海燕,周静东,杨磊,等.急性髓系白血病患者BAALC的表达及其临床意义[J].江苏大学学报(医学版),2015,25(2):169-174.
[11]Wen XM,Lin J,Yang J,et al.Double CEBPA mutations are prognostically favorable in non-M3 acute myeloid leukemia patients with wild-type NPM1 and FLT3-ITD[J].Int JClin Exp Pathol,2014,7(10):6832-6840.
[12]Lin J,Yao DM,Qian J,et al.Recurrent DNMT3A R882 mutations in Chinese patients with acute myeloid leukemia and myelodysplastic syndrome[J].PLoSOne,2011,6(10):e26906.
[13]Lin J,Yao DM,Qian J,et al.IDH1 and IDH2 mutation analysis in Chinese patientswith acutemyeloid leukemia andmyelodysplastic syndrome[J].Ann Hematol,2012,91(4):519-525.
[14]Su L,LiX,Gao SJ,etal.Cytogenetic and geneticmutation features of de novo acutemyeloid leukemia in elderly Chinese patients[J].Asian Pac J Cancer Prev,2014,15(2):895-898.
[15]Nigten J, Breems-de Ridder MC, Erpelinck-Verschueren CA,et al.ID1 and ID2 are retinoic acid responsive genes and induce a G0/G1 accumulation in acute promyelocytic leukemia cells[J]. Leukemia,2005,19(5):799-805.
[16]Suh HC,LeeanansaksiriW,JiM,et al.Id1 immortalizes hematopoietic progenitors in vitro and promotes amyeloproliferative disease in vivo[J].Oncogene,2008,27(42):5612-5623.
