miR-3691-3p靶向调控E2F3/PRDM1对前列腺癌细胞增殖、迁移和侵袭的影响
2018-01-18孙莉牛涵波徐义英孙茂民
孙莉,牛涵波,徐义英,孙茂民
(苏州大学实验动物中心,江苏苏州215123)
前列腺癌是老年男性泌尿生殖系统中最为多见的恶性肿瘤,从全球来看,前列腺癌的发生率居男性恶性肿瘤第二位,在欧美国家前列腺癌的死亡率仅仅低于支气管肺癌[1]。近年来,前列腺癌的死亡率逐渐增加,在美国,患者由2014年29 480例增加到2015年220 800例[2-3]。对于前列腺癌发生发展的分子机制已有一定了解,例如分子遗传机制扰乱了细胞增殖、细胞周期、细胞凋亡、自噬和衰老的平衡[4]。微小 RNA(microRNA,miRNA)是一类高度稳定的非编码小分子RNA,其分子大小约为21~23个核苷酸序列[5-6]。其通过与同源 mRNA的 3′非编码区(3′-untranslated region,3′-UTR)特异性序列的结合抑制多种蛋白的翻译[7]。miRNA靶基因蛋白的预测及验证是探讨其分子机制及其生物学特性的必要步骤。根据miRNA序列5′端的2-8位核苷酸基本上都能与靶基因mRNA序列3′-UTR完全互补这一原理[8],我们借助 Targetscan[9]、miRanda[10]、PicTar[11]和 DIANA-microT[12]等 miRNA靶基因预测软件,分别预测了miR-3691-3p前200个靶基因,求交集后仅剩下9个靶基因;进一步运用miR-Ontology Database数据库筛选出与肿瘤发生发展密切相关的靶基因,分别为PR结构域蛋白1(PR domain containing 1,PRDM1)和 E2F3(E2F transcription factor 3)。
锌指转录阻遏因子PRDM1也被称为Blimp-1,是PRDM家族中具有代表性的成员[13]。PRDM1在造血性肿瘤中具有重要作用[14]。E2F3属于E2F转录因子家族,能够参与调控细胞增殖,是一种与增殖强相关的癌基因[15]。但是有关E2F3和PRDM1作为靶基因在前列腺癌中作用的研究鲜有报道。DU145细胞株是从前列腺癌脑转移肿瘤中分离出来的,分化程度低,为雄激素非依赖的前列腺癌细胞,具有强大的转移潜能,缺乏内源性的雄激素受体的表达,主要用于研究晚期激素非依赖性前列腺癌的进展。本实验首先验证E2F3和PRDM1是否为miR-3691-3p的靶基因,再应用RNA干扰技术探究低水平的E2F3和PRDM1对DU145细胞株的增殖、迁移和侵袭能力的影响。现报告如下。
1 材料与方法
1.1 材料
DU145细胞株购自中国科学院典型培养物保藏委员会细胞库;胰蛋白酶(Gibco公司);二甲基亚砜(DMSO,Sigma公司);LipofectamineTM2000、DEPC水(Invitrogen公司);Trizol试剂、双染蛋白质标准品(天根生化科技有限公司);cDNA逆转录试剂盒、蛋白标准品(Thermo公司);PVDF膜(Millipore公司);兔抗E2F3多克隆抗体、羊抗PRDM1单克隆抗体、鼠抗β-肌动蛋白单克隆抗体(Abcam公司);胎牛血清、DMEM(高糖)培养基(Gibco公司)。
1.2 细胞株的培养和转染
将DU145细胞置于10%胎牛血清和1%双抗的DMEM高糖完全培养基中,置于37℃、5%CO2细胞培养箱中以进行后续实验。取将要进行传代的DU145细胞分别接种于96孔板、24孔板和6孔板中,待所铺细胞生长至50%~70%时,按照LipofectamineTM2000转染试剂说明书进行转染,每孔100 nmol/L。培养24 h或48 h后,收集细胞进行后续实验。E2F3 siRNA,PRDM1 siRNA和阴性对照siRNA及miR-3691-3p成熟体、阴性对照均购于广州市锐博生物科技有限公司。miR-3691-3p mimic序列:5′-ACCAAGUCUGCGUCAUCCUCUC-3′;阴性对照 mimic序列:5′-UUUGUACUACACAAAAGUACUG-3′;E2F3 siRNA序列:5′-GCACTACGAAGTCCAGATA-3′;PRDM1 siRNA序列:5′-GGACCTCGATGACTTTAGA-3′;阴性对照 siRNA序列:5′-UUUTGATCAUTGATGAAA-3′。
1.3 实时荧光定量PCR检测E2F3和PRDM1 mRNA的表达
将不同浓度梯度 miR-3691-3p mimic(10、50、100 nmol/L)转染入 DU145细胞内;Trizol一步法抽提DU145细胞中总RNA。根据逆转录试剂盒上所提供的方法,将所获得的总RNA进行逆转录反应。并按照实时荧光定量PCR试剂盒说明书进行PCR反应。E2F3引物序列上游:5′-GATGGGGTCAGATGGAGAGA-3′,下 游:5′-GAGACAC-CCTGGCATT-GTTT-3′;PRDM1引物序列上游:5′-AAAAGAAACATGACCGGCT-ACAAG-3′,下游:5′-GGTGGACCTTCAGATTGGAGA-3′;以 GAPDH为对照,引物序列上游:5′-CAAGGTCATCCATGACAACTTTG-3′,下游:5′-GTCCACCACCCTGTTGCTGTAG-3′。反 应 条 件:95℃预变性10 min,95℃ 10 s,60℃ 60s,40个循环。目的基因表达量按照2-ΔΔCt法计算。
将E2F3 siRNA、PRDM1 siRNA、阴性对照 siRNA转染入DU145细胞内,RNA的提取及实时荧光定量PCR反应同上。
1.4 蛋白质印迹法测定E2F3/PRDM1靶基因的蛋白水平
收集6孔板中已经进行转染的DU145细胞,实验分为 miR-3691-3p mimic 10 nmol/L、50 nmol/L、100 nmol/L和阴性对照组,并重复3次独立实验。收集细胞提取总蛋白,利用PMSF和裂解液的混合液裂解蛋白。取相应的总蛋白于10%聚丙烯酰胺凝胶中进行电泳。在PVDF膜上进行转膜反应,奶粉封闭后,于相应的一抗中4℃过夜(抗体稀释比为1∶1 000)。洗涤3次后,二抗在室温中放置1 h后,进行ECL液显影,并进行成像反应。
1.5 MTT法测定DU145细胞的增殖能力
取处于对数生长时期的DU145细胞,以1×103/孔的数量铺于96孔板,每组有5个复孔,分为阴性对照组、E2F3 siRNA组和PRDM1 siRNA组转染细胞,放置于培养箱中培养。分别于1、2和3 d后,于每孔中加20μL(5 g/L)新配置的 MTT液,放置4 h后去除板中液体,然后将150μL DMSO加入至每孔中,振荡10 min进行混匀,使蓝紫色结晶物甲臜充分溶解。随后酶标仪570 nm下测定其光密度(D)值。
1.6 划痕实验测定DU145细胞的迁移能力
将DU145细胞悬液(2×105个/mL)每孔2 mL铺于6孔板中。细胞分为阴性对照组、E2F3 siRNA组和PRDM1 siRNA组进行转染,6 h后换液。换液前用200μL的黄色灭菌枪头在每孔中央划一条直线,将板中原先液体吸去,缓冲液洗3次,洗去悬浮的细胞,并在每孔中加入新鲜纯DMEM。以设置划线为开始时间,在显微镜下拍摄0、24、48 h时DU145细胞的迁移情况,并统计划痕的愈合速率。
1.7 Transwell法测定DU145细胞侵袭能力
准备基质胶:将冻存于-20℃中的Matrigel胶放于4℃,使其融化。取300μL的纯DMEM,并将60μLMatrigel胶放入其中,混匀后各50μL放于上室;在37℃培养箱中放6 h,使其转变为固态。将制备的细胞悬液(1×105个/mL)500μL/孔接种于 24孔板中。实验分为阴性对照组、E2F3 siRNA组和PRDM1 siRNA组进行转染。每次实验重复3次。转染换液后,培养24 h。消化细胞后,纯DMEM洗涤3次,细胞计数板计数,并利用DMEM进行重悬。于上室中每孔加上述已配置的100μL细胞悬液(1×105个/mL),下室中加入500μL含10% 血清的DMEM。37℃培养箱中继续孵育24 h。利用PBS缓冲液将取出的小室轻轻冲洗3遍后,用4%低聚甲醛固定15 min。于0.2%结晶紫染色液中染色10 min,双蒸水洗2遍后,用棉签清除上表面残留的原有细胞,显微镜下观察。在镜下随机选择5个视野进行拍照,计算穿膜细胞数并取平均值以检测细胞的侵袭能力。
1.8 统计学处理
应用SPSS 19.0软件进行统计分析,各组数据采用均数±标准差(±s)表示,组间比较采用t检验,P<0.05表示差异有统计学意义。
2 结果
2.1 miR-3691-3p mimic转染后DU145细胞E2F3、PRDM1 mRNA的表达
随着miR-3691-3p mimic浓度的升高,在转染浓度为50 nmol/L时,DU145细胞株中 E2F3、PRDM1 mRNA的表达量均比阴性对照组明显降低(P<0.01或P<0.05)。见图1。
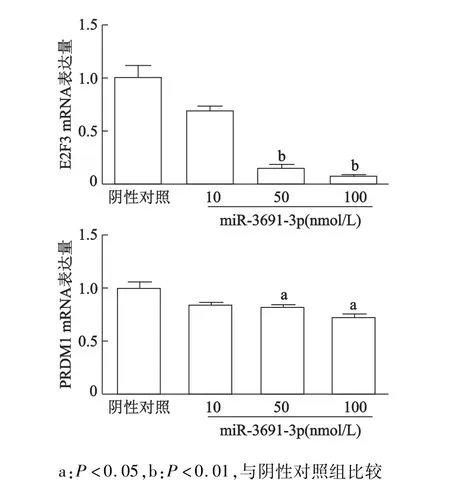
图1 实时荧光定量PCR检测DU145细胞E2F3、PRDM 1 mRNA的表达
2.2 miR-3691-3p mimic转染后DU145细胞E2F3、PRDM1蛋白的表达
蛋白质印迹结果显示,在DU145细胞中靶基因蛋白表达量均随着转染浓度的增大而降低,在转染浓度为100 nmol/L时,E2F3和PRDM1的蛋白表达量均最低(P<0.01),见图2。初步确认 E2F3和PRDM1为miR-3691-3p的靶基因。
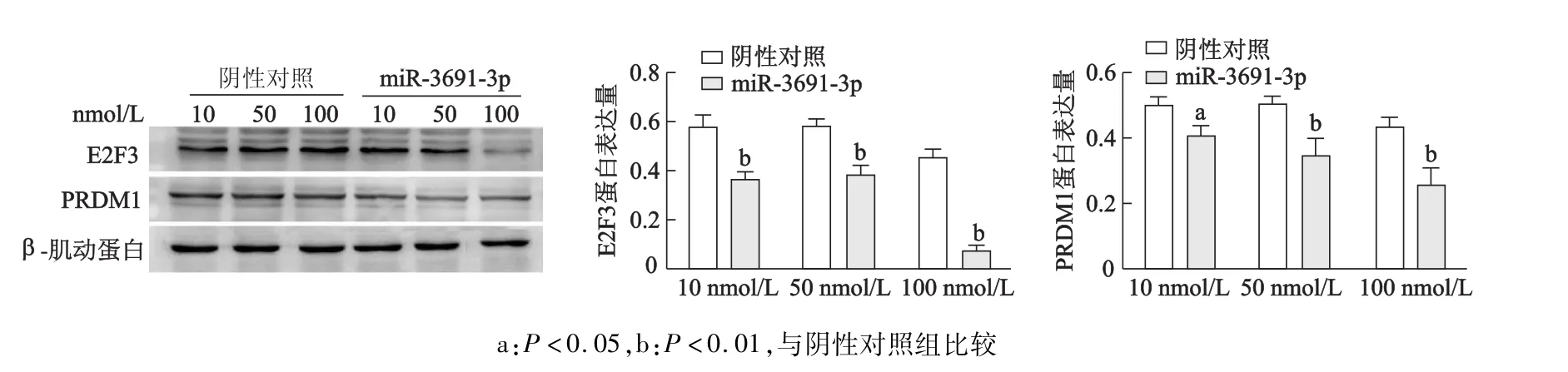
图2 蛋白质印迹检测DU145细胞E3F3、PRDM 1蛋白表达
2.3 实时荧光定量PCR验证E2F3 siRNA、PRDM1 siRNA转染效率
与阴性对照组细胞相比,转染E2F3 siRNA和PRDM1 siRNA后,DU145细胞中E2F3、PRDM1 mRNA表达水平显著降低(t=31,P<0.01;t=10.44,P<0.01),见图3。表明 E2F3和 PRDM1 siRNA能够敲低两个候选靶基因在DU145细胞株中的水平。
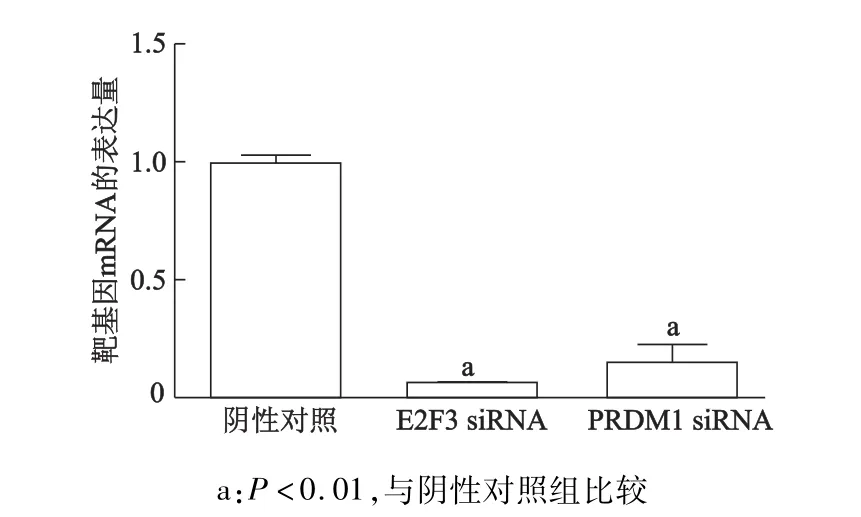
图3 实时定量PCR验证siRNA在细胞中的转染效率
2.4 低表达的E2F3和PRDM1对DU145细胞增殖能力的影响
MTT实验结果显示,相比于阴性对照组,转染E2F3 siRNA和PRDM1 siRNA后,DU145细胞的增殖能力明显被抑制(P<0.01,图4)。
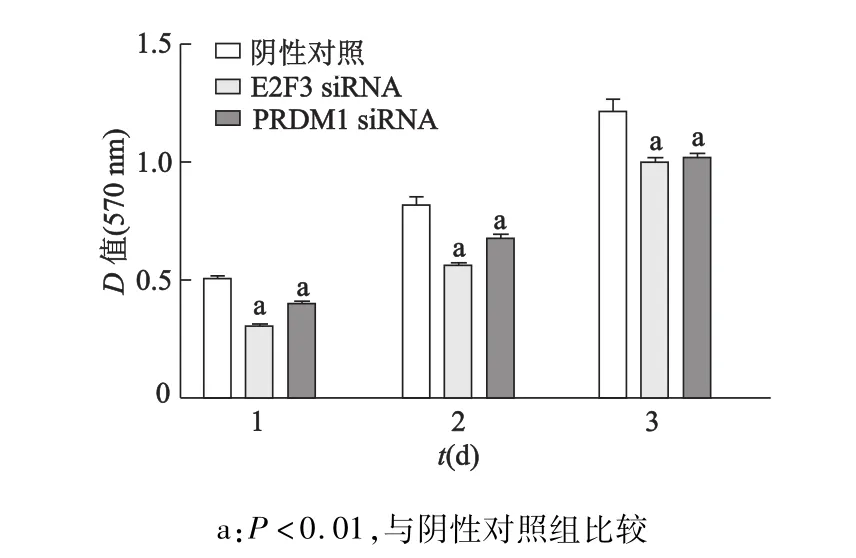
图4 M TT实验测定siRNA对DU145细胞增殖的影响
2.5 低表达的E2F3和PRDM1对DU145细胞迁移能力的影响
划痕实验结果显示,相比于阴性对照组,转染E2F3 siRNA的DU145细胞迁移被显著抑制(P<0.01),而PRDM1 siRNA转染后细胞迁移能力没有明显变化(P>0.05)。见图5、图6。
2.6 低表达的E2F3和PRDM1对DU145细胞侵袭能力的影响
Transwell实验结果显示,相比于阴性对照组,转染siRNA后DU145细胞的侵袭能力被明显抑制(E2F3:t=6.895,P<0.01;PRDM1:t=9.286,P<0.01)。见图7。
3 讨论
大量的体内、体外实验研究表明,miRNAs在多种癌症的发展进程中具有不可替代的作用[16-18]。在肿瘤中,miRNA主要通过其下游的靶蛋白来发挥自身的调节能力。miRNA能有效地与靶蛋白的3′-UTR区结合来抑制翻译作用或者降解mRNA,从而起到对靶蛋白表达的干扰作用[19]。为了探究miR-3691-3p在前列腺癌中的作用机制,我们利用三大生物信息学网站预测了miR-3691-3p的候选靶蛋白,最后筛选出E2F3和PRDM1。
E2F3属于E2F家族,能够调节细胞增殖和凋亡从而促进肿瘤的生长。E2F转录因子在癌症中占有重要地位,在视网膜母细胞瘤中可调控其下游靶基因来抑制肿瘤的增殖。不同的E2F在癌症的发生发展中具有不同程度的促进或抑制功能[20-21]。已有报道显示E2F3受多种miRNAs的调控。例如,miR-125b通过靶向调节E2F3来阻碍膀胱癌细胞的集落形成和裸鼠肿瘤发展。E2F3可作为miR-195的靶蛋白阻碍人胶质细胞瘤的增殖和侵袭。已有研究表明,PRDM1作为RAS/RAF/AP-1信号通路的调节因子,具有促进肺癌细胞迁移的作用;作为NF-κB下游效应因子,可激活RelB、Bcl-2和RAS通路,促进乳腺癌的迁移[22]。

图5 划痕实验检测3组DU145细胞的迁移能力(×100)
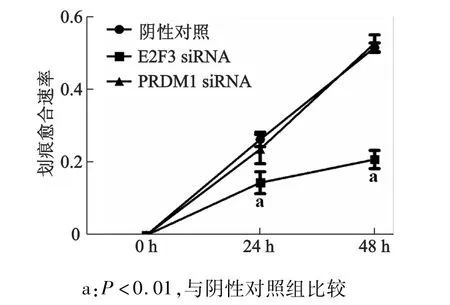
图6 siRNA对DU145细胞迁移能力的影响
本实验结果显示,随着miR-3691-3p mimic转染浓度的提高,DU145细胞E2F3和PRDM1 mRNA和蛋白表达水平逐渐下降。初步得出结论:在前列腺癌细胞中由于miR-3691-3p的降低,促使E2F3和PRDM1明显升高;而在miR-3691-3p高表达时,靶基因低表达。于是我们进一步将细胞中靶基因的表达量降低,观察细胞的生物学行为的变化。结果表明,敲低E2F3均抑制了DU145细胞的增殖、迁移和侵袭能力;而PRDM1的低表达只对细胞的增殖和侵袭能力有明显的抑制作用,对迁移能力的影响较弱,需要进一步的实验进行验证。
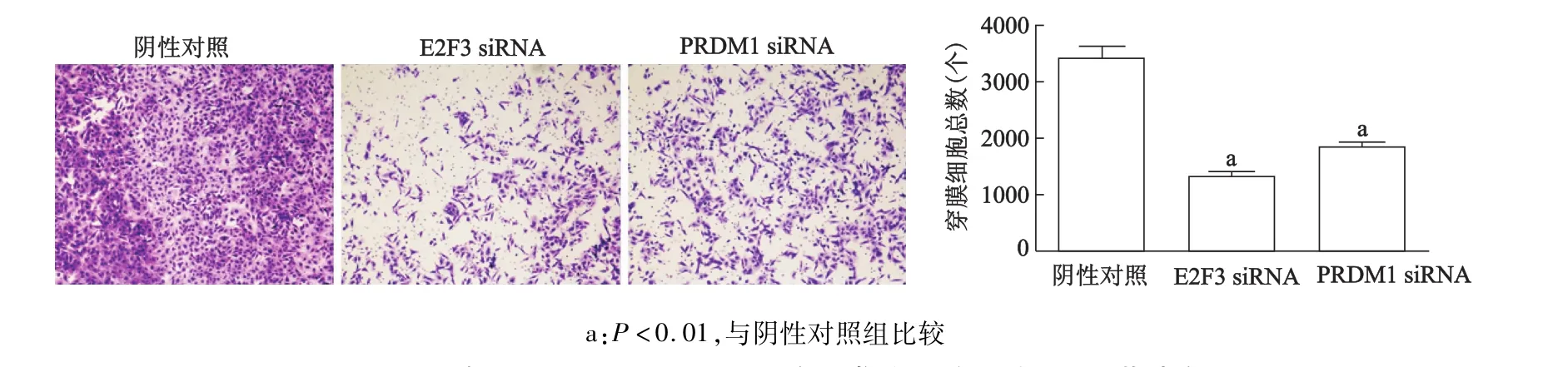
图7 Transwell检测siRNA对DU145细胞侵袭能力的影响(结晶紫染色×100)
综上所述,低表达水平的miR-3691-3p促进前列腺癌发生发展进程和恶性转化,其机制是通过调控下游靶基因E2F3和PRDM1表达量升高所致。沉默靶基因在DU145细胞中的表达,其增殖/迁移和侵袭能力被显著抑制。本研究初步阐明了miR-3691-3p调控前列腺癌发展进程的分子机制,为前列腺癌的临床诊断和靶向治疗提供了新思路。
[1]Filippou P,Ferguson JE 3rd,Nielsen ME.Epidemiology of prostate and testicular cancer[J].Semin Intervent Radiol,2016,33(3):182-185.
[2]Siegel R,Ma J,Zou Z,et al.Cancer statistics,2014[J].CA Cancer JClin,2014,64(1):9-29.
[3]Siegel RL,Miller KD,Jemal A.Cancer statistics,2015[J].CA Cancer JClin,2015,65(1):5-29.
[4]Hanahan D,Weinberg RA.Hallmarks of cancer:the next generation[J].Cell,2011,144(5):646-674.
[5]Ambros V.MicroRNA pathways in flies and worms:Growth,death,fat,stress,and timing[J].Cell,2003,113(6):673-676.
[6]Yates LA,Norbury CJ,Gilbert RJC.The Long and Short of MicroRNA[J].Cell,2013,153(3):516-519.
[7]Orellana EA,Kasinski AL.MicroRNAs in cancer:A historical perspective on the path from discovery to therapy[J].Cancers(Basel),2015,7(3):1388-1405.
[8]Lagan A.Computational prediction ofmicroRNA targets[J].Adv Exp Med Biol,2015,887:231-252.
[9]Lewis BP,Shih IH,Jones-Rhoades MW,et al.Prediction ofmammalian microRNA targets[J].Cell,2003,115(7):787-798.
[10]John B,Enright AJ,Aravin A,et al.Human MicroRNA targets[J].PLoSBiol,2004,2(11):e363.
[11]Krek A,Grun D,Poy MN,et al.Combinatorial microRNA target predictions[J].Nat Genet,2005,37(5):495-500.
[12]Kiriakidou M,Nelson PT,Kouranov A,et al.A combined computational-experimental approach predicts human microRNA targets[J].Genes Dev,2004,18(10):1165-1178.
[13]Hohenauer T,Moore AW.The Prdm family:expanding roles in stem cells and development[J].Development,2012,139(13):2267-2282.
[14]Boi M, Zucca E, Inghirami G, et al. PRDM1/BLIMP1:a tumor suppressor gene in B and T cell lymphomas[J].Leuk Lymphoma,2015,56(5):1223-1228.
[15]Xiao F,Zhang W,Chen L,et al.MicroRNA-503 inhibits the G1/S transition by downregulating cyclin D3 and E2F3 in hepatocellular carcinoma[J].J Transl Med,2013,11:195.
[16]Ohtsuka M,Ling H,Doki Y,etal.MicroRNA processing and human cancer[J].JClin Med,2015,4(8):1651-1667.
[17]Li F,Piao M,Piao Y,et al.A new direction of cancer classification:positive effect of low-ranking MicroRNAs[J].Osong Public Health Res Perspect,2014,5(5):279-285.
[18]Di Leva G,Croce CM.miRNA profiling of cancer[J].Curr Opin Genet Dev,2013,23(1):3-11.
[19]Bartel DP.MicroRNAs:Target recognition and regulatory functions[J].Cell,2009,136(2):215-233.
[20]Chen HZ,Tsai SY,Leone G.Emerging roles of E2Fs in cancer:an exit from cell cycle control[J].Nat Rev Cancer,2009,9(11):785-797.
[21]Kauer M,Ban J,Kofler R,et al.A Molecular function map of ewing′s sarcoma[J].PLoS One,2009,4(4):e5415.
[22]Yu Z,Sato S,Trackman PC,et al.Blimp1 activation by AP-1 in human lung cancer cells promotes a migratory phenotype and is inhibited by the lysyl oxidase propeptide[J].PLoSOne,2012,7(3):e33287.
