甲型副伤寒沙门菌特异性基因筛选及PCR检测体系建立
2018-01-04翟立公王俊颖张小雨赵婉晴
翟立公,王俊颖,张小雨,孟 欣,崔 葆,赵婉晴,牛 萍
(安徽科技学院食品药品学院,安徽 滁州 233100)
甲型副伤寒沙门菌特异性基因筛选及PCR检测体系建立
翟立公,王俊颖,张小雨,孟 欣,崔 葆,赵婉晴,牛 萍
(安徽科技学院食品药品学院,安徽 滁州 233100)
以甲型副伤寒沙门菌为检测目标,通过比较基因组和聚合酶链式反应(polymerase chain reaction,PCR)验证方法筛选到4 个该血清型的特异性基因,其中以gene_3105作为该血清型的检测靶点设计引物PA23;并结合沙门菌属特异性引物139-141,建立一种甲型副伤寒沙门菌的PCR检测方法。优化PCR反应体系,并对该检测体系的特异性、灵敏度、抗干扰能力及人工污染样品检出限等方面进行评价。结果表明,当样品中含有甲型副伤寒沙门菌时,该体系能扩增出2 条特异性条带,含有其他血清型的沙门菌仅能扩增出284 bp条带,不含沙门菌无扩增条带产生。灵敏度评价表明,基因组DNA和纯菌菌落检出限分别为32.4 pg/µL和4.3×103CFU/mL;抗干扰能力实验显示,当鸡肉背景菌群和猪肉背景菌群浓度在106CFU/mL和4.87×107CFU/mL时,检出限为6.43×104CFU/mL。当无菌的鸡肉和猪肉样品中添加N CFU/25 g甲型副伤寒沙门菌时,经10 h增菌,检测结果为阳性(0<N<10)。实验建立甲型副伤寒沙门菌PCR检测方法具有较好的特异性和灵敏度,有很好的应用价值,可在食品安全领域广泛应用。
甲型副伤寒沙门菌;比较基因组;血清型特异性基因;聚合酶链式反应
甲型副伤寒沙门菌(Salmonella Paratyphi A)是伤寒类沙门菌中重要的一种血清型,经粪口传播,临床表现类似伤寒类症状,常出现腹泻、肠胃炎、发热等情况,较严重者将出现肠出血和肠穿孔等症状[1-3]。据统计,全球每年引起的伤寒症状的患者多达2 200万 例,其中由甲型副伤寒沙门菌引起的占25%[4-6]。在我国的个别省份内也出现过大型的甲型副伤寒沙门菌的暴发和流行[7-8]。
我国的血清型鉴定和沙门菌的检测主要参照GB/T 4789.4—2010《食品微生物学检验 沙门氏菌检验》的传统培养的方法,需要5 步:预增菌、选择性培养、显示培养、生化鉴定和血清型鉴定[7,9-10]。血清型鉴定主要是通过沙门菌特异的鉴定血清与菌体的O抗原、H抗原和Vi抗原发生凝集反应,再与Kauffman-White血清表对照鉴定菌株的血清型[11-12]。此方法虽然直观,但操作较为繁杂,检测时间需要5 d以上[13]。而且,如果菌体表面的抗原发生丢失或被破坏将影响抗体对抗原决定簇的识别,沙门菌血清型达2 600多种,如果血清的特异性不强或凝集反应较为迟缓,都会造成分型的错误[14]。利用分子检测可有效弥补以上血清鉴定的不足,但是在基因水平上寻找到不同血清型之间的差异成为制约该技术发展的关键[15-16]。因此,血清型特异的检测靶点的鉴定在分子检测技术的发展扮演着重要的作用。Tennant等[17]以fl iC、sdfⅠ、16S RNA和 fliB- fl iA的间隔区域为目标,建立了针对沙门菌的肠炎、都柏林、斯坦利维尔、鼠伤寒及其变种血清型的多重聚合酶链式反应(polymerase chain reaction,PCR)检测系统,经实验证明该多重PCR体系具有较好的特异性。
比较基因组学是对不同物种的基因数据进行比较分析,揭示彼此间的相似性和差异性,将有助于了解物种形成的机制和基因或基因组上非编码区的功能[18-19]。随着沙门菌不同血清型基因测序的完成,通过BLAST比对程序,可以筛选到在血清型内的不同菌株中具有高度同源性,但对其他血清型差异较大的基因作为血清型特异性基因[18,20]。有研究对肠炎沙门菌和鼠伤寒沙门菌的全基因组进行分析分别获得8 个和38 个具有血清型特异的区域,PCR评估后确定由SEN1392和STM4495两个基因设计的引物的特异性和灵敏度最好,建立的PCR体系通过12 h富集培养后灵敏度达到2~3 CFU[21-22]。
本研究是利用比较基因组学技术筛选甲型副伤寒沙门菌的血清型特异性基因,并以此为模板设计甲型副伤寒沙门菌特异性引物与沙门菌属特异性引物139-141共同构建PCR检测体系,并对其实际应用性进行评估,以期能达到快速检测食品中甲型副伤寒沙门菌及沙门菌的目的。
1 材料与方法
1.1 材料与试剂
猪肉和鸡肉样品 市购。
本实验所涉及到菌株包括39 株沙门菌分属31 个不同的血清型,以上菌株与甲型副伤寒沙门菌的同源性较近。实验中选择的18 株非沙门菌均是在食品中检出率较高的食源性致病菌,具体菌株信息见表1。

表1 菌株及PCR检测结果Table 1 Strains used in the test of PCR specif i city

续表1
酵母提取物、胰蛋白胨 英国Oxoid公司;dNTP、DNA凝胶回收试剂盒和Ezup柱式细菌基因组DNA提取试剂盒、引物合成 生工生物工程(上海)股份有限公司;DNA标准分子质量DL 2000 Marker、Taq DNA聚合酶和6×loading buffer 广州东盛生物科技有限公司;2×Taq Master 南京诺唯赞生物科技有限公司。
1.2 仪器与设备
Life Touch PCR扩增仪 杭州博日科技有限公司;JS-780全自动数码凝胶成像分析仪 上海培清科技有限公司;NV3000C微量紫外分光光度计仪 北京凯奥科技发展有限公司;Ⅱ级生物安全柜 鑫北生物技术有限公司;HYL-A全温摇瓶柜 太仓市强乐实验设备厂;DHZ-D冷冻恒温振荡器 江苏太仓市实验设备厂;DK-8 D型电热恒温水槽 上海森信实验仪器有限公司;AY 120电子天平 日本岛津公司;单人单面净化工作台杭州和欣科技有限公司;手提式压力蒸汽灭菌器 上海申安医疗器械厂。
1.3 方法
1.3.1 特异性基因筛选
从NCBI(ftp://ftp.ncbi.nih.gov/genbank/genomes/Bacteria/)基因组公共数据库中,获取甲型副伤寒沙门菌CMCC50973的全基因组序列数据。将该株菌的全基因序列的每个基因在NCBI的BLASTN(https://blast.ncbi.nlm.nih.gov/Blast.cgi)系统中进行比对。在比对过程中,选取与代表菌株基因组同源性较高,即Query cover的值为100%并且匹配期望值E趋近于0(E值<10),同时与其他非该沙门菌血清型代表菌株基因组不具有同源性的基因作为该沙门菌血清型的准特异性靶点。
1.3.2 引物设计
引物设计通常使用Primer Premier 5.0软件(PremierBiosoft International Inc.,USA),获得的引物再通过Oligo 6.0软件和BLASTN进行分析,选择二聚体较少、自身无发夹结构、退火温度为60 ℃左右及与非模板DNA同源性较低的引物作为该基因的特异性引物并送往生工生物工程(上海)股份有限公司进行合成。本研究所需引物见表2。
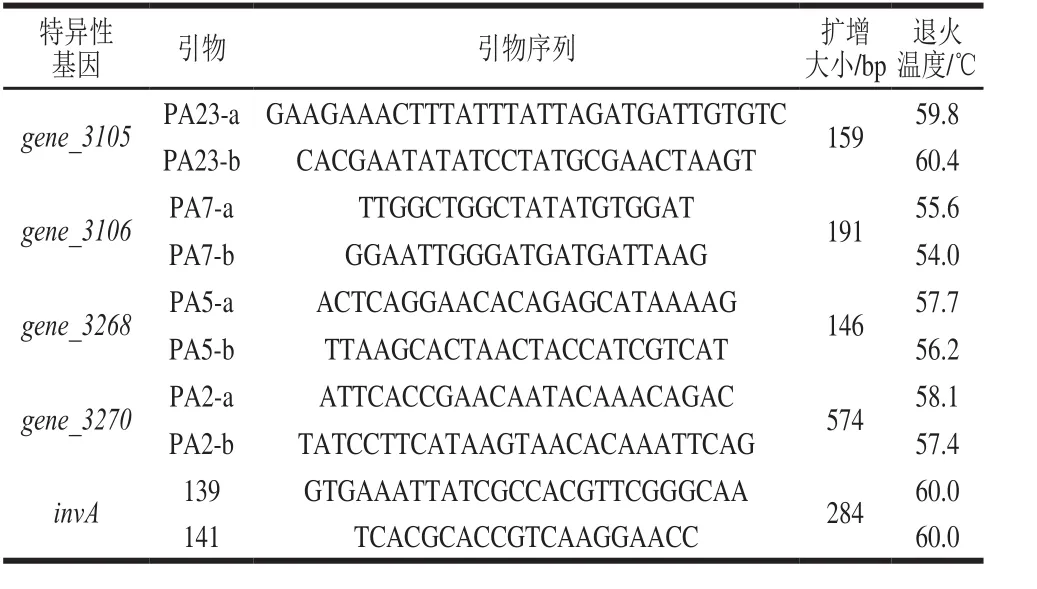
表2 引物序列及扩增片段大小Table 2 Primer sequences and the length of amplif i cation fragments
1.3.3 菌株培养及基因组提取
将菌株接种于LB或BHI液体培养基中,37 ℃过夜培养。利用热裂解法提取细菌基因组DNA和Ezup柱式细菌基因组DNA抽提试剂盒提取所有细菌基因组DNA。
1.3.4 沙门菌DNA浓度测定
将提取好的基因组DNA通过核酸浓度测定仪分析确定基因组DNA的质量及基因组DNA的浓度,然后置于-20 ℃冰箱保存备用。1.3.5 引物特异性验证
以34 株不同血清型的沙门菌和18 株非沙门菌的基因组DNA为模板,与之前设计的甲型副伤寒沙门菌特异性引物进行PCR扩增。在25 µL反应体系所包括物质的终浓度分别为:2×PCR Reaction Buffer 12.5 µL、Taq聚合酶0.1 U、引物0.4 µmol/L、DNA模板1 µL和ddH2O 8.5 µL。PCR反应条件:94 ℃预变性10 min;35 个循环,每个循环94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸45 s;最后72 ℃延伸10 min。PCR产物通过1.5%的琼脂糖凝胶进行电泳,凝胶成像仪下观察结果。
1.3.6 PCR检测体系的建立
根据前期筛选和特异性验证获得的甲型副伤寒沙门菌特异性基因作为模板设计引物,并且结合沙门菌属特异性引物139-141,进行优化。最终确定PCR的反应体系为:10×Taq Buffer(Mg2+free)2.5 µL、dNTP 0.45 mmol/L、MgCl24 mmol/L、Taq聚合酶0.2 U、引物139-141 0.2 µmol/L、引物PA23 1.2 µmol/L、DNA模板2 µL和ddH2O 3 µL。优化后的PCR反应条件:94 ℃预变性10 min;35 个循环,每个循环94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸45s;最后72 ℃延伸10 min。PCR产物通过1.5%的琼脂糖凝胶进行电泳,凝胶成像仪下观察结果。
1.3.7 特异性验证
用实验室所保存的甲型副伤寒沙门菌作为阳性菌株,其他血清型的沙门菌和非沙门菌的病原微生物作为阴性样本,分别提取各菌株的基因组DNA作为模板,应用以上PCR检测体系及反应参数进行扩增。
提取甲型副伤寒沙门菌CMCC50001的基因组DNA,并用核酸浓度测定仪分别测定基因组DNA浓度。将获得的甲型副伤寒沙门菌基因组DNA用无菌水分别进行10 倍的梯度稀释,每个稀释度分别取2 µL作为模板分别进行多重PCR扩增,琼脂糖凝胶电泳判断相应反应体系的DNA模板灵敏度。
过夜培养甲型副伤寒沙门菌CMCC50001,通过无菌水进行10 倍的梯度稀释。平板菌落计数10-6、10-7和10-8的稀释度,获得初始菌液的浓度。每个稀释度分别取1 mL菌液,通过热裂解法提取基因组DNA分别进行多重PCR扩增,电泳结果判断反应体系的菌落灵敏度。
1.3.9 抗干扰能力评估
超市购得鸡胸肉和猪肉样品,经标准细菌学方法GB 4789.4—2010验证无沙门菌。在无菌条件下分别取25 g样品加入到225 mL LB液体培养基中37 ℃过夜培养。获得的鸡肉背景增菌液和猪肉背景增菌液,通过平板计数法获得两种增菌初始浓度。同时将甲型副伤寒沙门菌标准菌株CMCC50001接种于LB液体培养基中进行过夜培养,平板菌落计数法获得菌液的初始浓度,再用无菌水进行梯度稀释。取500 μL不同稀释度的甲型副伤寒沙门菌的菌液加入到500 μL的鸡肉背景和猪肉背景增菌液中混匀,利用试剂盒法提取混合菌液的基因组DNA,进行PCR扩增,每个浓度3 次重复,同时以无菌水作为空白对照。
1.3.10 人工污染实验
猪肉和鸡肉样品,每种样品各称取3 份,每份25 g,进行巴氏灭菌(75~90 ℃处理15~20 s)。灭菌后的样品被1 mL较低浓度的甲型副伤寒沙门菌标准菌液CMCC50001进行污染,在均质袋内混匀,再转入225 mL的LB液体培养基中增菌4~10 h,每隔2 h取1mL的增菌液提取DNA,进行PCR反应,同时以无菌水作为空白对照。
2 结果与分析
2.1 甲型副伤寒沙门菌特异性基因验证
利用BLASTN系统对甲型副伤寒沙门菌CMCC50973的全基因组序列数据进行分析,共获得9个准特异性基因(与甲型副伤寒沙门菌具有同源性与其他微生物不具有同源性)分别gene_3037(321 bp)、gene_3044、gene_3057、gene_3056、gene_3269、gene_3270、gene_3268、gene_3106和gene_3105。将以上基因作为模板,利用Primer 5.0软件设计引物,退火温度值均在60 ℃左右,扩增片段大小在1 000 bp以下,并且分别以34 株不同血清型的沙门菌标准菌株和18 株非沙门菌的致病菌的基因组DNA为模板进行PCR验证。其中3 个基因的PCR结果无非特异性条带出现(表1),被认为甲型副伤寒沙门菌特异性基因(表2)。
1.4 衰弱指数(FI) 明确器官功能缺陷与临床结果相关性 基于健康缺陷理论,测评不健康指标在所有指标中比例,常用于预期寿命计算和流行病学健康评估。FI≥0.25为衰弱,FI≤0.12为无衰弱,0.12~0.25为衰弱前期[14-15]。在一项纳入33 706名60岁以上普外科患者的研究显示,随着FI增加,伤口发生率与感染率随之增加(P<0.001),并认为FI是急症手术患者术前衰弱评估的有效方法[16]。该评估需要专业人员进行,在一定程度上限制了临床应用。
2.2 PCR检测体系的建立
通过比较分析,PA23和139-141引物同时扩增时没有非特异性条带产生。因此,选取PA23引物作为甲型副伤寒沙门菌的特异性引物并与139-141引物共同构建PCR检测的反应体系,引物序列见表2。通过优化了两对引物的浓度、Mg2+浓度及Taq聚合酶浓度(优化过程数据没有呈现),最终反应体系见表3。该反应体系的阳性反应结果见图1。

表3 甲型副伤寒沙门菌特异性基因Table 3 Serotype-speci fi c genes of S. Paratyphi A
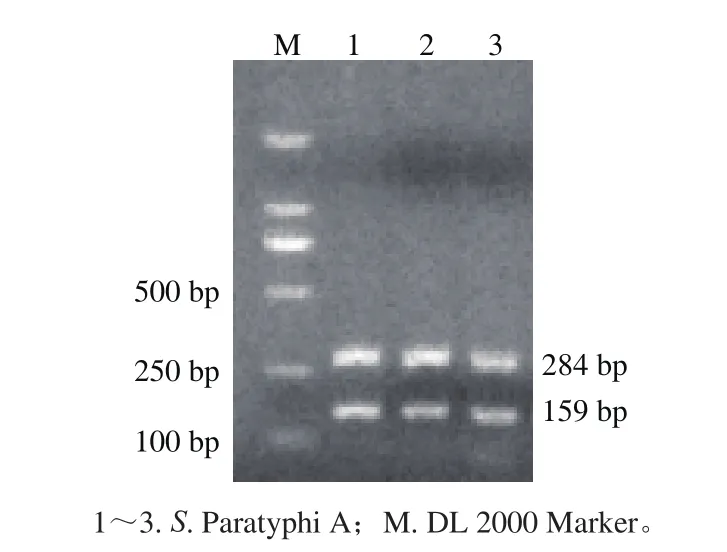
图1 检测S. Paratyphi A的PCR反应阳性结果Fig. 1 Positive results obtained in the PCR detection of S. Paratyphi A
2.3 PCR检测体系的特异性评价
通过对表1中菌株的DNA进行PCR检测,对该体系的特异性进行验证,结果见表2,139-141引物对于沙门菌模板均能获得284 bp的扩增条带,而对于非沙门菌的菌株无任何条带;同时甲型副伤寒沙门菌菌株还可以扩增出159 bp的条带,而该条带在其他沙门菌和非沙门菌菌株中均无条带产生。
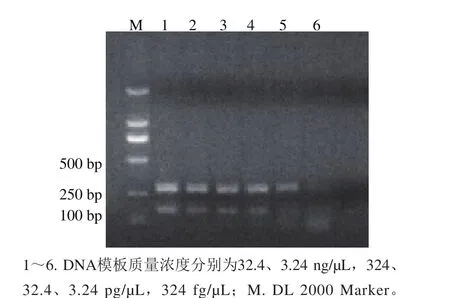
图2 S. Paratyphi A基因组DNA灵敏度检测结果Fig. 2 Detection DNA sensitivity of S. Paratyphi A
2.4 PCR检测体系的灵敏度评价
将甲型副伤寒沙门菌CMCC50001过夜培养,提取基因组DNA,测定其质量浓度为32.4 ng/µL,用无菌水做10 倍系列梯度稀释至324 fg/µL,每个稀释度各取2µL作为模板进行PCR扩增。结果显示,当模板浓度为32.4 pg/µL时,该反应体系的两对引物均可获得明显的扩增条带;当模板质量浓度为3.24 pg/µL时,139-141能获得明显的扩增条带,但PA23引物只能获得较为模糊的电泳条带,其结果较难判断。该结果表明,该反应体系的DNA灵敏度为32.4 pg/µL,结果见图2。
将过夜培养的甲型副伤寒沙门菌CMCC50001菌液,通过梯度稀释分别获得浓度为4.3×104CFU、4.3×103、4.3×102、4.3×101CFU/mL和4.3 CFU/mL的稀释液。每个稀释度的菌液通过热裂解法,提取基因组DNA进行PCR验证。结果表明,当菌液浓度为4.3×103CFU/mL和4.3×104CFU/mL时,该反应体系可以获得两条清晰电泳条带(PA23和139-141引物);而当菌液浓度为4.3×101CFU/mL,只有139-141引物扩增出目的条带,检测结果为阴性。因此,认为该反应体系的菌落灵敏度为4.3×103CFU/mL,结果见图3。
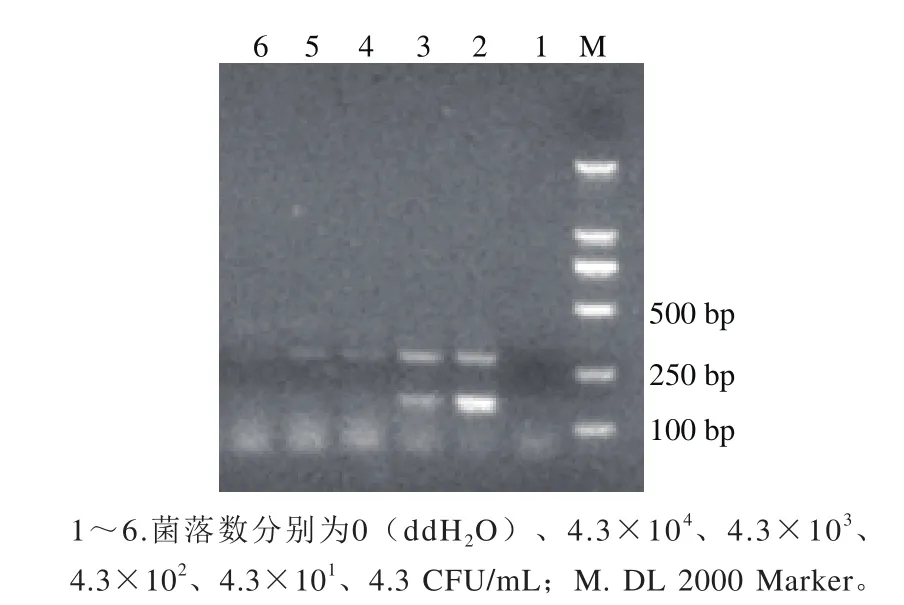
图3 S. Paratyphi A菌落灵敏度检测结果Fig. 3 Sensitivity of PCR for detection of cells from S. Paratyphi A
2.5 PCR检测体系的抗干扰能力评价
由超市购得鸡胸肉和猪肉样品,经标准细菌学方法GB 4789.4—2008验证无沙门菌。分别取25 g鸡胸肉和猪肉样品,经过夜增菌培养后,获得鸡肉增菌的浓度为6.72×106CFU/mL,猪肉增菌液的浓度为4.87×107CFU/mL。取不同浓度的甲型副伤寒沙门菌分别和两种增菌液混匀,提取混合菌的基因组DNA,进行PCR反应。如表4所示,当鸡肉背景菌群浓度为4.16×106CFU/mL的中混有6.43×104CFU/mL的甲型副伤寒沙门菌时,该反应体系可以有效的扩增2条条带,检测结果为阳性,当甲型副伤寒沙门菌浓度为6.43×103CFU/mL,只有139-141引物获得扩增条带,说明样品中含有沙门菌但不确定所含的沙门菌是甲型副伤寒沙门菌,检测结果为阴性。如表5所示,当猪肉背景菌群浓度为4.87×107CFU/mL的中混有6.43×104CFU/mL的甲型副伤寒沙门菌时,该反应体系可以有效的扩增2 条条带,检测结果为阳性。
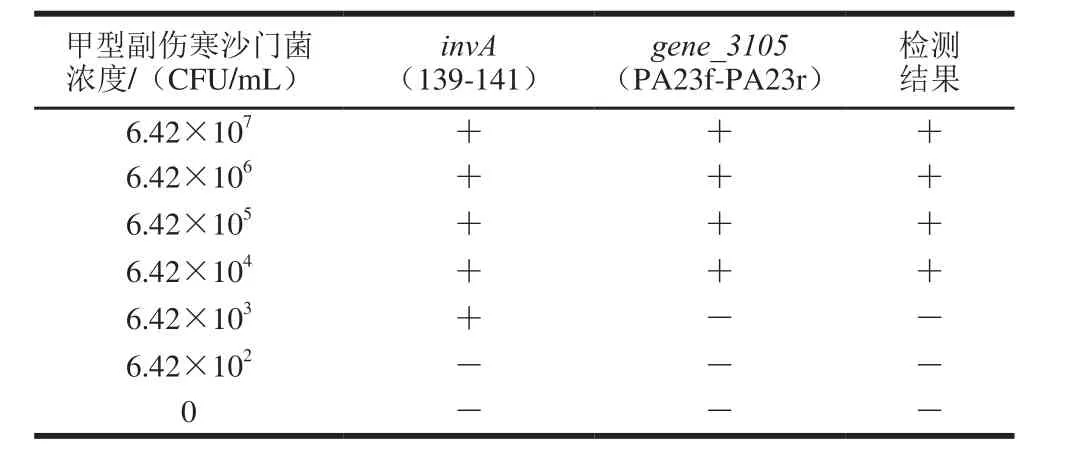
表4 鸡肉背景菌群的存在对甲型副伤寒沙门菌检测的影响Table 4 Detection of S. Paratyphi A from a natural background fl ora in chicken

表5 猪肉背景菌群的存在对甲型副伤寒沙门菌检测的影响Table 5 Detection of S. Paratyphi A from a natural background fl ora in pork
2.6 PCR检测体系的人工污染评价
将不同浓度的甲型副伤寒沙门菌分别接入无菌的鸡胸肉和猪肉样品,混匀后再转入到225 mL的LB液体培养基中,37 ℃、180 r/min增菌6、8、10 h和12 h。每份样品取1 mL增菌液,提取基因组DNA,进行PCR检测。在鸡肉样品,甲型副伤寒沙门菌污染的浓度分别7.53×103、7.53×102、7.53×101CFU和7.53 CFU,当增菌培养10 h以后,各个浓度污染的样品检出率均为100%。而增菌6 h,只能检测出初始污染浓度为75.3 CFU/25 g的鸡肉样品,结果见表6。在猪肉样品,甲型副伤寒沙门菌污染浓度分别为3.24×103、3.24×102、3.24×101CFU和3.24×100CFU,当增菌培养10 h以后,各个浓度污染的样品检出率均为100%。而增菌6 h,只能检测出初始污染浓度为3.24×102CFU/25 g的猪肉样品,结果见表7。综上所述,对鸡肉和猪肉样品增菌培养10 h,该PCR检测体系对甲型副伤寒沙门菌的检出限能达到N CFU/25 g(0<N<10)。
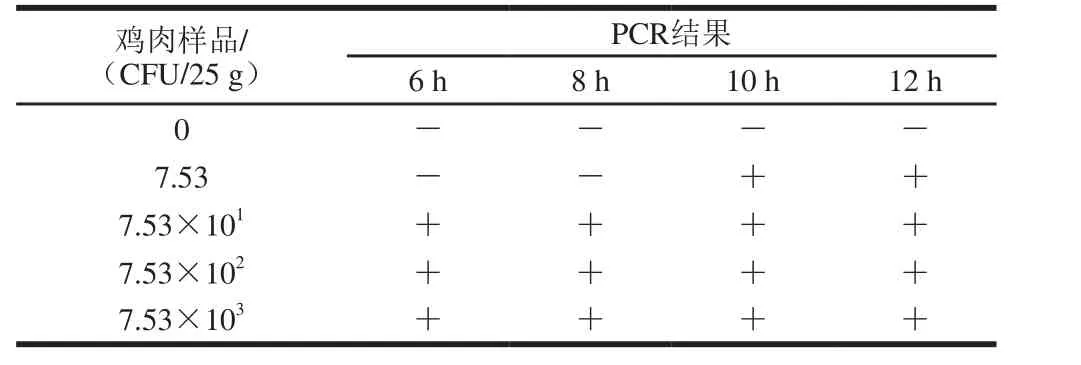
表6 甲副伤寒沙门菌的人工污染鸡肉样品的检测Table 6 Detection of S. Paratyphi A in artif i cially contaminated chicken samples

表7 甲副伤寒沙门菌的人工污染猪肉样品的检测Table 7 Detection of S. Paratyphi A in artif i cially contaminated pork samples
3 讨 论
根据沙门菌的流行学特点,针对血清型的分子检测技术已经成为现阶段沙门菌检测的研究热点,而该检测手段的关键是寻找特异的检测靶点[23]。研究表明,可利用O抗原和H抗原的合成基因作为检测靶点,进行PCR鉴定[2,15,24]。Levy等[25]根据O抗原、Vi抗原和H抗原的编码基因作为检测靶点,建立2 个多重PCR反应体系检测血液中的伤寒沙门菌、甲型副伤寒沙门菌和乙型副伤寒沙门菌,特异性和灵敏度方面均能达到100%。然而,该方法需要多个检测靶点才能鉴定某个血清型,而且检测过程中需要两个多重PCR反应增加了检测过程的操作,近而增加了检测结果的判断难度;而且对于亲缘关系比较近的血清型进行鉴定时,将会干扰目标血清型的鉴定,造成假阳性结果的出现。有研究通过扩增STY4220基因,鉴定伤寒沙门菌和甲型副伤寒沙门菌,但是该扩增目标不能区分以上两种血清型[26]。本研究利用比较基因组的方法,筛选了甲型副伤寒沙门菌的特异性基因gene_3268、gene_3270、gene_3106和gene_3105,只存在于该血清型的代表菌株CMCC50973中,设计引物在实验室保存的甲型副伤寒沙门菌中能获得正确PCR扩增条带,但其他菌株中均无扩增条带。139-141引物的靶点是invA基因,编码沙门菌吸附和侵袭上皮细胞表面的侵染蛋白[27]。Rahn等[28]将invA基因作为检测靶点建立了沙门菌检测PCR反应体系,应用于112 个不同的沙门菌血清型,在643 个已知菌株中检出率达到99%,对于33 株非沙门菌的肠道菌仅扩增出非目的大小的片段。在欧盟,将invA基因作为沙门菌检测的标准PCR方法的靶基因[29]。本研究利用实验保存的菌株特异性验证表明,只有甲型副伤寒沙门菌的检测能够出现两条扩增条带,其他沙门菌血清型仅出现一条扩增条带,非沙门菌无扩增条带产生。不同DNA浓度的检测可直接反映出体系中给引物与DNA模板的结合能力,本研究建立的检测体系在DNA模板质量浓度为32.4 pg/μL时能获得清晰的2个扩增条带,与前人建立的多重PCR检测结果相似[15-16,30]。Grace等[26]以STY4220基因为靶点同时检测甲型副伤寒沙门菌和伤寒沙门菌,其特异性能达到100%,菌落灵敏度为4.5×104~5.5×104CFU/mL,该菌落灵敏度低于本研究建立的PCR检测方法的菌落灵敏度。在猪肉和鸡肉这类生鲜样品中含有大量的非检测目的菌群,这群微生物的存在将会对该PCR反应体系的检出限有一定的影响作用。通过抗干扰性评价分析,当猪肉和鸡肉的背景菌群的浓度达到106CFU/mL和107CFU/mL时,该反应体系仍能检测出混有104CFU/mL的甲型副伤寒沙门菌,以上说明该检测体系具有较强的抗干扰能力。人工污染实验表明,在鸡肉和猪肉样品中,污染1 CFU/25 g的甲型副伤寒沙门菌经过10 h的增菌培养,PCR反应体系可获得阳性结果。但是为了保证检测结果的准确性,可将增菌时间延长到12 h。
综上所述,本研究所鉴定的甲型副伤寒沙门菌检测靶点特异性较好,建立的PCR检测体系具有较高的灵敏度,能够有效抵抗背景菌株的干扰,提供检测的准确性,为甲型副伤寒沙门菌的检测提供一种快速、灵敏、准确的方法。
[1] WAIN J, KEDDY K H, HENDRIKSEN R S, et al. Using next generation sequencing to tackle non-typhoidal Salmonella infections[J]. The Journal of Infection in Developing Countries, 2013,7(1): 1-5. DOI:10.3855/jidc.3080.
[2] CHESSA D, SPIGA L, DE RIU N, et al. Lipopolysaccharides belonging to different Salmonella serovars are differentially capable of activating Toll-like receptor 4[J]. Infection and Immunity, 2014,82(11): 4553-4562. DOI:10.1128/iai.02297-14 .
[3] DE OLIVEIRA ELIAS S, TOMASCO P V, ALVARENGA V O, et al.Contributor factors for the occurrence of salmonellosis during preparation, storage and consumption of homemade mayonnaise salad[J]. Food Research International, 2015, 78: 266-273.DOI:10.1016/j.foodres.2015.09.034.
[4] MAJOWICZ S E, MUSTO J, SCALLAN E, et al. The global burden of nontyphoidal Salmonella gastroenteritis[J]. Clinical Infectious Diseases, 2010, 50(6): 882-889. DOI:10.1086/650733.
[5] SCALLAN E, MAJOWICZ S E, HALL G, et al. Prevalence of diarrhoea in the community in Australia, Canada, Ireland, and the United States[J]. International Journal of Epidemiology, 2005, 34(2):454-460. DOI:10.1093/ije/dyh413.
[6] KOSEK M, BERN C, GUERRANT R L. The global burden of diarrhoeal disease, as estimated from studies published between 1992 and 2000[J]. Bulletin of the World Health Organization, 2003, 81(3):197-204. DOI:10.1097/00001648-200007000-00439.
[7] 梅鲜艳. 95 株甲型副伤寒沙门氏菌对21 种抗生素的药敏分析[J].大家健康(学术版), 2014, 8(8): 168. DOI:1009-6019(2014)04-0168-01.
[8] 周建芳, 杨珊明. 甲型副伤寒流行特点及细菌耐药性监测[J]. 中华预防医学杂志, 2004, 38(2): 26. DOI:10.3760/j:issn:0253-9624.2004.02.025.
[9] 陈冬娥, 陈冠武, 许如苏, 等. 沙门氏菌比对试验的检验分析[J].食品与生物技术学报, 2016, 35(5): 556-559. DOI:10.3969/j.issn.1673-1689.2016.05.017
[10] NIELSEN L R. Review of pathogenesis and diagnostic methods of immediate relevance for epidemiology and control of Salmonella Dublin in cattle[J]. Veterinary Microbiology, 2012, 162(1): 1-9.DOI:10.1016/j.vetmic.2012.08.003.
[11] 李昱辰, 程瞾, 蔡银强, 等. 沙门菌分型技术研究进展[J]. 中国动物检疫, 2014, 31(6): 32-35. DOI:10.3969/j.issn.1005-944X.2014.06.011.
[12] ISSENHUTH JEANJEAN S, ROGGENTIN P, MIKOLEIT M, et al. Supplement 2008-2010 (no. 48) to the White-Kauffmann-Le Minor scheme[J]. Research in Microbiology, 2014, 165(7): 526-530.DOI:10.1016/j.resmic.2014.07.004.
[13] ZHUANG L, GONG J, LI Q, et al. Detection of Salmonella spp. by a loop-mediated isothermal amplification (LAMP) method targeting bcfD gene[J]. Letters in Applied Microbiology, 2014, 59(6): 658-664.DOI:10.1111/lam.12328.
[14] LIU C C, YEUNG C Y, CHEN P H, et al. Salmonella detection using 16S ribosomal DNA/RNA probe-gold nanoparticles and lateral flow immunoassay[J]. Food Chemistry, 2013, 141(3): 2526-2532.DOI:10.1016/j.foodchem.2013.05.089.
[15] KUMAR P P, AGARWAL R K, THOMAS P, et al. Rapid detection of Salmonella enterica subspecies enterica serovar Typhimurium by loop mediated isothermal amplification (LAMP) test from field chicken meat samples[J]. Food Biotechnology, 2014, 28(1): 50-62.DOI:10.1016/j.foodchem.2013.05.089.
[16] SAEKI E K, ALVES J, BONFANTE R C, et al. Multiplex PCR (mPCR)for the detection of Salmonella spp. and the differentiation of the Typhimurium and Enteritidis serovars in chicken meat[J]. Journal of Food Safety, 2013, 33(1): 25-29. DOI:10.1111/jfs.12019.
[17] SHARAN R, CHHIBBER S, REED R H. Inactivation and sub-lethal injury of Salmonella Typhi, Salmonella Typhimurium and Vibrio cholerae in copper water storage vessels[J]. BMC Infectious Diseases,2011, 11(1): 204. DOI:10.1186/1471-2334-11-204.
[18] 程希, 胡跃明, 郭志荣, 等. 沙门氏菌Ⅲ型分泌候选基因yiiG的比较基因组分析[J]. 基础医学与临床, 2017, 37(1): 43-49. DOI:10.3969/j.issn.1001-6325.2017.01.015.
[19] 陈朋, 晏政, 李红玉. 嗜酸氧化亚铁硫杆菌基因组学研究进展[J]. 微生物学免疫学进展, 2017(2): 1-5. DOI:10.13309/j.cnki.pmi.2017.02.015.
[20] 向雪菲. 食品中沙门氏菌分子检测靶点的筛选与评价[D]. 上海:上海交通大学, 2008.
[21] 刘斌. 沙门氏菌血清分型分子靶点的发掘及鉴定体系的建立[D].上海: 上海交通大学, 2012.
[22] LIU B, ZHANG L D, ZHU X N, et al. PCR identification of Salmonella serogroups based on specific targets obtained by comparative genomics[J]. International Journal of Food Microbiology,2011, 144(3): 511-518. DOI:10.1016/j.ijfoodmicro.2010.11.010.
[23] LIU B, ZHANG L D, ZHU X N, et al. Development of a novel multiplex PCR assay for the identification of Salmonella enterica Typhimurium and Enteritidis[J]. Food Control, 2012, 27(1): 87-93.DOI:10.1016/j.foodcont.2012.01.062.
[24] LIM Y H, HIROSE K, LZUMIYA H, et al. Multiplex polymerase chain reaction assay for selective detection of Salmonella enterica serovar Typhimurium[J]. Japanese Journal of Infectious Diseases,2003, 56(4): 151-155.
[25] LEVY H, DIALLO S, TENNANT S M, et al. PCR method to identify Salmonella enterica serovars Typhi, Paratyphi A, and Paratyphi B among salmonella isolates from the blood of patients with clinical enteric fever[J]. Journal of Clinical Microbiology, 2008, 46(5):1861-1866. DOI:10.1128/JCM.00109-08.
[26] NGAN G J Y, NG L M, LIN R T P, et al. Development of a novel multiplex PCR for the detection and differentiation of Salmonella enterica serovars Typhi and Paratyphi A[J]. Research in Microbiology,2010, 161(4): 243-248. DOI:10.1016/j.resmic.2010.03.005.
[27] ANEJO-OKOPI J A, ISA S E, AUDU O, et al. Isolation and polymerase chain reaction detection of virulence invA gene in Salmonella spp. from poultry farms in Jos, Nigeria[J]. Journal of Medicine in the Tropics, 2016,18(2): 98. DOI:10.4103/2276-7096.192237.
[28] RAHN K, DEGRANDIS S A, CLARKE R C, et al. Amplicif i cation of an invA gene sequence of Salmonella Typhimurium by polymerase chain reaction as a specific method of detection of Salmonella[J]. Molecular and Cellular Probes, 1992, 6(4): 271-279.DOI:10.1016/0890-8508(92)90002-f .
[29] ZHAI L G, YU Q, BIE X M, et al. Development of a PCR test system for specific detection of Salmonella Paratyphi B in foods[J]. FEMS Microbiology Letters, 2014, 355(1): 83-89. DOI:10.1111/1574-6968.12443.
[30] GONZALEZ-ESCALONA N, BROWN E W, ZHANG G.Development and evaluation of a multiplex real-time PCR (qPCR)assay targeting ttrRSBCA locus and invA gene for accurate detection of Salmonella spp. in fresh produce and eggs[J]. Food Research International, 2012, 49(1): 618-618. DOI:10.1016/j.foodres.2012.03.009.
Mining of New Specif i c Molecular Targets and Development of a PCR Assay for Specif i c Detection of Salmonella Paratyphi A
ZHAI Ligong, WANG Junying, ZHANG Xiaoyu, MENG Xin, CUI Bao, ZHAO Wanqing, NIU Ping
(College of Food and Drug, Anhui Science and Technology University, Chuzhou 233100, China)
In this study, four serotype-specif i c genes of Salmonella Paratyphi A were identif i ed by comparative genomics and PCR. A PCR assay based on the gene_3105 and invA gene was developed and evaluated for the detection of S. Paratyphi A. The electrophoresis pattern showed only two bright specif i c bands at 284 bp and 384 bp in S. Paratyphi A. The PCR protocol was optimized and the specif i city, sensitivity, anti-jamming capability and limit of detection (LOD) for artif i cially contaminated food were evaluated.The specif i city results showed two bright specif i c bands for S. Paratyphi A, only one specific band at 284 bp for other Salmonella serotypes, no specific bands for non-Salmonella strains. The sensitivity of the PCR assay was 32.4 pg/µL and 4.3 × 103CFU/mL for genomic DNA and pure colonies, respectively. In the presence of natural background fl ora enriched from chicken and pork breast samples, the detection limit was 6.43 × 104CFU/mL.In artif i cially contaminated chicken and pork, the detection limit was N CFU/25 g after 10 h enrichment (0 < N < 10). In conclusion, the PCR assay for the detection of S. Paratyphi A is specif i c and sensitive, and has a good application value and can be widely used in the fi eld of food safety.
Salmonella Paratyphi A; comparative genomics; serotype-specif i c genes; PCR
10.7506/spkx1002-6630-201802024
TS201.6
A
1002-6630(2018)02-0151-07
翟立公, 王俊颖, 张小雨, 等. 甲型副伤寒沙门菌特异性基因筛选及PCR检测体系建立[J]. 食品科学, 2018, 39(2):151-157.
10.7506/spkx1002-6630-201802024. http://www.spkx.net.cn
ZHAI Ligong, WANG Junying, ZHANG Xiaoyu, et al. Mining of new specif i c molecular targets and development of a PCR assay for specif i c detection of Salmonella Paratyphi A[J]. Food Science, 2018, 39(2): 151-157. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201802024. http://www.spkx.net.cn
2017-04-17
安徽高校自然科学研究重点项目(KJ2016A182);安徽科技学院人才引进项目(SPYJ201602);
安徽科技学院大学生创新项目(16ZCX34)
翟立公(1983—),男,讲师,博士,研究方向为食源性致病菌检测。E-mail:gavin340@126.com
