干旱胁迫棉花转录组DNA损伤修复相关基因的分析
2017-12-28新疆大学生命科学与技术学院新疆生物资源基因工程重点实验室乌鲁木齐830046
(新疆大学生命科学与技术学院/新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
doi:10.6048/j.issn.1001-4330.2017.11.005
干旱胁迫棉花转录组DNA损伤修复相关基因的分析
包秋娟,张丽丽,海那尔·乌拉孜巴依,张富春
(新疆大学生命科学与技术学院/新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
目的研究干旱胁迫下,棉花 DNA 损伤修复基因的表达情况,从整体水平探讨棉花DNA损伤修复相关基因表达与抗旱的相关性。方法用2.5%PEG6000处理棉花幼苗,利用RNA-Seq技术对干旱胁迫下棉花幼苗的转录组进行测序。结果从棉花干旱胁迫响应的转录组中,筛选获得差异表达的 DNA损伤修复相关基因共51个,其中差异表达的上调基因23个,差异表达的下调基因28个,干旱胁迫能够影响棉花DNA损伤修复相关基因的表达。选取4个差异基因进行生物信息学分析及qRT-PCR验证,HMGB1、recA1、UDGs和GMP synthase基因的表达变幅有一定的差异,但基因的表达趋势一致,棉花转录组测序结果通过qRT-PCR验证是可靠的。结论DNA损伤修复相关基因可能与干旱胁迫有一定的相关性,干旱胁迫能够影响棉花DNA损伤修复相关基因的表达。
棉花;干旱胁迫;转录组测序;DNA损伤修复相关基因;差异表达
0 引 言
【研究意义】植物在正常生存条件下基因组DNA分子结构非常稳定,但在各种生物及非生物逆境胁迫下DNA 极易遭到损伤[1]。由于植物固着土壤的生存方式使植物不能像动物一样避逆,长期不断地暴露在各种非生物和生物胁迫之下,导致DNA受到损伤,包括DNA碱基氧化和烷基化,形成嘧啶二聚体,DNA交联,DNA单链断裂(SSB)和DNA双链断裂(DSB),使得植物维护基因组DNA完整性存在严峻挑战,为了确保基因组结构完整和化学性质稳定,植物已经进化出了一系列DNA损伤修复机制[2-6]。DNA损伤修复途径主要有直接修复途径、碱基切除修复途径(BER)、核苷酸切除修复途径(NER)、碱基错配修复途径(MMR)、同源重组修复途径(HR)和非同源的末端连接途径(NHEJ)[7-9]。其中BER是修复由于氧化、烷基化、脱氨和脱嘌呤/脱嘧啶引起的碱基损伤的主要修复途径,主要针对碱基改变较轻微的DNA损伤[10-12]。DSBs是细胞最严重的一种损伤类型,能直接导致细胞凋亡,甚至使细胞死亡[13-14]。修复DNA双链断裂损伤主要有两种方式: 同源重组修复(HR),主要是低等真核生物修复方式,另一种是非同源末端连接修复(NHEJ)主要是脊椎动物及高等植物修复方式[15-17]。转录组测序技术(RNA-Seq)可检测某一特定时间或处理的生物体中所有转录本,利用RNA-Seq技术可在整体水平研究干旱胁迫下DNA损伤修复基因的表达情况。【前人研究进展】陈欢[18]利用微阵列转录组数据分析表明耐辐射球菌DNA修复是呈阶段性的,并且氧化胁迫可使DNA修复基因上调表达。Mishra等[19]利用转录组学分析黑腹果蝇铬诱导的DNA链断裂及修复,证实了NHEJ途径参与DNA双链断裂修复。【本研究切入点】目前研究 DNA 损伤修复机制主要集中于动物和原核生物,植物DNA 损伤修复机制的研究起步较晚。研究棉花DNA损伤修复在抗旱中的作用,对于棉花抗逆性研究具有十分重要的作用。【拟解决的关键问题】研究采用RNA-seq 测序技术获得棉花干旱胁迫响应的转录组数据,从棉花DNA 损伤应激基因入手研究植物耐旱机制,并使用荧光定量PCR验证在干旱胁迫条件下基因的表达情况,为研究棉花DNA损伤修复途径响应干旱胁迫奠定基础。
1 材料与方法
1.1 材 料
选取籽粒饱满、大小一致的棉花新陆早17号种子用70%乙醇处理 1 min 后用灭菌水冲洗7~8次,再用15%过氧化氢浸泡5 h后用灭菌水冲洗5~6次,将洗净的棉种浸泡至灭菌水中24 h后,将棉花种皮剥去播种到固体MS培养基中,进行无菌培养,一周后将棉花幼苗转至Hoagland营养液中继续培养,一周更换一次营养液直至植株处于4~5片真叶时选长势相近的6株棉花,3株作为对照(在培养瓶中加入营养液),3株用2.5%PEG6000胁迫营养液处理,均处理36 h后迅速置于液氮中,于-80℃冰箱中保存。棉花种子由新疆农业科学院经济作物研究所提供。
1.2 方 法
1.2.1 转录组数据库中差异表达的DNA损伤修复相关基因
采用DESeq进行差异基因分析,将变化倍数(Fold Chang)≥2且错误发现率(FDR<0.05)作为筛选标准,检索棉花干旱转录组中参与DNA损伤修复途径相关的差异基因。
1.2.2 DNA损伤修复相关基因的生物信息学
用ProtParam (http://web.expasy.org/protparam/)分析氨基酸序列的理化性质; NCBI保守结构域数据库CDD(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsbcgiRID=1K7GNVX3015&mode=all)分析预测氨基酸序列的保守结构域;PSORTⅡ(http://www.genscript.com/tools/psort)预测蛋白质的亚细胞定位;SignalP4.1(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽;TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)分析蛋白质的跨膜结构;GOR4(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_gor4.pl)预测α-螺旋、β-折叠和无规则卷曲等在整体结构中所占的比例。
1.2.3 实时荧光定量PCR
用NCBI Primer-blast在线设计引物,以Gh-EF1α作为内参基因(引物见下表)。使用 ABI 7500 Real Time荧光定量PCR 仪,参照QIAGEN的 SYBR Green RT-PCR 试剂盒说明书进行荧光定量检测。Real-time PCR反应体系为20 μL:SYBR Green 10 μL,上下游引物各0.3 μL,模板c DNA 1.0 μL,RNase-free dd H2O 8.4 μL。荧光定量参数设置为: 95℃ 2 min;95℃15 s;60℃退火30 s;72℃延伸30 s(实时荧光信号采集);40个循环;采用 2-△△Ct相对定量法计算基因的相对表达量。
2 结果与分析
2.1 棉花DNA损伤修复途径响应干旱差异基因
从转录组数据共筛选获得差异表达的 DNA损伤修复相关基因51个,其中差异表达的上调基因23个,差异表达的下调基因28个。从DNA损伤修复途径中挑选4个差异表达基因进行后续分析。表1
表1 DNA损伤修复相关基因
Table 1 Genes of DNA damage repair

基因IDGeneID描述DescriptionFDR值FDRlog2变化倍数log2FC调控Regulatedevm.TU.Gh_D01G2231HighmobilitygroupB1(HMGB1)0.0084041.497295upevm.TU.Gh_A01G0250DNArepairproteinrecA1(recA1)0.000112.00427upevm.TU.Gh_D05G1136DNAglycosylasesuperfamilyprotein(UDGs)0.00875-1.6972downevm.TU.Gh_A05G1018GMPsynthase0.044075-1.97657down
2.2 DNA损伤修复相关基因的生物信息学
2.2.1氨基酸一级结构及理化性质预测
高迁移率族蛋白(HMGB1)ORF为519 bp,共编码172个氨基酸,预测分子量为19 522.37 Da,等电点为5.31,为不稳定的蛋白; DNA修复蛋白recA 1(recA1)ORF为2 115 bp,共编码704个氨基酸,预测分子量为77 553.62 Da,等电点为8.04,为稳定蛋白;DNA糖基化酶超家族蛋白(UDGs)ORF为1 140 bp,共编码379个氨基酸,预测分子量为41 082.74 Da,等电点为9.49,为不稳定的蛋白;鸟嘌呤一磷酸合成酶(GMP synthase)ORF为1 116 bp,共编码371个氨基酸,预测分子量为40 225.78 Da,等电点为9.38,为不稳定蛋白。表2
表2 氨基酸理化性质预测
Table 2 Prediction of Physical and Chemical Properties of Amino Acids
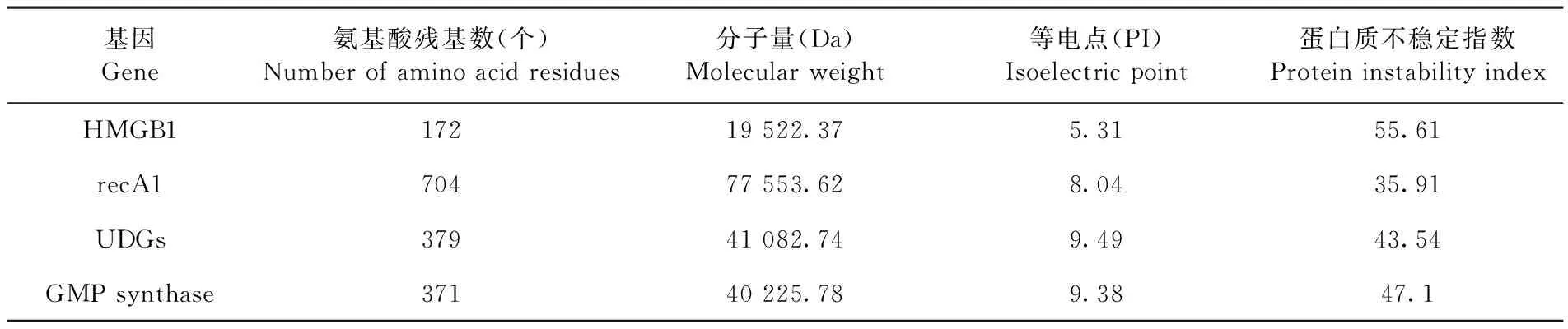
基因Gene氨基酸残基数(个)Numberofaminoacidresidues分子量(Da)Molecularweight等电点(PI)Isoelectricpoint蛋白质不稳定指数ProteininstabilityindexHMGB117219522.375.3155.61recA170477553.628.0435.91UDGs37941082.749.4943.54GMPsynthase37140225.789.3847.1
2.2.2 保守结构域预测
在线预测分析HMGB1、recA1、UDGs和GMP synthase的保守结构域,结果表明,HMGB1有一个HMG-box超家族,在55~110个氨基酸的位置有DNA结合位点,提示HMGB1作为转录因子可能对其他基因具有非常重要的调控作用。recA1有一个Rad51超家族和NAM超家族。UDGs和GMP synthase均有一个Ademine glyco超家族。
2.2.3 亚细胞定位、信号肽和跨膜结构域的预测
对HMGB1、recA1、UDGs和GMP synthase在线预测,结果表明,HMGB1主要定位于细胞核,占总体的82.6%,线粒体和分泌系统的囊泡也有分布,不具有信号肽,不存在跨膜螺旋区,为非跨膜蛋白。recA1主要定位于细胞核,占总体的65.2%,过氧化物酶体和线粒体中也有分布,不具有信号肽,为非跨膜蛋白。UDGs主要定位于细胞核,占总体的78.3%,细胞质和过氧化物酶体中也有分布,不具有信号肽,为非跨膜蛋白。GMP synthase主要定位于细胞核,占总体的78.3%,细胞质、过氧化物酶体和细胞骨架中也有分布,不具有信号肽,为非跨膜蛋白。
2.2.4 二级结构预测
蛋白质的二级结构主要有 α 螺旋、延伸链、 β折叠,无规则卷曲等。用GOR4在线预测二级结构(),HMGB1二级结构中,α 螺旋占的比例最高为 58.14%,延伸链占 38.37%,无规则卷曲占最少比例为3.49%。recA1二级结构中,无规则卷曲占的比例最高为 52.56%,延伸链占 18.18%,α 螺旋占29.06%。UDGs二级结构中,无规则卷曲占的比例最高为 53.56%,延伸链占 21.64%,α 螺旋占24.8%。GMP synthase二级结构中,无规则卷曲占的比例最高为 52.56%,延伸链占 23.18%,α 螺旋占24.26%。表3
表3 二级结构预测
Table 3 Secondary structure prediction

基因Geneα-螺旋α-helix延伸链Extensionchain无规则卷曲IrregularcurlHMGB1100(58.14%)66(38.37%)6(3.49%)recA1206(29.06%)128(18.18%)370(52.56%)UDGs94(24.8%)82(21.64%)203(53.56%)GMPsynthase90(24.26%)86(23.18%)195(52.56%)
2.2.5 实时荧光定量PCR
采用qRT-PCR方法, 分别检测对照和处理棉花叶片中HMGB1、recA1、UDGs和GMP synthase mRNA 转录水平的变化。结果表明,HMGB1和recA1在干旱胁迫后表达量呈上调,而UDGs和GMP synthase表达量呈下调。qRT-PCR与转录组测序结果比较表明,在干旱胁迫下棉花HMGB1、recA1、UDGs和GMP synthase基因的表达变幅有一定的差异,但基因的表达趋势一致,表明棉花转录组测序结果通过qRT-PCR验证是可靠的。图1,图2
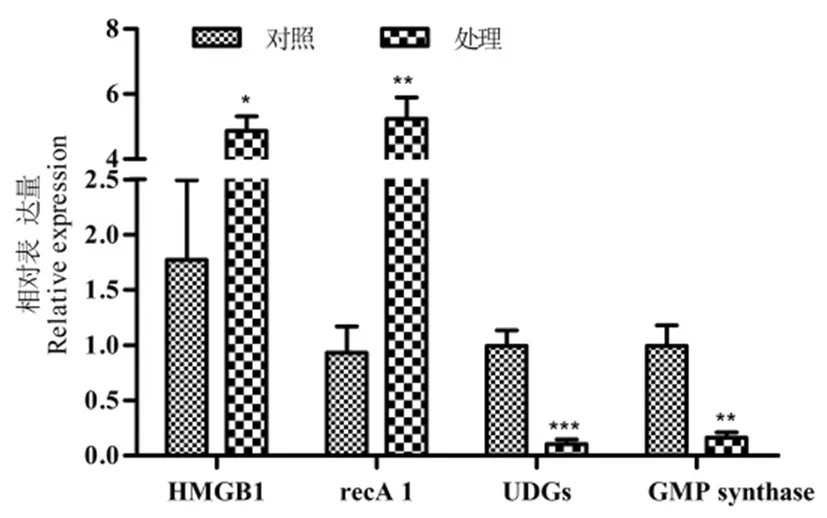
注:***表示P<0.001,**表示P<0.01,*表示P<0.05
Note:***indicateP<0.001,**indicateP<0.01,*indicateP<0.05
图1 差异表达基因的qRT-PCR验证
Fig.1 qRT-PCR validation of differentially expressed genes

图2 qRT-PCR验证RNA-Seq结果
Fig.2 qRT-PCR was used to validate RNA-Seq results
3 讨 论
植物在离子胁迫、氧化胁迫和渗透胁迫下都会导致DNA损伤变异[20],导致细胞和遗传毒害。目前的研究多集中在某一修复途径[21-22],而在整体水平研究DNA 损伤修复机制还鲜见报道。研究利用RNA-seq 测序技术,分析DNA损伤应激通路相关基因响应干旱胁迫的表达情况,从转录组数据共筛选获得差异表达的 DNA损伤修复相关基因51个,说明干旱胁迫对棉花DNA有一定的损伤,能够影响棉花DNA损伤修复相关基因的表达,破坏DNA的修复系统,提示DNA损伤修复相关基因可能与干旱胁迫有一定的相关性。其中被GO和KEGG注释的差异基因参与碱基切除修复的基因最多(8个),其次是参与同源重组修复(2个),参与核苷酸切除修复、非同源的末端连接途径和错配修途径中无差异表达基因,这表明2.5%PEG6000模拟干旱可能导致DNA发生轻微损伤,部分DNA发生双链断裂。能被注释到GO和KEGG数据库中参与BER、NER、MMR、HR和NHEJ途径的差异表达基因并不多,只占转录组数据库中与DNA损伤修复相关差异基因的10%,其余注释为DNA修复(DNA repair)、细胞响应DNA损伤(Cellular respense to DNA damage)和DNA损伤检验点(DNA damage chockpoint),说明目前对DNA损伤修复途径相关基因的注释还不全面。随机选取4个差异基因进行qRT-PCR验证,结果表明,干旱诱导HMGB1和recA1基因的表达,抑制UDGs和GMP synthase基因的表达。高迁移率族蛋白(HMGB)因其相对分子量小在电泳中迁移率高而得名[23],是三类高迁移率组(HMG)染色体蛋白之一,能弯曲DNA并优先结合扭曲的DNA,它作为核蛋白复合物装配中的建筑促进者可以实现重组和启动转录[24]。已有研究发现几乎所有的HMGB都可以修饰、弯曲或改变染色质/DNA的结构,从而调节基因转录[25-27]。Ito[28]等研究表明HMGB1参与小鼠核和线粒体DNA损伤修复,Mukherjee和Lange 等[29-30]说明了HMGB1参与NER、MMR、HNEJ和BER损伤修复途径,是一个精通DNA修复的全能蛋白。在转录组数据库中,对HMGB1的注释只参与了BER途径,说明注释数据库有待于进一步完善。recA1作为DNA重组修复蛋白能在重组修复蛋白recBCD或recFOR的作用下参与DNA的重组修复[31],它是同源重组的一个重要组成部分[32]。多种DNA 糖基化酶能够特异性识别、结合DNA的损伤部位,并移除受损伤的碱基,暴露出一个位点,即AP位点[33-34]。AP位点在其内切酶的作用下形成了核苷酸缝隙,之后在DNA 多聚酶 β作用下填补缝隙,最后在连接酶的复合体的作用下密封断开的DNA,完成BER修复[35]。在转录组中,只筛选到一个差异表达的DNA 糖基化酶基因,且为下调,提示在干旱胁迫36 h后,碱基切除修复过程已经完成了识别、结合过程。GMP synthase是一种能使细胞生长和DNA复制的重要核苷酸生物合成酶,Hosman等猜测核苷酸生物合成酶和转录调控之间的关联可能对细胞抵御基因组不稳定是重要的[40]。目前对GMP synthase如何参与DNA修复的过程还未见报道,转录组数据对GMP synthase的注释为参与碱基切除修复。对HMGB1、recA1、UDGs和GMP synthase的生物信息学分析表明HMGB1、UDGs和GMP synthase主要定位于细胞核,不含跨膜区域且没有信号肽,属于胞内不稳定蛋白,recA1主要定位于细胞核,是无信号肽稳定的非跨膜蛋白。HMGB1有一个HMG-box超家族,说明HMGB1可能作为转录因子发挥作用,二级结构预测其α螺旋占的比例最高,其次是延伸链,最后是无规则卷曲。recA1有Rad51超家族和NAM超家族,说明recA1参与减数分裂和DNA重组修复,二级结构预测其无规则卷曲占的比例最高,其次是延伸链,最后是α螺旋。UDGs和GMP synthase均有一个Ademine glyco超家族,推测其参与碱基切除修复,二级结构预测其无规则卷曲占的比例最高。实验的结果为干旱胁迫下棉花DNA损伤修复机制的研究提供了响应的数据。
DNA损伤修复是生物维护自身基因组稳定而做出的反应,DNA损伤修复过程是复杂的并呈现动态变化。虽然目前对DNA损伤修复的研究越来越成熟,但还是有很多问题没有得到解释。
4 结 论
研究采用RNA-seq 测序技术获得棉花干旱胁迫响应的转录组数据,分析了干旱胁迫对DNA损伤修复系统的影响,筛选出DNA损伤修复相关差异表达基因共51个,其中差异表达的上调基因23个,差异表达的下调基因28个,选取4个差异基因进行qRT-PCR验证,证明与转录组表达趋势一致,DNA损伤修复基因受干旱胁迫影响,其中下调基因略多于上调基因表明干旱对DNA损伤修复途径有一定的破坏。干旱胁迫能够影响棉花DNA的损伤修复,该研究的结果为干旱胁迫下棉花DNA损伤修复机制的研究提供了基础。
参考文献(References)
[1] Bian, X. Y., Rasheed, M. S., Seemanpillai, M. J., & Ali, R. M. (2006). Analysis of silencing escape of tomato leaf curl virus: an evaluation of the role of dna methylation.Molecularplant-microbeinteractions:MPMI, 19(6): 614.
[2] 张冀, 杜驰, 张富春. 盐胁迫下盐穗木DNA聚合酶λ基因的克隆和表达分析[J]. 新疆农业科学, 2017, 54(2):361-370.
ZHANG Ji, DU Chi, ZHANG Fu-chun. (2017). .Cloning and Expression Analysis of HcDNA pol λ Gene from Halostachys caspica under Salt Stress [J].XinjiangAgriculturalSciences, 54(2):361-370. (in Chinese)
[3] Kozak, J., West, C. E., White, C., Da, C. N. J., & Angelis, K. J. (2009). Rapid repair of dna double strand breaks in arabidopsis thaliana is dependent on proteins involved in chromosome structure maintenance.DnaRepair, 8(3): 413-419.
[4] Sujit, R. (2014). Maintenance of genome stability in plants: repairing DNA double strand breaks and chromatin structure stability.FrontiersinPlantScience, (5): 487.
[5] West, C. E., Waterworth, W. M., Sunderland, P. A., & Bray, C. M. (2004). Arabidopsis dna double-strand break repair pathways.BiochemicalSocietyTransactions, 32(6): 946-964.
[6] Roy, S., Choudhury, S. R., Singh, S. K., & Das, K. P. (2011). Atpolλ, a homolog of mammalian dna polymerase λ in arabidopsis thaliana, is involved in the repair of uv-b induced dna damage through the dark repair pathway.Plant&CellPhysiology, 52(2): 448-467.
[7] Furukawa, T., Angelis, K. J., & Britt, A. B. (2015). Arabidopsis dna polymerase lambda mutant is mildly sensitive to dna double strand breaks but defective in integration of a transgene.FrontiersinPlantScience, (6): 357.
[8] Amiard, S., Gallego, M. E., & White, C. I. (2013). Signaling of double strand breaks and deprotected telomeres in arabidopsis.FrontiersinPlantScience, 4(1): 405.
[9] Peng, Y., Allen, S., Millwood, R. J., & Jr, S. C. (2014). 'fukusensor:' a genetically engineered plant for reporting dna damage in response to gamma radiation.PlantBiotechnologyJournal, 12(9): 1,329-1,332.
[10] Robertson, A. B., Klungland, A., Rognes, T., & Leiros, I. (2009). DNA repair in mammalian cells: base excision repair: the long and short of it.Cellular&MolecularLifeSciencesCmls, 66(6): 981.
[11] Ho, T. V., & Schärer, O. D. (2010). Translesion DNA synthesis polymerases in dna interstrand crosslink repair.Environmental&MolecularMutagenesis, 51(6): 552.
[12] Dantzer, F., Rubia, G. D. L., Murcia, J. M., Zdenek Hostomsky, ≠., And, G. D. M., & Schreiber, V. (2000). Base excision repair is impaired in mammalian cells lacking poly (ADP-ribose) polymerase-1.Biochemistry, 39(25):7,559-7,569.
[13] 李蔚蔚,孔金昕,漆永梅,等. 非同源末端连接修复相关因子对DNA损伤修复调控及肿瘤治疗作用的研究进展[J]. 中国药理学与毒理学杂志,2015,29(4):607-613.
LI Wei-wei, KONG Jin-Xin, QI Yong-Mei, et al.(2015). Regulation of nonhomologous end-joining-related factors in DNA damage repair and tumor treatment [J].ChineseJournalofPharmacologyandToxicology,29(4):607-613. (in Chinese)
[14] Yamtich, J., & Sweasy, J. B. (2010). DNA polymerase family x: function, structure, and cellular roles.BiochimicaEtBiophysicaActaProteins&Proteomics, 1804(5): 1,136-1,150.
[15] Breimer, L. H. (1990). Molecular mechanisms of oxygen radical carcinogenesis and mutagenesis: the role of dna base damage.MolecularCarcinogenesis, 3(4): 188-197.
[16] 李雨民. DNA损伤修复与细胞凋亡[J]. 国外医学(放射医学核医学分册),1999,23(3):17-20.
LI Yu-ming. (1999). DNA damage repair and apoptosis [J].ForeignMedicalSciences(SectionofRadiationMedicineandNuclearMedicine) , 23(3): 17-20. (in Chinese)
[17] Martin, M. J., Garcia-Ortiz, M. V., Gomez-Bedoya, A., Esteban, V., Guerra, S., & Blanco, L. (2013). A specific n-terminal extension of the 8 kda domain is required for dna end-bridging by human polμ and polλ.NucleicAcidsResearch, 41(19): 9,105-9,116.
[18] 陈欢. 耐辐射球菌极端条件下和重要基因突变后转录组的研究[D]. 杭州:浙江大学博士学位论文, 2008.
CHEN Huan.(2008).AnalysisofDeinococcusradiodurans'stranscriptionalresponsetoionizingradiation,hydrogenperoxide,andsomeimportanttranscriptregulatorsdisruption[D]. PhD Thesis. Zhejiang University, Hangzhou. (in Chinese)
[19] Mishra, M., Sharma, A., Shukla, A. K., Pragya, P., Murthy, R. C., & Pomerai, D. D., et al. (2013). Transcriptomic analysis provides insights on hexavalent chromium induced dna double strand breaks and their possible repair in midgut cells of drosophila melanogaster, larvae.MutationResearch, (1): 28-39.
[20] DeRose-Wilson L J. (2010). Genome Evolution and the Genetics of Abiotic Stress Tolerance in Arabidopsis thaliana and Arabidopsis lyrata .DissertaytionofUniversityofCalifornia,Irvine.
[21] Hirakawa, T., Hasegawa, J., White, C. I., & Matsunaga, S. (2017). Rad54 forms DNA repair foci in response to DNA damage in living plant cells.PlantJournal.
[22] Vu, G. T. H., Cao, H. X., Reiss, B., & Schubert, I. (2017). Deletion‐bias in dna double‐strand break repair differentially contributes to plant genome shrinkage.NewPhytologist, 214(4): 1,712.
[23] 冀芦沙, 于守超, 赵燕,等. 拟南芥高迁移率族蛋白B族基因表达模式分析[J]. 西北植物学报, 2012, 32(3):447-453.
JI Lu-sha, YU Shou-chao, ZHAO Yan.(2012)Expression Analysis of the Arabidopsis High Mobility Group Protein B Family Genes (AtHMGB) [J].ActaBotanicaBoreali-OccidentaliaSinica, 32(3): 447-453. (in Chinese)
[24] Thomas JO, & Travers AA. (2001). HMG1 and 2, and related 'architectural' dna-binding proteins.TrendsinBiochemicalSciences, 26(3): 167.
[25] And, J. L., & Grasser, K. D. (2001). Differential chromatin association and nucleosome binding of the maize hmga, hmgb, and ssrp1 proteins.Biochemistry, 40(26):7,860-7,867.
[26] Christian Stemmer., Silvia Fernández, §., Gema Lopez, §., And, J. C. A. §., & , K. D. G. (2002). Plant chromosomal hmgb proteins efficiently promote the bacterial site-specific β-mediated recombination in vitro and in vivo?.Biochemistry, 41(24): 7,763-7,770.
[27] Grasser, K. D., Launholt, D., & Grasser, M. (2007). High mobility group proteins of the plant hmgb family: dynamic chromatin modulators.BBA-GeneStructureandExpression, 1769(5): 346-357.
[28] Ito, H., Fujita, K., Tagawa, K., Chen, X., Homma, H., & Sasabe, T., et al. (2015). HMGB1 facilitates repair of mitochondrial dna damage and extends the lifespan of mutant ataxin-1 knock-in mice.EmboMolecularMedicine, 7(1):78.
[29] Mukherjee, R. M., Shravanti, G. V., Jakkampudi, A., Kota, R., Jangala, A. L., & Reddy, P. B., et al. (2013). Reduced expression of dna damage repair genes high mobility group box1 and poly(adp-ribose) polymerase1 in inactive carriers of hepatitis b virus infection-a possible stage of viral integration.JournalofClinical&ExperimentalHepatology, 3(2): 89-95.
[30] Lange, S. S., & Vasquez, K. M. (2009). HMGB1: the jack-of-all-trades protein is a master dna repair mechanic.MolecularCarcinogenesis,48(7): 571-580.
[31] 邱洁芳, 潘学峰. 功能型DNA重组修复蛋白质RecR的体内分布示踪 [J]. 自然科学进展, 2009, 19(7):704-710.
QIU Jie-fan, PAN Xue-feng. (2009). In vivo biodistribution of recombinant DNA repair protein RecR [J].ProgressinNaturalScience, 19(7): 704-710. (in Chinese)
[32] Odahara, M., Inouye, T., Fujita, T., Hasebe, M., & Sekine, Y. (2007). Involvement of mitochondrial-targeted reca in the repair of mitochondrial dna in the moss, physcomitrella patens.Genes&GeneticSystems,82(1):43-51.
[33] Lindahl, T. (1974). An n-glycosidase from escherichia coli that releases free uracil from dna containing deaminated cytosine residues.ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 71(9): 3,649-3,653.
[34] Sobol, R. W., Norton, J. K., Kühn, R., Gu, H., Singhal, R. K., & Prasad, R., et al. (1996). Requirement of mammalian dna polymerase-β in base-excision repair.Nature, 379(6561): 183.
[35] 刘博雅, 杨鑫, 任梦梦,等. DNA损伤修复机制-解读2015年诺贝尔化学奖[J]. 中国生物化学与分子生物学报, 2015, 31(12):1 322-1 329.
LIU Bo-ya, YANG Xin, RENG Meng-meng,et al.(2015). The Repair Mechanism for DNA Damage Understanding the 2015 Nobel Prize in Chemistry [J].ChineseJournalofBiochemistryandMolecularBiology, 31(12): 1,322-1,329. (in Chinese)
[36] Hosman, L. (2013). Abstract ia09: nucleotide biosynthetic enzyme gmp synthase is a relay of p53 stabilization in response to genomic stress.CancerResearch, 73(13 Supplement), IA09-IA09.
AnalysisofDNADamageRepairRelatedGenesinDroughtStressCottonTranscriptome
BAO Qiu-juan, ZHANG Li-li, Hainar Wulazibai, ZHANG Fu-chun
(CollegeofLifeScienceandTechnology,XinjiangKeyLaboratoryofBiologicalResourcesandGeneticEngineering,Urumqi830046,China)
ObjectiveThe study aims to investigate the expression of DNA damage repair related genes in cotton under drought stress, and to explore the correlation between the expression of DNA damage repair related genes and drought resistance at the whole level.Method2.5% PEG 6000 was used to treat cotton, and transcriptome was sequenced using RNA-Seq technology of cotton seedlings under drought stress.ResultA total of 51 genes with differentially expressed DNA damage were filtered from the transcripts of cotton drought stress response, among which there were 23 differentially up-regulated expressed genes and 28 differentially down-regulated expressed genes, which showed that drought stress could affect the expression of DNA repair related genes in cotton. 4 genes were selected by bioinformatics analysis and verification results showed that the expression of qRT-PCR, HMGB1, recA1, UDGs amplitude and GMP synthase gene displayed some differences, but the gene expression trends were consistent, showing that cotton transcriptome sequencing results verified by qRT-PCR were reliable.ConclusionThe DNA damage repair related genes may be related to drought stress, so drought stress could affect the expression of DNA damage related genes.
cotton; drought stress; transcriptome sequencing; DNA damage repair related gene; differential expression
Supported by: Joint Key Fund of National Natural Science Foundation of China and Xinjiang "Exploration of key genes related to high-efficiency water utilization in cotton and creation of drought resistant germplasm"(U1303282)
ZHANG Fu-chun (1962-), male, native place: Urumqi, Xinjiang. Professor,research field: Molecular biology. (E-mail)zfcxju@xju.edu.cn
S562;Q756
A
1001-4330(2017)11-1999-07
2017-09-28
国家自然科学基金-新疆联合基金重点项目“棉花水分高效利用的关键基因的挖掘及抗旱种质材料的创制”(U1303282)
包秋娟(1993-),女,新疆麦盖提人,硕士研究生,研究方向为植物分子生物学,(E-mail)1515643021@qq.com
张富春(1962-),男,新疆乌鲁木齐人,教授,博士,研究方向为分子生物学,(E-mail)zfcxju@xju.edu.cn
