茶树miR156a靶基因SPL6和SPL9的克隆及表达分析
2017-12-11刘亚芹田坤红孙琪璐潘铖李叶云蒋家月江昌俊
刘亚芹,田坤红,孙琪璐,潘铖,李叶云,蒋家月,江昌俊,2*
1. 安徽农业大学茶树生物学与资源利用国家重点实验室,安徽 合肥,230036;2. 信阳师范学院河南省茶树生物学重点实验室,河南 信阳,464000
茶树miR156a靶基因SPL6和SPL9的克隆及表达分析
刘亚芹1,田坤红1,孙琪璐1,潘铖1,李叶云1,蒋家月1,江昌俊1,2*
1. 安徽农业大学茶树生物学与资源利用国家重点实验室,安徽 合肥,230036;2. 信阳师范学院河南省茶树生物学重点实验室,河南 信阳,464000
MicroRNA156a-SQUAMOSA promoter binding-like protein(miR156a-SPLs)参与植物生长阶段转变、叶片形态建成和逆境胁迫等复杂的生理过程。基于茶树转录组数据,从茶树中克隆出 2个 SPL家族成员——CsSPL6和CsSPL9。CsSPL6cDNA全长2 318 bp,ORF框长1 668 bp,编码555个氨基酸;CsSPL9cDNA全长1 954 bp,ORF框长1 116 bp,编码371个氨基酸;CsSPL6和CsSPL9都有典型SBP结构域和Csn-miR156a识别位点。时空表达特性分析表明,Csn-miR156a在芽中表达量最高,第 7叶中最低,与CsSPL6和CsSPL9呈负调控关系;不同品种(系)表达特性分析表明,Csn-miR156a的表达模式与新梢生长量、光合指标结果一致,与CsSPL6和CsSPL9表达模式相反,说明 Csn-miR156a、CsSPL6和CsSPL9参与茶树生长过程,Csn-miR156a和CsSPLs可作为初步判断品种生长势强弱的分子手段;高温处理4个品种(系)表达特性表明Csn-miR156a与CsSPL9呈负相关,推断茶树在高温胁迫下Csn-miR156a可能通过负调控CsSPL9来增加耐高温性。Csn-miR156a负调控CsSPLs在茶树生长发育、逆境胁迫中发挥作用,为茶树生长和抗逆机制提供理论依据。
茶树;生长势;高温;miR156a;SPL
SQUAMOSA 启动子结合蛋白(SQUAMOSA promoter binding protein-like,SPL)是植物特有和保守的一个转录因子,最先在金鱼草[1]中分离。研究发现,miR156通过转录后水平抑制拟南芥 SPL转录因子的表达从而调控植株生长发育、形态建成、花青素积累、孢囊发育、赤霉素合成、信号转导,以及胁迫响应等生理过程[2-7]。目前在拟南芥中已鉴定出 17个 SPL家族成员,其中11个成员——SPL2、SPL3、SPL4、SPL5、SPL6、SPL9、SPL10、SPL11、SPL13a/b和SPL15具有miR156识别位点[8-10],并且受其负调控。不同的SPL成员在拟南芥生长阶段发挥着不同的作用,ATSPL3、ATSPL4、ATSPL5调控阶段转变和开花[3,10];ATSPL8参与花药发育和赤霉素合成[11];AtSPL9和AtSPL15调控叶原基长度、器官大小、芽叶成熟[5,12];ATSPL2、ATSPL10、ATSPL11在生殖生长阶段调控芽叶生长[13]。
在逆境胁迫下,Cui等认为miR156可被作为信号分子,通过miR156-SPL9-DFR的途径参与植物生长和非生物胁迫[14]。拟南芥在响应高温胁迫时,miR156表达量会急剧增加,SPL2和SPL11则表现降低趋势[15]。苜蓿[16]在干旱胁迫下,miR156的过表达使其有更强的耐旱性,miR156通过下调SPL13的表达来提高抗旱性。对棉花进行盐胁迫和干旱胁迫发现,miR156通过负调控SPL2来提高耐受力[17]。磷酸盐胁迫下,拟南芥的根际酸化能力与 miR156呈正调控关系,与SPL3呈负调控关系[18]。
近年来,SPL转录因子在越来越多的物种如枳[19]、水稻[8,20]、花生[21]、陆地棉[22]、草莓[23]等植物中被克隆。miR156-SPLs的功能与分子机制的研究成为热点,越来越多的功能被挖掘。然而目前研究的植物大多是草本植物,对于茶树这样的木本植物研究较少。本研究基于茶树转录组数据,采用cDNA末端快速扩增(rapid amplification of cDNA ends,RACE)方法克隆出CsSPL6和CsSPL9的基因全长,检测 Csn-miR156a和CsSPLs在茶树不同叶位、不同品种(系)和高温胁迫中的表达量,为揭示 Csn-miR156a-CsSPLs在茶树生长发育、抗逆性中的作用提供依据。
1 材料与方法
1.1 实验材料及处理
植物材料选取生长势不同的东北抗、黄山白茶1号(C. sinensiscv. Huangshanbaicha)、舒茶早(C. sinensiscv. Shuchazao)、特香早(C.sinensiscv. Texiangzao)4个茶树品种(系)。茶树新株系东北抗是本课题组从祁门槠叶群体种中筛选的优良单株,通过多年观察,发现该株系具有生长旺盛、速度快和耐高温的特性。
不同叶位:选取芽、第1叶、第3叶、第5叶、第7叶。高温胁迫处理参数:温度为38℃/28℃,相对湿度为80%/70%,平均光强为600 μmol·m-2·s-1,光周期为14 h/10 h。以处理0、1、2、3、4 d时间点的芽下第2叶为材料。每个样品均为3个生物学重复,样品采集后立即投入液氮处理,冷冻后–80℃储存备用。
1.2 茶树生长势特性测定
新梢生长量测定:腋芽萌发时选取生育期基本一致的进行挂牌,每个品种(系)10株,每隔3 d测定芽叶生长量[24]。
光合作用的测定:取芽下第3叶为测试材料,采用LI-COR 6400便携式光合测定系统分析仪(LI-COR,美国)在光强1 200 μmol·m-2·s-1,气体流速500 μmol·s-1,CO2浓度 500 μmol·mol-1,叶温(25±1)℃,空气湿度(70±5)%条件下测定净光合速率Pn、蒸腾速率Tr、气孔导度Gs。
1.3 总RNA提取及cDNA的合成
实验材料经液氮研磨后,使用 RNAiso Plus提取试剂盒及Fruit-mate for RNA Purification(TaKaRa,大连)提取 RNA。用核酸定量仪(Thermo Electron,美国)测定总RNA样品的浓度、纯度,用1.2%变性琼脂糖凝胶电泳检测总RNA完整性,-80℃储存备用。总RNA反转录为cDNA使用PrimeScriptTM1st Strand cDNA Synthesis Kit(TaKaRa,大连)试剂盒。
1.4 CsSPL6和CsSPL9基因全长克隆
从茶树转录组数据中获得2个SPL转录因子序列,利用Primer 5.0设计引物(表1),验证片段后通过 SMART-RACE-PCR技术对CsSPL6和CsSPL9的 3′和 5′端进行扩增。第一轮扩增以SMARTTMRACE cDNA Amplification Kit(Clontech,美国)合成 3′和5′cDNA第一链为模板,扩增体系为50 μL:10×LATaqbufferⅡ(Mg2+plus)5 μL,UPM( 10 μmol·L-1) 5 μL , dNTP Mixure (2.5 mmol·L-1) 4 μL , RACE GSP primer( 10 μmol·L-1)1 μL,cDNA 1 μL,LA Taq(5 U·μL-1)0.3 μL,ddH2O 33.7 μL。PCR 扩增参数为:94℃3 min;94℃ 30 s、72℃ 2 min,5 个循环;94℃30 s、70℃ 30 s、72℃ 2 min,5个循环;94℃30 s、65℃ 30 s、72℃ 2 min,25 个循环;72℃延伸 10 min。第二轮扩增以第一轮稀释 50倍的扩增产物为模板,反应体系同第一轮扩增。反应参数:94℃ 3 min,94℃ 45 s,67℃ 45 s,72℃ 2 min,30个循环;72℃ 10 min。产物用AxyPrep DNA Gel Extraction Kit(Axygen,美国)试剂盒进行回收,连接pMD 19-T载体,转化至大肠杆菌DH5α感受态细胞,菌落PCR挑选阳性克隆(至少3个重复),测序由上海生工有限公司完成。获得全长序列后,跨开放阅读框(ORF)设计特异引物,验证序列完整性。
1.5 序列生物信息学分析
用EditSeq和MegAlign软件查询ORF框、氨基酸序列。蛋白质等电点和分子量用EditSeq软件分析,ExPASY-ProtScale(http://web.expasy.org/protscale)预测亲/疏水性,SignalP(http://www.cbs.dtu.dk/services/SignalP-2.0)预测信号肽,TMHMM (http://www.cbs.dtu.dk/services/TMHMM-2.0)预测跨膜螺旋,NetPlos2.0 PredictProtein (https://ppopen.informatik.tu-muenchen.de)预测蛋白质的亚细胞定位,SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sop ma.html)预测蛋白质的二级结构,SWISS
MODEL (https://swissmodel.expasy.org/interactive)模拟蛋白质的三维结构,保守结构域的预测在NCBI(https://blast.ncbi.nlm. nih.gov/Blast.cgi)中进行,用Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo)比对蛋白质的同源性序列,拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、葡萄(Vitis vinifera)、小立碗藓(Physcomitrella patens)SBP转录因子基因序列参照文献[21],利用MEGA5.1[25]软件并通过邻接(N-J)算法构建系统进化树。
1.6 茶树Csn-miR156a和SPLs的表达分析
荧光定量分析东北抗不同叶位、4个品种(系)间和高温胁迫下 Csn-miR156a和SPLs的表达。根据 miR156a成熟序列设计具有茎环结构的反转录引物和 miR156a荧光定量正反向引物[26]。Csn-miR156a荧光定量内参为Csn-pc-3p-222[27],CsSPLs的内参为 GAPDH。荧光定量实验体系和反应参数按照SYBR®Premix Ex TaqTMII试剂盒(TaKaRa,大连)说明进行。采用 CFX96TMReal-Time PCR Detection System进行PCR扩增,每个样品3次生物学重复。验证目标基因和内参基因的扩增效率和扩增范围,采用2-ΔΔCT法进行相对表达量的计算。
2 结果与分析
2.1 CsSPL6和CsSPL9的全长克隆和序列分析
以 CsSPL6-3′RACE和 CsSPL6-5′RACE引物分别扩增出约1 000 bp(图1-A)和750 bp(图1-B)的片段,拼接后利用引物CsSPL6-F/R验证ORF框完整性(图1-C)。经测序和分析,得到CsSPL6全长为2 318 bp,包含1个1 688 bp完整的ORF框,编码1个由 555个氨基酸组成的蛋白(图 2)。预测蛋白分子量为 60.44 kDa,等电点为 6.58。以引物 CsSPL9-3′RACE扩增出 500 bp(图 1-D)左右的片段,CsSPL9-5′RACE扩增出1 200 bp(图 1-E)左右的条带,利用 CsSPL9-F/R验证ORF框完整性(图1-F)。测序分析表明,茶树CsSPL9基因全长1 954 bp,完整ORF框大小为1 116 bp,编码371个氨基酸(图2)。预测的蛋白质分子量是 39.62 kDa,等电点为8.3。CsSPL6和CsSPL9序列提交至GenBank,登录号KX808499和KY569510。
CsSPL6和 CsSPL9蛋白为亲水性蛋白,Score值小于0.5,不存在信号肽。CsSPL6不存在跨膜结构,不属于跨膜蛋白。在CsSPL9上发现1处由内到外的跨膜螺旋和1处由外到内的跨膜螺旋,存在跨膜区域,属于跨膜蛋白,CsSPL6和CsSPL9定位于细胞核。CsSPL6由9.55%的 α-螺旋(Alpha helix)、11.89%的 β-折叠(Beta sheet)和 55.41%的无规则卷曲(Random coil)组成,CsSPL9由19.41%的α-螺旋、8.63%的β-折叠和52.02%的无规则卷曲组成。CsSPL6和CsSPL9属于α/β型(图3),具有高等植物转录因子特有的SBP结合域。
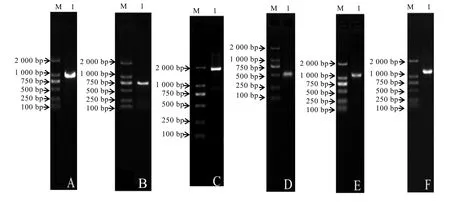
图 1 茶树 CsSPL6和 CsSPL9全长 cDNAFig. 1 Full cDNA sequences of CsSPL6 and CsSPL9 in C. sinensis
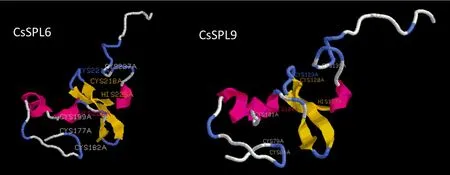
图3 预测的CsSPL6/CsSPL9蛋白三级结构Fig. 3 Tertiary structure prediction of protein CsSPL6/CsSPL9

图2 CsSPL9和CsSPL6 cDNA全长与推导的氨基酸序列Fig. 2 Full cDNA sequences of CsSPL9 and CsSPL6 and their deduced amino acids

续图2 CsSPL9和CsSPL6 cDNA全长与推导的氨基酸序列Continued Fig. 2 Full cDNA sequences of CsSPL9 and CsSPL6 and their deduced amino acids
2.2 CsSPL6和CsSPL9的进化分析和氨基酸序列的比对
为进一步分析CsSPL6和CsSPL9转录因子的进化,将CsSPL6和CsSPL9的氨基酸序列与 34条双子叶植物(拟南芥和葡萄)、19条单子叶植物(水稻)、13条无花植物(小立碗藓)的SBP序列进行比对,参考Li[21]的分类方法,构建同源进化树,SPL转录家族被分成 6组(图 4)。CsSPL6与AtSPL6(At1g69170)、VvSBP1(XM002273498.1)、VvSBP16(XM002265167.1)聚在一类,属于G2组;CsSPL9与VvSBP8(XM002278476.1)、AtSPL9(At2g42200)、AtSPL15(At3g57920)同处一个分支,属于G1组,推测CsSPL9与VvSBP8、AtSPL9、AtSPL15具有相似的生物学功能。而小立碗藓SBP转录因子没有聚类在G1和G2,说明茶树CsSPL6和CsSPL9转录因子和小立碗藓亲缘关系比较远,而和拟南芥、葡萄较近。不同物种的 SBP转录因子并没有聚在一起,演化情况可能比较复杂。
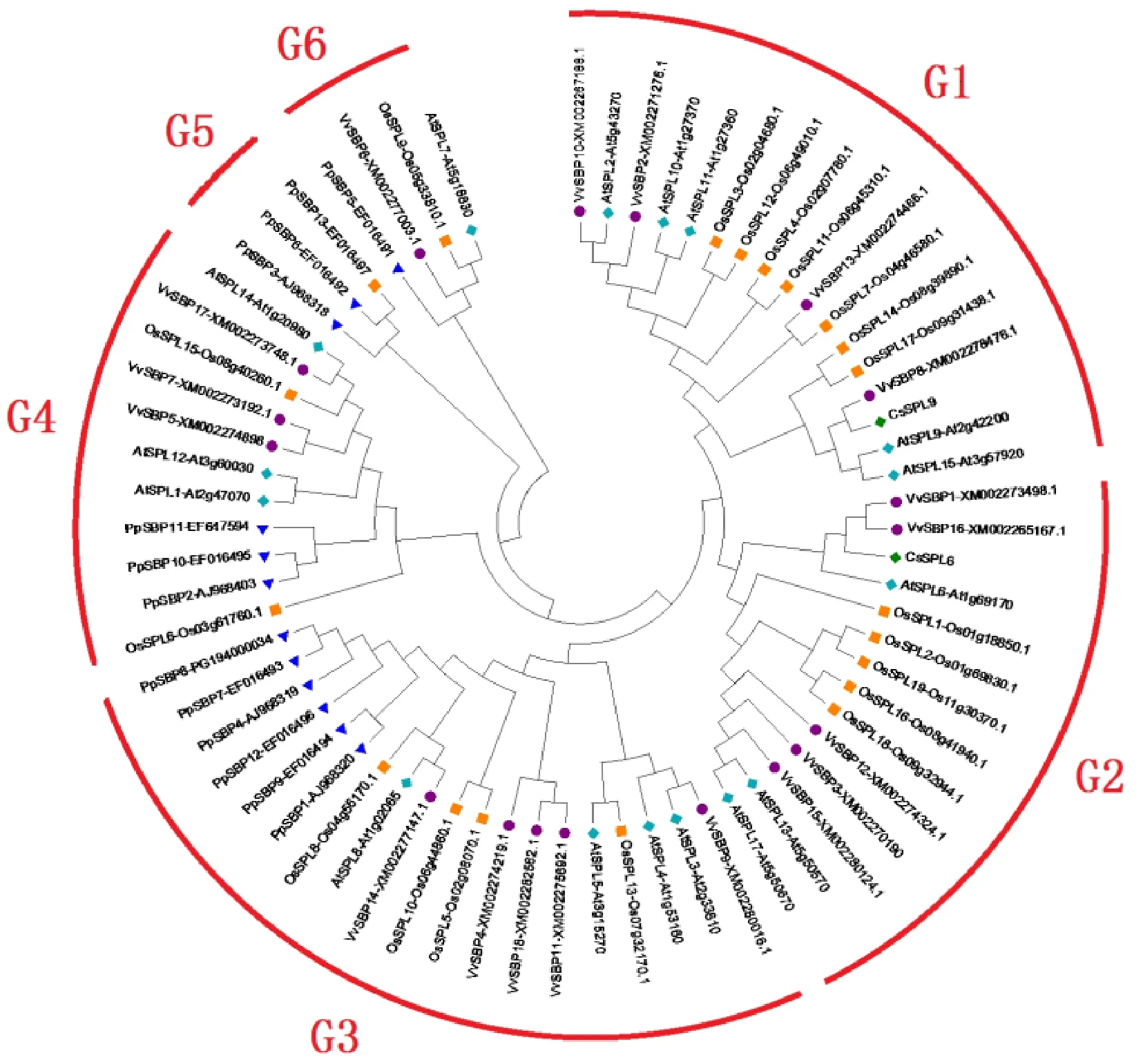
图4 茶树CsSPL6和CsSPL9转录因子的进化分析Fig. 4 Phylogenetic analysis of CsSPL6 and CsSPL9 transcription factors in C. sinensis
对茶树CsSPL6和CsSPL9转录因子进行保守结构域预测发现CsSPL6和CsSPL9都含有高度保守的SBP结构域(图5-A和图5-B)和 miR156a的识别位点。通过 BlastP同源序列检索与多序列比对,CsSPL6与葡萄(XP_010663469.1)、可可(XP_017973789.1)、枣(XP_015883292.1)相似度分别为61.10%、61.63%和59.15%;CsSPL9与葡萄(ADG36380.1)、番薯(XP_019186327.1)和可可(XP_017977243.1)的相似度达65.51%、54.59%和59.31%,而且SBP结构域在植物中高度保守(图5-C)。

图5 茶树CsSPL6(A)和CsSPL9(B)转录因子的保守域预测及和同源物种SBP结构域比对(C)Fig. 5 Prediction of CsSPL6 (A) and CsSPL9 (B) conserved domains and alignment of SBP domain in C. sinensis and homologous proteins in other species (C)
2.3 茶树Csn-miR156a和SPLs表达机制分析
2.3.1 不同叶位时序表达
如图 6显示,CsSPL6和CsSPL9以及Csn-miR156a在不同叶位中均有所表达,但表达量存在差异。Csn-miR156a在芽中表达量最高,随着叶位的增加,表达量依次降低;CsSPL6和CsSPL9的表达情况一致,表达量在芽中最低,第7叶中最高,分别是芽的2.3和2.9倍,与Csn-miR156a的表达模式相反。
2.3.2 不同生长势品种(系)表达
右图7可见,CsSPL6、CsSPL9和Csn-miR156a在4个品种(系)间表达量差异明显。Csn-miR156a在4个品种(系)的表达量东北抗最高,特香早最低;而CsSPL6和CsSPL9的表达趋势一致,与Csn-miR156a呈负相关关系。
新梢生长势的测定结果表明(图 8),4个品种(系)生长速度东北抗最快,特香早最慢。光合指标中(表2),Pn和Tr结果一致,东北抗最高,特香早最低,而Gs结果相反。生长势高的植物Pn和Tr高,Gs低[28-29],即植物光合能力强而呼吸作用弱,有利于干物质积累,增加植物产量。新梢生长势和光合作用指标测定结果与 Csn-miR156a的表达量的趋势一致。东北抗生长速度最快,Csn-miR156a的表达量最高;特香早生长最慢,Csn-miR156a的表达量最低。

图6 茶树不同叶位Csn-miR156a-CsSPLs表达水平的qRT-PCR分析Fig. 6 Expression profiles of the Csn-miR156a-CsSPLs in different leaf positions

图7 茶树不同品种(系)Csn-miR156a-CsSPLs表达水平的qRT-PCR分析Fig. 7 Expression analysis of Csn-miR156a-CsSPLs in different varieties (strains) by qRT-PCR

图8 不同品种(系)的新梢长度和生长速率比较Fig. 8 Comparison of new shoots length and growth rate of different tea cultivars(strains)

表2 不同茶树品种(系)光合指标Table 2 Photosynthesis indexes of different tea cultivars (strains)
2.3.3 响应高温胁迫表达
从图 9可以看出,高温影响CsSPL6、CsSPL9和Csn-miR156a的表达,东北抗和黄山白茶1号Csn-miR156a在处理的第1天表达量达到最高,之后表达水平下降,Csn-miR156a的表达量在4个品种(系)顺序为东北抗>黄山白茶 1号>特香早>舒茶早。黄山白茶 1号和舒茶早在处理 1天后CsSPL6表达量达最高,分别达到对照的1.7和2.4倍,随后呈下降趋势,仍维持很高水平;特香早经高温处理后CsSPL6呈波动下降趋势并在处理第3天降至最低。东北抗在处理的第 2天和第 4天CsSPL9的表达量急剧增加,是对照的3.9倍;舒茶早高温胁迫 1 d后,CsSPL9剧增达最大值,是对照4.3倍,之后表达量下降仍高于处理前水平。高温胁迫下,CsSPL6的表达不受Csn-miR156a调控,而CsSPL9受Csn-miR156a负调控。
3 讨论
3.1 CsSPL6和CsSPL9的克隆及序列分析
从茶树中克隆出 SPL家族中 2个成员——CsSPL6和CsSPL9的 cDNA全长序列。序列分析发现,CsSPL6编码555个氨基酸,CsSPL9编码371个氨基酸。CsSPL6和CsSPL9编码蛋白序列都含SBP结构域和miR156a的识别位点。SBP结构域由79个保守的氨基酸残基组成,结合 2个锌离子形成锌指结构(Zinc-finger domain)[30]。2个锌指结构均由3个半胱氨酸和1个组氨酸残基形成四面体并结合1个锌离子,从而形成稳定的构象。其中N-端构成是Cys-Cys-Cys-His,C-端是Cys-Cys-His-Cys,C末端带有1个双向核定位信号(Nuclear Localization Signal)KRXXXRRRK[1],与第2个锌指结构有部分重叠。Birkenbihl[31]等研究表明,该信号肽序列可以引导 SBP蛋白进入细胞核,从而调控下游相关基因的转录表达。系统进化树发现茶树CsSPL6和CsSPL9转录因子分别属于G2组和G1组,并且和小立碗藓亲缘关系比较远,而和拟南芥、葡萄较近;这可能和 SBP转录因子序列长度、结构域起始位置、外显子数量及miRNA识别位点的差异性有关[19]。
3.2 Csn-miR156a和CsSPLs的表达分析
miR156a在拟南芥和大豆第1、2叶表达水平高,随着叶位增加,表达水平大幅降低[32-34]。miR156a在顶芽中表达量最高并在植物生长发育过程中与 SPL呈负相关关系[35]。目前研究证明 miR156a随发育进程表达量降低的时序降低机制可能涉及到内源信号分子响应[36]。在本研究中Csn-miR156a与CsSPL6和CsSPL9呈负调控,推测Csn-miR156a通过负调控CsSPL6和CsSPL9参与茶树生长过程[37],在茶树生长阶段发育和转变中起重要作用。
在不同茶树品种(系)的表达模式中,Csn-miR156a表达趋势与新梢生长量以及光合指标的测定结果趋势一致,与CsSPLs呈负调控关系。目前这方面的研究较少,初步推断Csn-miR156a和CsSPLs表达机制可能是茶树生长过程中的重要组成部分,Csn-miR156a表达量越高,CsSPLs表达量越低,植物生长可能越快,反之,则越慢,推测 Csn-miR156a和CsSPLs表达机制可作为判断植物生长快慢的重要分子指标。
高温胁迫下不同茶树品种(系)、不同处理时间 Csn-miR156a和CsSPLs表达有所差异,说明在高温胁迫下存在调控关系。在胁迫处理第1天,Csn-miR156a通过急剧上调减少高温对植物体的伤害,与Xin等[38]、Stief等[15]研究结果一致。芸薹属蔬菜作物受高温胁迫后,miR156也是下调SPL2来增加耐受性[39]。干旱条件下研究miR156与其呈负调控关系的靶基因SPL6、SPL12、SPL13的表达模式时,发现SPL6、SPL12表达不显著,无规律,而SPL13表达量明显下调从而增加气孔导度、减少水分缺失。推测在干旱胁迫中,miR156调控SPL13占主导部分从而提高抗旱能力[16]。SPL基因家族在响应盐胁迫后会刺激二级代谢产物如 VATP、GRP、NHX1、RCI2、H+-ATP酶和SOS1等的合成,从而直接或间接地参与逆境胁迫[17,40-41]。在本研究中,不同茶树品种(系)在高温胁迫下,CsSPL9与Csn-miR156a呈负相关,可能是因为茶树在高温胁迫下Csn-miR156a通过下调CsSPL9来增加耐高温性,而其他CsSPLs成员的作用则可能是通过刺激二级代谢产物的合成来达到抵抗高温的作用,但目前这些产物及其作用机制还是未知的,需进一步研究。
本研究通过茶树生长势测定或者高温胁迫初步揭示 Csn-miR156a-CsSPL的表达机制可以反映植物的生长快慢以及耐高温程度。但关于 miR156-SPLs处于怎样的调控网络,miR156的表达受哪些上游基因的时序调控,及SPLs的下游调控基因是什么等问题都有待更多研究去揭示。
[1]Klein J, Saedler H, Huijser P. A new family of DNA binding proteins includes putative transcriptional regulators of the Antirrhinum majus floral meristem identity gene SQUAMOSA [J]. Mol Gen Genet, 1996, 250(1): 7-16.
[2]Yu S, Galvao V C, Zhang Y C, et al. Gibberellin regulates theArabidopsisfloral transition through miR156-targeted SQUAMOSA promoter binding-like transcription factors [J].Plant Cell, 2012, 24(8): 3320-3332.
[3]Wu G, Poethig R S. Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its target SPL3 [J]. Development, 2006, 133(18): 3539-3547.
[4]Wang J W, Czech B, Weigel D. miR156-Regulated SPL transcription factors define an endogenous flowering pathway inArabidopsis thaliana[J]. Cell, 2009, 138(4):738-749.
[5]Schwarz S, Grande A V, Bujdoso N, et al. The microRNA regulated SBP-box genesSPL9andSPL15control shoot maturation inArabidopsis[J]. Plant Molecular Biology,2008, 67(1/2): 183-195.
[6]Jung J H, Seo P J, Kang S K, et al. miR172 signals are incorporated into the miR156 signaling pathway at theSPL3/4/5genes inArabidopsisdevelopmental transitions [J].Plant Molecular Biology, 2011, 76(1/2): 35-45.
[7]Eriksson M, Moseley J L, Tottey S, et al. Genetic dissection of nutritional copper signaling in chlamydomonas distinguishes regulatory and target genes [J]. Genetics, 2004,168(2): 795-807.
[8]Yang Z, Wang X, Gu S, et al. Comparative study of SBP-box gene family in Arabidopsis and rice [J]. Gene, 2008,407(1/2): 1-11.
[9]Rhoades M W, Reinhart B J, Lim L P. Prediction of plant microrna targets [J]. Cell, 2002, 110(4): 513-520.
[10]Gandikota M, Birkenbihl R P, Höhmann S, et al. The miRNA156/157 recognition element in the 3' UTR of theArabidopsisSBP box gene SPL3 prevents early flowering by translational inhibition in seedlings [J]. Plant Journal for Cell amp; Molecular Biology, 2007, 49(4): 683-693.
[11]Zhang Y, Schwarz S, Saedler H, et al. SPL8, a local regulator in a subset of gibberellin-mediated developmental processes inArabidopsis[J]. Plant Molecular Biology, 2007, 63(3):429-439.
[12]Wang J W, Schwab R, Czech B, et al. Dual effects of miR156-targeted SPL genes and CYP78A5/KLUH on plastochron length and organ size inArabidopsisthaliana[J].Plant Cell, 2008, 20(5): 1231-1243.
[13]Shikata M, Koyama T, Mitsuda N, et al.ArabidopsisSBP-box genes SPL10, SPL11 and SPL2 control morphological change in association with shoot maturation in the reproductive phase [J]. Plant amp; Cell Physiology, 2009,50(12): 2133-2145.
[14]Cui L, Shan J, Shi M, et al. The miR156-SPL9-DFR pathway coordinates the relationship between development and abiotic stress tolerance in plants [J]. Plant Journal for Cell amp;Molecular Biology, 2014, 80(6): 1108-1117.
[15]Stief A, Altmann S, Hoffmann K, et al.ArabidopsismiR156 regulates tolerance to recurring environmental stress through spl transcription factors [J]. The Plant Cell, 2014, 26(4):1792-1807.
[16]Arshad M, Feyissa B A, Amyot L, et al. MicroRNA156 improves drought stress tolerance in alfalfa (Medicago sativa) by silencing SPL13 [J]. Plant Science, 2017,258:122-136.
[17]Wang M, Wang Q, Zhang B. Response of miRNAs and their targets to salt and drought stresses in cotton (Gossypium hirsutumL.) [J]. Gene, 2013, 530(1): 26-32.
[18]Lei K J, Lin Y M, An G Y. miR156 modulates rhizosphere acidification in response to phosphate limitation inArabidopsis[J]. Journal of Plant Research, 2016, 129(2):275-284.
[19]宋长年, 钱剑林, 房经贵, 等. 枳 SPL9和 SPL13全长cDNA克隆、亚细胞定位和表达分析[J]. 中国农业科学,2010, 43(10): 2105-2114.
[20]Miura K, Ikeda M, Matsubara A, et al. OsSPL14 promotes panicle branching and higher grain productivity in rice [J].Nature Genetics, 2010, 42(6): 545-549.
[21]Li M, Zhao S Z, Zhao C Z, et al. Cloning and characterization of SPL-family genes in the peanut (Arachis hypogaeaL.) [J]. Genetics and Molecular Research, 2015,14(1): 2331-2340.
[22]Zhang X, Dou L, Pang C, et al. Genomic organization,differential expression, and functional analysis of the SPL gene family inGossypium hirsutum[J]. Molecular Genetics and Genomics, 2015, 290(1): 115-126.
[23]赵晓初, 李贺, 代红艳, 等. 草莓 miR156靶基因 SPL9的克隆与表达分析[J]. 中国农业科学, 2011, 44(12):2515-2522.
[24]李磊. 不同肥料处理对茶树生长和茶叶品质的影响[D].泰安: 山东农业大学, 2010.
[25]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods [J].Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[26]Erika V G. Stem-Loop qRT-PCR for the detection of plant microRNAs [M]. Totowa N J: Humana Press, 2016.
[27]谢小芳. 茶树 miRNA靶基因的鉴定及在低温胁迫下的表达分析[D]. 合肥:安徽农业大学, 2016.
[28]张永福, 任禛, 莫丽玲, 等. 葡萄品种生长势差异的相关机制研究[J]. 北方园艺, 2015 (2): 1-5.
[29]杨雨华, 宗建伟, 杨风岭. 不同生长势马尾松光合日变化研究[J]. 中南林业科技大学学报(自然科学版), 2014, (8):25-29.
[30]Yamasaki K, Kigawa T, Inoue M, et al. A novel zinc-binding motif revealed by solution structures of DNA-binding domains of Arabidopsis SBP-family transcription factors [J].Journal of Molecular Biology, 2004, 337(1): 49-63.
[31]Birkenbihl R P, Jach G, Saedler H, et al. Functional dissection of the plant-specific sbp-domain: overlap of the dna-binding and nuclear localization domains [J]. Journal of Molecular Biology, 2005, 352(3): 585-596.
[32]Yang L, Conway S R, Poethig R S. Vegetative phase change is mediated by a leaf-derived signal that represses the transcription of miR156 [J]. Development, 2011, 138(2):245-249.
[33]Yoshikawa T, Ozawa S, Sentoku N, et al. Change of shoot architecture during juvenile-to-adult phase transition in soybean [J]. Planta, 2013, 238(1): 229-237.
[34]Gou J Y, Felippes F F, Liu C J, et al. Negative regulation of anthocyanin biosynthesis inArabidopsisby a miR156-targeted SPL transcription factor [J]. Plant Cell,2011, 23(4): 1512-1522.
[35]Salinas M, Xing S, Höhmann S, et al. Genomic organization,phylogenetic comparison and differential expression of the SBP-box family of transcription factors in tomato[J]. Planta,2012, 235(6): 1171-1184.
[36]冯圣军. MicroRNA156调控烟草发育阶段转变的功能研究[D]. 临安: 浙江农林大学, 2014.
[37]虞莎, 王佳伟. miR156介导的高等植物年龄途径研究进展[J]. 中国科学, 2014, 59(15): 1398-1404.
[38]Xin M M, Wang Y, Yao Y Y, et al. Diverse set of microRNAs are responsive to powdery mildew infection and heat stress in wheat [J]. BMC Plant Biology, 2010, 10: 123-133.
[39]Yu X, Wang H, Lu Y Z, et al. Identification of conserved and novel microRNAs that are responsive to heat stress in Brassica rapa [J]. Journal of Experimental Botany, 2012,63(2): 1025-1038.
[40]Arshad M, Gruber M Y, Wall K, et al. An insight into microrna156 role in salinity stress responses of alfalfa [J].Frontiers in Plant Science, 2017, 8(658):356-370.
[41]Yu Z X, Wang L J, Zhao B, et al. Progressive regulation of sesquiterpene biosynthesis inArabidopsisand patchouli(Pogostemon cablin) by the miR156-Targeted SPL transcription factors [J]. Molecular Plant, 2014, 8(1): 98-110.
Cloning and Expression Analysis of miR156a-targeted Genes SPL 6 and SPL9 in Camellia sinensis
LIU Yaqin1, TIAN Kunhong1, SUN Qilu1, PAN Cheng1,LI Yeyun1, JIANG Jiayue1, JIANG Changjun1,2*
1. State Key Laboratory of Tea Plant Biology and Utilization, Anhui Agricultural University, Hefei 230036, China;2. Henan Key Laboratory of Tea Biology, Xinyang Normal University, Xinyang 464000, China
MicroRNA156a-SQUAMOSA promoter binding-like protein(miR156a-SPLs)play important roles in growth process, formation of leaves and response to stress in tea plant (Camellia sinensis). Based on transcriptome data, two cDNACsSPL6(2 318 bp) andCsSPL9(1 954 bp) were cloned from tea plant. The nucleotide and amino acid sequences, biological information, phylogenetic tree and molecular expression models of theCsSPL6andCsSPL9were analyzed. Sequence analysis showed that the open reading frames ofCsSPL6andCsSPL9are 1 668 bp and 1 116 bp, encoding 555 and 371 amino acids respectively. Both genes contain typical SBP domain and the recognition sites of microRNA156a. Quantitative real-time PCR analysis showed that the expression profile of Csn-miR156a was the highest in the buds, and the lowest in the 7th leaves, which was negatively correlated withCsSPL6andCsSPL9. For different varieties(strains), the expression of Csn-miR156a was consistent with the growth of new shoots and the value of Photosynthesis index, but negatively correlated with the expression ofCsSPL6 and CsSPL9,indicating. Csn-miR156a,CsSPL6andCsSPL9were involved in the growth of tea plant and can be used as markers to assess growth abilities among varieties. In addition, the expression ofCsSPL9was negatively correlatedwith Csn-miR156a under heat stress in four varieties (strains), which might be associated with the enhanced heat tolerance ability. The results showed that the expression ofCsSPLswere negatively regulated by Csn-miR156a,which played an important role in the growth, development of tea plant and the stress resistance,providing a theoretical basis for understanding the growth and resistance mechanism of tea plant.
Camellia sinensis, growth potential, high temperature, miR156a, SPL
TS272.2;Q52
A
1000-369X(2017)06-551-14
2017-07-04
2017-08-05
茶树响应低温胁迫的microRNA发掘及其调控机制研究(31270729)、国家自然科学基金(31270729)
刘亚芹,女,硕士研究生,主要从事茶树种质资源与育种。*通讯作者:jiangcj@ahau.edu.cn
