茶树MADS-box转录因子基因的克隆与非生物胁迫响应分析
2017-12-11沈威滕瑞敏李辉刘志薇王永鑫王文丽庄静
沈威,滕瑞敏,李辉,刘志薇,王永鑫,王文丽,庄静
南京农业大学园艺学院,茶叶科学研究所,江苏 南京 210095
茶树MADS-box转录因子基因的克隆与非生物胁迫响应分析
沈威,滕瑞敏,李辉,刘志薇,王永鑫,王文丽,庄静*
南京农业大学园艺学院,茶叶科学研究所,江苏 南京 210095
本研究基于茶树转录组数据库,以茶树龙井43为试验材料,通过RT-PCR方法从该茶树的cDNA中克隆得到1个CsMADS1基因。序列分析表明:茶树CsMADS1基因开放阅读框长度为657 bp,编码218个氨基酸,是典型的植物MADS-box家族转录因子。序列多重比对显示,该序列与多个相关物种的MADS-box序列一致性为65.65%,含有高度保守的MADS结构域和半保守的K结构域。氨基酸理化性质、亲疏水性、亚细胞定位预测、无序化分析,以及二级和三级结构分析显示,CsMADS1转录因子是亲水性蛋白,可能定位于细胞核中,无序化程度明显,以 α-螺旋结构为主,并与人 MEF2蛋白具有相似的三级结构。利用实时荧光定量PCR方法分析了茶树龙井43中CsMADS1基因在非生物胁迫下的表达。结果表明,茶树中CsMADS1基因对高温、低温、干旱和高盐等不同非生物胁迫有响应,且表达存在差异。
茶树;MADS-box;转录因子;进化分析;非生物胁迫;表达分析
MADS-box基因广泛存在于植物中,它所编码的蛋白是一类转录因子,由于在植物生长发育方面发挥着非常重要的作用而被广泛研究。在拟南芥(Arabidopsis thaliana)[1]、水稻(Oryza sativa)[2]、葡萄(Vitis vinifera)[3]、杨树(Populus)[4]、二穗短柄草(Brachypodium distachyon)[5]、和芜菁(Brassica rapa)[6]中,MADS-box家族转录因子分别有107、75、93、105、57和167个成员。
目前,MADS-box基因研究的热点主要集中在植物花发育过程中,该基因在ABCDE模型中发挥重要的调控作用[7]。除此之外,MADS-box基因还在植物的根发育[8]、芽休眠[9]、果实成熟[10]及细胞衰老[11]等方面起着重要作用。MADS-box基因在植物逆境响应等方面也起着重要作用,例如:Cooper等建立的基因网络证实了 MADS-box转录因子参与水稻的逆境调控[12];矮牵牛中的PMADS9基因在多种逆境胁迫条件下表达量均显著提高[13];Yu等[14]发现在多种逆境处理下,MADS-box基因的表达显著影响植物根系的生长与发育。但是,目前该基因在茶树逆境方面的研究还鲜有报道。茶树(Camellia sinensis)属于山茶科山茶属,是一种喜酸惧碱,喜温怕寒的多年生常绿木本植物,在我国已经有几千年的栽培历史[15]。茶树一生中受到多种非生物胁迫伤害,其中高温、低温、干旱、高盐等严重影响着茶树的生长发育,从而影响茶叶的产量和品质。因此,研究 MADS-box转录因子在茶树生长发育和逆境调控中的作用具有重要意义。
本研究以茶树材料龙井43的cDNA为模板克隆得到1个茶树CsMADS1基因,并对该基因编码的氨基酸序列、理化性质、进化树和三级结构等方面进行了分析。利用荧光定量PCR,分析了龙井43中CsMADS1基因在高温、低温、干旱和高盐等4种非生物胁迫条件下的表达特征。本研究以期通过生物技术,探寻与茶树逆境响应相关的基因,为茶树抗逆遗传育种和生长发育调控提供理论基础。
1 材料与方法
1.1 试验材料和胁迫处理
试验材料为茶树龙井 43两年生扦插幼苗,种植于南京农业大学茶叶科学研究所温室。摘取健康茶树植株的幼嫩叶片,进行总RNA提取并反转录为cDNA。对龙井43两年生植株进行 4种非生物胁迫处理,分别为:38℃高温、4℃低温、20% PEG、200 mmol·L-1NaCl,处理时间分别为1、4、8、24 h;以未做任何处理的龙井43植株叶片为空白对照,每个处理设3个重复。其中温度处理由光照培养箱控制,分别设为38℃、4℃,湿度设为70%;干旱和盐处理采用叶面喷施的方式,分别在各时间段采集不同处理的第2叶,保存在-80℃冰箱,于第2天统一提取RNA后合成cDNA,作为实时荧光定量检测的模板。
1.2 茶树总RNA的提取和cDNA的合成
茶树龙井43的RNA提取参考Quick RNA Isolation Kit试剂盒说明书完成(北京华越洋生物科技有限公司),RNA的样品浓度和质量分别用微量紫外检测仪Nano-Drop和1.2%琼脂糖凝胶电泳检测。RNA反转录成 cDNA按照Prime Script RT Reagent Kit试剂盒(大连TaKaRa公司)完成。
1.3 茶树CsMADS1基因的克隆
根据本实验室茶树转录组数据[16],设计1对引物(CsMADS1-F:5′-ATGGTGAGACA GAGAATTCAGATC-3′和 CsMADS1-R:5′-GC TCTGATAAGGTAGGCCCAA-3′) 。 以 龙 井43叶片cDNA为模板进行PCR扩增。扩增体系为 20 μL:CsMADS1-F/R 引物各 1 μL、10 μLEx TaqMix 酶、1 μL 模板、7 μL ddH2O。PCR扩增程序为:94℃、5 min,94℃、30 s,52℃、30 s,72℃、1 min,35个循环;72℃,10 min。根据DNA回收试剂盒将经过1.2%的琼脂糖凝胶电泳分离的 PCR产物回收,随后连接到pMD19-T载体上,转化至大肠杆菌DH5α。质粒酶切和鉴定之后送至通用生物系统(安徽)有限公司测序。
1.4 生物信息学分析
通过BioXM2.6软件查找基因的开放阅读框以及编码氨基酸序列。在 NCBI网站(http://www.ncbi.nlm.nih.gov)对氨基酸序列进行同源性搜索和保守域预测。利用ExPASy-ProtParam(http://web.expasy.org/prot-param)和SMS(http://www.bio-soft.net/sms)分析相关物种MADS-box转录因子的氨基酸理化性质、理论等电点及组成成分等。使用最新版本MEGA 7软件构建分子系统发育进化树[17]。借助于 DNAMAN 8.0软件进行茶树CsMADS1基因推导氨基酸的多重对比和亲疏水性分析。通过 LocTree3(https://rostlab.org/services/loctree3)和SoftBerry ProtComp 9.0(http://linux1. softberry.com/berry.phtml)完成亚细胞定位预测[18-19]。无序化特征分析利用Fold Index[20]程序(http://bip.weizmann.ac.il/fldbin/findex)完成。利用SOPMA[21]程序(http://npsa-pbil.
ibcp.fr/cgi-bin/npsa)和Swiss-Model[22]程序(https://swissmodel.expasy.org)进行蛋白质二级结构及三级结构预测分析,并在Swiss-Pdb Viewer软件上编辑生成茶树CsMADS1转录因子的三维结构图形。
1.5 茶树CsMADS1基因的表达特性分析
按照SYBR PremixEx Taq试剂盒(大连TaKaRa公司)操作说明,使用 Bio-CFX96 real-time PCR system仪器完成实时荧光定量PCR。茶树GAPDH基因作为内参基因[23],与目标基因CsMADS1同时扩增。扩增体系为15 μL:酶 7.5 μL,模板 1.5 μL,上下游引物各 0.3 μL,ddH2O 5.4 μL。扩增程序为:95℃ 5 min;95℃5 s,55℃ 30 s,65℃ 10 s,40个循环。内参基因和目标基因CsMADS1的表达检测引物分别 为 : 5′-TTGGCATCGTTGAGGGTCT-3′和5′-CAGTGGGAACACGGAAAGC-3′;5′-GGC ATAATCTACAACCACAG-3′和 5′-CCACGC ATTCTCTTCCAT-3′。用2-ΔΔCT法计算分析茶树龙井43中CsMADS1相对表达量[24]。
2 结果与分析
2.1 龙井43茶树中CsMADS1基因的克隆
以龙井43茶树材料叶片的cDNA为模板,利用引物 CsMADS1-F和 CsMADS1-R进行PCR扩增反应,对扩增片段进行克隆、测序。将该序列在NCBI上进行Blast分析,结果显示该序列与多种植物的MADS-box基因有较高同源性,为所需的目的序列。通过BioXM2.6软件分析显示,该基因开放阅读框长度为657 bp,编码 218个氨基酸(图 1),命名为CsMADS1。

图1 茶树CsMADS1基因核苷酸序列与推测的氨基酸序列Fig. 1 The nucleotide and amino acid sequences of CsMADS1 gene from C. sinensis
2.2 茶树CsMADS1基因编码氨基酸序列比对及理化性质分析
对茶树CsMADS1基因编码氨基酸序列进行分析,结果显示(图2-A),该氨基酸序列在3~60位含有高度保守的MADS结构功能域,在94~169位含有半保守的结构功能域K-box,具有植物MADS转录因子家族典型的MIKC结构域。为进一步分析茶树 CsMADS1的保守结构域,将它与其他相似度较高的多个物种MADS-box氨基酸序列进行多重比对(图2-B),结果显示一致性为65.65%,它们均含有高度保守的MADS结构域和半保守的K结构域。
对上述植物 MADS-box转录因子蛋白氨基酸进行理化性质分析。结果显示(表1),这些植物中MADS-box蛋白残基数在210~241之间,相对分子质量在23.72~27.26 kDa之间;酸性氨基酸比例略少于碱性氨基酸,理论等电点在 5.75~8.96之间。芳香族氨基酸占 4%左右,明显少于脂肪族氨基酸;总平均疏水性在-0.7左右,表现出较强的亲水性。

图2 茶树CsMADS1转录因子保守域及与其他植物氨基酸序列的多重比对Fig. 2 The conserved domain of CsMADS1 transcription factor and alignment of amino acid sequences from C. sinensis and other plant species

表1 不同植物中MADS-box家族转录因子氨基酸组成成分及理化性质分析Table 1Comparison of compositionand physicochemical properties of aminoacidsequences of MADS-box transcription factors amongplant species

表2 CsMADS1亚细胞定位预测Table 2The predictedsubcellular localization of CsMADS1
2.3 茶树CsMADS1氨基酸序列进化树分析
为了进一步分析茶树 CsMADS1与MADS-box家族相关转录因子之间的进化关系,参考 Annette Becker等[25]的 MADS-box转录因子家族分类方法,并利用MEGA7构建茶树CsMADS1与拟南芥MADS-box家族转录因子的同源进化树(图3),结果显示,茶树CsMADS1转录因子属于MADS-box家族转录因子中的STMADS11亚族。
2.4 茶树CsMADS1蛋白氨基酸亲疏水性分析
利用DNAMAN 8.0软件对茶树CsMADS1转录因子进行亲疏水性分析。结果显示,茶树CsMADS1转录因子亲水性最强的位点是第155位的亮氨酸(Leu)(图4-A);疏水性最强的位点是45位的缬氨酸(Val)(图4-B)。由此可知,亲水性氨基酸明显占更多比例,推测CsMADS1转录因子属于亲水性蛋白。
2.5 茶树CsMADS1蛋白亚细胞定位预测
通过在线预测软件 SoftBerry ProtComp 9.0对CsMADS1蛋白进行亚细胞定位预测。结果显示(表2),茶树CsMADS1蛋白定位在细胞核的相应预测数值最高。进一步利用在线预测软件LOCTREE3对CsMADS1蛋白进行亚细胞定位,结果显示CsMADS1蛋白定位在细胞核。由此判断,CsMADS1蛋白可能定位于细胞核中。
2.6 茶树CsMADS1蛋白固有无序化预测与分析
利用FoldIndex程序对CsMADS1氨基酸序列进行了无序化分析。结果表明(图5),茶树CsMADS1氨基酸序列中无序化区域有2个,总的无序化氨基酸数目为143,无序化比例为 65.60%。所以 CsMADS1有明显的无序化程度。
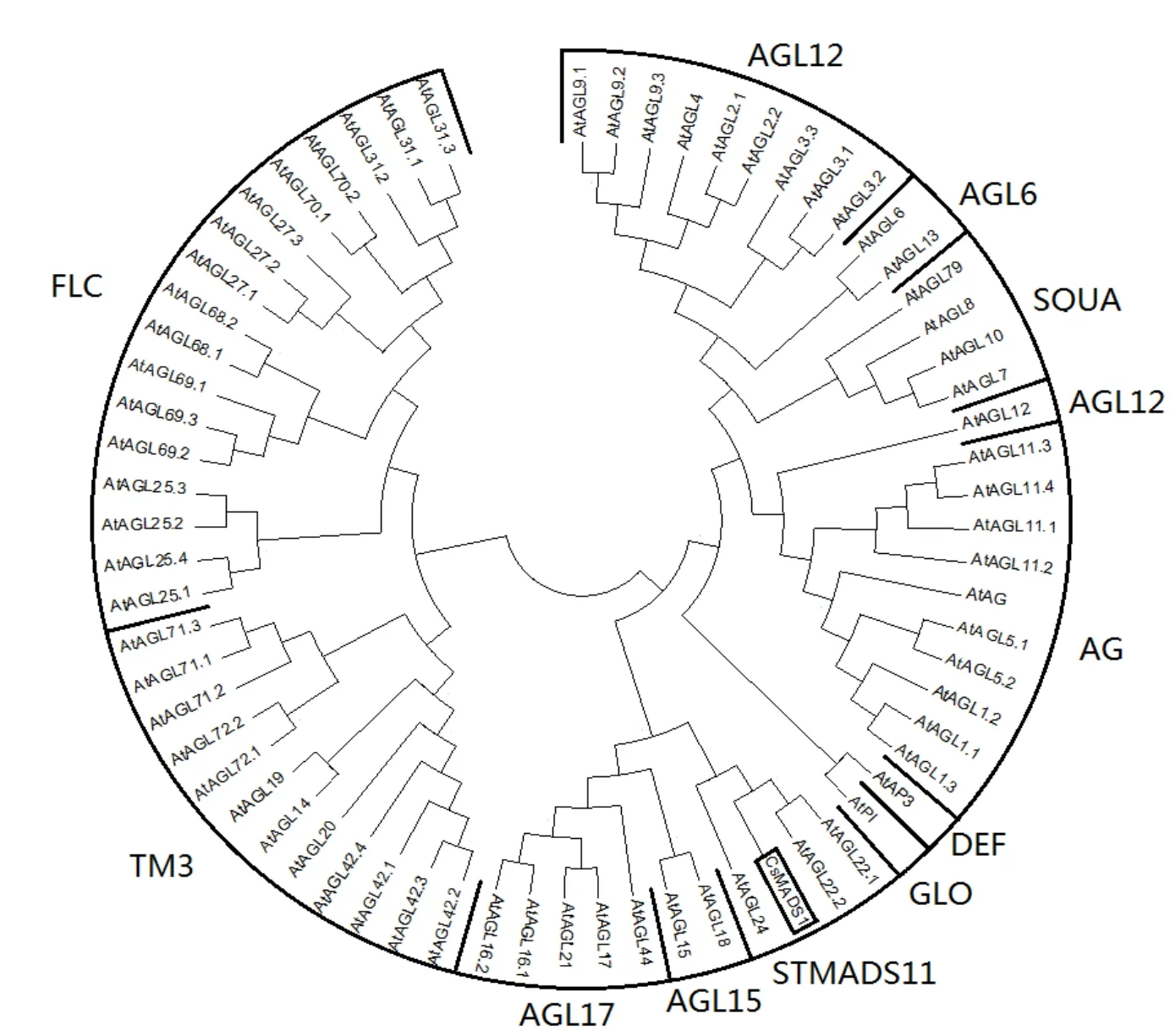
图3 茶树CsMADS1转录因子的进化分析Fig. 3 Phylogenetic analysis of CsMADS1 transcription factors from C. sinensis

图4 茶树CsMADS1氨基酸序列亲疏水性分析Fig. 4 Analysis of hydrophilicity and hydrophobicity of CsMADS1 from C. sinensis

图5 茶树CsMADS1折叠状态的分析Fig. 5 Analysis of the folding state of CsMADS1 from C. sinensis
2.7 茶树CsMADS1蛋白推导的二级结构和三级结构预测分析
从茶树 CsMADS1蛋白序列预测的二级结构中可以看出(图 6),CsMADS1蛋白由53.21%的 α-螺旋(Alpha helix),14.68%的延伸主链(Extended strand),6.42%的 β-转角(Beta turn)和25.69%的随机卷曲(Random coil)组成。其中α-螺旋结构是CsMADS1蛋白的主要组成部分,而延伸主链和随机卷曲结构则散布于蛋白序列中。
以人 MEF2转录因子为模板,对茶树CsMADS1转录因子的三级结构进行建模分析,结果显示相似度为48.65%。在结合域的α螺旋、β-折叠部位,CsMADS1与MEF2的差异位点共有19个,其中第1个和第2个α-螺旋分别有9个和8个差异位点,另外2个差异位点分别在两个β-折叠部位(图7),但这些差异位点对其三级结构并没有太大影响。
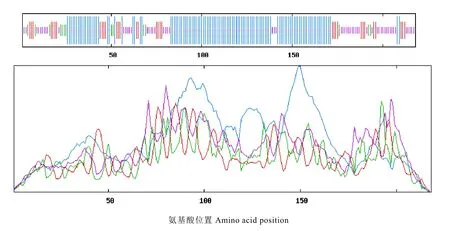
图6 茶树CsMADS1的二级结构预测Fig. 6 The secondary structure of CsMADS1 from C. sinensis

图7 茶树CsMADS1的三维结构预测Fig. 7 The tertiary structure of CsMADS1 from C. sinensis
2.8 茶树CsMADS1基因在不同非生物胁迫下的表达分析
通过实时荧光定量 PCR方法,检测了龙井43茶树中CsMADS1基因在高温(38℃)、低温(4℃)、干旱(20% PEG)和高盐(200 mmol/L NaCl)4种非生物胁迫条件下的表达情况。由图8可知,茶树中CsMADS1基因的表达在不同胁迫时间和不同胁迫处理间存在差异。在高温、干旱和高盐胁迫处理下,CsMADS1表达量均表现出先降低后增加再降低的趋势,其中高温和干旱胁迫下的表达量均在8 h时达到了最大值,且较对照差异显著,分别为对照的1.34和2.22倍。在高盐胁迫下的表达量较对照均表现出一定程度下调,1 h时表达量最低,随处理时间增加,到8 h时表达量有一定的回升,此时与对照无显著差异。低温胁迫处理下,CsMADS1表达量先增加后降低再增加,在1 h和4 h时的CsMADS1表达量显著高于对照,分别为对照的2.29和1.88倍,而在8 h和24 h时CsMADS1较对照均无显著差异。
3 讨论
植物生长发育过程中常受到高低温、干旱、高盐等非生物胁迫的伤害,严重时会导致植株死亡。茶树是极易受到恶劣环境影响的经济作物之一。目前,关于 MADS-box转录因子对茶树在生长发育和逆境调控中的作用机制等研究尚未见报导。因此本文开展茶树中MADS-box转录因子的克隆、序列分析、结构特征、功能及特性等研究,及其对茶树逆境胁迫的响应,具有较为重要的意义。
本文通过RT-PCR方法从茶树龙井43的cDNA中克隆得到1个MADS-box基因,命名为CsMADS1,该基因编码218个氨基酸,通过Blast分析表明,它具有MADS-box转录因子家族典型的 MIKC结构域[26]。序列比对和系统进化树分析结果表明,茶树CsMADS1转录因子属于MADS-box家族的STMADS11亚族,与多种植物有较高的同源性。亚细胞定位预测显示,CsMADS1定位于细胞核中。其无序化比例为65.60%,可能与其亲水性氨基酸比例较高有关[20]。茶树CsMADS1二级结构主要表现为α-螺旋结构。通过三级结构建模显示,茶树CsMADS1虽与人MEF2蛋白有19个差异位点,但对空间结构影响较小[27]。

图8 茶树CsMADS1在不同处理条件下的表达Fig. 8 Expression patterns of CsMADS1 under different stress treatments
实时荧光定量 PCR分析表明,高低温、干旱、高盐等非生物胁迫明显影响着茶树CsMADS1转录因子的表达量,但不同逆境之间响应存在一定的差异。已有的研究证明,在二穗短柄草中,多个MADS-box基因在低温、干旱和盐胁迫处理后出现不同的响应[5];银杏叶在盐、干旱和冷胁迫后,MADS-box转录因子基因的表达量显著高于对照[28];Guo等[29]发现,番茄SlMBP11基因在盐和受伤处理下的表达较为相似,都是先上升再下降,表达量分别在4 h和8 h时达到最大值,在干旱处理时,是一个逐渐上升的趋势,直到 36 h时到达最大值;水稻[2,30]和大白菜[31]在经过盐、干旱、低温、GA3和BAP等处理后,MADS-box转录因子基因的表达量也出现不同程度的上调和下调。本试验中,茶树CsMADS1基因受低温胁迫后能够快速诱导表达,在盐胁迫下主要表现为被抑制,在高温和干旱胁迫下,其表达量先降低,随后逐渐上升后又下降。以上结果显示,MADS-box转录因子在不同物种间的响应有明显的差异性。
MADS-box转录因子拥有范围广泛的靶目标基因,它的家族成员均是通过 C区的保守序列与其他因子形成复合体而行使功能的。对于高等植物而言,任一逆境环境的改变不仅仅导致植株简单的生理变化,而是通过影响其他因子的响应来影响 MADS蛋白复合体的功能[13]。Hill等[32]证明与 MADS-box亚家族成员 AGL15作用的 SIN3/HDAC1复合体成员SAP18在低温、干旱和高盐胁迫下,其表达量将会增加。由于 MADS-box转录因子自身拥有靶目标的广泛性和复合体多样性等特点,同时不同物种间在生理途径和响应逆境具有差异性,CsMADS1基因在响应茶树逆境胁迫时也表现出特有的表达模式。CsMADS1基因在茶树中可能作为调控基因直接参与抗逆相关生理途径,或者通过调控抗逆相关调控因子间接影响茶树的生长发育。由于植物体内逆境调控的复杂性,以上基于生物信息学分析的预测结果有待更多的试验研究加以验证,以便进一步了解茶树 CsMADS1转录因子的调节途径和作用机理。
[1]Parenicová L, De F S, Kieffer M, et al. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis: new openings to the MADS world [J]. Plant Cell, 2003, 15(7): 1538-1551.
[2]Arora R, Agarwal P, Ray S, et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress [J]. BMC Genomics, 2007, 8(1): 242-263.
[3]Díazriquelme J, Lijavetzky D, Martínezzapater J M, et al.Genome-Wide analysis of MIKCC-type mads box genes in grapevine [J]. Plant Physiology, 2009, 149(1): 354-369.
[4]Leseberg C H, Li A, Kang H, et al. Genome-wide analysis of the MADS-box gene family inPopulus trichocarpa[J].Gene, 2006, 378(1): 84-94.
[5]Wei B, Zhang R Z, Guo J J, et al. Genome-wide analysis of the MADS-Box gene family inBrachypodium distachyon[J].Plos One, 2014, 9(1): e84781.
[6]Rameneni J J, Dhandapani V, Paul P, et al. Genome-wide identification, characterization, and comparative phylogeny analysis of MADS-box transcription factors inBrassica rapa[J]. Genes amp; Genomics, 2014, 36(4): 509-525.
[7]赵兴富, 朱永平, 肖靖译, 等. 植物 MADS-box基因多样性及进化研究进展[J]. 北方园艺, 2015(11): 180-186.
[8]Yu C, Liu Y, Zhang A, et al. MADS-box Transcription factor OsMADS25 regulates root development through affection of nitrate accumulation in rice [J]. Plos One, 2015,10(8): e0135196.
[9]Yamane H, Tao R, Ooka T. Comparative analyses of dormancy-associated MADS-box genes, PpDAM5 and PpDAM6, in low- and high-chill peaches (PrunuspersicaL.)[J]. Journal of the Japanese Society for Horticultural Science,2011, 80(3): 276-283.
[10]Shima Y, Kitagawa M, Fujisawa M, et al. Tomato FRUITFULL homologues act in fruit ripening via forming MADS-box transcription factor complexes with RIN [J].Plant Molecular Biology, 2013, 82(4): 427-438.
[11]Xie Q, Hu Z, Zhu Z, et al. Overexpression of a novel MADS-box geneSlFYFLdelays senescence, fruit ripening and abscission in tomato [J]. Scientific Reports, 2014,4(7491): 4367. DOI: 10.1038/srep04367.
[12]Cooper B, Clarke J D, Budworth P, et al. A network of rice genes associated with stress response and seed development[J]. Proceedings of the National Academy of Sciences, 2003,100(8): 4945-4950.
[13]陈璟, 李名扬, 闫明旭, 等. 矮牵牛 PMADS9基因的结构特征和 mRNA的表达分析[J]. 园艺学报, 2011, 38(1):108-116.
[14]Yu L H, Miao Z Q, Qi G F, et al. MADS-box transcription factor AGL21 regulates lateral root development and responds to multiple external and physiological signals [J].Molecular Plant, 2014, 7(11): 1653-1669.
[15]童启庆. 茶树栽培学[M]. 北京: 中国农业出版社, 2007:4-33.
[16]Wu Z J, Li X H, Liu Z W, et al. De novo assembly and transcriptome characterization: Novel insights into catechins biosynthesis inCamelliasinensis[J]. BMC Plant Biology,2014, 14(1): 277. DOI: 10.1186/s12870-014-0277-4.
[17]Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology amp; Evolution, 2016, 33(7):1870-1874.
[18]Goldberg T, Hecht M, Hamp T, et al. LocTree3 prediction of localization [J]. Nucleic Acids Research, 2014, 42(Web Server issue): W350-W355.
[19]李慧峰, 贾厚振, 董庆龙, 等. ‘鲁星’桃中 10个MADS-box基因克隆和表达分析[J]. 中国农业科学, 2016(23):4593-4605.
[20]Prilusky J, Felder C E, Zeevbenmordehai T, et al. Foldindex:a simple tool to predict whether a given protein sequence is intrinsically unfolded [J]. Bioinformatics, 2005, 21(16):3435-3438.
[21]Combet C, Blanchet C, Geourjon C, et al. NPS@: network protein sequence analysis [J]. Trends in Biochemical Sciences, 2000, 25(3): 147-150.
[22]Schwede T, Kopp J, Guex N, et al. SWISS-MODEL: an automated protein homology-modeling server [J]. Nucleic Acids Research, 2003, 31(13): 3381-3385.
[23]Wu Z J, Tian C, Jiang Q, et al. Selection of suitable reference genes for qRT-PCR normalization during leaf development and hormonal stimuli in tea plant (Camellia sinensis) [J]. Scientific Reports, 2016, 6: 19748. DOI:10.1038/srep19748.
[24]Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR [J]. Nucleic Acids Research, 2001, 29(9): e45.
[25]Becker A, Theissen G. The major clades of MADS-box genes and their role in the development and evolution of flowering plants [J]. Molecular Phylogenetics amp; Evolution,2004, 29(3): 464-489.
[26]Ren Z Y, Yu D Q, Yang Z E, et al. Genome-wide identification of the MIKC-type MADS-box gene family inGossypiumhirsutumL. unravels their roles in flowering [J].Frontiers in Plant Science, 2017, 8: 384.https://doi.org/10.3389/fpls.2017.00384.
[27]Han A, Pan F, Stroud J C, et al. Sequence-specific recruitment of transcriptional co-repressor cabin1 by myocyte enhancer factor-2 [J]. Nature, 2003, 422(6933):730-734.
[28]Yang F, Xu F, Wang X, et al. Characterization and functional analysis of a MADS-box transcription factor gene(GbMADS9) fromGinkgobiloba[J]. Scientia Horticulturae,2016, 212: 104-114.
[29]Guo X, Chen G, Cui B, et al.Solanum lycopersicum,agamous-like MADS-box protein AGL15-like gene,SlMBP11,confers salt stress tolerance [J]. Molecular Breeding, 2016,36(9): 125. DOI: 10.1007/s11032-016-0544-1.
[30]Puig J, Meynard D, Khong G N, et al. Analysis of the expression of the AGL17 - like, clade of MADS-box transcription factors in rice [J]. Gene Expression Patterns,2013, 13(5): 160-170.
[31]Duan W, Song X, Liu T, et al. Genome-wide analysis of the MADS-box gene family inBrassicarapa(Chinese cabbage)[J]. Molecular Genetics and Genomics, 2015, 290(1):239-255.
[32]Hill K, Wang H, Perry S. A transcriptional repression motif in the MADS factor AGL15 is involved in recruitment of histone deacetylase complex components [J]. Plant Journal for Cell amp; Molecular Biology, 2008, 53(1): 172-185.
Cloning of a MADS-box Transcription Factor Gene from Camellia sinensis and its Response to Abiotic Stresses
SHEN Wei, TENG Ruimin, LI Hui, LIU Zhiwei, WANG Yongxin, WANG Wenli, ZHUANG Jing*
Tea Science Research Institute, College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China
In this study, based on the transcriptome database of tea plant, theCsMADS1gene was cloned from cDNA of ‘Longjing43’ by RT-PCR method. The length of open reading frame ofCsMADS1gene was 657 bp, encoding 218 amino acids, which was a typical transcription factor of MADS-box family. Multiple alignments of CsMADS1 with related species showed that the identity of them was 65.65%, with a highly conserved MADS domain and a semi-conserved K domain. The physicochemical properties, hydrophilicity and hydrophobicity, subcellular localization, disordered feature, secondary and tertiary structure were also analyzed. CsMADS1 transcription factor is a hydrophilic protein, may be located in nucleus. The disordered feature of CsMADS1 protein was obvious, which was mainly composed of alpha helix structure, and had similar tertiary structure with MEF2 of human. Quantitative real-time PCR was used to analyze the expression profiles of theCsMADS1gene under abiotic stress treatments of tea cultivar ‘Longjing43’. The results showed that theCsMADS1gene responded to high and low temperatures, drought and high salinity. Different expression patterns ofCsMADS1gene were observed under those abiotic stress treatments.
Camellia sinensis, MADS-box, transcription factor, phylogenetic analysis, abiotic stress, expression analysis
TS272.5+4;Q946.88
A
1000-369X(2017)06-575-11
2017-04-28
2017-07-03
国家自然科学基金面上项目(31570691)
沈威,男,硕士研究生,主要从事茶树分子生物学研究。*通讯作者:zhuangjing@njau.edu.cn
