烤烟烟碱含量的全基因组关联分析
2017-11-16孙鑫文柳璎尹鹏嘉罗成刚樊文强任民
孙鑫,文柳璎,尹鹏嘉,罗成刚,樊文强,3,任民
1 中国农业科学院烟草研究所,中国农业科学院烟草遗传改良与生物技术重点开放实验室,山东青岛科苑经四路11号 266101;2 中国农业科学院研究生院,北京海淀中关村南大街12号 100081;3 苍溪县扶贫互助社管理办公室,四川苍溪刘家巷3号 628400
烤烟烟碱含量的全基因组关联分析
孙鑫1,2,文柳璎1,尹鹏嘉1,2,罗成刚1,樊文强1,3,任民1
1 中国农业科学院烟草研究所,中国农业科学院烟草遗传改良与生物技术重点开放实验室,山东青岛科苑经四路11号 266101;2 中国农业科学院研究生院,北京海淀中关村南大街12号 100081;3 苍溪县扶贫互助社管理办公室,四川苍溪刘家巷3号 628400
烟碱是烟草属特有的一类生物碱,为加深对烟碱遗传调控机制的解析,充分挖掘高烟碱优异等位。本研究选取219份烤烟品种开展了RAD重测序研究,获得了384,904个高质量SNP位点的群体分型数据。同时在四川西昌和山东诸城2个试验点,于2012—2015年相同栽培管理条件下对供试品种的烟叶烟碱含量进行了表型鉴定。利用全基因组关联分析的策略,共发掘到2个与烟碱含量显著关联的SNP位点,分别位于1号染色体的69,912,063bp处和2号染色体的81,115,794bp处。基于上述SNP位点预测到2个候选基因,并将目标SNP位点转化3对可通过PCR检测的功能标记。通过以上研究,加深了烟碱遗传调控机制的解析,为高烟碱优异等位基因型的发掘和高烟碱新品种的分子标记辅助选择提供了可用的标记和基因资源。
烟草;烟碱;全基因组关联分析;重测序;单核苷酸多态性
烟碱即尼古丁(nicotine),是烟草生物碱中的主要成分,约占生物碱总含量的90%~95%[1]。其含量高低是烟草和卷烟工业中重要的品质指标[2]。同时,烟碱在医学上也可用于阿尔茨海默氏病和帕金森综合症的治疗[2-3];在农业上,可作为绿色农药用于生产杀虫剂、除草剂和植物生长调节剂[4];此外,烟碱在动物疾病防治等方面也有着一定的用途[2]。
烟碱在烟草根部合成,最终转运至叶部细胞液泡内进行贮存积累[5]。烟碱含量属于典型的数量性状,虽然受到多种因素的影响[6],但遗传因素是决定品种间含量差异的基础[5,7]。因此可利用分子标记直接从基因水平上检测高、低烟碱基因型,并可有效避免常规表型鉴定中存在的周期长、成本高、易受环境影响等问题,进而提高检测效率,降低研究成本。近年来,已有研究对烟草烟碱含量进行了QTL分析、关联分析以及相关代谢途径的基因功能解析,定位到多个QTL位点,明确了部分基因在合成代谢途径中的作用[8-13]。但是,仍然存在着定位区间较大,功能基因遗传多样性对品种烟碱含量表型变异的影响不明确等的局限性[14],需要进一步开展品种间遗传差异解析和基因发掘研究。
近年来,随着高通量测序技术即二代测序技术[15](Next-generation sequencing, NGS)的快速发展。限制性酶切位点相关DNA(Restriction-site associated DNA, RAD)测序成为当前简化基因组测序策略中运用较为广泛的测序技术,具有基因组覆盖均匀、成本低、周期短等特点[16]。与全基因组关联分析(genome-wide association study, GWAS)[17-18]方法结合可以取得较高的研究效率,在数量遗传性状研究方面潜力巨大。本研究对219份烤烟品种开展了RAD重测序,采用全基因组关联分析的方法,发掘与烟碱含量变异显著关联的单核苷酸多态性(Singlenucleotide polymorphism, SNP)位点,并预测候选基因,开发功能标记,为烟草高烟碱品种的选育,提供可用的标记资源及相应的理论支持。
1 材料与方法
1.1 供试材料的构建
本研究所用219份烤烟品种(见附表1,因版面原因不在纸质版刊登,详见电子版)由国家烟草种质资源中期库提供。从2012—2015年连续在四川西昌和山东诸城2个实验基地开展了田间种植和制样。采用随机区组实验设计,每个品种种植2行,3次重复。每行20株,行距1.0 m,株距0.5m。西昌和诸城实验点的栽培管理措施一致,待烟叶成熟后,采收烟株中部叶片,三重复混收。经烘烤调制制备烟叶样品,烘烤方法参照杨立均等[19]的方法。
1.2 供试材料烟碱含量的测定
烤后烟叶样品烟碱含量测定由农业部烟草产业产品质量监督检验测试中心进行。检测的仪器为SAN++连续流动分析仪(荷兰SKALAR公司)和410型火焰光度计(英国Sherwood公司)。检测方法参照行业标准《YC/T 217—2007》。
1.3 简化基因组重测序(RAD-Seq)及SNP鉴定
采用TIANGENE™ Plant Genomic DNA Kit(天根生化科技(北京)有限公司)提取供试品种的全基因组DNA,并用1.0%的琼脂糖凝胶电泳和NanoDrop 2000 分光光度计对DNA 质量进行检测。RAD简化基因组重测序及SNP鉴定与华大基因有限公司合作开展,采用EcoR I限制性内切酶对供试品种DNA进行酶切并随机打断,电泳回收目标DNA片段,加上接头后进行cluster制备,最后上机测序,测序仪器为IlluminaHiseq 2000;从测序后Clean Data数据中采用GATK-3.2-2流程(https://www.broadinstitute.org/gatk/)进行SNP鉴定。
1.4 数据分析
1.4.1 表型数据统计分析
对供试材料8个环境点的烟碱含量检测数据进行整理后,利用SAS 9.3[20]软件计算各品种的烟碱含量最佳无偏预测值(BLUP)[21],代表每个供试品种的烟碱表型变异。表型差异显著性检验采用SPSS 18.0[20,22-23]软件的one-way ANOVA进行分析。
1.4.2 群体遗传学分析
利用MEGA 7.0[24]软件依据Maximum Composite Likelihood模型计算供试品种间的遗传相似性,并用Neighbor-Joining[25]法绘制供试群体聚类树。采用DnaSP 5.0[26]计算供试群体的核苷酸多态性(Nucleotide Polymorphism)π值和 π(JC)值。SNP间的连锁不平衡(LD,Linkage Disequilibrium)分析采用Haploview 4.2[27]软件,SNP位点间距小于500kb,忽略缺失率超过50%的个体。
1.4.3 全基因组关联分析
全基因组关联分析采用TASSEL V5.0[28]的混合线性模型MLM (PCA+K)进行,统计模型为 y = marker e ff ect + PCA + K + residual。依次过滤掉稀有等位频率(MAF)≤5.0%、无多态性和缺失率高于10.0%的SNP位点。PCA分析设置Components为5。家系关系分析(K)Kinship method为Centered_IBS,Max Alleles为6。MLM 模型的Compression Level为 Optimum Level,Variance Component Estimation选择了P3D。关联位点显著性阈值的判别采用基于FWER (Family-Wise Error Rate)标准的Bonferroni校正法,p = a / n (a = 0.05,n为实际用到的SNP位点数 )。
1.5 候选基因预测
基于关联结果,借助烟草基因组数据库(http://218.28.140.17/),将关联位点定位到烟草连锁群上,确定其物理位置,继而确定关联位点上下游相邻基因。借助烟草基因组数据库及tair网站(http://www.arabidopsis.org/)相应基因的功能注释,进行候选基因预测。
1.6 SNP标记转化
得到目标SNP位点后,首先利用烟草基因组数据库(http://218.28.140.17/),截取SNP位点上下游各300bp的序列;然后通过WASP[29]网站(http://bioinfo.biotec.or.th/WASP)设计AS-PCR引物,并合成引物;最后用1.0%的琼脂糖凝胶电泳检测以确定实际扩增效果。
2 结果与分析
2.1 烟草种质烟碱含量的表型变异统计分析
对供试品种8个年点的烟碱含量表型变异进行统计分析,结果显示各年点的变异系数均表现出较高的幅度,表明品种间表型变异丰富(表1)。进而根据8个年点的表型变异数据,计算每个供试品种的烟碱含量最佳无偏预测值(BLUP),以代表每个供试品种的烟碱含量表型变异水平。烟碱含量表型变异BLUP值平均为3.087,变幅从1.196到5.732(图1A),经w检验符合正态分布(图1B)。烟碱含量最高的5个品种为福泉朝天立、8022、开阳团鱼叶、毕金一号和长葛柳叶,其BLUP值分别达到4.442、4.512、4.546、4.563和 5.732;最低的 5个品种为大柳条、9201、OX2028、福泉高脚黄和TI1112,其 BLUP值 分 别 为 1.196、1.366、1.636、1.639和1.651。
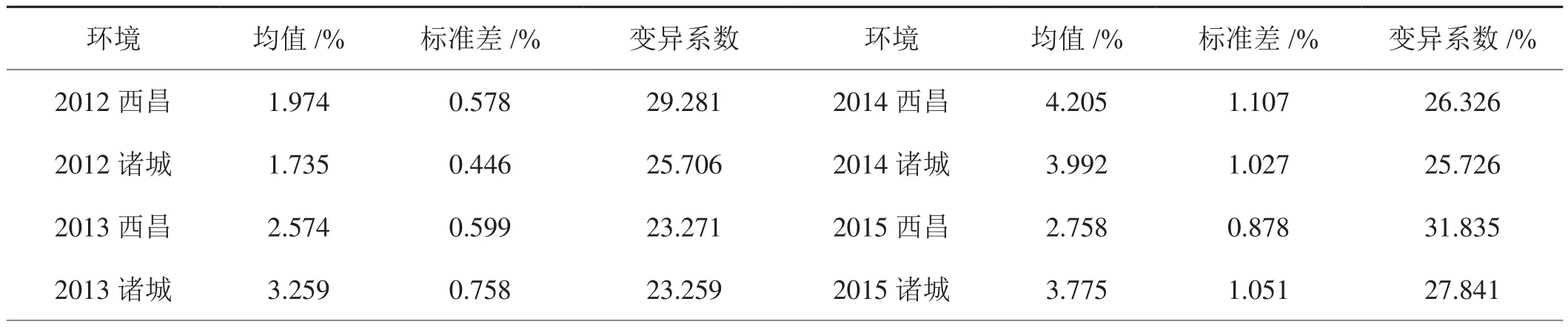
表1 各年点烟碱含量的描述统计Tab.1 Descriptive statistics of nicotine content of different years/at different sites

图 1 供试材料各年点烟碱含量表型变异分析图Fig.1 Variation analysis of nicotine content from the tested materials of different years/at different sites
2.2 供试群体简化基因组重测序
通过简化基因组(RAD)重测序在供试群体上一共检测到1,935,878个SNP位点,其中定位到染色体序列上的有1,167,614个SNP位点,在染色体序列上平均SNP间距2,738 bp。从上述SNP数据中进一步过滤得到了384,904个有高质量分型的SNP位点,平均间距8,311 bp,占发掘到SNP位点的33%。分析了24条染色体上SNP的密度分布情况,发现在17号染色体上SNP的密度较高,高密度的部分主要集中在17号染色体的前半部(图2)。基因序列SNP的突变类型中错义突变11142个,占59.567%,无义突变654个,占3.496%,同义突变6909个,占36.937%。
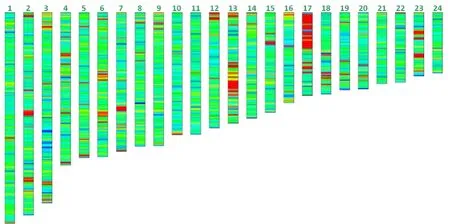
图2 24条染色体的SNP密度分布热图Fig.2 SNP density heat mapof 24 chromosomes
2.3 供试群体遗传多样性分析
通过计算供试群体的核苷酸多态性(Nucleotide Polymorphism)可知,总群体的π值为:0.04051,π(JC)值为:0.04051;国内育成品种的π值为:0.05868,π(JC)值为:0.06217;国外引进品种的π值为:0.04231,π(JC)值为:0.04501;国内地方品种的π值为:0.02574,π(JC)值为:0.02653。表明,供试群体中国内育成品种和国外引进品种的遗传多样性较高,国内地方品种的遗传多样性较低。供试品种的NJ聚类分析(图3)也反映出上述结论,国内育成品种和国外引进品种的聚类关系更加发散,而国内地方品种则集中在少数分枝。并且从图3中还可以发现国内育成品种和国外引进品种在多个聚类分枝上互相交错,表明有一定的基因交流。上述现象与我国当前育种中大量采用优异引进品种作亲本材料的育种实践是相符的。但国内地方品种与育成品种的交错较少,上述现象与其他作物有较大区别。
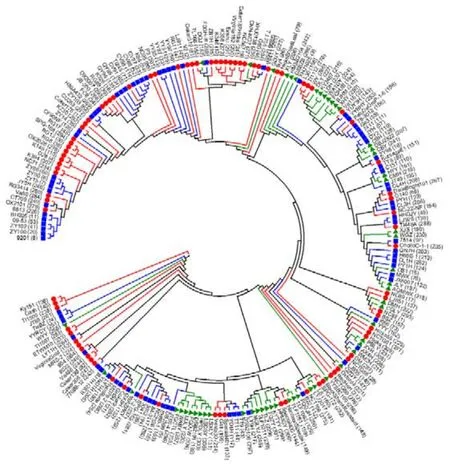
图3 供试群体的N-J聚类树Fig.3 Neighbor-joining cluster of the tested materials
2.4 供试群体SNP变异位点与烟碱含量的全基因组关联分析
在依据材料与方法1.4.3的标准进行了数据处理后,将供试群体的烟碱含量表型数据与SNP数据进行关联分析。实际进入关联分析运算的SNP位点共274,652处。根据Bonferroni[30]法可知,关联位点的显著性p-value应当低于1.82×10-7,即本研究的显著性阈值应为6.7(-log10p-value)。据此阈值共发掘到2个关联SNP位点(图4),分别位于1号和2号染色体。其中1号染色体上的SNP位点位于该染色体的69,912,063bp处(C1_69,912,063),-log10p-value= 9.667,能够解释24.5%的表型变异,2号染色体上的SNP位点位于2号染色体的81,115,794bp处(C2_81,115,794),-log10p-value= 7.198,能够解释17.92%的表型变异。
2.5 高烟碱含量优异等位变异的筛选
对关联到的SNP位点进行了不同基因型间烟碱含量的统计分析。结果表明,SNP位点C1_69,912,063在群体中的等位变异为A和C。A基因型烟碱含量平均BLUP值为3.502,C基因型BLUP值为2.768。方差分析结果表明,该SNP不同等位变异间烟碱含量差异达到极显著水平(图5A),A基因型烟碱含量高,为该位点的优异等位变异,占供试品种总数的36.1%;SNP位点C2_81,115,794,在群体中的等位变异为T和C,T基因型烟碱含量平均BLUP值为3.413,C基因型BLUP值为2.736。方差分析结果表明,该SNP不同等位变异间烟碱含量差异达到极显著水平(图5B),T基因型烟碱含量高,为该位点的优异等位变异,占供试品种总数的44.7%。
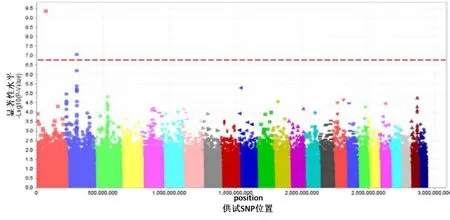
图4 烟碱含量全基因组关联分析Fig. 4 Manhattan plot for genome-wide association analysis of nicotine contento
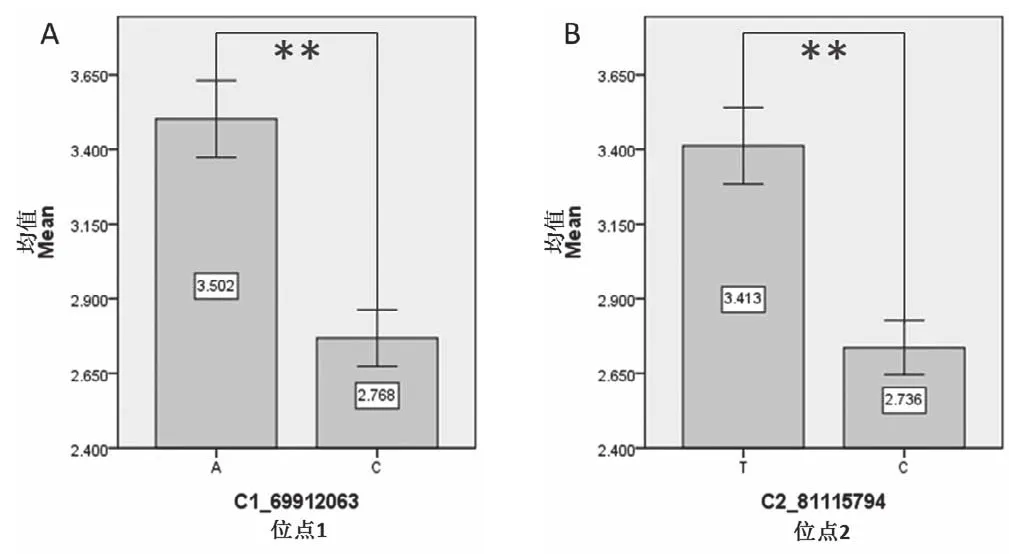
图5 不同SNP位点基因型所对应的烟碱含量Fig.5 Different average nicotine content between two alleles
2.6 烟碱含量相关候选基因预测
通过中国烟草基因组数据库分析了上述2个关联位点上下游的基因注释、功能预测,同时结合关联区段的连锁不平衡(LD)分析,进行了烟碱含量相关候选基因的预测。依据与关联位点处于同一个LD区段,基因的功能注释可能与烟碱代谢调控相关等原则,在紧邻关联位点的位置预测到2个烟碱含量候选基因,编号分别为Ntab0691660和Ntab0574880。编号为Ntab0691660的基因位于关联位点C1_69,912,063的下游28kb处,该基因与拟南芥ATCYS6基因同源。在拟南芥中,ATCYS6编码具有半胱氨酸蛋白酶抑制剂活性的蛋白质,过度表达可增加对非生物胁迫因素的耐受能力,故推测Ntab0691660可能通过调控生物胁迫抗受能力相关通路参与了烟碱合成代谢的调控;候选基因Ntab0574860位于关联位点C2_81,115,794的下游约19kb处(图6),该基因与关联位点共同位于一个单体型(Haplotype)中,与水稻中的OSJMJ706基因同源。JMJ706基因编码一个异染色质相关的去甲基化酶,有较弱的去甲基化酶的活性,相关位点的甲基化修饰与转录抑制相关,功能缺失突变体表现为甲基化修饰程度积累,影响小穗及花发育[31-32]。推测在烟草中,Ntab0574860可能与烟碱合成关键酶腐胺N-甲基转移酶[1](PMT,putrescine-N-methyltransferase)相关基因的表达调控有关,从而影响了最终烟碱含量的表型变异。
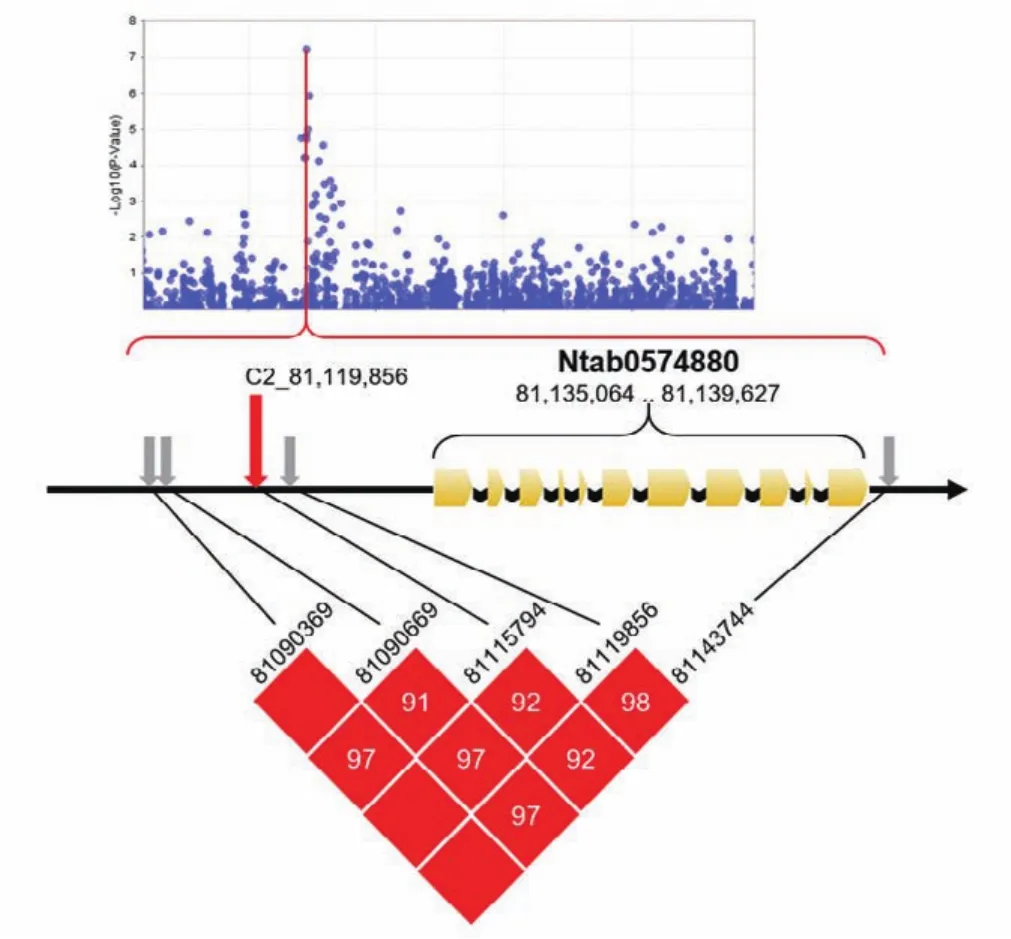
图6 高烟碱含量候选基因预测Fig. 6 Candidate genes prediction of high nicotine content
2.7 SNP位点的功能标记开发
针对关联位点和候选基因设计了相应的等位变异特异PCR(AS-PCR)[33]标记,相关序列见表2。关联位点C1_69,912,063筛选到1个可用的标记AP014,实际扩增检验表明,其扩增带型清晰,分型结果与测序一致(图7)。以供试材料41和8分别为A、C等位变异的对照品种,依据扩增带型可方便快捷的鉴定供试材料在该位点上的碱基类型。
SNP位点C2_81,134,669紧邻关联位点C2_81,115,794,位于候选基因Ntab0574860的上游5Kb以内的调控区域,由于和关联位点C2_81,115,794同处于一个紧密的LD区间(见图6),因而对烟碱含量的选择有着相同的效率。该位点在供试群体上等位变异为T和G,分别占供试群体的40.0%和60.0%。从该位点上筛选到2组扩增带型清晰的AS-PCR标记(图7):AP068和AP072,均以供试材料41和8为T、C等位变异的对照品种。

图7 SNP位点的功能标记开发Fig.7 Functional markers development of SNP loci

表2 引物序列Tab.2 AS-PCR primer sequences
3 讨论与结论
3.1 烟碱含量定位结果比较分析
本研究通过全基因组关联分析策略,在烟草基因组内定位到2个与烟碱含量变异显著关联的位点或区段,峰值SNP分别在1号染色体的69,912,063 bp处(C1_69,912,063),和2号染色体的81,115,794 bp处(C2_81,115,794)。在已有的报道[9-10,12,34-35]中,主要通过构建分离群体的方法定位到了若干烟碱含量QTL位点,数量从2到8个不等。所用分子标记主要为以PCR为基础的第二代标记技术,包括了RAPD、SRAP、ISSR、AFLP和SSR等。与以往研究相比,得益于近年来快速发展的高通量测序技术,本研究实现了更高密度的基因组覆盖,同时通过SNP位点直接将目标性状的QTL区段定位到物理图谱,在以下2个方面取得了显著的优势,首先,关联SNP位点有唯一的物理位置,方便了今后不同研究之间的比较和补充;其次,自然群体携带了更多的染色体交换事件[36],加之高密度的SNP位点,可以获得更加精细的遗传定位信息,从而为下一步的基因发掘和分子育种应用奠定了更好的基础。
3.2 关联位点在烟草分子育种中的应用
鉴于通过高通量测序得到的SNP位点难以直接用于烟草分子育种,本研究还开展了SNP到AS-PCR标记转化的研究。AS-PCR标记是基于PCR技术的新型分子标记,通过2条竞争性引物,可对模板DNA中目标SNP位点的碱基组成进行特异检测[37-38]。本研究针对2个关联位点,设计出了3组可用的ASPCR标记。上述标记的特异性强,扩增条带易于识别,方便应用于烟草分子育种。研究加深了对烟碱含量相关基因遗传变异的理解,为优异基因的聚合提供了实验证据,为高烟碱材料的选育提供了分子依据。
3.3 问题与展望
在开展烟叶成熟期烟碱含量表型鉴定的过程中,由于烟草植株大,生长周期长,导致实验用地面积大,研究成本高,限制了供试群体规模的扩大。下一步,拟开展苗期的表型鉴定研究,以降低研究成本,方便扩大供试群体规模。同时,拟增加表型鉴定的范围,从烟碱含量的检测扩大到相关基因的表达分析,即表达基因组关联分析[39](expression genome-wide association study, eGWAS)研究。通过表达基因相关位点的关联分析,进一步挖掘烟碱含量相关位点,继而开展基于表达基因的SNP位点发掘及候选基因预测等后续研究。
另外,下一步对预测的候选基因还将继续开展功能验证,比如进行过表达[40]和CRISPR/CAS9敲除[41]等,进一步分析明确候选基因功能,为高烟碱品种的选育提供坚实的理论依据。
[1] 张洪博. 烟草重要基因篇:3.烟草烟碱合成代谢相关基因[J]. 中国烟草科学,2014, 35(3):117-120.ZHANG Hongbo. Important gene of tobacco: 3.Nicotine biosynthesis and metabolism related genes in tobacco[J].Chinese Tobacco Science, 2014, 35(3):117-120.
[2] 薛小平,陈懿,王茂胜等. 烟碱的研究现状[J]. 安徽农业科学,2008,36(10):4121-4122,4143.XUE Xiaoping, CHEN Yi, WANG Maosheng, et al. Research status of nicotine[J]. Journal of Anhui Agri,2008,36(10):4121-4122,4143.
[3] 张真娜,张桂治. 烟碱对帕金森病治疗的潜在作用[J].中国烟草学报,2013,19 (06):114-119.ZHANG Zhenna, ZHANG Guizhi. On nicotine’s potential role in treating Parkinson’s disease[J].Acta Tabacaria Sinica, 2013,19 (06):114-119.
[4] 张先海,史丽岩,史景明. 烟碱的研究现状[J]. 海峡药学,2010,22(11):16-18.ZHANG Xianhai, SHI Liyan, SHI Jingming. Research status of nicotine[J].Strait Pharmaceutical Journal,2010,22(11):16-18.
[5] 金云峰,李军营,张建波等. 烟草烟碱代谢的生化和分子机制及其调控[J]. 基因组学与应用生物学,2015,34(04):882-891.JIN Yunfeng, LI Junying, ZHANG Jianbo, et al.Biochemical and molecular mechanism of nicotine metabolism in tobacco plants[J]. Genomics and Applied Biology, 2015, 34(04):882-891.
[6] 周齐志. 烟草烟碱含量研究进展[J]. 安徽农业科学,2008,36(06):2359-2361.ZHOU Qiping. Research progress of nicotine content in tobacco[J]. Journal of Anhui Agri, 2008, 36(06):2359-2361.
[7] Saitos F, Nona M, Kawashima N. The alkaloid contents of sixty Nicotiana species[J]. Phytochemistry, 1985,24(3):477-480.
[8] 谭效磊,徐秀红,王暖春等. 烤烟易烤性QTL定位分析[J]. 分子植物育种,2012,10( 02):201-206.TAN Xiaolei, XU Xiuhong, WANG Nuanchun, et al.QTLs analysis of the easy curing potential in flue-cured tobacco[J]. Mol Plant Breed, 2012,10(02):201-206.
[9] 蔡长春,柴利广,王毅等. 白肋烟分子标记遗传图谱的构建及部分性状的遗传剖析[J].作物学报,2009,35(9):1646-1654.CAI Changchun, CHAI Liguang, WANG Yi, et al.Construction of genetic linkage map of burley tobacco(Nicotiana tabacum L.)and genetic dissection of partial traits[J], Acta Agron Sin, 2009,35(9):1646-1654.
[10] 肖炳光,卢秀萍,焦芳蝉等. 烤烟几种化学成分的QTL初步分析[J]. 作物学报,2008,34(10):1762-1769.XIAO Bingguang, LU Xiuping, JIAO Fangchan, et al.Preliminary QTL analysis of several chemical components in flue-cured tobacco (Nicotiana tabacum L.)[J]. Acta Agron Sin, 2008, 34(10):1762-1769.
[11] 王莹,王艺,黄薇. 全基因组关联分析在人类复杂疾病研究中的应用[J]. 诊断学理论与实践,2010, 9(5):430-432.WANG Ying, WANG Yi, HUANG Wei. The application of whole genome association analysis in the study of human complex diseases[J]. J Diagn Concepts Pract, 2010,9(5):430-432.
[12] 李华丽,陈美霞,周东新等. 烟草六个重要性状的QTL定位[J]. 作物学报,2011,37(9):1577-1584.LI Huali, CHEN Meixia, ZHOU Dongxin, et al.QTL analysis of six important traits in tobacco (Nicotiana tabacum L.) [J]. Acta Agron Sin,2011,37(9):1577-1584.
[13] 牟建英,钱玉梅,任民等. 烟草白粉病抗性基因的QTL定位[J]. 中国烟草学报,2013,19(4):105-108.MU Jianying, QIAN Yumei, REN Min, et al. QTL analysis of resistance gene to powdery mildew in tobacco[J].Acta Tabacaria Sinica, 2013,19(4):105-108.
[14] 晁毛妮,郝德荣,印志同等. 大豆生物量与产量组分间的相关及关联分析[J]. 作物学报,2014,40(1):7-16.CHAO Maoni, HAO Derong, YIN Zhitong, et al.Correlation and association analysis between biomass and yield component sin soybean[J].ActaAgron Sin, 2014,40(1):7-16.
[15] 王洋坤,胡艳,张天真. RAD-seq技术在基因组研究中的现状及展望[J]. 遗传,2014,36(1):41-49.WANG Yangkun, HU Yan, ZHANG Tianzhen. Current status and perspective of RAD-seq in genomic research[J].Hereditas, 2014,36(1):41-49.
[16] 陈勋. 基于简化基因组测序的油菜高通量SNP分析及白菜基因组DNA甲基化解析[D].华中农业大学,2014.CHEN Xun. High-throughput SNP analysis in oilseed(Brassica Napus L.) andgenome-Scale DNA methylation profiling in brassica rapa based on reduced representation sequencing[D]. Huazhong Agricultural University, 2014.
[17] 段忠取,朱军. 全基因组关联分析研究进展[J]. 浙江大学学报(农业与生命科学版),2015,41(4):385-393.DUAN Zhongqu, ZHU Jun. Research progress of genomewide association study[J].Journal of Zhejiang University(Agric&Life Sci),2015,41(4):385-393.
[18] 任民,张长静,蒋彩虹等. 基于高密度SSR连锁群的烟草致香物质关联分析[J]. 中国烟草学报,2014,20(4):88-93.REN Min, ZHANG Changjing, JIANG Caihong, et al.Association analysis of tobacco aroma constituents based on high density SSR linkage group[J]. Acta Tabacaria Sinica,2014,20(4):88-93.
[19] 杨立均, 宫长荣. 烤烟三段式烘烤操作技术[J]. 烟草科技, 2003(7):46-48.YANG Lijun, GONG Changrong. The Operating Technique of Three-stage Curing of Flue-cured Tobacco[J]. Tobacco Science &Technology, 2003(7):46-48.
[20] 陈广凤,陈建省,田纪春. 小麦株高相关性状与SNP标记全基因组关联分析[J]. 作物学报,2015,41(10):1500-1509.CHEN Guangfeng, CHEN Jiansheng, TIAN Jichun.Genome-Wide association analysis between SNP markers and plant height related traits in wheat[J].Acta Agron Sin,2015,41(10):1500-1509.
[21] SUN Qi, LI Wenlan, CHEN Litao, et al. Research Progress on Plant Genomic Selection(GS) and Its Application in Maize Breeding[J].Acta Botanica Boreali-Occidentalia Sinica,2016,36(6):1269-1277.
[22] 张文彤, 阎洁. SPSS 统计分析基础教程[M]. 北京: 高等教育出版社, 2004.pp257-278.ZHANG Wentong, YAN Jie. A foundation course for SPSS statistical analysis[M]. Beijing: Higher Education Press,2004. pp257-278.
[23] 樊文强,孙鑫,杨爱国等. 基于关联分析发掘烟草高钾优异等位变异[J]. 中国烟草学报,2016,22(2):100-107.FAN Wenqiang, SUN Xin, YANG Aiguo, et al. Exploring Elite Alleles for high potassium by Association Analysis[J].Acta Tabac Sin, 2016, 22(2):100-107.
[24] Baker K R. Scheduling the production of components at a common facility [ J]. IIE Transactions, 1988, 20( 1):32-35.
[25] Letunic I and Bork P. Interactive Tree Of Life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees[J]. Nucleic Acids Res, 2016,44(1):242-245.
[26] Librado P, Rozas J.DnaSP v5: A software for comprehensive analysis of DNA polymorphism data[J].Bio informatics, 2009,25(11):1451-1452.
[27] Barrett JC, Fry B, Maller J, Daly MJ. Haploview:analysis and visualization of LD and haplotype maps.Bioinformatics, 2005, 21(2): 263–265.
[28] Bradbury P J, Zhang Z, Kroon D E, et al. TASSEL: software for association mapping of complex traits in diverse samples[J].Bioinformatics, 2007, 23(19): 2633-2635.
[29] Wangkumhang P, Chaichoompu K, Ngamphiw C, et al.WASP: a Web-based Allele-Speci fi c PCR assay designing tool for detecting SNPs and mutations[J]. BMC Genomics,2007, 8(1):275.
[30] Bland JM, Altman DG. Multiple significance tests: the Bonferroni method [ J] . Br Med J, 1995, 310 ( 6986) : 1073-1075.
[31] SunQW,ZhouDX.Rice jmjC domain-containing gene JMJ706 encodes H3K9 demethylase required for floral organ development[J]. PANS, 2008, 105(36):136-179.
[32] 王彬. emf2b-jmj706双突变体表观修饰与花发育基因的关联性分析[D].四川农业大学,2013.WANG Bin. Analysis of modification in emf2b-jmj706 double mutants and flower development genes in rice[D].Sichuan Agricultural University, 2013.
[33] Landegren U, Kaiser R, Caskey C T, et al. DNA diagnosticsmolecular techniques and automation[J]. Science, 1988,242(4876):229-237.
[34] 李茜. 烟草遗传图谱构建与农艺性状和化学成分的QTL定位[D].西南大学,2015.LI Qian. Construction of linkage genetic map and QTL mapping for agronomic characters and chemical quality traits in tobacco [D].Southwest University, 2015.
[35] 柴利广. 白肋烟遗传连锁图谱的构建及烟碱含量QTL的定位[D].华中农业大学,2008.CHAI Liguang. Construction of genetic linkage map and mapping QTL of nicotine content in burley tobacco[D].Huazhong Agricultural University,2008.
[36] 张焕欣,翁建峰,张晓聪等. 玉米穗行数全基因组关联分析[J]. 作物学报,2014,40(1):1-6.ZHANG Huanxin, WENG Jianfeng, ZHANG Xiaocong, et al. Genome-wide association analysis of kernel row number in maize[J].Acta Agron Sin,2014,40(1):1-6.
[37] 吴明根,金万赫,许勇男. 采用ASPCR技术检测抗药性雨久花ALS突变基因碱基种类的探讨[J]. 安徽农业大学学报,2011,38(2):296-298.WU Minggen, JIN Wanhe, XU Yongnan. Development of allele-speci fi c PCR for detection of herbicide-resistance in Monochoriakorsakowii Regel et Maack[J]. Journal of Anhui Agricultural University, 2011,38(2):296-298.
[38] 曹西蓉,吴德生. AS-PCR在ADH2、ALDH2基因多态型分析中的应用[J]. 环境与职业医学,2004,21(5):371-373.CAO Xirong, WU Desheng. AS-PCR—a Convenient and Cost-saving Method for Analyzing the Genotypes of Alcohol Dehydrogenase2(ADH2)and Aldehyde Dehydrogenase 2(ALDH2)Loci with High Accuracy[J]. J EnvironOccup Med, 2004,21(5):371-373.
[39] Zou F, Chai H S, Younkin C S, et al. Brain Expression Genome-Wide Association Study (eGWAS) Identifies Human Disease-Associated Variants[J]. Plos Genetics,2012, 8(6):e1002707.
[40] 张黎, 牛向丽, 张惠莹,等. 水稻木葡聚糖内糖基转移酶基因OsXTH11过表达的作用分析[J]. 中国农业科学,2012, 45(16):3231-3239.ZHANG Li, NIU Xiangli, ZHANG Huiying,et al.Functional Analysis via Overexpressing Xyloglucan Endotransglycosylase Gene OsXTH11 in Rice[J].ScientiaAgriculturaSinica, 2012, 45(16):3231-3239.
[41] 朱金洁. CRISPR-Cas9介导的玉米基因组定点编辑研究[D].中国农业大学,2015.ZHU Jinjie. Targetedgenome editing in maize using CRISPR-Cas9[D].China Agricultural University,2015.

附表 1 本研究所用的219份供试品种Supplementary Table1 219 tested materials in this study
续表
续表
:SUN Xin, WEN Liuying, YIN Pengjia, et al. Genome-wide association study of nicotine content in fl ue-cured tobacco [J]. Acta Tabacaria Sinica, 2017, 23(1)
*Corresponding author. Email:renmin@caas.cn
Genome-wide association study of nicotine content in fl ue-cured tobacco
SUN Xin1,2, WEN Liuying1, YIN Pengjia1,2, LUO Chenggang1, FAN Wenqiang3, REN Min1*
1 Tobacco Research Institute, Chinese Academy of Agricultural Sciences/ Key Laboratory of Tobacco Genetic Improvement and Biotechnology, Qingdao 266101, Shandong, China;2 Post Graduate School, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3 Poverty Alleviation and Mutual Aid Cooperative Management Office, Cangxi County, Cangxi 628400, Sichuan, China
Nicotine is a species-speci fi c alkaloid in Nicotiana.219 fl ue-cured tobacco varieties were selected as tested materials and a high quality SNP genotyping dataset of 384,904 loci were obtained via whole genome RAD resequencing. For phenotyping of nicotine content of cured leaves, all varieties studied were planted in 2 di ff erent experimental sites (Xichang of Sichuan province and Zhucheng of Shandong province) under same cultivation and management conditions from 2012 to 2015. Genome-wide association study showed that there were 2 peak SNPs signi fi cantly correlated with nicotine content (p<0.000001) which was located on 69,912,063 bp of chromosome 1 (C1_69,912,063) and 81,115,794 bp of chromosome 2 (C2_81,115,794) respectively. Based on the above mentioned SNPs, 2 candidate genes were predicted and 3 novel Allele-Speci fi c PCR markers were veri fi ed. This study gave a comprehensive understanding of nicotine genetic variation and provided available gene and molecular marker resources for the high-nicotine tobacco varieties breeding.
tobacco; nicotine; genome wide association study; resequencing; single nucleotide polymorphism
孙鑫,文柳璎,尹鹏嘉,等. 烤烟烟碱含量的全基因组关联分析[J]. 中国烟草学报,2016,23(1)
中国农业科学院科技创新工程(ASTIP-TRIC01) ;国家烟草专卖局烟草基因组计划重大专项 (110201301008);中国农业科学院烟草研究所青年科学基金(2015A01)
孙鑫(1992—),硕士,主要从事烟草烟碱含量遗传变异研究,Tel:18563934302,Email:sunxincaas@163.com
任民(1979—),Tel:0532-88702209,Email:renmin@caas.cn
2016-09-12;< class="emphasis_bold">网络出版日期:
日期:2017-01-25
