大肠杆菌合成羟基脂肪酸的研究进展
2017-09-30何乔飞
何乔飞,吴 辉
(华东理工大学 生物反应器工程国家重点实验室,上海 200237)
大肠杆菌合成羟基脂肪酸的研究进展
何乔飞,吴 辉
(华东理工大学 生物反应器工程国家重点实验室,上海 200237)
羟基脂肪酸(hydroxy fatty acids,HFAs)具备羟基和羧基功能团,是聚酯纤维和聚酰胺的前体,是当今绿色聚合物材料的理想单体之一,在医疗、工业和食品等方面有着广泛应用。本文中,笔者对利用大肠杆菌生物催化合成羟基脂肪酸的不同策略、研究进展及其成果进行综述,并介绍了大肠杆菌中利用代谢工程策略合成羟基脂肪酸的
研究方向与进展。
羟基脂肪酸;脂肪酸;生物催化;代谢工程;大肠杆菌
羟基脂肪酸(hydroxy fatty acids,HFAs)是由一个或多个羟基和一端具有羧基的长碳链组成的饱和或不饱和脂肪酸,根据其碳链长、羟基数和羟基位置的不同而种类繁多。羟基脂肪酸中的羟基使其具有特殊的性质,例如较高的熔点、沸点、黏度和反应活性;易倾向于形成二聚物;亲水性高,但不溶于水[1]。这些特殊的性质都赋予了羟基脂肪酸重要的应用价值,如它可作为聚酯纤维和聚酰胺的前体,合成绿色环保的聚合物材料[2];作为药物中间体和合成前体[3];作为化妆品成分、表面活性剂、泡沫改进剂、乳化剂和除臭棒原料等[4];且具有很强的抗真菌活性,在食品生物防腐剂方面的应用十分广阔[5]。
目前,羟基脂肪酸的生产方法主要有化学合成法和微生物合成法。但是化学合成法有生产成本高、反应过程和提取工艺复杂以及环境污染较为严重等缺点,导致该工艺的商业化生产困难。因此,探索操作简便、成本低廉和绿色环保的生物合成路线是今后羟基脂肪酸发展的趋势。生物催化技术是指利用微生物代谢过程中所产生的酶或生物有机体作为催化剂对外源化合物进行化学转化的技术。该技术因绿色环保、操作简便及特异性强等优点受到国内外学者的广泛青睐。已有研究报道多种微生物能够将脂肪酸催化合成羟基脂肪酸。目前,大多数羟基脂肪酸以野生菌发酵和工程菌生产为主。野生菌发酵主要有假单胞菌属、芽孢菌属、微菌属、酵母菌和曲霉属等多种类型,其中以芽孢菌属生产羟基脂肪酸为主[6]。吴立新等[7]利用短小芽孢杆菌(Bacillus pumilus)突变株M-F641发酵生产ω-羟基脂肪酸,对发酵条件进行优化,ω-羟基脂肪酸的产率达 22.3%。Durairaj等[8]选用来自尖孢镰孢(FoCYP)的两种细胞色素P450单加氧酶FoCYP539A7和FoCYP655C2与异源酿酒酵母CPR(ScCPR)和同源尖孢镰孢CPR(FoCPR)还原酶系统重组,并敲除Saccharomyces cerevisiae的pox1基因以降低β-氧化,实验发现,同源CYP539A7-FoCPR和 CYP655C2-FoCPR 重组系统分别产生 73.8、52.2 mg/L 的10-羟基癸酸以及72.2、51.9 mg/L的12-羟基十二烷酸和45.1 mg/L羟基辛酸。由于大肠杆菌具备遗传背景清楚、操作简便以及生长周期短等优点,已经被广泛用于生产脂肪酸及其衍生物。本文中,笔者主要阐述大肠杆菌生物催化转化生产羟基脂肪酸(HFAs)。近些年,代谢工程技术快速发展,利用代谢工程技术对大肠杆菌代谢途径改造合成羟基脂肪酸也成为重要研究方向,因此,本文也对大肠杆菌代谢合成羟基脂肪酸的相关研究进行系统综述。
1 羟基脂肪酸生物催化策略
羟基脂肪酸生物催化策略的核心是以脂肪酸为底物的羟化酶,现已经发现多种酶系可以使游离脂肪酸特定碳链位置发生羟基化(图1):①使用羟化酶和P450酶系(P450s)可直接在特定位置引入羟基;②减少特定位置双键的产生并随后用去饱和酶和水合酶在双键中进行水合;③减少特定位置双键的产生并随后进行环氧化,然后使用去饱和酶、环氧化酶和环氧化物水解酶进行水解;④在(顺式,顺式)-双键和双键生成二羟基后使用脂氧合酶和二醇合酶[9]。
已知参与脂肪酸羟基化的酶有P450单加氧酶、水合酶、脂氧合酶、烷烃羟化酶和二醇合酶,总结于表1。
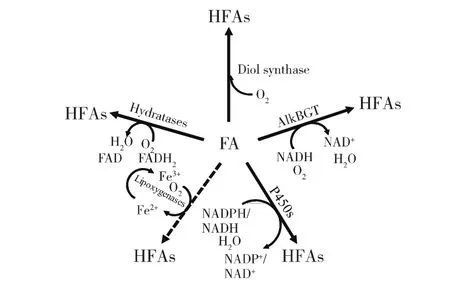
图1 羟基脂肪酸合成代谢流程Fig.1 Schematic representation of the metabolic process of synthetic hydroxyl fatty acid

表1 脂肪酸羟基化酶系简表Table 1 Summary of fatty acid hydroxylase system
1.1 P450酶系
在已开发的几种策略中,P450酶系是最有效且应用最为广泛的。细胞色素P450酶作用于底物的非活性甲基端的碳氢键,即主要作用于C12~C22的脂肪酸末端第2个碳的羟基化反应,其对底物有较强的专一性,并依赖于NADPH,且催化底物的速度与酶系统来源有很大的相关性[2]。Wang等[20]比较了3种不同的P450基因(LCR、YetO和P450BM3)在大肠杆菌中表达的差异,结果发现,含有LCR基因的菌株仅产生少量的羟基脂肪酸(HFAs),而含有P450BM3基因的菌株产生了 60.5 mg/L的 HFAs,比含有YetO基因的菌株高1.2倍,产物大多为ω-3-羟基十二烷酸(ω-3-C12:0)、ω-2-羟基十二烷酸(ω-2-C12:0)、ω-1-羟基十二烷酸(ω-1-C12:0)、3-羟基十四烷酸(ω-3-C14:0)、ω-2-羟基十四烷酸(ω-2-C14:0)和ω-1-羟基十四烷酸(ω-1-C14:0)。由此可以证明,P450BM3基因具有最高的催化能力。
脂肪酸羟基化P450酶系可分为羧基末端羟化酶(α-羟化酶)和末端第二位羟化酶(ω-羟化酶),α-羟化酶催化饱和脂肪酸(C12~C18)和不饱和脂肪酸羧基末端的α位和β位,分别转化为2-羟基和3-羟基脂肪酸,具有高催化活性[21]。绝大部分P450酶系的催化位点为ω-位,而不同来源的同种酶的催化位点不尽相同,其中来自精氨酸单胞菌的CYP152仅在脂肪酸的 α-位催化羟基化[15];然而,来自枯草芽孢杆菌的CYP152可同时催化脂肪酸中α-位(40%)和 β-位(60%)的羟基化[22]。目前,研究中最具有挑战性的是控制长链游离脂肪酸(free fatty acids,FFA)的末端位置羟基化,即 ω-羟基化。负责脂肪酸ω-羟基化的细菌单加氧酶已经从几种芽孢杆菌属中克隆出来,包括巨大芽孢杆菌(B.megaterium)[23]、枯草芽孢杆菌(B.subtilis)[24]、炭疽芽孢 杆 菌 (B.anthracis)[25]和 蜡 状 芽 孢 杆 菌(B.cereus)[26]。其中,来自巨大芽孢杆菌的细胞色素P450BM3是这些酶中研究最透彻的成员。Scheps等[27]将来自 Marinobacter aquaeloei 的单加氧酶CYP153A与来自B.megaterium的P450BM3还原酶结构域融合,并在天然连接域前加上3xGly-Gly-Ser(GGS),组成CPR2,同时对CYP153A(G307A)进行单点突变后得到CPR2mut,结果表明,与CPR2相比,CPR2mut具有更高的ω-羟化活性。
P450s不仅具有较高的催化活性,还可催化多种有机底物,其反应通常具有较高的区域和立体选择性,因此,P450酶系具有高度的底物特异性。Munday等[28]比较了CYP102家族的两种单加氧酶Krac0936和Krac9955的特性,结果发现:Krac0936使用NADPH作为辅因子,主要在直链饱和脂肪酸ω-1和ω-2位进行羟基化,且Krac0936对较短的不饱和脂肪酸活性较低,而Krac9955更倾向于氧化较短的脂肪酸,并在ω-4、ω-5和ω-6位进行氧化。
1.2 水合酶
水合酶(hydratase)作用于不饱和脂肪酸的双键,将水分解成氢和羟基,形成羟基脂肪酸。目前已知10-羟基硬脂酸可通过肠球菌(Enterococcus gallinarum)、乳酸杆菌(Pediococcus acidilactici)、乳杆菌 属 (Lactobacillus sp)[29]、化 脓 性 链 球 菌(Streptococcus pyogenes)[30]、大肠球菌(Macrococcus caseolyticus)[31]以及赖氨酸芽孢杆菌(Lysinibacillus fusiformis)[32]等的水合酶催化合成。来源不同的水合酶对不同底物具有不同的催化活性。双歧杆菌(Bifidobacterium breve)[33]和 植 物 乳 杆 菌(Lactobacillus plantarum)[34]对油酸具有最高水合活性,而嗜酸乳杆菌NBRC13951的脂肪酸双键水合酶与亚油酸或γ-亚麻酸作用活跃[35]。在黄素腺嘌呤二核苷酸(FAD)和 NADH存在下,植物乳杆菌AKU1009a的亚油酸Δ9水合酶通过重组大肠杆菌可将亚油酸转化为(S)-10-羟基-顺式-12-十八碳烯酸(HYA),转化率达99%(mol/mol)。同样地,可使得95%(mol/mol)的具有Δ9碳双键的C18不饱和脂肪酸转化成相应的10-羟基脂肪酸[36]。化脓性链球菌M49的肌球蛋白交叉反应性抗原(myosin cross reactive antigens,MCRA)是 FAD 酶,其作为 C16、C18非酯化脂肪酸的(9Z)-和(12Z)-双键上的水合酶,可生产 10-羟基和 10,13-二羟基脂肪酸[30]。因此,水合酶对于10-羟基硬脂酸的生产是至关重要的。
1.3 脂氧合酶
脂氧合酶(lipoxygenase,LOX)催化具有一个或多个(1Z,4Z)-戊二烯的游离多不饱和脂肪酸(PUFA),形成相应的氢过氧化脂肪酸后再还原成HFAs。脂氧合酶(LOX)在真核生物体中基本上是普遍存在的,并且已被证明存在于许多植物和动物的组织中。目前,作用于花生四烯酸特异性位置的LOX有5-LOX、8-LOX、12-LOX和 15-LOX等,而9-LOX、11-LOX和13-LOX催化亚油酸产生9-HODE、11-HODE和13-HODE。此外,LOX可以与其他蛋白融合,作为融合蛋白中的结构域发生其催化作用,第一个LOX融合蛋白被发现于珊瑚Plexaura homomalla中,在该珊瑚体内,生产8R-氢过氧二十碳四烯酸(8R-HpETE)的LOX结构域与含血红素的过氧化物酶结构域连接,该融合蛋白将脂肪酸过氧化物转化成氧化烯,这种不稳定的中间体可进一步转化为环戊烯酮二十烷酸[17]。近年来,对脂氧合酶的探究已取得一定的进展,但对其结构和功能上仍有许多不确定因素还有待研究。
1.4 烷烃羟化酶
另一种催化脂肪酸及其衍生物ω-羟基化的重要酶系是来源于恶臭假单胞菌的烷烃羟化酶(即AlkBGT)。AlkB是膜结合型烷烃羟化酶,AlkG是红素氧还蛋白,可将电子传递到AlkB以进行ω-羟基化,AlkT是红素氧还蛋白还原酶。与脂肪酸相比,AlkBGT可以更加有效地ω-官能化脂肪酸甲酯,且外膜蛋白AlkL可促进ω功能化。Van Nuland等[37]研究发现,表达AlkBGT系统的大肠杆菌可以ω-氧化壬酸乙酯(NAEE);AlkBGT可以ω-氧化C6~C10脂肪酸的乙基、丙基和丁基酯。此外,增加烷基链的长度还可提高其对C6和C7脂肪酸酯的ω-氧化活性,且AlkBGT和AlkL的共表达使得NAEE的ω-氧化活性提高 1.7 倍[37]。
1.5 二醇合酶体系
二醇合酶(diol synthase,DS)通常也被称为脂肪酸二羟基化酶,可以通过组合使用相同或不同的酶,包括P450、脂氧化酶、水合酶和羟化酶,形成多种类型的二羟基脂肪酸。Kaprakkaden等[38]通过使用3种不同类型的脂肪酸修饰酶,分别为来自S.cerevisiae的脂肪酸去饱和酶(fatty acid desaturase,FAD)、来自Caenorhabditis elegance的环氧化物水解酶(epoxide hydrolase,EH)和来自 Stokasialaevis的环氧化酶(epoxidase,EPOX),最终成功合成了 9,10-二羟基十六烷酸。在巨大芽孢杆菌中,发现环氧化的双键可以被相同的P450单加氧酶催化生成长链ω-羟基脂肪酸,进一步利用水合酶可生成二羟基脂肪酸[39]。三羟基脂肪酸在脂肪酰基链中比二羟基脂肪酸多一个羟基,它们比其他HFAs具有更高的反应活性和特殊的生理功能。铜绿假单胞菌PR3也可以将蓖麻油酸转化为7,10,12-三羟基油酸(7,10,12-THOD),产率为约 45%[40]。曲霉属菌能表达具有脂肪酸双加氧酶活性的蛋白或非功能性氢过氧化物异构酶/细胞色素P450结构域融合蛋白,如5,8-亚油酸二醇合酶(LDS)和 10R-双加氧酶(DOX)。7,8-LDS 和 5,8-LDS 的 N-末端氢过氧化物异构酶结构域显示与P450酶系具有同源性[41],表明其与P450的催化作用相似。来自Nostoc sp.的二醇合酶PCC7120由脂氧合酶和烯丙氧基合酶组成,它可将亚油酸转化为9,14-二羟基亚油酸(9,14-diHODE)、9,10-diHODE、8,11-diHODE 和 9-HODE;将 α-亚麻酸转化为 9,16-二羟基亚麻酸(9,16-diHOTE)和 9,13-diHOTE、9-HOTE;将 γ-亚麻酸转化为9,14-diHOTE和9-HOTE;将花生四烯酸转化为11,16-二羟基花生四烯酸(11,16-diHETE)和11-HETE[42]。
以上概述的是羟基脂肪酸合成途径中的关键酶,这些酶的表达直接影响到最终产物羟基脂肪酸的种类及其产量,而影响最终产量的另一关键因素是前体脂肪酸的通量。因此,工程化整体代谢途径将碳通量导向脂肪酸是非常重要的。
2 羟基脂肪酸的从头生物合成途径
利用代谢工程手段改造大肠杆菌生产脂肪酸及其衍生物也成为当今研究的热点。大肠杆菌中羟基脂肪酸的合成途径中涉及脂肪酸合成、脂肪酸β-氧化、α-氧化和ω-氧化等。大肠杆菌使用II型脂肪酸合成酶(FAS)催化脂肪酸生物合成途径。II型FAS的直接产物是脂肪酰-ACP,其需要通过硫酯酶(thioesterase,TE)水解以释放游离脂肪酸,并且随后通过脂肪酰-CoA连接酶活化为脂肪酰基-CoA。然而,野生型大肠杆菌并不能积累脂肪酸并进一步转化为HFAs。为了实现工程大肠杆菌利用葡萄糖从头转化HFAs的目标,需要①通过过表达硫酯酶来过量产生游离脂肪酶(FFA),表达外源性脂肪酸羟化酶将FFA转化为HFAs;②直接利用脂肪酸生产途径中的中间代谢产物,通过硫酯酶直接催化生产HFAs。图2(a)为大肠杆菌利用葡萄糖合成羟基脂肪酸的整体代谢途径。
2.1 增加前体脂肪酸的合成
基因工程大肠杆菌脂肪酸生物合成(FASII)途径是生产脂肪酸衍生物的关键途径。此途径中的关键酶包括乙酰CoA羧化酶(ACCase)、丙二酰辅酶A-ACP转酰酶(FabD)、β-酮酰基-ACP合成酶 III(FabH)、β-酮酰基-ACP 还原酶(FabG)、β-羟基酰基-ACP脱水酶(FabA、FabZ)以及硫酯酶等。对以上几个关键酶的组合表达是提高脂肪酸合成量的一个有效策略,如图2(b)所示。其中,硫酯酶的表达对于在大肠杆菌中产生FFAs是最为关键的。首先,它是连接前体和脂肪酸的直接桥梁。其次,它消耗长链酰基ACP,直接增加导向脂肪酸合成的通量,并降低由脂酰-ACP引起的反馈抑制。最后,硫酯酶的底物特异性决定了产物链长,大多数硫酯酶对C16~C18酰基链具有最高的特异性。与此同时,在大肠杆菌天然体系中,脂肪酸会通过β-氧化循环进行降解,这显然不利于脂肪酸的积累,可通过敲除或下调β-氧化循环中的关键基因fadD和fadE以阻止或减缓脂肪酸的降解,从而提高总脂肪酸产量。Zhang等[43]过表达来自4种不同生物的 fabD基因,最终提高中长链游离脂肪酸产量约11%。Jawed等[44]过表达 FabZ、ACCase和硫酯酶,同时抑制FabR的表达后,得到总短链游离脂肪酸产量为17.5 g/L。Cao 等[45]过表达硫酯酶基因 tesA’后,合成了大约108.5 mg/L的脂肪酸,为了增加脂肪酸前体的供应,其共表达TesA和ACCase后,脂肪酸产量达到188.6 mg/L;在摇瓶条件下,过表达E.coli BL21(DE3)硫酯酶(TesA)和乙酰辅酶A羧化酶(ACCase),同时敲除fadD,并插入细胞色素P450酶(P450BM3),最终产生 58.7 mg/L 的 HFAs。Xu等[46]通过表达tesA’,脂肪酸产量与亲本菌株相比,增长了近90%。Bowen等[47]选用了2种不同的硫酯酶(UcFatB2和TesA),最终分别得到230 mg/L C14 FFA和367 mg/L C12 FFA。不同物种的酰基-ACP硫酯酶具有不同程度的链长特异性。Zhang等[48]研究了来自 Diploknema butyracea、Gossypiumhirsutum,R.communis和 Jatropha curcas的酰基-ACP硫酯酶基因过表达对游离脂肪酸产量的影响,结果表明:脂肪酸的积累量很大程度上取决于所选用的硫酯酶,其中来自 R.communis和 J.curcas的酰基-ACP硫酯酶产生FFA超过2.0 g/L,且主要产物为C14、C16:1和C16 直链FFA。Zhang等[49]通过调整工程化大肠杆菌宿主中FadR的表达,将脂肪酸质量浓度增加 7.5倍,达到了 5.2 g/L。大肠杆菌FadL为外膜游离脂肪酸转运蛋白,它可由横向扩散通过外膜转运外源性游离脂肪酸。Jin等[50]研究过表达fadL以增加膜内游离脂肪酸的量,结果发现:与没有过表达fadL的菌株相比,过表达fadL菌株的ω-羟基棕榈酸产量增加了80.7%,该结果表明fadL的过表达增强了棕榈酸转运到细胞中的能力。Tan等[51]发现OmpF蛋白作为三聚体存在于外膜中,并参与外膜上的糖、离子、抗生素和蛋白质的转运。外膜蛋白基因ompF的缺失可以显著增加膜完整性,脂肪酸耐受性,同样可以提高脂肪酸产量。实验数据表明,与仅过表达 fadL的菌株相比,敲除ompF的同时过表达fadL菌株的脂肪酸产量高出20%。Shin等[52]将弗氏藤杆菌丙酸杆菌的甲基丙二酰CoA羧基转移酶(methylmalonyl CoA carboxytransferase,MMC)基因导入大肠杆菌中表达,可绕过调节乙酰CoA羧化的复合体系,引入MMC旁路将碳通量从TCA循环重新导向到FFA合成,减少细胞生长抑制并减少乙酸盐形成,最终FFA产量提高2.5 倍[52]。

图2 大肠杆菌合成脂肪酸及羟基脂肪酸的代谢途径(a)及代谢工程改造策略(b)Fig.2 Schematic representation of the metabolic pathway of fatty acid and hydroxyl fatty acid biosynthesis in Escherichia coli(a)and strategies of metabolic engineering(b)
此外,奇数链 FFA合成的研究也为奇数链HFAs创造了可能。Wu等[53]通过表达酰基ACP硫酯酶(TE),丙酰 CoA合成酶(PrpE)和 β-酮酰基-ACP合成酶III(FabH),最终工程大肠杆菌利用葡萄糖和丙酸盐产生奇数直链FFA的最高总质量浓度达1 205 mg/L,其中85%为C15 FFA。另外,特定的硫酯酶可以直接催化脂肪酸合成循环中的中间代谢产物 β-羟酰基-ACP 生成 HFAs。Zheng 等[54]利用PhaG,以葡萄糖底物生产了196 mg/L的3-羟基癸酸。Wang等[55]也证明了PhaG可作为β-羟酰基-ACP硫酯酶,将β-羟酰基-ACP直接催化为β-羟基脂肪酸。
最近有报道提出逆β-氧化循环(r-BOX)可作为合成中链脂肪酸及其衍生物(medium chain fatty acids,MCFA)的潜在平台。与传统的脂肪酸合成途径不同的是,r-BOX直接利用乙酰CoA进行延伸,通过硫解酶、羟酰基CoA脱氢酶和硫酯酶等关键酶形成反转β-氧化循环。Wu等[56]通过表达硫解酶(bktB)、羟酰基 CoA脱氢酶/烯酰 CoA水合酶(fadB)、反式-烯醇CoA还原酶(ter)、硫酯酶(ydiI)和乙酰CoA合成酶(acs)基因,得到MCFA产量为2.8 g/L。Clomburg 等[57]结合反转 β-氧化循环和ω-氧化途径,选择硫解酶(bktB)、硫酯酶(ydiI)和来自恶臭假单胞菌的烷烃羟化酶系AlkBGT,产生大于0.8 g/L 的 C6~C10 ω-HFAs。r-BOX 循环已被证明可用于脂肪酸及其衍生物的生产,该循环的建立将有助于进一步扩展代谢工程策略来从头合成羟基脂肪酸。
此外,随着合成生物学的发展,近年来新兴的一种基因编辑技术CRISPR/Cas系统因其快速、简单、高效的优点受到广泛青睐。Xia等[58]利用CRISPR/Cas系统敲除了β-氧化循环的第一个关键酶基因fadD和催化丙酮酸进入蛋白质合成途径的基因ppc,结果显示,敲除fadD基因的菌株产生835.4 mg/L脂肪酸,相对于野生型来说,脂肪酸含量增长了4.9%,而ppc基因的敲除对于脂肪酸合成并无显著影响。CRISPR/Cas系统在大肠杆菌合成脂肪酸领域中的这一应用为脂肪酸合成策略拓宽了思路。
2.2 减少副产物的形成
在构建从头合成羟基脂肪酸代谢途径中,消除或减少途径中代谢副产物的积累是增强代谢通量的关键一步,脂肪酸合成途径以乙酰CoA为起始单位、丙二酰-ACP为扩增单位进入循环。在大肠杆菌中,丙酮酸可通过多条代谢途径产生乳酸、乙酸等代谢副产物,可通过对相关基因的敲除或下调以实现副产物通路的阻遏,以此来增加目标产物的积累。途径中形成的乙酸盐是一种有毒副产物,也是最主要的代谢副产物,可抑制细胞生长,在大肠杆菌中主要的两种乙酸盐生产途径是通过乙酸激酶/磷酸转乙酰酶(AckA-Pta)和丙酮酸氧化酶(PoxB)。Li等[59]研究了 fadD 突变菌株(ML103)、ack-pta 和poxB双突变株(ML112)以及fadD、ack-pta和 poxB三突变株(ML115)中乙酸盐途径的阻遏对FFA产量及其组成的影响,结果发现:fadD的缺失对FFA产量的影响很小,消除乙酸盐生产途径确实使乙酸盐浓度降低,但却几乎没有改善FFA的产量,这可能是由于从乙酸“回收”的一些碳被转移到了丙酮酸盐。Lin等[60]将敲除pta基因的菌株与未敲除pta基因的菌株相比,乙酸浓度降低约30%,FFA产量达84.9 mg/g(以细胞干质量计),增加约26%,且总脂肪酸组分中C12和C14脂肪酸的比例明显提高。由于敲除乙酸盐生成途径可能导致产物特异性发生变化,因此,可以考虑是否通过乙酸盐利用途径中乙酰CoA合成酶的过表达将碳通量重新导向乙酰CoA以改善FFA产量。Wu等[56]研究发现,过表达acs的菌株中,乙酸盐的浓度明显降低,且细胞中乙酰CoA的量增加了271.3%。该结果证明,过表达乙酸盐利用途径是增加FFA合成起始单位乙酰CoA的有效策略。另外,他们利用CRISPRi系统抑制乙醇脱氢酶(adhE)、磷酸转乙酰酶(pta)、丙酮酸脱氢酶(poxB)、乳酸脱氢酶(ldhA)和琥珀酸脱氢酶(frdA)的基因,观察各靶基因对应的副产物浓度发现,CRISPRi系统确实起到控制基因表达的作用,抑制 adhE、pta、poxB、ldhA和 frdA基因表达,分别提高中链 FFA 产量 11.5%、20.4%、5.6%、7.9%、10.7%,其中抑制pta基因的菌株表达产量最高达3.2 g/L。
3 总结与展望
羟基脂肪酸(HFAs)在各领域内都有广泛的应用,包括聚合材料、食品添加剂、化妆品、表面活性剂和药品等,具有较高的商业价值,因此也越来越受到人们的关注。羟基脂肪酸种类众多,利用微生物法合成可以增加产物特异性,降低产物分离纯化的成本,在制备工艺上具有一定的优势。目前,大多数微生物合成羟基脂肪酸的研究主要集中于对催化酶系的开发,转化外源脂肪酸。本文中,笔者针对大肠杆菌中采用生物催化与代谢工程合成羟基脂肪酸的不同策略、研究进展及其成果进行综述,而通过大肠杆菌利用葡萄糖等底物从头生产羟基脂肪酸的探索还不成熟,对其进行代谢工程改造以适应大规模、高产率的工业化生产仍是今后研究的主要方向之一。
[1] 川岛正毅.羟基脂肪酸的开发和应用[J].日用化学工业译丛,1992(4):23-25.
[2] 翁佩芳,吴祖芳.脂肪酸微生物转化生产羟基脂肪酸的研究进展[J].中国粮油学报,2008,23(1):203-206.
[3] CROSS H R,CLARKE K,OPIE L H,et al.Is lactate-induced myocardial ischaemic injury mediated by decreased pH or increased intracellular lactate[J].J Mol Cell Cardiol,1995,27(7):1369-1381.
[4] KOAY G F,CHUAH T G,ZAINAL-ABIDIN S,etal.Development,characterization and commercial application of palm based dihydroxy stearic acid and its derivatives:an overview[J].J Oleo Sci,2011,60(5):237-65.
[5] 程璐,缪铭,张涛,等.食品生物防腐剂抗真菌乳酸菌研究进展[J].食品与发酵工业,2010,36(9):129-133.
[6] KUO T M,NAKAMURA L K,LANSER A C.Conversion of fatty acids by Bacillus sphaericus-like organisms[J].Curr Microbiol,2002,45(4):265-271.
[7] 吴立新,吴祖芳.短小芽孢杆菌脂肪酸羟基化发酵特性与培养条件优化[J].食品与生物技术学报,2011,30(4):602-608.
[8] DURAIRAJ P,MALLA S,NADARAJAN S P,et al.Fungalcy to chrome P450 monooxygenases of Fusarium oxysporum for the synthesis of ω-hydroxy fatty acids in engineered Saccharomyces cerevisiae[J].Microb Cell Fact,2015,14(1):1-16.
[9] SUNG C,JUNG E,CHOI K Y,et al.The production of ω-hydroxy palmitic acid using fatty acid metabolism and cofactor optimization in Escherichia coli[J].Appl Microbiol Biotechnol,2015,99(16):1-10.
[10] FER M,CORCOS L,DRÉANO Y,et al.Cytochromes P450 from family 4 are the main ω-hydroxylating enzymes in humans:CYP4F3B is the prominent player in PUFA metabolism[J].J Lipid Res,2008,49(11):2379-2389.
[11] TIJET N,HELVIG C,PINOT F,et al.Functional expression in yeastand characterization of a clofibrate-inducible plant cytochrome P-450(CYP94A1)involved in cutin monomers synthesis[J].Biochem J,1998,332(2):583-589.
[12] BENVENISTE I,TIJET N,ADAS F,et al.CYP86A1 from Arabidopsis thaliana encodes a cytochrome P450-dependent fatty acid omega-hydroxylase[J].Biochem Biophys Res Commun,1998,243(3):688-693.
[13] LI H,PINOT F,SAUVEPLANE V,et al.Cytochrome P450 family member CYP704B2 catalyzes the omega-hydroxylation of fatty acids and is required for anther cutin biosynthesis and pollen exine formation in rice[J].Plant Cell,2010,22(1):173-190.
[14] LENTZO,URLACHER V,SCHMID R D.Substrate specif i city of native andmutated cytochrome P450(CYP102A3)from Bacillus subtilis[J].J Biotechnol,2004,108(1):41-49.
[15] MATSUNAGA I,SUMIMOTO T,UedaA,et al.Fatty acid-specif i c,regiospecif i c,and stereospecif i c hydroxylation by cytochrome P450(CYP152B1) from Sphingomonas paucimobilis:substrate structure required for α-hydroxylation[J].Lipids,2000,35:365-371.
[16] BEVERS L,PINKSE M,VERHAERT P,et al.Oleate hydratase catalyzes the hydration of a nonactivated carbon-carbon bond[J].J Bacteriol,2009,191:5010-5012.
[17] IVANOV I,HEYDECK D,HOFHEINZ K,et al.Molecular enzymology of lipoxygenases[J].Arch Biochem Biophys,2010,503:161-174.
[18] VAN DE LOO F J,BROUN P,TURNER S,et al.Anoleate 12-hydroxylase from Ricinus communis L.is a fatty acyl desaturasehomolog[J].Proc Natl Acad Sci USA,1995,92:6743-6747.
[19] LANG I,GÖBEL C,PORZEL A,et al.A lipoxygenase with linoleate diol synthase activity from Nostoc sp.PCC 7120[J].Biochem J,2008,410:347-357.
[20] WANG X W,LI L L,ZHENG Y N,et al.Biosynthesis of long chain hydroxy fatty acids from glucose by engineered Escherichia coli[J].Bioresour Technol,2012,114(114):561-566.
[21] KIM K R,OH D K.Production of hydroxy fatty acids by microbial fatty acid-hydroxylation enzymes[J].Biotechnol Adv,2013,31(8):1473-1485.
[22] MATSUNAGA I,UEDA A,FUJIWARA N,et al.Characterization of the ybdT gene product of Bacillus subtilis:novel fatty acid βhydroxylating cytochrome P450[J].Lipids,1999,34(8):841-846.
[23] RUETTINGER R T,WEN L P,FULCO A J.Coding nucleotide,5'regulatory,anddeduced amino acid sequences of P-450BM-3,a single peptide cytochrome P-450:NADPH-P-450 reductase from Bacillus megaterium[J].J Biol Chem,1989,264:10987-10995.
[24] GUSTAFSSON M C U,ROITEL O,MARSHALL K R,et al.Expression,purification,and characterization of Bacillus subtilis cytochromes P450 CYP102A2 and CYP102A3:flavocytochrome homologues ofP450 BM3from Bacillusmegaterium [J].Biochemistry,2004,43:5474-5487.
[25] FURUYA T,SHIBATA D,KINO K.Phylogenetic analysis of Bacillus P450 monooxygenases and evaluation of their activity towards steroids[J].Steroids,2009,74:906-912.
[26] CHOWDHARY P K,ALEMSEGHED M,HAINES D C.Cloning,expression and characterization of a fast self-sufficient P450:CYP102A5 from Bacillus cereus[J].Arch Biochem Biophys,2007,468:32-43.
[27] SCHEPS D,HONDA MALCA S,RICHTER S M,et al.Synthesis of ω-hydroxydodecanoic acid based on an engineered CYP153A fusion construct[J].Microb Biotechnol,2013,6(6):694-707.
[28] MUNDAY SD,MADDIGANNK,YOUNGRJ,etal.Characterisation of two self-sufficient CYP102 family monooxygenases from Ktedonobacter racemifer DSM44963 which have new fatty acid alcohol product profiles[J].Biochim Biophys Acta,2016,1860(6):1149-1162.
[29] MORVAN B,JOBLIN K N.Hydration ofoleicacid by Enterococcus gallinarum,Pediococcus acidilactici and Lactobacillus sp.isolated from the rumen[J].Anaerobe,1999,5(6):605-611.
[30] VOLKOV A,LIAVONCHANKA A,KAMNEVA O,et al.Myosin cross-reactive antigen of Streptococcus pyogenes M49 encodes a fatty acid double bond hydratase that plays a role inoleic acid detoxification and bacterial virulence[J].J Biol Chem,2010,285(14):10353-10361.
[31] JOO Y C,JEONG K W,YEOM S J,et al.Biochemical characterization and FAD-binding analysis of oleate hydratase from Macrococcus caseolyticus[J].Biochimie,2012,94(3):907-915.
[32] KIM B N,JOO Y C,KIM Y S,et al.Production of 10-hydroxystearic acid from oleic acid and olive oil hydrolyzate by anoleate hydratase from Lysinibacillusfusiformis[J].Appl Microbiol Biotechnol,2012,95(4):929-937.
[33] O'CONNELLK J,MOTHERWAY M O,HENNESSEY A A,et al.Identification and characterization of anoleate hydratase-encoding gene from Bifidobacterium breve[J].Bioengineered,2013,4(5):313-321.
[34] TAKEUCHI M,KISHINO S,HIRATA A,et al.Characterization of the linoleic acid Δ9 hydratase catalyzing the first step of poly unsaturated fatty acid saturation metabolism in Lactobacillus plantarum AKU 1009a[J].J Biosci Bioeng,2015,119(6):636-641.
[35] OH H J,KIM S U,SONG J W,et al.Biotransformation of linoleic acid into hydroxy fatty acids and carboxylic acids using a linoleate double bond hydratase as key enzyme[J].Adv Synth Catal,2015,357(2/3):408-416.
[36] TAKEUCHI M,KISHINO S,PARK S B,et al.Efficient enzymatic production of hydroxy fatty acids by linoleic acid Δ9 hydratase from Lactobacillus plantarum AKU 1009a[J].J Appl Microbiol,2016,120(5):1282-1288.
[37] VAN NULAND Y M,EGGINK G,WEUSTHUIS R A.Application of AlkBGT and AlkL from Pseudomonas putida GPo1 for selective alkyl ester ω-oxyfunctionalization in Escherichia coli[J].Appl Environ Microbiol,2016,82(13):3801-3807.
[38] KAPRAKKADEN A,SRIVASTAVA P,BISARIA V S.In vitro synthesis of 9,10-dihydroxy hexadecanoic acid using recombinant Escherichia coli[J].Microb Cell Fact,2017,16(1):85.
[39] NIEHAUS W G,KISIC A,TORKELSON A,et al.Stereospecific hydration of cis-and trans-9,10-epoxyocta-decanoic acids[J].J Biol Chem,1970,245(15):3802-3809.
[40] KUO T M,KIM H,HOU C T.Production of a novel compound,7,10,12-trihydroxy-8(E)octadecenoic acid from ricinoleic acid by Pseudomonas aeruginosa PR3[J].Curr Microbiol,2001,43(3):198-203.
[41] JERNERÉN F,GARSCHA U,HOFFMANN I,et al.Reaction mechanism of 5,8-linoleate diol synthase,10R-dioxygenase,and 8,11-hydroperoxide isomerase of Aspergillus clavatus[J].Biochim Biophys Acta,2010,1801(4):503-507.
[42] LANG I,GÖBEL C,PORZEL A,et al.A lipoxygenase with linoleate diol synthase activity from Nostoc sp.PCC 7120[J].Biochem J,2008,410:347-357.
[43] ZHANG X,AGRAWAL A,SAN K Y.Improving fatty acid production in Escherichia coli through the overexpression of malonyl coA-Acyl carrier protein transacylase[J].Biotechnol Prog,2012,28(1):60-65.
[44] JAWED K,MATTAM A J,TATMA Z,etal.Engineered production of short chain fatty acid in Escherichia coli using fatty acid synthesis pathway[J].PloS ONE,2016,11(7):e0160035.
[45] CAO Y J,CHENG T,ZHAO G,et al.Metabolic engineering of Escherichia coli for the production of hydroxy fatty acids from glucose[J].BMC Biotechnol,2016,16(1):26.
[46] XU P,GU Q,WANG W,et al.Modular optimization of multi-gene pathways for fatty acids production in E.coli[J].Nat Commun,2013,4(1):1409.
[47] BOWEN CH,BONINJ,KOGLERA,etal.Engineering Escherichia coli for conversion of glucose to medium-chain ωhydroxy fatty acids and α,ω-dicarboxylic acids[J].ACS Synth Biol,2015,5(3):200-206.
[48] ZHANG X,LI M,AGRAWAL A,et al.Efficient free fatty acid production in Escherichia coli using plant acyl-ACP thioesterases[J].Metab Eng,2011,13(6):713-722.
[49] ZHANG F,OUELLET M,BATTH T S,et al.Enhancing fatty acid production by the expression of the regulatory transcription factor FadR[J].Metab Eng,2012,14(6):653-660.
[50] JIN HB,PARK B G,JUNG E,et al.fadD deletion and fadL over expression in Escherichia coli increase hydroxy long-chain fatty acid productivity[J].Appl Microbiol Biotechnol,2014,98(21):8917-8925.
[51] TAN Z,BLACK W,YOON J M,et al.Improving Escherichia coli membrane integrity and fatty acid production by expression tuning of FadL and OmpF[J].Microb Cell Fact,2017,16(1):38.
[52] SHIN K S,LEE S K.Introduction of an acetyl-CoA carboxylation bypass into Escherichia coliforenhanced free fatty acid production[J].Bioresour Technol,2017,31(5):160.
[53] WU H,SANKY.Efficient odd straight medium chain free fatty acid production by metabolically engineered Escherichia coli[J].Biotechnol Bioeng,2014,111(11):2209-2219.
[54] ZHENG Z,ZHANG M J,ZHANG G,et al.Production of 3-hydroxydecanoic acid by recombinant Escherichia coli HB101 harboring phaG gene[J].Anton Leeuw,2004,85(2):93-101.
[55] WANG Q,TAPPEL RC,ZHU C,et al.Development of a new strategy for production of medium-chain-length polyhydroxyalkanoates by recombinant Escherichia coli via inexpensive non-fatty acid feedstocks[J].Appl Environ Microb,2012,78(2):519-527.
[56] WU J,ZHANG X,XIA X,et al.A systematic optimization of medium chain fatty acid biosynthesis via the reverse betaoxidation cycle in Escherichia coli[J].Metab Eng,2017,41:115-124.
[57] CLOMBURG J M,BLANKSCHIEN M D,VICK J E,et al.Integrated engineering of β-oxidation reversal and ω-oxidation pathways for the synthesis of medium chain ω-functionalized carboxylic acids[J].Metab Eng,2015,28:202-212.
[58] XIA J,WANG L,ZHU J B,et al.Expression of Shewanella frigidimarina fatty acid metabolic genes in E.coli by CRISPR/cas9-coupled lambda Red recombineering[J].Biotechnol Lett,2016,38(1):117-122.
[59] LI M,ZHANG X,AGRAWAL A,et al.Effect of acetate formation pathway and long chain fatty acid CoA-ligase on the free fatty acid production in E.coli expressing acyl-ACP thioesterase from Ricinus communis[J].Metab Eng,2012,14(4):380-387.
[60] LIN F,CHEN Y,LEVINE R,et al.Improving fatty acid availability for bio-hydrocarbon production in Escherichia coli by metabolic engineering[J].PloS ONE,2013,8(10):e78595.
(责任编辑 荀志金)
Advances in biosynthesis of hydroxy fatty acids by Escherichia coli
HE Qiaofei,WU Hui
(State Key Laboratory of Bioreactor Engineering,East China University of Science and Technology,Shanghai,200237,China)
Hydroxy fatty acids containing hydroxyl and carboxyl functional groups are ideal precursors of green polymer materials,poly ester fibers and poly amides.They are widely used in medical,industrial and food fields.We review here the research progress of hydroxy fatty acid biosynthesis by Escherichia coli.Metabolic engineering strategies to improve hydroxy fatty acid synthesis in engineered Escherichia coli are also reviewed.
hydroxy fatty acids;fatty acid;biocatalysis;metabolic engineering;Escherichia coli
Q819
A
1672-3678(2017)05-0071-09
10.3969/j.issn.1672-3678.2017.05.009
2017-07-23
国家自然科学基金青年基金(21406065)国家自然科学基金面上基金(21776083);国家重点研发计划(2017YFB0309302);上海市“科技创新行动计划”基础研究领域项目(17JC1404800);工业生物催化教育部重点实验室开放课题(2015101);中央高校基本科研业务费探索研究基金(222201313007、22A201514042);生物反应器工程国家重点实验室开放课题
何乔飞(1994—),女,浙江台州人,研究方向:生物催化与生物转化;吴 辉(联系人),副教授,E-mail:hwu@ecust.edu.cn
