植物底盘:天然产物合成生物学研究的新热点
2017-09-30李建华王凯博
邵 洁,李建华,王凯博,3,王 勇
(1.中国科学院 上海生命科学研究院 植物生理生态研究所 中国科学院分子植物科学
卓越创新中心中国科学院合成生物学重点实验室,上海 200032;2.中国科学院大学,北京 100049;3.河南大学 植物逆境生物学重点实验室,河南 开封 475000)
植物底盘:天然产物合成生物学研究的新热点
邵 洁1,2,李建华1,王凯博1,3,王 勇1
(1.中国科学院 上海生命科学研究院 植物生理生态研究所 中国科学院分子植物科学
卓越创新中心中国科学院合成生物学重点实验室,上海 200032;2.中国科学院大学,北京 100049;3.河南大学 植物逆境生物学重点实验室,河南 开封 475000)
天然产物广泛地存在于植物体内,是药物、食品添加剂和新型生物燃料等开发的主要来源,具有重要的商业价值,该类化合物也一直是合成生物学研究的热点之一。随着研究的深入,近年来以植物为底盘的天然产物研究日益兴起。本文中,笔者综述了近年来以植物为底盘的天然产物合成生物学研究的进展,包括该类代谢物代谢途径的解析、以植物为底盘的遗传操作技术和方法等,为相关研究者提供参考。
天然产物;合成生物学;植物底盘
天然产物由自然界长期进化而来,是生物活性物质和药物发现的重要源泉。植物来源的天然产物具有抗肿瘤、抗过敏及抗菌等多种生物活性[1],在食品、日化和医疗等领域受到广泛关注,展现出巨大的应用潜力和广阔的市场前景。但是该类化合物成分复杂,且在自然界的含量往往极低,传统的研究和开发方法已不能满足人们的需求[2]。近年来,合成生物学为天然产物的开发提供了新的思路,基于该思想建立的微生物细胞工厂已实现了多种天然化合物的高效合成,有效提高了天然产物总体的生产水平[3]。
随着科研工作者的努力,合成生物学研究的对象已从最初的单细胞原核微生物体系逐渐涉足到复杂的多细胞真核体系,其中以植物为底盘的合成生物学研究逐渐为人瞩目[4]。与单细胞的微生物相比,植物富含各种内膜系统和细胞器,复杂的空间特性,为不同代谢物的合成提供了所需的最适环境,也为合成生物学研究提供了绝佳的模式体系[5]。本文中,笔者将综述近年来以植物为底盘的天然产物合成生物学研究的进展,包括该类代谢物代谢途径的解析、以植物为底盘的遗传操作技术和方法等。
1 植物次生代谢物合成基因簇的发现
微生物产生的次生代谢物,如在放线菌和真菌中,其编码基因往往成簇存在于基因组中。但近年来,随着植物基因组学研究的深入,特别是一些模式植物基因组测序的完成,人们注意到植物次生代谢相关的基因也会成簇排列[6]。这一发现为植物次生代谢物合成途径的解析及其合成生物学设计提供了便利。目前已经在植物中发现了一些负责天然化合物合成的基因簇[7],例如番茄中的单萜α-蒎烯(α-pinene)、β-罗勒烯(β-ocimene),水稻中的二萜类化合物稻壳酮A(momilactone A)及植物卡生(phytocassanes),燕麦中的三萜类化合物燕麦根皂苷(avanacin)。Boutanaev等[8]对 17种不同模式植物来源的萜类化合物的多样性进行系统分析时发现,萜类合成的两类关键基因萜类合酶(TPS)的基因和细胞色素氧化酶(CYP)的基因通常也是成簇存在的。在对十字花科植物的牻牛儿基法呢基焦磷酸合酶(GFPPS)结构与功能的研究过程中,Wang等[9]也发现GFPPS的基因通常与TPS的基因串联形成一个基因簇,这与先前在茄科植物单萜和倍半萜合成途径中发现共同存在的异戊烯基转移酶(CPT)-TPS现象[10]很是类似。笔者所在课题组利用萜类代谢途径改造的大肠杆菌底盘细胞,对其中来自拟南芥的基因簇的代谢产物进行了研究,成功获得了两个结构新颖的二倍半萜基本碳氢骨架化合物 (+)-thalianatriene 和 (-)-retigeranin B[11],首次完成对植物来源的二倍半萜合成基因簇及其功能性代谢产物的研究。King等[12]通过对蓖麻基因组数据的分析发现,有8个细胞色素P450酶、2个乙醇脱氢酶、1个乙酰转移酶和1个单萜合成酶位于同一个基因簇上。经鉴定,其中3个细胞色素P450酶(CYP726A14、CYP726A17和 CYP726A18)能催化蓖麻烯生成5-羟基蓖麻烯或5-酮基蓖麻烯。随着更多植物天然产物合成基因簇的发现,基于基因组测序的基因簇发掘已成为这些天然产物合成途径解析的主要方法之一。
2 以植物为底盘的合成生物学研究
以微生物为底盘的天然产物合成生物学研究已经取得了巨大的成功:青蒿酸在酵母中的异源合成是该领域的标志性成果。2013年,Keasling实验室的Paddon等[13]以酿酒酵母为底盘,实现了青蒿酸的微生物合成,产量达到25 g/L。随后赛诺菲公司收购了该项专利并宣布将基于该成果启动大规模生产半合成青蒿素的产业化研究。但是这一合成生物学商业化应用的里程碑事件的发展并不像预期那样顺利,2015年赛诺菲并未生产任何“半合成”青蒿素,该公司却出售位于意大利的青蒿素加工厂。这主要是由于黄花蒿供过于求,植物来源的青蒿素每公斤销售额不到250美元,低于赛诺菲“不盈利-不损失”标准下每公斤350~400美元的销售价格。所以Keasling教授也认为如果价格已经足够低,有大量产青蒿素的植物,就没必要再利用发酵设备来生产青蒿酸[14]。这说明,与微生物底盘细胞相比,植物底盘仍有其天然优势,如植物仅以CO2和水为原料,经光合作用就可以合成各类复杂的天然产物,而无需高耗能、高耗氧的发酵过程;植物底盘可以突破微生物底盘对细胞色素P450酶表达性差、对活性产物的耐受性差的局限性;同时,作为具有复杂分化的多细胞生物,植物底盘本身的复杂分区化、不同器官的精细化分工和协作也为实现复杂功能人工设计提供了可能。因此,以植物为底盘进行天然产物的合成生物学研究(图1)有其优越性和必要性,并已取得了一些进展。2011年Farhi等[15]将紫穗槐-4,11-二烯合酶(ADS)、细胞色素P450还原酶(CPR)、细胞色素 P450羟化酶 CYP71AV1(CYP71AV1)以及青蒿醛 Δ11(13)双键还原酶(DBR2)4个基因构建在pSAT载体上,并通过农杆菌介导的稳定转化技术首次实现了青蒿素在烟草中的异源合成。再进一步通过过表达甲羟戊酸(MVA)途径限速酶3-羟基-3-甲基戊二酸单酰辅酶A还原酶(HMGR),运用信号肽ADS定位到线粒体上的方式,使烟草中青蒿素含量达到 6.8 μg/g(以1 g烟草干质量计)。2016年Lv等[16]使用RNA干扰技术在黄花蒿中导入cDNA反义链,分别下调β-石竹烯合成酶(CPS)、β-法呢基焦磷酸合成酶(BFS)和角鲨烯合成酶(SQS)等萜类生物合成途径中影响青蒿素生物合成竞争支路上的关键基因,结果和对照组相比,所有转基因黄花蒿中,青蒿素的含量均明显上升,达70%左右。接下来,他们通过基因共表达分析策略和转录组高通量测序分析策略,先后筛选出了能正调控青蒿素生物合成的转录因子 AaMYC2[17]、AaNAC1[18]和 AaHD1[19],并在此基础上培育出可在废弃的盐碱地生长,具有抗除草剂性能,适合规模化、基地化种植,青蒿素含量达到1.5%~2%的新一代黄花蒿产品。
叶绿体是植物细胞和真核藻类细胞所特有的进行光合作用的场所,其中存在大量的代谢途径,通常被形象地称为“植物细胞生物合成中心”。叶绿体基因组具有与原核微生物类似的表达多顺反子的潜力。Fuentes等[20]将青蒿酸的完整合成途径的基因整合到烟草叶绿体的基因组中,同时引入一些使系统更加稳定的基因,最终获得了120 mg青蒿酸(以1 kg生物量计)。
天然产物在植物细胞内不同的细胞器或组织中合成,催化反应合成的酶定位于不同的细胞器中可以降低中间体对细胞的毒害作用。通过细胞区分化设计可以有效提高产物的产量。Malhotra等[21]在烟草细胞中进行了青蒿素合成途径分区化设计:通过信号肽cox4融合将ADS定位在线粒体中,通过质体导肽将CYP71AV1、CPR、DBR2定位在叶绿体内,同时在叶绿体中引入酵母的整条MVA途径来增加萜类前体供应,最终获得了0.8 mg/g(以细胞干质量计)的青蒿素。烟草因其生物安全性和遗传转化易操作性,已经成为植物合成生物学研究的常用底盘。上述青蒿素的植物异源合成研究均是在烟草中开展的。
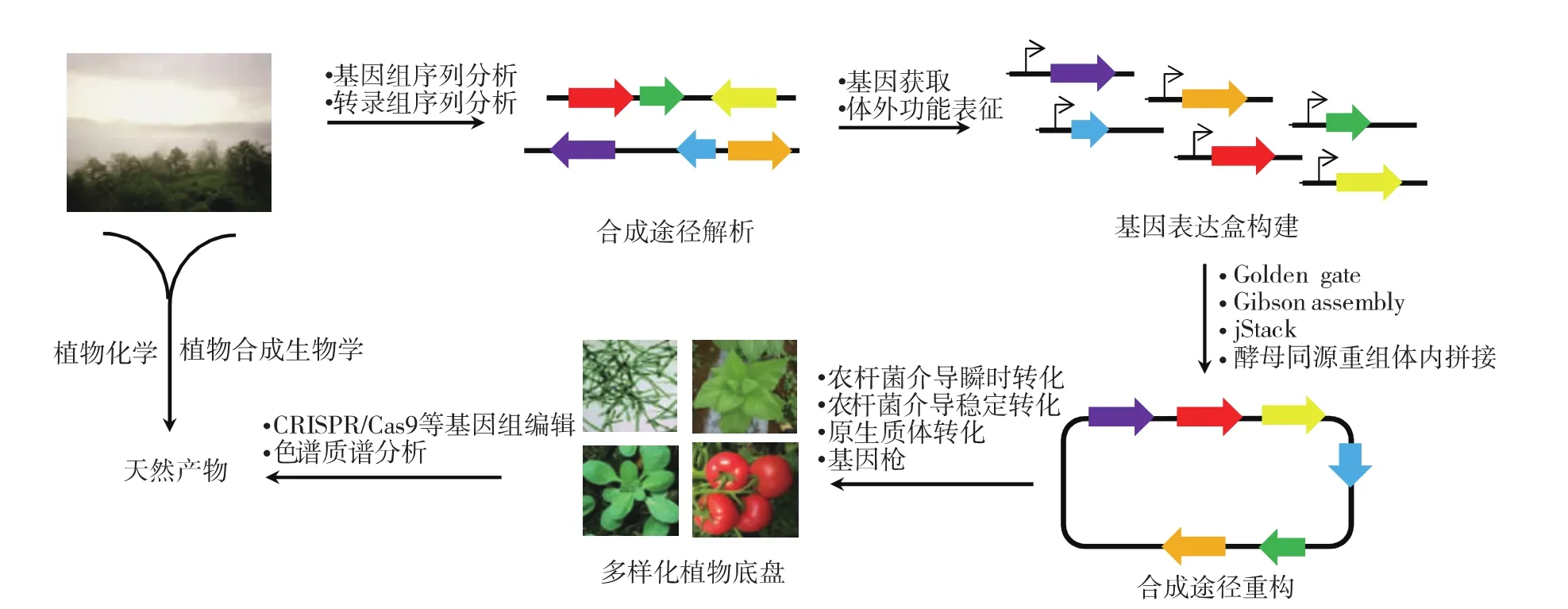
图1 植物底盘的天然产物合成生物学研究思路Fig.1 Research on natural product synthetic biology of plant chassis
其他植物甚至藻类也具有成为植物底盘的潜力。小立碗藓是一种低等苔藓植物,它的基因组仅有500 Mb,而且全基因组信息已经公布。清晰的遗传背景也方便对其进行基因组编辑和外源基因导入。Anterola等[22]在小立碗藓中表达红豆杉来源的紫杉二烯合酶,获得了紫杉二烯5 μg/g(以1 g干细胞质量计)。类似的工作在拟南芥[23]、番茄[24]和人参毛状根[25]等中也有报道。Tohge等[26]在番茄中引入金鱼草来源的转录因子Delila和Rosea1后,检测到花青素和苯丙类黄酮衍生物的含量都有所增加。随后该团队通过在番茄中引入拟南芥来源的黄酮特异性调控因子AtMYB12后,检测到黄酮类化合物和对羟基肉桂酸乙酯的含量达到了果实干质量的10%。此外,AtMYB12的表达不仅可以促进黄酮类化合物的合成,还能增加初级代谢源碳、能量和还原力的供应,这可能是AtMYB12的表达会刺激莽草酸和苯丙氨酸途径,为次生代谢提供更多的芳香族氨基酸。通过表达AtMYB12并增加系统的前体、能量和还原力可用于在番茄中生产高含量的苯丙类化合物,这也为生产其他有价值的天然化合物提供了新的思路[27-28]。
3 应用于植物底盘的使能技术

表1 应用于植物底盘的合成生物学技术Table 1 The synthetic biology technology applied in plant chassis
与微生物中的情况类似,各类针对植物底盘合成生物学的使能技术研究方兴未艾(表1)。Engler等[29-30]报道了基于II S型限制性内切酶的Golden Gate克隆方法,II S型限制性内切酶可以特异性地识别DNA上的靶点,并在靶点下游非特异性地切割DNA序列,利用II S型限制内切酶的这一特性可以灵活设计实验方案,即通过引物设计实现前一个片段的C端与后一个片段的N端的II S型限制内切酶位点尾巴是相同的,经过II S型限制内切酶酶切之后就可以用连接酶把多个片段按照既定的顺序实现无缝拼接。Shih等[31]报道了基于酵母同源重组的jStack方法,该方法将紫色杆菌素的整个合成途径中包括的5个基因装配到pYB载体上,已经在烟草叶片中表达成功,可以检测到紫色杆菌素和一些中间体。同时,该团队还尝试将此方法应用于植物基因簇的研究并成功装配了10个10.6~20.5 kb的基因簇相关片段。
除了一些DNA拼接方法的改进,基因组编辑方法也有很大进步。2016年,高彩霞研究员团队的Zhang等[32]在小麦中建立了基于CRISPR/Cas9瞬时表达的基因组编辑系统。该研究通过CRISPR/Cas9 DNA或RNA瞬时表达,在六倍体小麦和四倍体小麦中进行了基因的定点敲除,并在T0代得到了不含外源基因的小麦纯合敲除突变体,突变效率为1.0%~9.5%,且无脱靶效应。另外此系统的瞬时表达CRISPR/Cas9 RNA的方法中不涉及外源DNA,避免了植物中基因组编辑导致的生物安全问题。但是,CRISPR/Cas9 DNA进入到细胞以后仍然有可能会降解产生小的DNA片段整合到基因组中,而CRISPR/Cas9 RNA瞬时表达技术操作较为严格,但生产成本较高。为了克服这一缺陷,该研究团队于2017年1月又报道了在小麦中的DNA-free基因组编辑方法研究新进展[33],通过将CRISPR/Cas9蛋白和gRNA在体外组装成核糖核蛋白复合体(RNP),再利用基因枪法将CRISPR/Cas9 RNP转入小麦细胞中,研究人员在2个六倍体小麦品种中分别对2个不同基因进行定点编辑,成功地在小麦中建立了全程无外源DNA的基因组编辑系统。相比于CRISPR/Cas9的DNA或RNA瞬时表达体系,CRISPR/Cas9的RNP可以明显降低脱靶效应(DNA-free)。这种DNA-free的基因组编辑方法具有精准度和特异性高、操作简单、成本低廉的优势,并且成功避免了外源DNA片段整合到基因组中的潜在风险,使得在植物底盘中高效地进行基因编辑成为可能。
植物中表达系统的优化也有很大进展,Sainsbury等[34]建立了基于CPMV-HT的高效瞬时表达系统,可在植物侵染后5 d内获得高水平的重组蛋白;而Kanagarajan等[35]利用这套系统成功地在本氏烟草中纯化到了有活性的青蒿素生物合成途径中的ADS蛋白。
与微生物细胞相比,植物细胞中存在复杂的膜结构和细胞器,代谢途径中的酶经常分布在不同的内膜系统中,基于这些因素,在植物底盘中进行代谢途径设计的时候应该进行定位化设计。2016年,德国马普所的Fuentes等[20]开发了一种新的合成生物学方法,即COSTREL(combinatorial supertransformation of transplastomic recipient lines),他们以此方法将青蒿酸的完整合成途径基因整合定位到烟草叶绿体的基因组中,筛选出最优的转化烟草后,在细胞核DNA中再导入一系列可调节物质代谢的其他基因,最终从每千克生物量中获得了120 mg青蒿酸。此研究不仅可以满足大批量廉价生产抗疟疾药物的需要,而且所用的定位化设计也为在植物底盘中生产有价值的天然产物提供了思路。
4 生物合成途径的解析仍是难点和瓶颈
生物合成途径的解析是合成生物学工程化设计的基础。迄今为止,植物来源的天然产物得到完整解析的还很少。虽然植物的基因组庞大,但目前仅有部分植物物种的基因组公布(表2)。因此,如何高效地从这些基因组资源中挖掘鉴定到参与天然产物合成的基因,特别是参与后修饰的基因(如细胞色素P450酶基因、糖基转移酶基因和氧化还原酶基因)仍是现阶段的工作难点。

表2 已完成基因组测序的植物信息汇总Table 2 Summary of plants that have been sequenced
目前,鉴定这些未知基因的主要方法包括候选基因的异源表达和体外酶促反应,而由于植物底盘在天然产物合成方式和蛋白翻译后修饰上与候选基因来源的植物具有更多的相似性,因此如何鉴定出上述的这些关键酶,会直接影响植物底盘的进一步开发和应用。
长期以来,天然产物的合成途径主要根据已知的化学结构、化学反应及合成机制等进行推测。这些推测的合理性与准确性会直接影响未知功能基因的发现。现在,同位素示踪法[39]已经用于跟踪植物体内代谢物,以此可以进一步验证推测的途径是否准确。次丹参酮二烯作为丹参酮合成的中间体,其后修饰过程及下游途径的解析在先前研究得较少。Guo等[40]将丹参类贝壳杉烯合酶(SmKSL)、丹参柯巴基焦磷酸合酶(SmCPS)和牻牛儿基牻牛儿基焦磷酸合酶(GGPPS)基因转入大肠杆菌中进行共表达,在培养过程中通过添加13C标记的葡萄糖,获得了被标记的次丹参酮二烯。在后续培养丹参毛状根时,加入13C标记的次丹参酮二烯,可以检测到相应标记的铁锈醇和隐丹参酮,由此证明了两个关键中间体的存在,为进一步的解析奠定基础。
高通量测序的快速发展,使获得一个物种的基因信息成为可能。最近黄三文团队通过基因组学的研究,在黄瓜苦味素的形成机制上取得了突破,Shang等[41]研究发现负责催化合成葫芦素(Cucurbitacin C)的酰基转移酶(ACT)、氧化鲨烯环化酶(OSC)和 3个 P450酶基因 CYP81Q58、CYP89A140、CYP97D19成簇于6号染色体中,而负责进一步羟基化修饰的 CYP712D8、CYP88L2和CYP88L3成簇在3号染色体,CYP87D2单独存在于1号染色体中。同时他们还发现这9个基因由两个“开关”基因Bl和Bt负责控制黄瓜不同组织的苦味,Bl控制叶片苦味,而Bt控制果实苦味。
但是限于植物基因组和性状的复杂性以及高昂的测序成本,现阶段完全获得一个物种的基因组信息还较为困难。针对特定代谢产物途径的解析,物种的转录组数据往往更有帮助,且也更容易获得。从数万个基因中筛选可能参与特定天然产物合成的候选基因具有很大的挑战性。通过在植物底盘中进行诱导信号刺激使之产生代谢物成分或种类差异,或挖掘具有代谢物差异的植物突变株,对差异样本进行差异转录分析,从而获得候选基因序列,并进一步进行功能分析,从而鉴定出特定基因成为可能。如诺斯卡品(Noscapine)是来源于罂粟,具有抗癌活性的生物碱。Winzer等[42]对含Noscapin的HN1和不含该生物碱的HM1、HT1 3个品种进行转录分析,发现10个基因仅在HN1中表达,通过对这10个基因的功能验证解析出Noscapin的生物合成途径。
代谢途径中酶促反应顺序的难以确定以及真实代谢中间体的难以获得也是天然产物合成途径解析中的难题之一。现在,可以通过植物表达载体共同瞬时表达,将候选基因分别构建成植物表达载体,转化到农杆菌中后进行组合表达候选蛋白,以此策略通过分析最终代谢产物进而来表征未知基因的功能。如鬼臼毒素是抗癌药物依托泊苷的前体,其生物合成途径没有得到完全解析,Lau等[43]通过分析盾叶鬼臼叶片损伤之后转录组数据差异,得出29个候选基因,将这29个候选基因在烟草里组合共表达,鉴定出6个有功能的基因,其中包括2个甲基转移酶、3个P450酶和1个双加氧酶。再将鉴定的6个基因和之前明晰功能的4个基因在烟草里面共表达,成功地在烟草叶片中检测到了依托泊苷的前体糖苷配基。
由于无法获得准确的代谢物中间体进行体外的功能验证,因此在植物体内建立功能验证方法显得尤为重要。目前,植物体内功能验证的主要思路是通过基因删除或基因表达抑制全部或部分缺失酶的功能,然后检测植物体内次生代谢产物含量的变化,以此来鉴定特定基因在原植物体内的生物功能。目前应用较为广泛的是病毒诱导的基因沉默(virus induced gene silencing,VIGS)技术和 RNA 干扰(RNAi)技术。Salim等[44]在长春花的转录组数据中筛选到参与环烯醚萜类合成的候选基因,利用VIGS技术和代谢分析,鉴定到了 1个羟化酶CrDL7H,能够参与裂环马钱子苷的第三步到最后一步合成。Zhao等[45]在解析人参皂苷的合成途径时,利用RNAi鉴定到的β-香树素(β-amyrin)合成酶基因;当该基因表达被抑制,β-香树素含量下降,同时齐墩果烷型人参皂苷R0含量上升,表明其具有重要作用。Wang等[46]将西洋参中克隆到的1个原人参三醇合酶基因(CYP6H)进行了过表达和RNAi研究,通过RT-PCR分析发现:在过表达的西洋参毛状根中该基因表达量显著增加,毛状根中原人参二醇型皂苷含量下降,原人参三醇型皂苷含量上升,结合RNAi实验结果证明在西洋参中CYP6H能对原人参二醇的6位进行羟基化,将原人参二醇转化为原人参三醇。
5 结论与展望
植物中含有的天然产物类型多样,其合成途径也十分复杂。以植物为底盘进行复杂的工程化设计,是合成生物学研究由简单到复杂、由单细胞到多细胞的必然,这不仅有助于我们了解复杂生命体再造的工程化原理,也有望变革传统的种植业,实现医药、食品等传统行业的革命性升级。
但这一领域仍有许多瓶颈亟待突破:代谢途径的解析仍是最大的瓶颈。尽管植物天然产物基因簇的发现为植物天然产物代谢途径的设计提供了便利,但是这对于大量尚未阐明合成机制的天然产物来说仅是冰山一角。许多参与天然产物生物合成的基因仅部分成簇存在,负责结构修饰的基因仍有可能存在于染色体的其他位置[47]。植物天然产物积累方式具有时空特异性,表明合成基因的表达受到植物体内或外部生长环境的严格调控。这些现象使得植物源天然产物的途径解析变得更加棘手。新一代的基因组学及多组学技术的结合是今后发展的重要方向。除此之外,开发新的植物底盘以应对不同的需求也是今后一个值得关注的方向。基于植物底盘的遗传操作技术和方法仍然不够完善。可应用于植物底盘的新一代基因组合成及基因组编辑的技术有待开发。植物是相对复杂的系统,代谢途径高度区域化的特点使得在进行底盘的复杂设计、定量描述和数学建模的时候面临更为复杂的局面,这些也都待开发新的方法。
[1] THOLL D.Biosynthesis and biological functions of terpenoids in plants[J].Adv Biochem Eng/Biotechnol,2015,148:63-106.
[2] KATZ L,BALTZ R.Natural product discovery:past,present,and future[J].J Ind Microbiol Biotechnol,2016,43(2):155-176.
[3] XIAO H,ZHONG J J.Production of useful terpenoids by higherfungus cell factory and synthetic biology approaches[J].Trends Biotechnol,2016,34(3):242-255.
[4] LIU W,STEWARTCN J R.Plant synthetic biology[J].Trends Plant Sci,2015,20(5):309-317.
[5] 王勇.新本草计划:基于合成生物学的药用植物活性代谢物研究[J].生物工程学报,2017,33(3):478-485.
[6] NÜTZMANN H W,OSBOURM A.Gene clustering in plant specialized metabolism[J].Curr Opin Biotechnol,2014,26:91-99.
[7] NÜTZMANN H W,HUANG A,OSBOURM A.Plant metabolic clusters:from genetics to genomics[J].New Phytol,2016,211(3):771-789.
[8] BOUTANAEV A M,MOSES T,ZI J,et al.Investigation of terpene diversification across multiple sequenced plant genomes[J].Proc Natl Acad Sci USA,2015,112(1):E81-E88.
[9] WANG C,CHEN Q,FAN D,etal.Structural analyses of shortchain prenyltransferasesidentifyan evolutionarily conserved GFPPS clade in Brassicaceae plants[J].Mol Plant,2016,9(2):195-204.
[10] FIELD B,FISTON-LAVIER A S,KEMENA,et al.Formation of plant metabolic gene clusters within dynamic chromosomal regions[J].Proc Natl Acad Sci USA,2011,108:16116-16121.
[11] SHAO J,CHEN Q W,LV H J,et al.(+)-Thalianatriene and(-)-retigeranin B catalyzed by sesterterpene synthases from Arabidopsis thaliana[J].Org Lett,2017,19(7):1816-1819.
[12] KING A J,BROWN G D,GILDAY A D,et al.Production of bioactive diterpenoid in the euphorbiaceae depend on evolutionarily conserved gene clusters[J].Plant Cell,2014,26(8):3286-3298.
[13] PADDON C J,WESTFALL P J,PITERA D J,et al.High-level semi-synthetic production of the potent antimalarial artemisinin[J].Nature,2013,496:528-532.
[14] PEPLOW M.Synthetic biology's first malaria drug meets market resistance[J].Nature,2016,530:389-390.
[15] FARHI M,MARHEVKA E,BEN-ARI J,et al.Generation of the potent anti-malarialdrug artemisinin in tobacco[J].Nature Biotechnol,2011,29(12):1072-1074.
[16] LV Z,ZHANG F,PAN Q,et al.Branch pathway blocking in Artemisia annua is a useful method for obtaining high yield artemisinin[J].Plant Cell Phisiol,2016,57(3):588-602.
[17] SHEN Q,LU X,YAN T,et al.The jasmonate-responsive AaMYC2 transcription factor positively regulates artemisinin biosynthesis in Artemisia annua[J].New Phytol,2016,210:1269-1281.
[18] LV Z,WANG S,ZHANGF,etal.Overexpression of a novel NAC domain-containing transcription factor gene(AaNAC1)enhances the content of artemisinin and increases tolerance to drought and Botrytis cinerea in Artemisia annua[J].Plant Cell Phisiol,2016,57(9):1961-1971.
[19] YAN T,CHEN M,SHEN Q,et al.HOMEODOMAIN PROTEIN 1 is required for jasmonate-mediated glandular trichome initiation in Artemisia annua[J].New Phytol,2016,213:1145-1155.
[20] FUENTES P,ZHOU F,ERBAN A,et al.A new synthetic biology approach allows transfer of an entire metabolic pathway from a medicinal plant to a biomass crop[J].ELife,2016,5:e13664.
[21] MALHOTRA K,SUBRAMANIYANM,RAWATK,etal.Compartmentalized metabolic engineering for artemisinin biosynthesis and effective malaria treatment by oral delivery of plant cells[J].Mol Plant,2016,9(11):1464-1477.
[22] ANTEROLA A,SHANLE E,PERROUD P F,et al.Production of taxa-4(5),11(12)-diene by transgenic Physcomitrella patens[J].Transgenic Res,2009,18(4):655-660.
[23] BESUMBES O,SAURET-GUETO S,PHILLIPS M A,et al.Metabolic engineering of isoprenoid biosynthesis in Arabidopsis for the production of taxadiene,the first committed precursor of taxol[J].Biotechnol Bioeng,2004,88(2):168-175.
[24] CHA M,SHIM S H,KIM S H,et al.Production of taxadiene from cultured ginseng roots transformed with taxadiene synthase gene[J].BMB Rep,2012,45(10):589-494.
[25] KWVACS K,ZHANG L,LINFORTH R S,et al.Redirection of carotenoid metabolism for the efficient production of taxadiene[taxa-4(5),11(12)-diene]in transgenic tomato fruit[J].Transgenic Res,2007,16(1):121-126.
[26] TOHGE T,ZHANG Y,PETEREK S,et al.Ectopic expression of snapdragon transcription factors facilitates the identification of genes encoding enzymes of anthocyanin decoration in tomato[J].Plant J,2015,83(4):686-704.
[27] ZHANG Y,BUTELLIE,ALSEEKH S,etal.Multi-level engineering facilitates the accumulation of bioactive compounds in tomato[J].Nature Commun,2015,6:8635.
[28] ZHANG Y,DE STEFANO R,ROBINE M,et al.Different reactive oxygen species scavenging properties of flavonoids determine their abilities to extend the shelf life of tomato[J].Plant Physiol,2015,169(3):1568-1583.
[29] ENGLER C,KANDZIA R,MARILLONNET S.A one pot,one step,precision cloning method with high-throughput capability[J].PLoS ONE,2008,3(11):e3647.
[30] ENGLER C,YOULES M,GRUETZNER R,et al.A golden gate modular cloning toolbox for plants[J].ACS Synth Biol,2014,3(11):839-843.
[31] SHIH P M,VUU K,MANSOORI N,et al.A robust gene-stacking method utilizing yeast assembly for plant synthetic biology[J].Nature Commun,2016,7:13215.
[32] ZHANG Y,LIANG Z,ZONG Y,et al.Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA[J].Nature Commun,2016,7:12617.
[33] LIANG Z,CHEN K,LI T,et al.Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes[J].Nature Commun,2017,8:14261.
[34] SAINSBURY F,LOMONOSSOFF G P.Extremely high-level and rapid transient protein production in plants without the use of viral replication[J].Plant Physiol,2008,148(3):1212-1218.
[35] KANAGARAJAN S,MUTHUSAMY S,GLISZCZYNSKA A,et al.Functionalexpression and characterization ofsesquiterpene synthases from Artemisia annua L.using transient expression system in Nicotiana benthamiana[J].Plant Cell Rep,2012,31(7):1309-1319.
[36] MATTOZZI MD,VOGES MJ,SILVERPA,et al.Transient gene expression in tobacco using Gibson assembly and the Gene Gun[J].J Vis Exp,2014,doi:10.3791/51234.
[37] ZHANG F,MAEDER M L,UNGER-WALLACE E,et al.High frequency targeted mutagenesis in Arabidopsis thaliana using zine finger nuclease[J].Proc Natl Acad Sci USA,2010,107:12028-12033.
[38] LI T,LIU B,SPALDING M H,et al.High-effency TALEN-based gene editing produces disease-resistant rice[J].Nature Biotechnol,2012,30(5):390-392.
[39] DI P,ZHANG L,CHEN J,et al.13C tracer reveals phenolic acids biosynthesis in hairy root cultures of Salvia miltiorrhiza[J].ACS Chem Biol,2013,8(7):1537-1548.
[40] GUO J,ZHOU Y J,HILLWING M L,et al.CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts[J].Proc Natl Acad Sci USA,2013,110:12108-12113.
[41] SHANG Y,MA Y,ZHOU Y,et al.Plant science:biosynthesis,regulation,and domestication of bitterness in cucumber[J].Science,2014,346:1084-1088.
[42] WINZER T,GAZDA V,HE Z,et al.A Papaver somniferum 10-gene cluster for synthesis of the anticancer alkaloid noscapine[J].Science,2012,336:1704-1708.
[43] LAU W,SATTELY E S.Six enzymes from mayapple that complete the biosynthetic pathway to the etoposide aglycone[J].Science,2015,349:1224-1228.
[44] SALIM V,YU F,ALTAREJOS J,et al.Virus-induced gene silencing identifies Catharanthus roseus 7-deoxyloganic acid-7-hydroxylase,a step in iridoid and monoterpene indole alkaloid biosynthesis[J].Plant J,2013,76:754-765.
[45] ZHAO C,XU T,LIANG Y,et al.Functional analysis of β-amyrin synthase gene in ginsenoside biosynthesis by RNA interference[J].Plant Cell Rep,2015,34(8):1307-1315.
[46] WANG L,ZHAO S J,LIANG Y L,etal.Identification of the protopanaxatriol synthase gene CYP6H for ginsenoside biosynthesis in Panax quinquefolius[J].Funct Integr Genomics,2014,14(3):559-570.
[47] BOYCHEVA S,DAVIET L,WOLFENDER J L,et al.The rise of operon-like gene clusters in plants[J].Trends Plant Sci,2014,19(7):447-459.
(责任编辑 荀志金)
Plant chassis:new hotspots of natural product synthetic biology
SHAO Jie1,2,LI Jianhua1,WANG Kaibo1,3,WANG Yong1
(1.Key Laboratory of Synthetic Biology,CAS Center for Excellence in Molecular Plant Sciences,Institute of Plant Physiology and Ecology,Shanghai Institutes for Biological Sciences,Chinese Academy of Sciences,Shanghai 200032,China;2.University of Chinese Academy of Sciences,Beijing 100049,China;3.Key Laboratory of Stress Plant Biology,Henan University,Kaifeng 475000,China)
Natural products are widely produced in plants and become major source of pharmaceuticals,aromatics and potential next-generation biofuels,with great commercial value.These compounds have also been the hotspots of synthetic biology.With deeper investigation,plant chassis have been increasingly applied to natural product biosynthesis research.In this paper,we reviewed the recent advances and achievements of synthetic biology based on plant chassis,including metabolic pathway elucidation,genetic manipulation technology and methods.
natural product;synthetic biology;plant chassis
Q819;TQ041
A
1672-3678(2017)05-0024-08
10.3969/j.issn.1672-3678.2017.05.003
2017-06-29
中国科学院分子植物科学卓越创新中心部署项目(CEMPS2016004);国家重点基础研究发展计划(973计划)(2012CB721104);国家自然科学基金(31170101)
邵 洁(1993—),女,江苏南通人,博士研究生,研究方向:天然产物合成生物学;王 勇(联系人),研究员,E-mail:yongwang@sibs.ac.cn
