酿酒酵母醛酮还原酶的克隆表达和酶学性质研究
2017-09-12应向贤毛王伟
应向贤,高 亮,张 丽,毛王伟,赵 嫚,汪 钊
(浙江工业大学 生物工程学院,浙江 杭州 310014)
酿酒酵母醛酮还原酶的克隆表达和酶学性质研究
应向贤,高 亮,张 丽,毛王伟,赵 嫚,汪 钊
(浙江工业大学 生物工程学院,浙江 杭州 310014)
从酿酒酵母(Saccharomycescerevisiae) CICC 1002基因组中克隆获得醛酮还原酶基因,并在大肠杆菌BL21 (DE3)中实现过量表达.重组醛酮还原酶经Ni-NTA亲和层析分离纯化获得高纯度目的蛋白,并对其进行酶学性质表征.该重组酶经SDS-PAGE检测为单一条带,分子量为38 kDa.该酶的最适pH和最适温度分别为6.0,60 ℃,且具有良好的稳定性.1 mmol/L金属离子Co2+或Ni2+显著提高酶活力.底物特异性分析表明:该重组酶对邻位二酮具有较高活力,其中对酮基泛解酸内酯的比酶活可达20.53 U/mg.
酿酒酵母;醛酮还原酶;克隆表达;酶学性质;酮基泛解酸内酯
醛酮还原酶(Aldo-keto reductase; AKR)是氧化还原酶超家族成员之一,广泛存在于真核与原核生物中[1].AKR超家族目前由15 个家族组成,家族成员约有150 个[2],它们的催化中心为D-Y-K-H,且以β-折叠的羧基端与辅酶结合,这与短链脱氢/还原酶(Short-chain dehydrogenases/reductases; SDR)不同,后者是通过Rossmann折叠区域和NAD (P) (H)结合[3].鉴于醛酮还原酶表现出较高的催化活性和立体选择性,其在手性醇不对称合成上的应用备受关注[4].D-泛解酸内酯是D-泛酸合成过程中重要的手性前体,工业上由化学法羟醛缩合作为起始反应合成混旋泛解酸内酯[5-6],再通过生物酶水解拆分而获得[7-8].生物酶选择性水解拆分泛解酸内酯需要D-泛解酸和L-泛解酸内酯的分离,D-泛解酸内酯化以及L-泛解酸内酯的消旋化后再利用[9].为了克服拆分法的不足,研究者开发了多种新的催化工艺,其中又以氧化还原酶法合成D-泛解酸内酯的工艺最具应用潜力[9].氧化还原酶法利用L-泛解酸内酯脱氢酶催化L-泛解酸内酯的脱氢生成酮基泛解酸内酯,酮基泛解酸内酯再通过酮基泛解酸内酯还原酶催化的不对称还原反应生成D-泛解酸内酯[10],实现手性翻转.来自Candidaparapsilosis中的CPR-C2是目前已知的活力较高的酮基泛解酸内酯还原酶[11],但其催化效果仍达不到工业化应用的要求.因而,发现新型的高活力酮泛解酸内酯还原酶具有积极意义.
以酿酒酵母(SaccharomycescerevisiaeCCIC 1002)为出发菌株,对其基因组中的醛酮还原酶基因进行分子克隆和诱导表达,并利用Ni-NTA亲和层析实现目的蛋白的分离纯化,确定其可以用于不对称合成D-泛解酸内酯.在此基础上,表征了该重组酶的酶学性质,如最适温度、最适pH、pH和温度稳定性、金属离子的影响和底物特异性等,为后续酶法不对称还原酮基泛解酸内酯的工业应用奠定了基础.
1 材料与方法
1.1 材 料
1.1.1 菌株与载体
酿酒酵母(S.cerevisiaeCCIC 1002)由本实验室保藏;宿主菌EscherichiacoliBL21(DE3)为实验室已有资源;E.coliTrans-T1克隆宿主菌和载体pEASY-Blunt E1购自北京全式金(TransGen Biotech)生物技术有限公司.
1.1.2 试 剂
PfuDNA聚合酶及配套材料,北京全式金生物技术有限公司;Taq DNA聚合酶及其对应dNTPs,南京诺唯赞生物科技有限公司;用于质粒提取、PCR产物纯化试剂盒,大连宝生物工程有限公司;基因组提取试剂盒、辅酶NAD(P)(H)、蛋白标准溶液BSA等,上海生工生物工程股份有限公司.
1.1.3 引 物
根据S.cerevisiaeS288c中D-阿拉伯糖1-脱氢酶基因设计PCR扩增醛酮还原酶基因引物,引物如下:
上游引物SceAKR3C1 F:5′- ATGTCTTCTTCAGTAGCCTCAAC-3′
下游引物SceAKR3C1 R:5′-TTAATACTTTAAATTGTCCAAGTTTG-3′
1.1.4 培养基
YPD培养基(g/L):酵母膏 10,蛋白胨 20,葡萄糖 20.调节pH至7.0,培养基于115 ℃下高压蒸汽灭菌20 min.
LB培养基(g/L):蛋白胨10,酵母膏5,NaCl 10.调节pH至7.0,固体培养基中添加2.0%的琼脂粉,培养基于121 ℃下高压蒸汽灭菌20 min.
LB/Amp培养基(g/L):在LB培养基中加入氨苄青霉素(Ampicillin; Amp)至终质量浓度100 μg/mL.
1.2 方 法
1.2.1 醛酮还原酶SceAKR3C1的基因克隆
以S.cerevisiaeCCIC 1002基因组为DNA模板,进行醛酮还原酶SceAKR3C1的基因克隆,PCR扩增参数:95 ℃预变性2 min;95 ℃变性20 s;55 ℃复性20 s;72 ℃延伸1 min;反应循环数为35;72 ℃复延伸5 min.PCR扩增产物用0.8%的琼脂糖凝胶电泳检测,并回收纯化.纯化后PCR产物与载体pEASY-Blunt E1连接,并将连接产物转入50 μLE.coliTrans-T1感受态细胞中.采用菌落PCR筛选阳性克隆子,选取阳性克隆子送至上海桑尼生物科技有限公司进行测序确证.将测序所得的基因序列与NCBI数据库中已有的基因序列进行比对,并将含有目的片段的质粒命名为pEASY-Blunt E1-SceAKR3C1.
1.2.2 基因重组菌E.coliBL21 (DE3)/pEASY-Blunt E1-SceAKR3C1的构建及诱导表达
利用质粒提取试剂盒提取重组质粒pEASY-Blunt E1-SceAKR3C1,取50~100 ng将其转入50 μL刚解冻的感受态细胞E.coliBL21 (DE3)中,孵育1 h后将其涂布于LB/Amp固体平板上,37 ℃恒温培养箱中过夜培养.挑取阳性克隆子,接种于含Amp抗性的LB液体培养基中,37 ℃,200 r/min摇瓶培养12 h,作为种子液.按体积分数2%接种量扩大培养至Amp抗性的150 mL LB液体培养基中,37 ℃,200 r/min摇瓶培养至菌体浓度OD600=0.6~0.8时,添加诱导剂异丙基-β-D-硫代吡喃半乳糖苷(IPTG;0.1 mmol/L).然后22 ℃,150 r/min诱导10 h.
1.2.3 SceAKR3C1的分离纯化
诱导结束后,4 ℃,8 000 r/min离心10 min收集菌体,并用超纯水反复重悬清洗,再离心收集菌体.用50 mmol/L Tris-HCl缓冲液(pH 8.0)重悬菌体(菌体最终质量浓度为50 g/L),然后在冰浴条件下超声破碎细胞.超声破碎参数:工作时间1 s,间歇时间1 s,有效破碎时间为10 min.细胞裂解液在4 ℃下12 000 r/min离心10 min,收集上清即为含SceAKR3C1粗酶液.
Ni-NTA亲和层析柱经含5 mmol/L咪唑的Tris-HCl缓冲液(50 mmol/L, pH 8.0)预平衡后,加入SceAKR3C1粗酶液上样.Ni-NTA亲和层析柱上结合的蛋白通过5~250 mmol/L的咪唑浓度梯度进行逐步梯度洗脱,并对各洗脱组分进行SDS-PAGE检测.将洗脱出的含有目的蛋白SceAKR3C1的溶液用50 mmol/L Tris-HCl (pH 8.0)通过超滤脱盐,保存在-20 ℃下进一步使用.以牛血清白蛋白作为标准蛋白,利用Bradford试剂测定蛋白质浓度.
1.2.4 醛酮还原酶SceAKR3C1的酶学性质研究
以酮基泛解酸内酯为底物,利用紫外分光光度计检测NADPH在波长340 nm下1 min内的吸光度变化值来测定酶活力.标准酶活力测定体系为2.5 mL,包括:10 mmol/L酮基泛解酸内酯,5.9 μg SceAKR3C1,0.1 mmol/L NADPH,0.2 mol/L PBS缓冲液(pH 7.0)补至2.5 mL,反应温度60 ℃.NADPH的摩尔消光系数为6.22×103L/(mol·cm),酶活力单位(U)定义为每分钟消耗1 μmol NADPH所需的酶量.
通过测定pH 5.0~8.5缓冲液中SceAKR3C1的酶活力确定该目的蛋白的最适pH,其中pH 5.0缓冲液为0.2 mol/L KH2PO4-Na2HPO4溶液.在最适pH条件下,通过测定25~65 ℃下SceAKR3C1的酶活力确定该目的蛋白的最适温度.在pH稳定性方面,在最适温度下将该酶孵育在不同pH的缓冲液中,间隔一定时间测定残余活力,孵育前的酶活力为100%.在热稳定性方面,在最适pH下测定该酶在最适温度下孵育一定时间后的残余活力,孵育前的酶活力为100%.在金属离子影响方面,在标准反应体系中分别添加终浓度均为1 mmol/L的金属离子或金属螯合剂EDTA,在酶的最适反应条件下测定其酶活力,以不添加金属离子时的酶活力为100%.在底物特异性方面,在标准反应体系中分别添加终浓度均为10 mmol/L的不同的醛、酮化合物作为底物,在最适反应条件下测定其酶活力,以酮基泛解酸内酯为底物时的酶活力为100%.
2 结果与讨论
2.1 SceAKR3C1的表达和纯化
以S.cerevisiaeCCIC 1002基因组为DNA模板,经PCR扩增后获得目的基因,该基因序列与S.cerevisiaeS288c中D-阿拉伯糖1-脱氢酶基序列一致.成功构建E.coliBL21 (DE3)/pEASY-Blunt E1-SceAKR3C1重组菌,并实现了重组菌中醛酮还原酶SceAKR3C1的过量表达.粗酶液经Ni-NTA亲和层析进行分离纯化,目的蛋白用含40 mmol/L咪唑的Tris-HCl缓冲液(50 mmol/L,pH 8.0)洗脱,并通过超滤进行脱盐处理.重组菌的诱导表达前后以及SceAKR3C1纯酶经SDS-PAGE检测结果如图1所示,相比于诱导前,诱导后菌体在大小约为38 kDa处出现明显的加粗条带,并与纯化的目的蛋白位置一致.通过标准酶活力检测和蛋白质浓度测定,该酶的比活力为20.53 U/mg.

M—蛋白Marker;1—诱导前粗酶液;2—诱导后粗酶液;3—纯化后SceAKR3C1图1 SceAKR3C1的SDS-PAGE分析Fig.1 SDS-PAGE analysis of SceAKR3C1
2.2 最适温度和最适pH
不同pH和温度对酶活的影响分别如图2,3所示.醛酮还原酶SceAKR3C1在pH 5.0~7.0条件下,酶活力相对较高,在pH 6.0条件下达到最大,但随着pH升高,酶活力迅速下降,在pH 8.5下几乎没有酶活.温度为25~60 ℃时,醛酮还原酶SceAKR3C1酶活随着温度的上升而提高,当温度为60 ℃时,酶活力达到最高,但继续增加温度,酶活急剧下降.因此,醛酮还原酶的最适pH为6.0,最适温度为60 ℃.

图2 pH对醛酮还原酶SceAKR3C1活力的影响 Fig.2 Effect of pH on the activity of the aldo-keto reductase SceAKR3C1
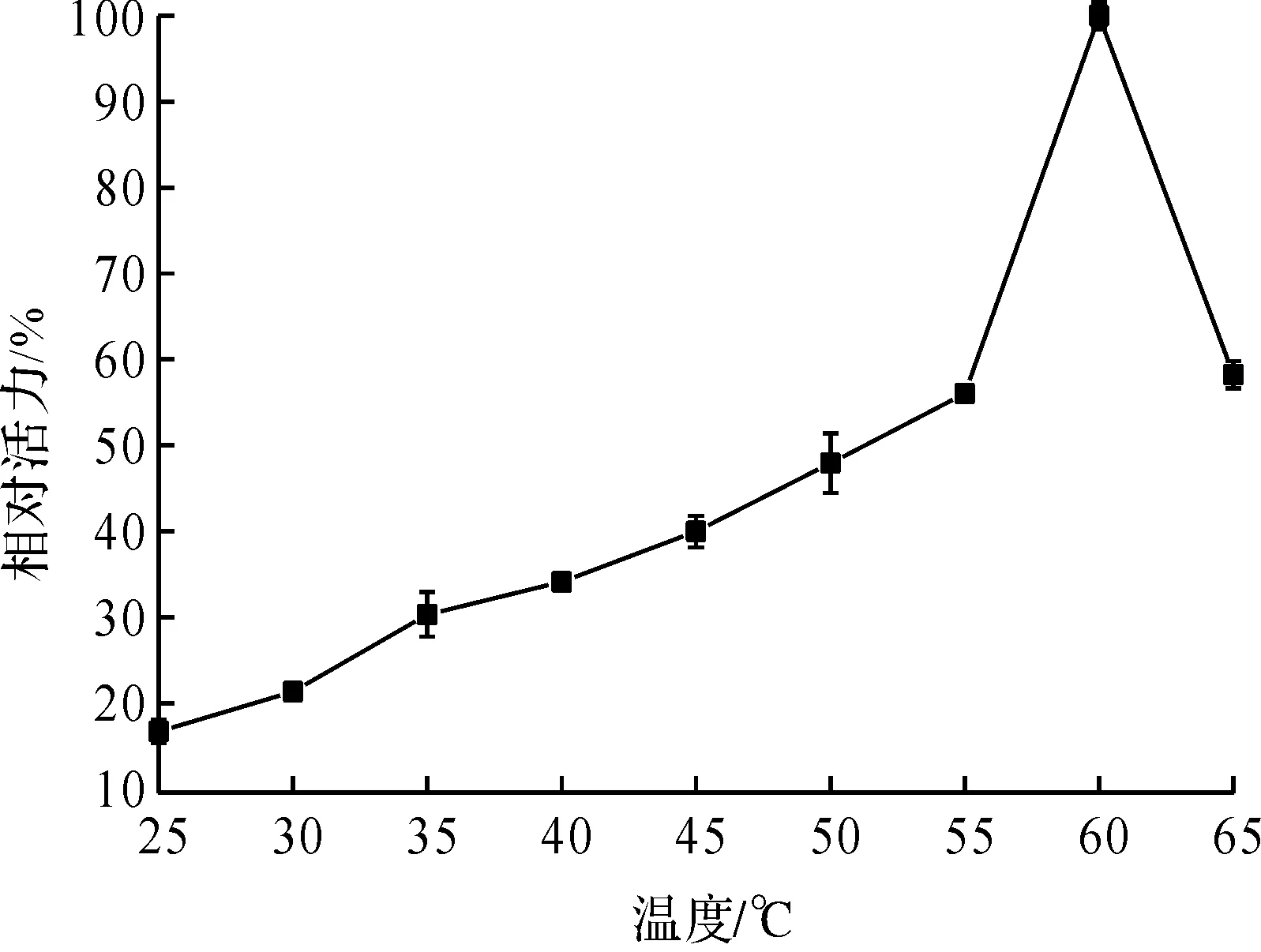
图3 温度对醛酮还原酶SceAKR3C1活力的影响 Fig.3 Effect of temperature on the activity of the aldo-keto reductase SceAKR3C1
2.3 热稳定性和pH稳定性
在pH分别为5.0,6.0,7.0的0.2 mol/L PBS缓冲液环境下考察醛酮还原酶SceAKR3C1的稳定性(图4).结果表明:偏酸性的条件对酶的稳定性影响并不显著,当在pH 5.0条件下放置60 h后酶活力还能保持在初始活力的83.6%.此外,在最适温度60 ℃下考察了酶的热稳定性.在60 ℃水浴中孵育10 h,酶活力基本不变,随着放置时间的延长,酶活力快速下降,约40 h后酶活力基本消失,相对于初始活力只剩下10.4%(图5).

图4 醛酮还原酶SceAKR3C1的pH稳定性Fig.4 The pH stability of the aldo-keto reductase SceAKR3C1

图5 醛酮还原酶SceAKR3C1的热稳定性Fig.5 Thermostability of the aldo-keto reductase SceAKR3C1
2.4 金属离子的影响
终浓度为1 mmol/L的金属离子可影响醛酮还原酶SceAKR3C1的活力(图6).Co2+,Ni2+均可不同程度地提高SceAKR3C1的酶活力,在含有1 mmol/L Co2+和Ni2+体系中其相对活力分别达到了163%和149%.而Ba2+,Mn2+,K+,Mg2+,Na+或EDTA对酶活力没有显著影响,但Ca2+,Al3+或Zn2+则对该酶活力存在一定的抑制作用.低浓度的Co2+或Ni2+能有效地提高还原酶SceAKR3C1的酶活力,该特性可在后续催化工艺的构建中予以考虑.
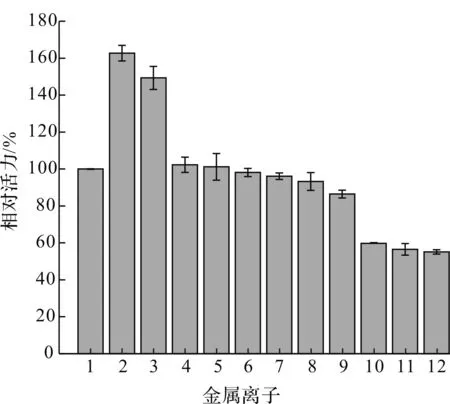
1—对照;2—Co2+;3—Ni2+;4—Ba2+;5—Mn2+;6—K+;7—Mg2+;8—Na+;9—EDTA;10—Ca2+;11—Al3+;12—Zn2+图6 金属离子对醛酮还原酶SceAKR3C1的影响 Fig.6 Effect of metal ions on the activity of the aldo-keto reductase SceAKR3C1
2.5 酮基泛解酸内酯还原酶的底物特异性
以酮基泛解酸内酯的比酶活力(20.53 U/mg)为对照,考察醛酮还原酶SceAKR3C1的底物特异性,结果如表1所示.SceAKR3C1对酮基泛解酸内酯(Entry 1)具有高酶活力,但对其自发水解产物酮基泛解酸(Entry 6)、以及D-泛解酸内酯、L-泛解酸内酯(Entry 7,8)均无活力.同时SceAKR3C1以邻位二酮(Entry 2)为底物时,相对活力达到78.70%,但对间位酮基底物活力则相对较低(Entry 3~5).
表1 醛酮还原酶Sce-AKR3C1的底物特异性
Table 1 Substrate specificity of the aldo-keto reductase SceAKR3C1
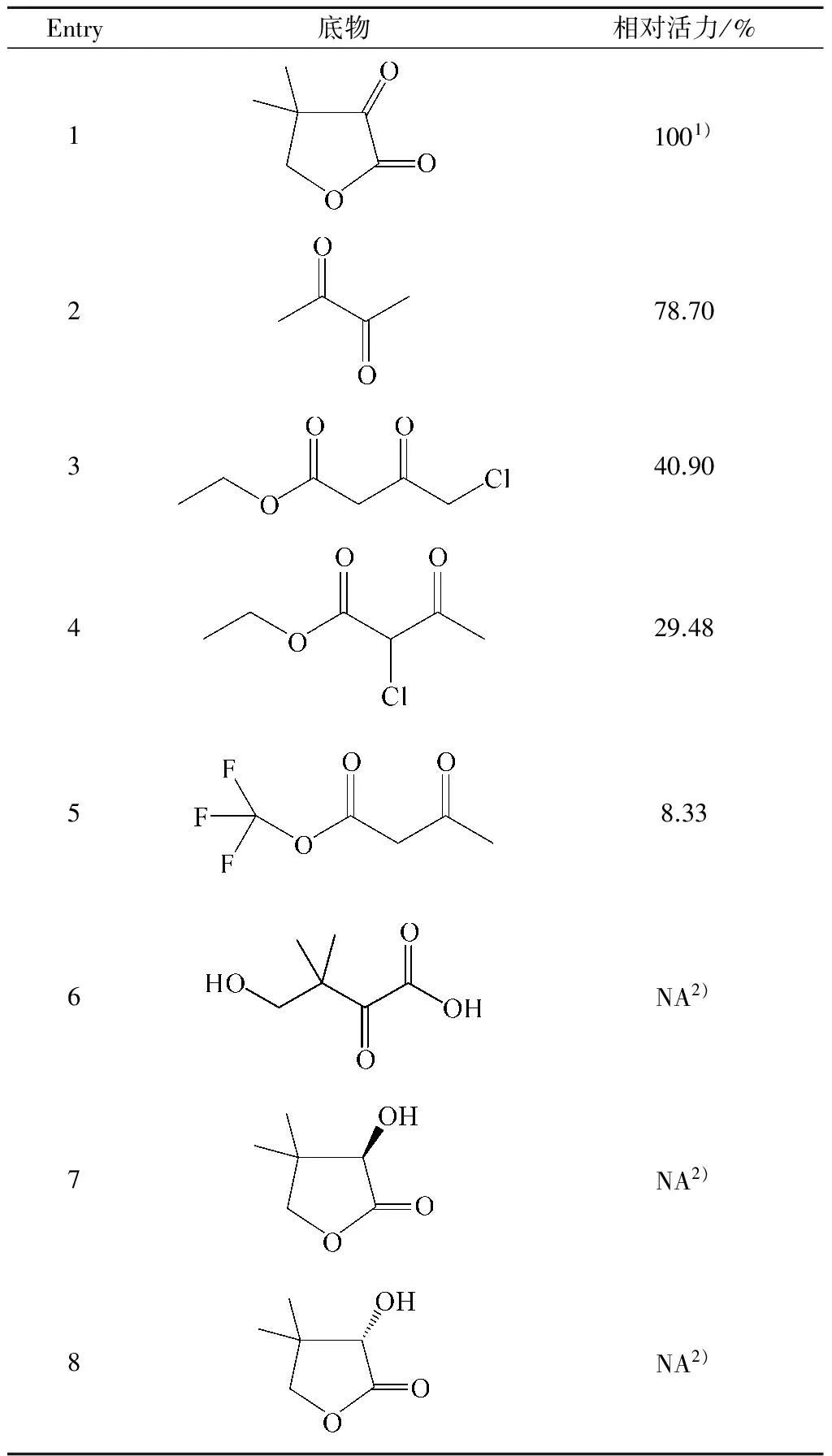
Entry底物相对活力/%11001)278.70340.90429.4858.336NA2)7NA2)8NA2)
注:1) 当SceAKR3C1的相对活力为100%时,其比酶活力为20.53 U/mg;2) NA表示未检测到活力.
3 结 论
以S.cerevisiaeCCIC 1002基因组为DNA模板,利用基因工程技术成功克隆表达并分离纯化获得了一种醛酮还原酶SceAKR3C1.该酶能利用辅酶NADPH催化不对称还原酮基泛解酸内酯反应,并且有较好的温度稳定性及pH稳定性.低浓度的Co2+,Ni2+可有效地提高还原酶SceAKR3C1的酶活力.在底物特异性方面,该重组酶对邻位二酮具有较高的酶活力,其中对酮基泛解酸内酯活力最高.重组醛酮还原酶SceAKR3C1与当前研究较多的酮基泛解酸内酯还原酶CPR-C2 (19.30 U/mg)活力相近[12],在酶法不对称还原酮基泛解酸内酯形成D-泛解酸内酯中具有应用潜力.
[1] ELLIS E M. Microbial aldo-keto reductases[J]. FEMS microbiology letters, 2002, 216(2): 123-131.
[2] HYNDMAN D, BAUMAN D R, HEREDIA V V, et al. The aldo-keto reductase superfamily homepage[J]. Chemico-biological interactions, 2003, 143(2): 621-631.
[3] KAMITORI S, IGUCHI A, OHTAKI A, et al. X-ray structures of NADPH-dependent carbonyl reductase fromSporobolomycessalmonicolorprovide insights into stereoselective reductions of carbonyl compounds[J]. Journal of molecular biology, 2005, 352(3): 551-558.
[4] 李凌凌, 吕早生, 吴敏, 等. 醛酮还原酶及其在不对称合成手性醇中的应用[J]. 化学与生物工程, 2009, 26 (11): 62-67.
[5] EVANS D A, WU J, MASSE C E, et al. A general method for the enantioselective synthesis of pantolactone derivatives[J]. Organic letters, 2002, 4(20): 3379-3382.
[6] HEIDLINDEMANN M, HAMMEL M, SCHEFFLER U, et al. Chemoenzymatic synthesis of vitamin B5-intermediate (R)-pantolactone via combined asymmetric organo-and biocatalysis[J]. Journal of organic chemistry, 2015, 80(7): 3387-3396.
[7] SHIMIZU S, MORIKAWA T, NITTA K, et al. Biocatalytic optical resolution of DL-pantolactone on an industrial scale[J]. The chemical society of Japan, 2002(1): 1-9.
[8] KATAOKA M, SHIMIZU K, SAKAMOTO K, et al. Optical resolution of racemic pantolactone with a novel fungal enzyme, lactonohydrolase[J]. Applied microbiology and biotechnology, 1995, 43(6): 974-977.
[9] 汪钊,黄美娟,高亮, 等. 化学酶法合成D-泛解酸内酯的研究进展[J]. 发酵科技通讯, 2016, 45(4): 193-198.
[10] SHIMIZU S, HATTORI S, HATA H, et al. One-step microbial conversion of a racemic mixture of pantoyl lactone to optically active D-(-)-pantoyl lactone[J]. Applied and environmental microbiology, 1987, 53(3): 519-522.
[11] KATAOKA M, DELACRUZ-HIDALGO A R G, AKOND M A, et al. Gene cloning and overexpression of two conjugated polyketone reductases, novel aldo-keto reductase family enzymes, ofCandidaparapsilosis[J]. Applied microbiology and biotechnology, 2004, 64(3): 359-366.
[12] HATA H, SHIMIZU S, HATTORI S, et al. Ketopantoyl lactone reductase is a conjugated polyketone reductase[J]. FEMS microbiology letters, 1989, 58(1): 87-90.
(责任编辑:朱小惠)
Cloning, over-expression and characterization of an aldo-keto reductase fromSaccharomycescerevisiaeCICC 1002
YING Xiangxian, GAO Liang, ZHANG Li, MAO Wangwei, ZHAO Man, WANG Zhao
(College of Biotechnology and Bioengineering, Zhejiang University of Technology, Hangzhou 310014, China)
The gene encoding an aldo-keto reductase fromSaccharomycescerevisiaeCICC 1002 (SceAKR3C1) was cloned and over-expressed inEscherichiacoliBL21 (DE3). The recombinant aldo-keto reductase SceAKR3C1 was purified by Ni-NTA affinity chromatography and a single band corresponding to a molecular weight of 38 kDa was observed on the SDS-PAGE. The enzymatic properties were characterized. The optimum pH and temperature of SceAKR3C1 were 6.0 and 60 ℃, respectively, and the metal ions Co2+and Ni2+significantly increased the enzymatic activity. The substrate spectrum analysis indicated that the recombinant SceAKR3C1 had high activity on vicinal diketones, with a specific activity of 20.53 U/mg using ketopantolactone as substrate.
Saccharomycescerevisiae; aldo-keto reductase; cloning and expression; enzymatic properties; ketopantolactone
2017-03-29
浙江省新苗人才计划项目(2016R403074);浙江省自然科学基金资助项目(LY17B020012)
应向贤(1975—),男,浙江永康人,副教授,博士,研究方向为生物催化,E-mail:yingxx@zjut.edu.cn.
Q55
A
1674-2214(2017)03-0129-05
