新疆塔城传统酸奶中乳酸菌的多样性及发酵特性分析
2017-09-03蒋艾廷李宝坤乔传丽赵利利
蒋艾廷,李宝坤,金 丹,乔传丽,杨 婕,赵利利
(石河子大学食品学院,新疆石河子 832000)
新疆塔城传统酸奶中乳酸菌的多样性及发酵特性分析
蒋艾廷,李宝坤*,金 丹,乔传丽,杨 婕,赵利利
(石河子大学食品学院,新疆石河子 832000)
本实验通过观测发酵液的pH与溴甲酚紫平板的变色时间,从新疆塔城传统酸奶中筛选出产酸快的30株乳酸菌。通过形态特征观察与生理生化鉴定等传统方法,结合rep-PCR基因分型和16S rDNA基因序列分析对其进行鉴定,并分析不同种属之间乳酸菌的蛋白酶活性、自溶度与产酸特性的关系。结果表明:这30株乳酸菌分别为植物乳杆菌(lactobacillusplantarum)12株、海氏肠球菌(Enterococcushirae)9株、耐久肠球菌(Enterococcusdurans)3株、发酵乳杆菌(lactobacillusfermentum)与屎肠球菌(Enterococcusfaecium)各2株、乳酸片球菌(Pediococcusacidilactici)与粪肠球菌(Enterococcusfaecalis)各1株。7种乳酸菌在牛乳中均能正常生长,可以降低牛乳的pH,蛋白酶活性在3~14 U之间,自溶度在3%~29%之间,其发酵特性与菌株的种属有着密切的关系。
乳酸菌,16s rDNA,酸奶,多样性,发酵特性
传统酸奶是少数民族日常生活中不可或缺的一种食品,与市面工业化生产的酸奶相比,制作工艺过程更加生态、自然,其中的益生菌种类也更加丰富。一般是由当地农家妇女制作将牛乳挤在羊胃中,放在阳光下晾晒,获得发酵剂,随后将发酵剂转移至塑料桶中,将牛乳挤入桶中发酵3~5 d,获得这种天然发酵乳制品。这些发酵乳制品中的微生物经过几千年的自然驯化,已经适应了这种生存环境并且有着不同的发酵特性[1]。2015年Xiao Ping[2]等人首次从腌制的茶叶中筛选出了8株产酸快的乳酸菌,经鉴定均为植物乳杆菌,极大地缩短了发酵茶的生产周期。在发酵乳制品生产过程中,产酸快的乳酸菌可以缩短发酵时间,提高生产效率,从而降低生产成本,提高经济效益[3-4]。此外研究[5-6]发现,乳酸菌的蛋白酶活性和自溶对发酵乳制品的发酵时间和凝乳质地以及风味形成也有一定关系。Hannon[7]等人发现乳酸菌具有较高的蛋白酶活性与适当的自溶度可以缩短干酪的成熟周期,增加产品风味。孙洁等[8]通过N+注入的诱变方式获得了自溶度较高的唾液链球菌和德氏乳杆菌,对控制产品的成熟周期及感官品质具有良好实用价值。目前,研究学者[9-11]针对发酵食品中某些优良特性乳酸菌的筛选作了大量研究,但大多采用传统的鉴定方法,操作相对复杂且成本过高。重复片段基因指纹分析(Repetitive-element PCR genome fingerprinting,rep-PCR)是一种以核糖核酸为基础的分子生物学分型方法[12],被广泛地运用在许多农业、环境与医药中[13]。Dirk等[14]发现rep-PCR能够快速、可靠地对发酵食品中的乳杆菌和其他乳酸菌进行基因分型。
本研究通过对传统发酵酸奶中的乳酸菌的产酸能力进行评价,筛选出在酸奶发酵过程中产酸快的菌株,采用生理生化实验与现代分子生物学的方法进行种属鉴定,探讨传统酸奶中产酸快乳酸菌的菌相构成,分析不同种属乳酸菌的产酸特性、自溶度、蛋白酶活性,为优良发酵特性的发酵剂生产菌株的选择提供参考。
1 材料与方法
1.1 材料与仪器
实验菌株 由石河子大学食品学院筛选并保藏;细菌基因组总DNA提取试剂盒 北京Trans Gen公司;琼脂糖、溶菌酶 美国Sigma公司;2×Taq PCR Master Mix 广州东盛生物有限公司;其他试剂及培养基均为国产分析纯。
高速冷冻离心机(5417R) 德国Eppendorf公司;PCR扩增仪(TC-512) 英国Techne公司;凝胶成像仪 美国Bio-Rad公司;恒温恒湿培养箱 德国Binder公司;超净工作台 苏州苏洁净化设备有限公司;电热蒸汽灭菌锅 上海申安医疗器械厂。
1.2 实验方法
1.2.1 菌种活化与产酸快菌株的筛选 采用液体MRS培养基将冻藏的乳酸菌活化两次,稀释涂布于含有0.06%溴甲酚紫指示剂的MRS琼脂平板中,置于37 ℃恒温培养箱内培养24 h,观察并筛选出使平板变黄的菌株。谢静等[15]研究发现,乳酸菌的产酸特性与生长周期有关,处于对数期的乳酸菌生长与产酸的速率最大。将能使溴甲酚紫变黄的菌株活化后,接入MRS液体培养基中,培养16 h到达对数生长末期,用pH计测定培养基的pH,ΔpH表示乳酸菌从0 h生长至16 h的产酸量。挑出ΔpH较大的菌株于4 ℃保存备用。
1.2.2 生理生化实验 对筛选出来产酸快的乳酸菌进行吲哚实验、硝酸盐还原实验、明胶液化实验、葡萄糖产气等生理生化实验,实验方法参照《常见细菌系统鉴定手册》[16]、《乳酸菌分类鉴定及实验方法》[17]。
1.2.3 16S rDNA 基因序列分析
1.2.3.1 乳酸菌基因组DNA的提取 应用Gen Elute TM细菌总DNA提取试剂盒对乳酸菌分离菌株的DNA进行提取,按照说明书操作。
1.2.3.2 rep-PCR指纹图谱分析 rep-PCR所用的引物为单引物(GTG)5,序列为(5′-GTG GTG GTG GTG GTG-3′)[18],PCR反应体系为25 μL,引物1 μL,DNA稀释液1 μL,预混液12.5 μL,补充10.5 μL dd H2O至终体积为25 μL。扩增的反应程序为:95 ℃预变性7 min;94 ℃变性30 s,40 ℃退火1 min,65 ℃延伸8 min,执行35个循环;65 ℃维持16 min。1.5%的琼脂糖凝胶(含EB)电泳(电压55 V,电泳时间2 h),检测扩增结果,UVI凝胶成像系统照相,记录条带谱。
电泳结果通过比对每条条带的有(1)或无(0)构建n×t数据矩阵,如果条带存在,则记为1,不存在,则记为0。数据矩阵使用Jaccard系数(Jaccard’s coefficient)检测不同菌株间的相似性[19]。使用Ntsys 2.1软件进行非加权组平均法(unweighted pair groupmethod with arithmetic mean,UPGMA)归类分析构建树形图,并检测共性分类相关系数。
1.2.3.3 16S rDNA基因序列测定 应用细菌16S rDNA基因通用引物对细菌16S rDNA的V6~V8区段进行PCR扩增。上游引物为U968(5′-AACGCGAAGAACCTT AC-3′);下游引物为L1401(5′-CGGTGTGTACAAGACCC-3′)[20]。PCR反应体系为25 μL,引物各1 μL,1 μL的DNA稀释液,预混液12.5 μL,补充9.5 μL dd H2O至终体积为25 μL。PCR扩增程序:94 ℃预变性5 min,30个循环(94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s),72 ℃延伸10 min。PCR扩增产物经1%琼脂糖凝胶电泳检测,
1.2.3.4 同源性分析 采用MEGA5.0中的邻接法(Neighbour-joining)将测序所得序列与NCBI中BLAST获得的标准菌株16S rDNA基因序列进行系统发育树的构建。
1.2.4 发酵牛乳实验 将7株乳酸菌用MRS培养基活化2代,使其恢复活力,按4%的接种量已灭菌的牛乳中,每隔2 h测定pH。
1.2.5 自溶度的测定 活化菌株MRS肉汤培养基37 ℃培养至稳定期,离心收集菌体细胞(4000 r/min,10 min),以无菌水洗涤两次,所得菌体细胞重悬于PBS缓冲液(50 mmol/L,pH6.5)中,调整菌液吸光值(OD650nm)至1.0左右,置于37 ℃下进行培养,于0、24 h取样测定。自溶度计算公式如下[21]。
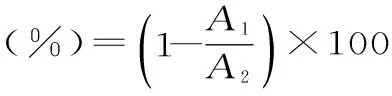
式(1)
式(1)中:A1为菌液吸光值;A2为菌液初始吸光值。
1.2.6 蛋白酶活性测定 蛋白酶活性的检测采用Folin酚法[22],于680 nm波长下测定吸光值,以不加酶液的体系设置空白对照。酶活力定义:在40 ℃、pH7.5条件下,1 min水解酪蛋白产生1 μg 酪氨酸所需的酶量为一个酶活性单位U。计算公式为:
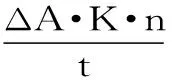
式(2)
式(2)中:ΔA为样品与空白对照的吸光值之差;K为吸光常数;n为反应总体积,mL;t为反应时间,min。根据本次实验中酪氨酸标准曲线,确定K=83.15。
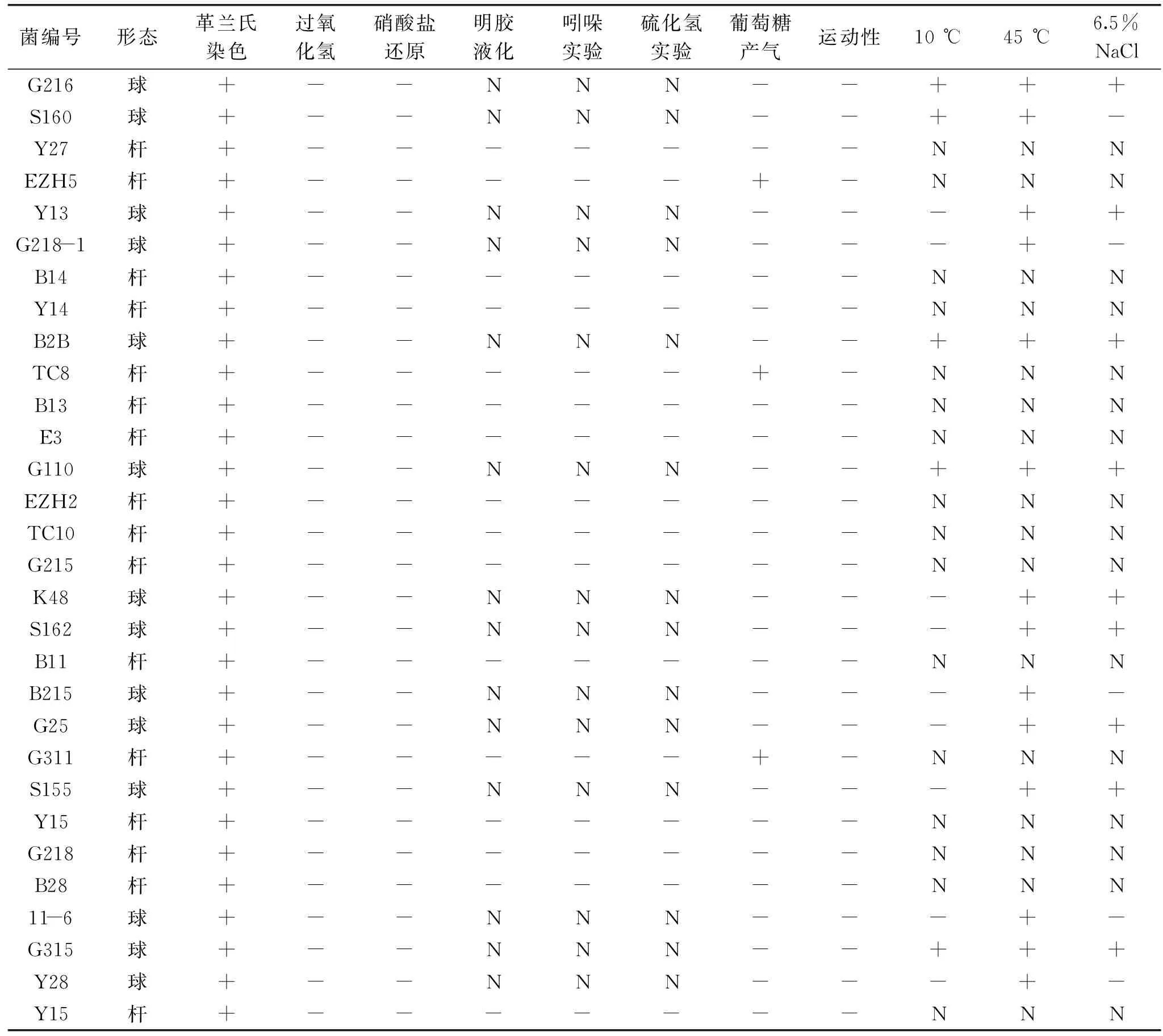
表1 生理生化鉴定结果Table 1 Result of identification physico-chemical properties
注:“+”代表阳性反应,“-”代表阴性反应,“N”代表未测定。)
1.3 数据处理分析
实验数据采用Excle 2007与origin 2016软件处理并制作图表。
2 结果与分析
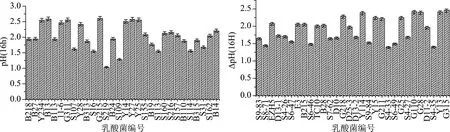
图1 培养16h 发酵液pH的变化Fig.1 The changing of fermentation broth pH after culture 16 hours
2.1 产酸快乳酸菌的筛选
如图1所示,培养16 h后,不同菌株的发酵液,ΔpH均增加1.0以上,说明大部分菌株都能产生有机酸,但产酸能力差异较大,产酸能力最弱的菌株为S219,ΔpH为1.04,产酸能力最强的菌株为B13,ΔpH为2.59。大多数菌株的ΔpH都集中在1.5~2.5。选取ΔpH>2.0的30株乳酸菌,近一步研究。
2.2 生理生化实验特征
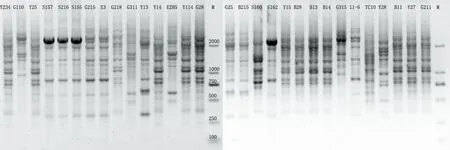
图3 rep-PCR产物电泳条带Fig.3 Electrophoresis banding of rep-PCR
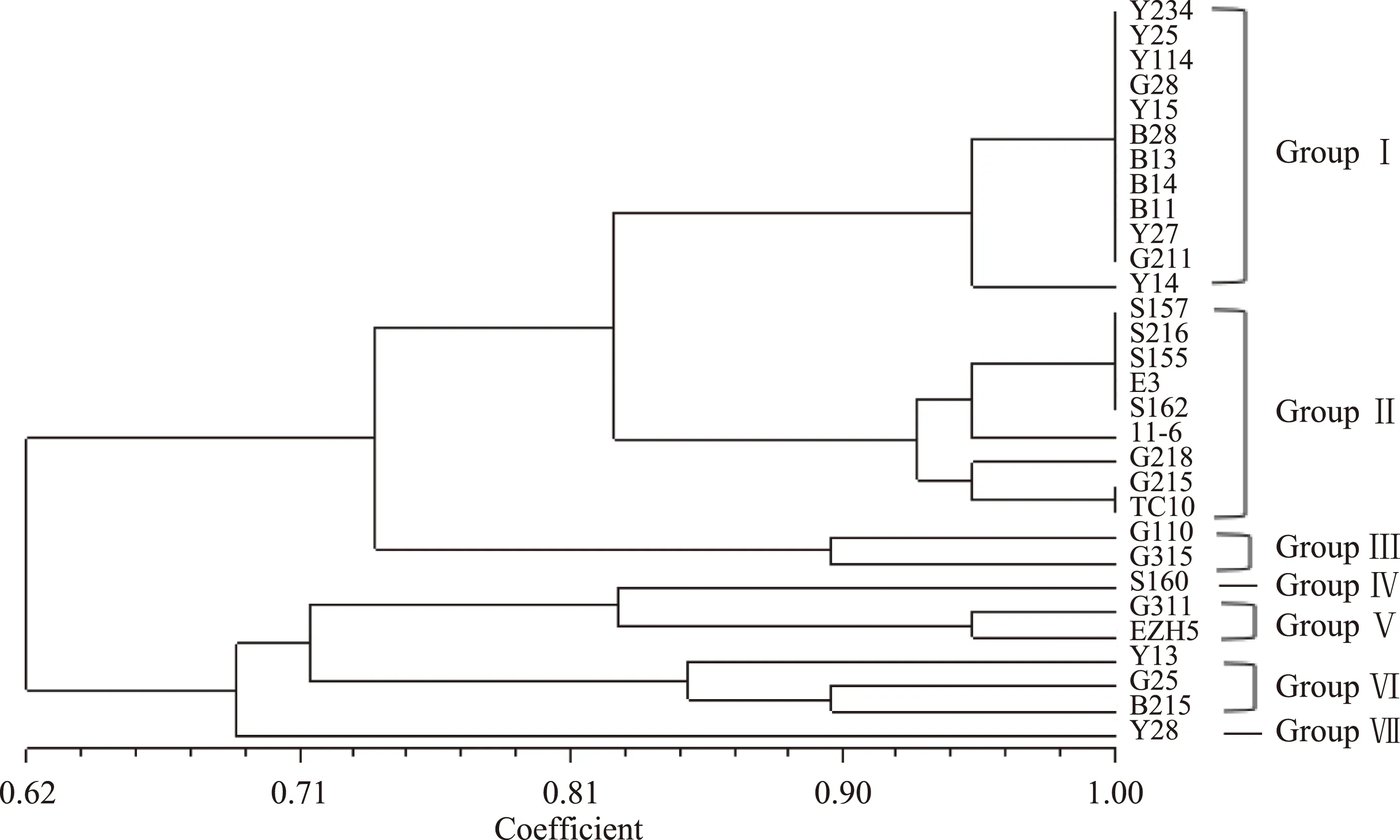
图4 基于rep-PCR指纹构建的相关性树状图Fig.4 Dendrogram representing relationships based on rep-PCR fingerprints
如图2所示,平板中的菌落多为表面光滑、边缘整齐、中央有凸起的白色或半透明状。杆菌显微形态为长杆状或短杆状,多数呈链状排列。球菌呈圆形或卵圆形,一般以单个、成链或成片排列。如表1所示,16株杆菌经过硝酸盐还原实验、明胶液化实验、吲哚实验、H2S实验,结果均为阴性,可初步鉴定为乳杆菌属;其余14株球菌的实验结果为:硝酸盐还原实验阴性、运动性实验阴性、6.5%NaCl生长实验阳性、10 ℃、45 ℃生长实验阳性、葡萄糖产气实验阴性,初步确定为肠球菌属。
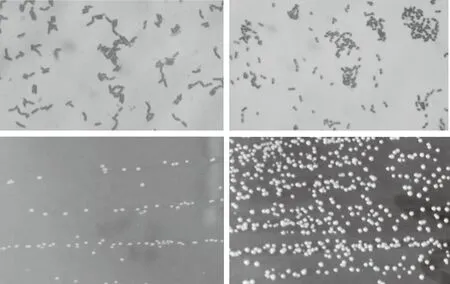
图2 乳酸菌显微形态与菌落形态Fig.2 Microscopic morphology and colony morphology of lactic acid bacteri
2.3 分子生物学鉴定
2.3.1 指纹图谱相关性分析 图谱共产生7种不同类型的可重复条带(图3),产生条带最大为2000bp左右,最短长度为200 bp左右。实验结果可以根据菌株的相关性树状图(图4)被分为7组,第一组为Y234、Y25、Y114、G28、Y15、B28、B13、B14、B11、Y27、G211与Y14,第二组为S157、S216、S155、E3、S162、11-6、G218、G215、TC10;第三组为G110、G315;第四组为S160;第五组为G311、EZH5;第六组为Y13、G25、B215第七组为Y28,其相关系数均在85%~100%之间。
2.3.2 16S rDNA PCR扩增 经过DNA指纹图谱聚类分析将30株乳酸菌分为7类,每一类选出1株菌作为代表菌株进行16s rDNA扩增,产物经1%琼脂糖凝胶电泳检测,获得约450 bp的特异性扩增条带(图5),符合测序要求。
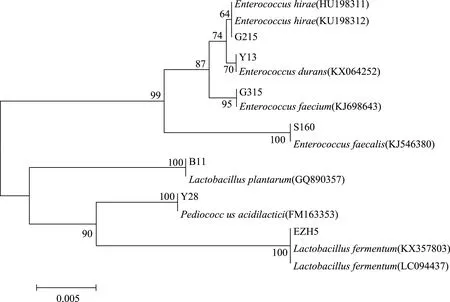
图6 代表菌株与标准菌株16S rDNA基因序列建立的系统发育树Fig.6 Phylogenetic tree establishment of representative Lactobacillus and standard strain 16S rDNA gene sequences
图5 PCR扩增的16S r DNA序列琼脂糖凝胶电泳图Fig.5 PCR amplification agarose gel electrophoresis of 16S rDNA partial sequenc
2.3.3 系统发育树的构建和乳酸菌的鉴定 从图5可以看出菌株G215与标准菌株Enterococcushirae处于同一分支,且相似性为100%,故将其鉴定为Enterococcushirae。菌株Y13与标准菌株Enterococcusdurans处于同一分支,且相似性为99%,因此可将其归为Enterococcusdurans。菌株G315与标准菌株Enterococcusfaecium处于同一分支,且相似性为99%,故将其鉴定为Enterococcusfaecium。

表2 产酸快乳酸菌的16S rRNA基因序列分析结果Table 2 Analysis results of 16S rRNA gene sequences from rapid acidification actic acid bacteria
菌株S160与标准菌株Enterococcusfaecalis处于同一分支,且相似性为100%,故将其鉴定为Enterococcusfaecalis。菌株B11与标准菌株Lactobacillusplantarum处于同一分支,且相似性为99%,因此可将其归为Lactobacillusplantarum。菌株Y28与标准菌株Pediococcusacidilactici处于同一分支,且相似性为100%,故将其鉴定为Pediococcusacidilactici。菌株EZH5与标准菌株Lactobacillusfermentum处于同一分支,且相似性为99%,故将其鉴定为Lactobacillusfermentum。
2.4 产酸快乳酸菌的多样性分析
由表2可知,新疆传统酸奶中产酸快的乳酸菌主要包括乳杆菌属(Lactobacillus)、肠球菌属(Enterococcus)和片球菌属(Pediococcus),在筛选出的30株产酸快的乳酸菌中,乳杆菌属占46.7%,其中植物乳杆菌(Lb.plantarum)12株,发酵乳杆菌(Lb.fermentim)2株。肠球菌属占50%,包括屎肠球菌(E.faecium)2株、粪肠球菌(E.faecalis)1株、耐久肠球菌(E.durans)3株和海氏肠球菌(E.hirae)9株。片球菌属仅包含乳酸片球菌(P.acidilactici),所占比例最低为3.3%。
2.5 牛乳发酵实验
如图7所示,7种乳酸菌在牛乳中均能正常生长,由于种子液的活力较强,从发酵第0 h时,牛乳的pH开始急剧的下降,发酵16 h后牛乳的pH由6.55降低至4.7以下,形成凝乳但不同的菌株在牛乳中的产酸能力有一定的差异,发酵16 h后,接种植物乳杆菌B11的牛乳pH最低为4.21,其次是接种海氏肠球菌G215的牛乳,pH为4.28,接种发酵乳杆菌EZH5、粪肠球菌S160、乳酸片球菌Y28、耐久肠球菌Y13的牛乳pH分别为4.5、4.6、4.63、4.65,接种屎肠球菌G315 的牛乳pH最高为4.70。
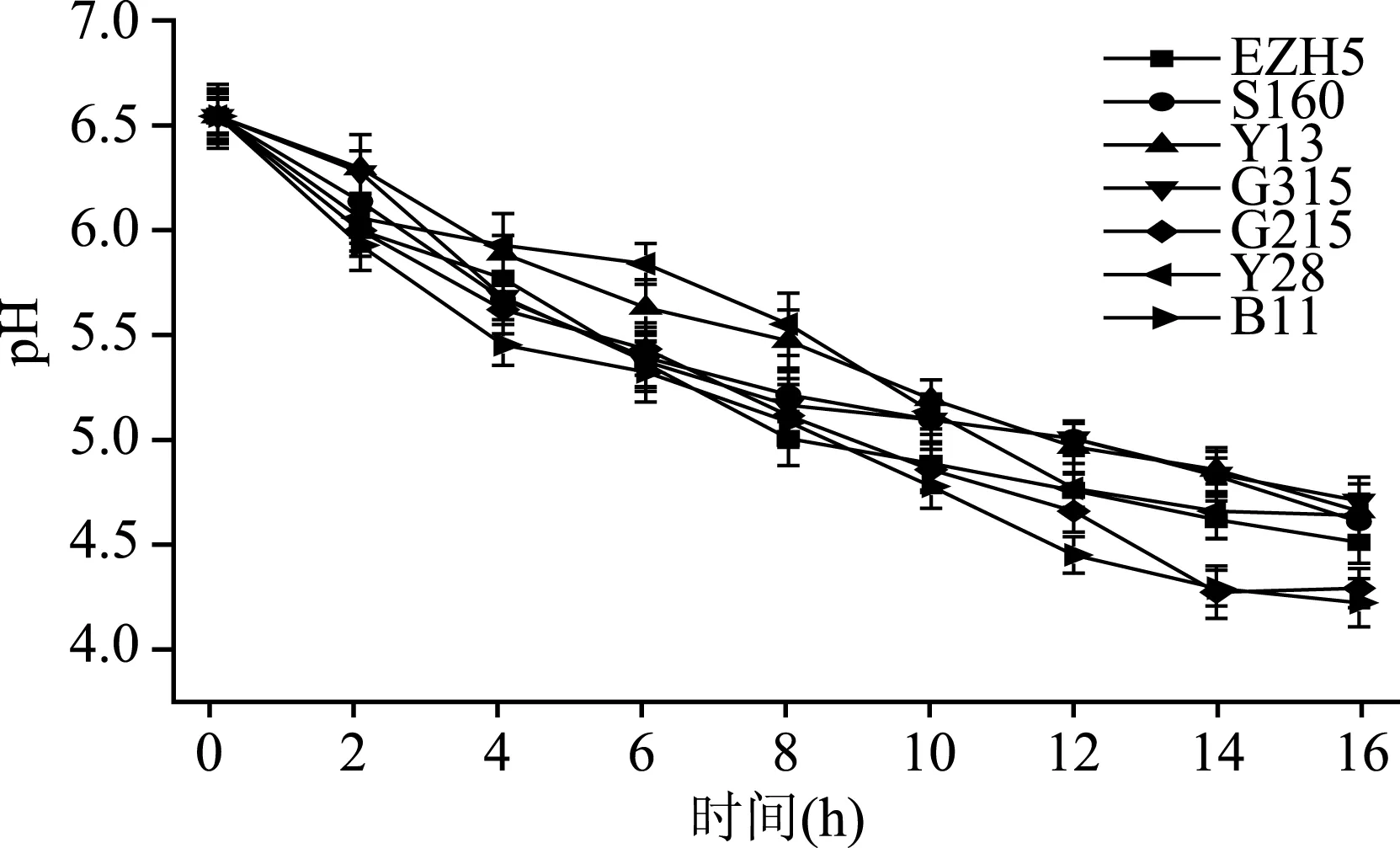
图7 不同乳酸菌在牛乳中的产酸能力Fig.7 Different lactic acid bacteria acid producing ability in milk
2.6 不同乳酸菌的自溶度与蛋白酶活性
如图8所示,乳酸菌的自溶度和蛋白酶活性与菌株的种类有很大的关系。发酵乳杆菌的自溶度最高为28.5%,其次为乳酸片球菌与植物乳杆菌分别为18.3%与12.4%,海氏肠球菌、耐久肠球菌、屎肠球菌、粪肠球菌的自溶度均低于10%,其中耐久肠球菌的自溶度最低为3.8%,几乎不发生自溶。植物乳杆菌的蛋白酶活性最高为13.71 U,其次是耐久肠球菌和屎肠球菌,其蛋白酶活性分别为7.16 U与7.07 U,海氏肠球菌与发酵乳杆菌的蛋白酶活性分别为5.41 U和5.33 U,粪肠球菌的蛋白酶活性为4.63 U,乳酸片球菌的蛋白酶活性最低为3.40 U。
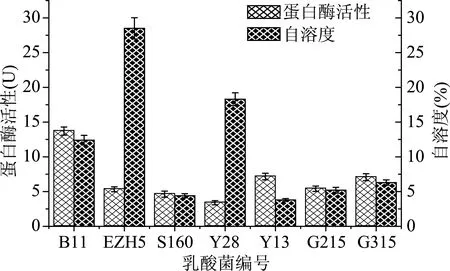
图8 不同乳酸菌的蛋白酶活性与自溶度Fig.8 The protease activity and autolysis of different lactic acid bacteria
3 结论与讨论
新疆传统酸奶是一个复杂的微生物体系,其中的乳酸菌资源具有无可比拟的生物多样性和基因多样性[23]。研究表明,传统酸奶在发酵过程中起主要作用的乳酸菌属主要有乳杆菌属、链球菌属、明串珠菌属、肠球菌属与乳球菌属[24]。袁雪林等[25]通过传统的方法分离了新疆喀什地区传统发酵酸乳中的乳酸菌,共56株7个种,分别为德氏乳杆菌、发酵乳杆菌、瑞士乳杆菌、嗜酸乳杆菌、鸡乳杆菌、屎肠球菌与耐久肠球菌,其优势菌株为德氏乳杆菌。呼斯楞等[26]对内蒙古呼伦贝尔地区传统发酵乳制品中乳酸菌的生物多样性进行了研究,分离出的24株乳酸菌分别属于乳球菌属、肠球菌属、乳杆菌属、链球菌属、明串株菌属。本实验筛选出的30株乳酸菌通过DNA指纹图谱与序列分析共鉴定出3个属7个种的快速产酸乳酸菌。其中植物乳杆菌、耐久肠球菌、发酵乳杆菌、粪肠球菌、屎肠球菌与前人的研究相一致。此外,本实验未检测出德氏乳杆菌与嗜热链球菌但检测出了海氏肠球菌与乳酸片球菌,这可能与传统酸奶的加工环境与地理位置有关。
在发酵乳制品生产中,乳酸菌除了对健康有益外,还具有酸化、形成特有组织结构和风味物质等功能[27]。不同乳酸菌组成的发酵剂的酸化能力、自溶度以及蛋白酶活性等发酵特性各不相同,Hebert[28]提出,快速产酸乳酸菌应在16 h内将牛乳pH降低至酪蛋白等电点并凝乳。本实验筛选出的7种乳酸菌在牛乳中发酵的凝乳时间均小于16 h,属于快速产酸乳酸菌,但不同种乳酸菌的蛋白酶活性与自溶度相差很大。实验发现,植物乳杆菌的产酸速度最快且蛋白酶活性最高而乳酸片球菌的产酸速度最慢且蛋白酶活性最低,可能是在发酵牛乳过程中蛋白酶能够将牛乳中大分子蛋白质分解成小分子的多肽,有利于乳酸菌的生长。
本实验通过微生物生理生化实验与DNA指纹图谱技术筛选出能快速产酸的30株7个种的乳酸菌,发现在传统酸奶的制作过程中植物乳杆菌与海氏肠球菌为优势菌种。乳酸菌株之间的特异性决定了其发酵特性,复合菌株发酵对发酵乳制品的品质可能有一定的促进作用。
[1]董晓婉,李宝坤,李开雄,等. 新疆蒙古族和哈萨克族传统乳制品中乳酸菌多样性的比较[J]. 食品工业科技,2013,21(59):162-166.
[2]Xiao P,Huang Y,Yang W,et al. Screening lactic acid bacteria with high yielding-acid capacity from pickled tea for their potential uses of inoculating to ferment tea products[J]. J Food Sci Technol,2015,52(10):6727-6734.
[3]赵婧,李慧,张玉玉,等. 高产酸乳酸菌的筛选、鉴定和生长特性研究[J]. 食品工业科技,2013,34(3):173-176.
[4]Tetsuya Masuda,Ayako Hidaka,Naoko Kondo,et al. Intracellular Enzyme Activities and Autolytic Properties of Lactobacillus Acidophilus and Lactobacillus Gasseri[J]. Food Sci. Technol,2005,11(3):328-331.
[5]张爽,张兰威,韩雪. 乳酸菌蛋白酶与发酵乳制品质量相关性研究进展[J]. 微生物学报,2015,55(12):1530-1536.
[6]孙卓,吕加平,张佳程,等. 乳酸菌自溶对切达干酪成熟中蛋白质分解的影响[J]. 中国乳品工业,2010,38(12):12-14.
[7]Hannon J A,Wilkinson M G,Delahunty C M,et al. Use of autolytic starter systems to accelerate the ripening of Cheddar cheese[J]. 2003,13(4):313-323.
[8]孙洁,吕加平,刘鹭,等. N+注入诱变高自溶度的乳酸菌突变株[J]. 核农学报,2010,24(4):684-688.
[9]何捷,曾小群,吕鸣春,等. 新疆酸奶中高产蛋白酶与产脂肪酶乳酸菌的筛选[J]. 食品科学,2015,36(17):130-133.
[10]王刚,田丰伟,刘小鸣,等. 2株具有优良体外抗氧化能力乳酸菌的筛选与鉴定[J]. 食品工业科技,2013,34(15):149-153.
[11]张旭,赵斌,张香美,等. 产细菌素乳酸菌的筛选及细菌素相关基因的分析[J]. 中国农业大学学报 2013,18(4):168-177.
[12]Dombek P E. Use of repetitive DNA sequences and the pcr to differentiate Escherichia coli isolates from human and animal sources[J]. Applied and Environmental Microbiology,2000,66(6):2572-2577.
[13]Ishii S,Sadowsky M J. Applications of the rep-PCR DNA fingerprinting technique to study microbial diversity,ecology and evolution[J]. Environ Microbiol,2009,11(4):733-740.
[14]Dirk Gevers. Applicability of rep-PCR fingerprinting for identification of Lactobacillus species[J]. FEMS Microbiology Letters,2001,205(1):31-36.
[15]谢静,熊善柏,曾令彬,等. 分离自腊鱼的乳酸菌生长及产酸特性[J]. 食品科学,2012,33(11):147-150.
[16]凌代文. 乳酸菌分类鉴定及实验方法[M]. 北京:中国轻工业出版社,1999:1-25.
[17]东秀珠,蔡妙英. 常见细菌系统鉴定手册[M]. 北京:科学出版社,2001:289-294.
[18]Versalovic J,Schneider M,De Bruijin F J,et al. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction[J].Methods in Molecular and Cellular Biology,1994,5(1):25-40.
[19]Waltenbury,D.R,L. Leduc,G. Ferrooni. The use of RAPD genomic fingerprinting to study reatedness in strains of Acidithiobacillus ferrooxidans[J]. Journal of micoobiological methods,2005,62(1):103-112.
[20]Pelt-Verkuil,Elizabeth van,Belkum,et al. Principles and Technical Aspects of PCR Amplification[M]. Springer Netherlands,2008:63-90.
[21]Ken-ji Yokoia b,Ken-Ichi Kawasakib,Akira Taketoc,et al.Characterization of lytic activities of Latobacillus gasseri. with special reference to autolysis[J]. Int J Food Microbiol,2004,96(03):273-279.
[22]卢燕云,林建国,李明. 复合诱变选育酸性蛋白酶高产菌株[J]. 中国酿造,2009,202(1):49-51.
[23]Cludia N,Manuel A C,Jorge S,et al. Study of the volatile components of a candied plum and estimation of their contribution to the aroma[J]. Food Chemistry,2008,111(4):897-905.
[24]Beukes,Elisabeth M. Bester,Bernie H. Mostert,et al.The microbiology of South African traditional fermented milks[J]. International Journal of Food Microbiology,2001,63(3):189-197.
[25]袁雪林,杨洁,胡敏,等. 新疆喀什地区传统发酵酸乳中乳酸菌多样性的初步分析[J]. 食品工业科技,2015,36(10):202-204.
[26]呼斯楞,刘红新,于洁,等. 内蒙古呼伦贝尔地区传统发酵乳中乳酸菌的多样性分析[J]. 微生物学通报,2016,43(5):984-990.
[27]孟祥晨. 乳酸菌与乳品发酵[M]. 北京:科学出版社,2009:350-360.
[28]Hebert E M,Raya R R,Tailliez P,et al. Characterization of natural isolates of Lactobacillus strains to be used of Lactobacillus strains to be used as starter cultures in dairy fermentation[J]. International Journal of Food Microbiology,2000,59(2):19-27.
Analysis of the diversity and fermentation characteristics of lactic acid bacteria in traditional yogurts from TaCheng,Xinjiang
JIANG Ai-ting,LI Bao-kun*,JIN Dan,QIAO Chuan-li,YANG Jie,ZHAO Li-li
(College of Food Engineering,Shihezi University,Shihezi 832000,China)
In this experiment,30 lactic acid bacterias with fast acid-producting capacity were screened from the traditional yogurt of Tacheng in Xinjiang by observing the pH of the fermentation broth and the discoloration time of bromocresol purple plate. Identified them in different ways,for example,morphological characteristics,physiological and biochemical identification,rep-PCR genotyping,16S rDNA gene sequence analysis and so on,and furtherly analysised the relationship between the acid production characteristics and protease activity and autolysis rates of different kinds of lactic acid bacteria. The results showed that there were 12 strains ofLactobacillusplantarum,9 strains ofEnterococcushirae,3 strains ofEnterococcusdurans,2 strains ofLactobacillusfermentumandEnterococcusfaeciumrespectively,1 strain ofPediococcusacidilacticiandEnterococcusfaecalisrespectively. It was worth mentioning that all of 7 kinds of lactic acid bacteria could normally grow in the milk and reduce the pH of milk,the protease activity was between 3 and 14 U,the autolysis rates was between 3% and 29%,and there was a close relationship between the fermentation characteristics and the species of strains.
Lactic acid bacteria;16s rDNA;yogurt;finger printing;fermentation characteristics
2017-02-21
蒋艾廷(1993-),男,硕士研究生,研究方向:乳制品加工与安全,E-mail:jiangaitingw@163.com。
*通讯作者:李宝坤(1979-),男,博士,副教授,研究方向:畜产品加工与安全,E-mail:libaokun1998@163.com。
国家自然科学基金地区项目(31560444);石河子大学重点科技攻关(gxjs2014-2dggo7)。
TS201.3
A
1002-0306(2017)15-0122-07
10.13386/j.issn1002-0306.2017.15.024
