亚科棒花鱼属鱼类的分类与系统发育关系
2017-08-16李玉火李明月傅萃长
何 欢 李玉火 曹 恺 李明月 傅萃长
(复旦大学生命科学院生物多样性科学研究所, 生物多样性与生态工程教育部重点实验室, 上海 200438)
亚科棒花鱼属鱼类的分类与系统发育关系
何 欢 李玉火 曹 恺 李明月 傅萃长
(复旦大学生命科学院生物多样性科学研究所, 生物多样性与生态工程教育部重点实验室, 上海 200438)
逗亚科棒花鱼属鱼类的分类与系统发育关系尚未解决。研究选择线粒体Cyt b基因和核基因RAG1、MLH3及MSH6作为分子标记, 重建了棒花鱼属鱼类的系统发育关系, 并结合形态证据对该属鱼类进行了分类厘定。结果表明: 棒花鱼与钝吻棒花鱼是单系种。拉林棒花鱼与辽宁棒花鱼是并系种, 一起形成单系群。拉林棒花鱼+辽宁棒花鱼一起嵌套在小鳔逗属鱼类内部, 钝吻棒花鱼嵌套在片唇逗属鱼类内部。拉林棒花鱼+辽宁棒花鱼+钝吻棒花鱼+片唇逗属+胡逗属+小鳔逗属+琵琶逗属鱼类组成单系群, 它们一起与棒花鱼形成姊妹群关系。可量性状与可数性状均不能区分拉林棒花鱼和辽宁棒花鱼。结合系统发育关系与形态证据,棒花鱼属鱼类的分类厘定如下: 棒花鱼属鱼类包括棒花鱼和平江棒花鱼(未取样); 辽宁棒花鱼是拉林棒花鱼的同物异名, 后者的分类位置有待进一步校订; 钝吻棒花鱼校订为钝吻片唇逗。
鲤科; 逗亚科; 棒花鱼属; 片唇逗属; 小鳔逗属; 系统发育
依据Tang等[1]建立的鲤科(Cyprinidae)逗亚科(Gobioninae)鱼类分类系统, Jiang和Zhang[2]进一步把逗族(Gobionini)划分为逗亚族(Gobionina)、鳅鰥亚族(Gobiobotiina)与似刺蛇逗亚族(Armatogobionina)。其中, 似刺蛇逗亚族包括7个属[2]: 似逗属(Pseudogobio)、蛇逗属(Saurogobio)、棒花鱼属(Abbottina)、胡逗属(Huigobio)、片唇逗属(Platysmacheilus)、琵琶逗属(Biwia)与小鳔逗属(Microphysogobio)。先前放在逗族的突吻逗属(Rostrogobio)是小鳔逗属的同物异名[1]。似刺蛇逗亚族鱼类中, 棒花鱼属鱼类的系统发育关系以及分类争议尚未很好地解决。
棒花鱼属包括5种鱼类[3]: 棒花鱼[Abbottina rivularis (Basilewsky, 1855)], 模式地是中国北方,广泛分布于东亚地区各主要水系; 钝吻棒花鱼[Abbottina obtusirostris(Wu & Wang, 1931)], 模式地是四川成都, 分布于长江上游水系; 辽宁棒花鱼(Abbottina liaoningensis Qin, 1987), 模式地是辽宁营口、丹东, 分布于辽河与鸭绿江水系; 拉林棒花鱼(Abbottina lalinensis Huang & Li, 1987), 模式地是黑龙江省五常市, 分布于黑龙江流域松花江水系;平江棒花鱼(Abbottina binhi Nguyen, 2001), 模式地越南平江河(流入我国称为水口河), 分布于珠江流域西江水系左江。
棒花鱼属鱼类的分类争议主要包括2方面: (1)拉林棒花鱼和辽宁棒花鱼是否为同物异名。乐佩琦[4]描述棒花鱼属鱼类时没有提及拉林棒花鱼。解玉浩[5]把拉林棒花鱼作为辽宁棒花鱼的同物异名, 但并没有提供校订依据。(2)钝吻棒花鱼的分类归属有待进一步证实。Bănărescu和Nalbant[6,7]与Bănărescu[8]将钝吻棒花鱼放在小鳔逗属, 而罗云林等[9]与乐佩琦[4]将钝吻棒花鱼放在棒花鱼属。
基于头部侧线与骨骼特征的逗亚科鱼类系统发育关系研究, Hosoya[10]认为棒花鱼属是似逗属的姐妹群。俞利荣和乐佩琦[11]使用外部与骨骼特征对似逗类(=似刺蛇逗亚族)鱼类进行了系统发育关系研究, 结果表明棒花鱼属是琵琶逗属+胡逗属+片唇逗+小鳔逗属+突吻逗属鱼类的姊妹群, 它们一起与蛇逗属鱼类形成姊妹群关系。Yang等[12]与Liu等[13]使用线粒体细胞色素b基因对逗亚科鱼类进行了系统发育关系研究, 揭示棒花鱼属和蛇逗属是姊妹群关系, 它们一起与似逗属+胡逗属+突吻逗属+片唇逗属+小鳔逗属+琵琶逗属鱼类形成姊妹群关系。Tang等[1]使用线粒体细胞色素b与细胞色素氧化酶COⅠ基因和核基因Rag1与Rhodopsin对逗亚科鱼类进行了系统发育关系研究,他们的研究发现棒花鱼属鱼类是琵琶逗属+胡逗属+片唇逗属+小鳔逗属鱼类的姊妹群, 它们一起与蛇逗属鱼类形成姊妹群关系。上述基于形态证据与分子证据的逗亚科鱼类的系统发育关系研究只探讨了棒花鱼属鱼类的系统发育位置, 没有涉及棒花鱼属鱼类种间的系统发育关系。
本研究选择线粒体细胞色素b基因(Cyt b), 核基因重组激活基因1(RAG1)、错配修复蛋白MutL基因3(MLH3)与错配修复蛋白MutS基因6(MSH6)作为分子标记, 重建了棒花鱼属鱼类种间的系统发育关系, 并结合形态证据对该属鱼类进行了分类厘定。
1 材料与方法
1.1 物种取样和分子标记
本研究采集了棒花鱼属鱼类4种31尾(表 1), 包括黑龙江、辽河、海河、黄河、长江和珠江水系的棒花鱼13尾; 黑龙江水系的拉林棒花鱼4尾; 鸭绿江和辽河水系的辽宁棒花鱼7尾; 长江上游水系的钝吻棒花鱼7尾。同时, 还包括了胡逗属、片唇逗属、琵琶逗属、小鳔逗属、蛇逗与似逗属鱼类15种23尾(表 1)。选择了2种逗属鱼类作为外类群。分子标记选择线粒体基因Cyt b和核基因RAG1、MLH3及MSH6。

表 1 种名、采样地点、个体编号及GenBank登录号Tab.1 Species, sampling sites, code and GenBank accession number of all individuals
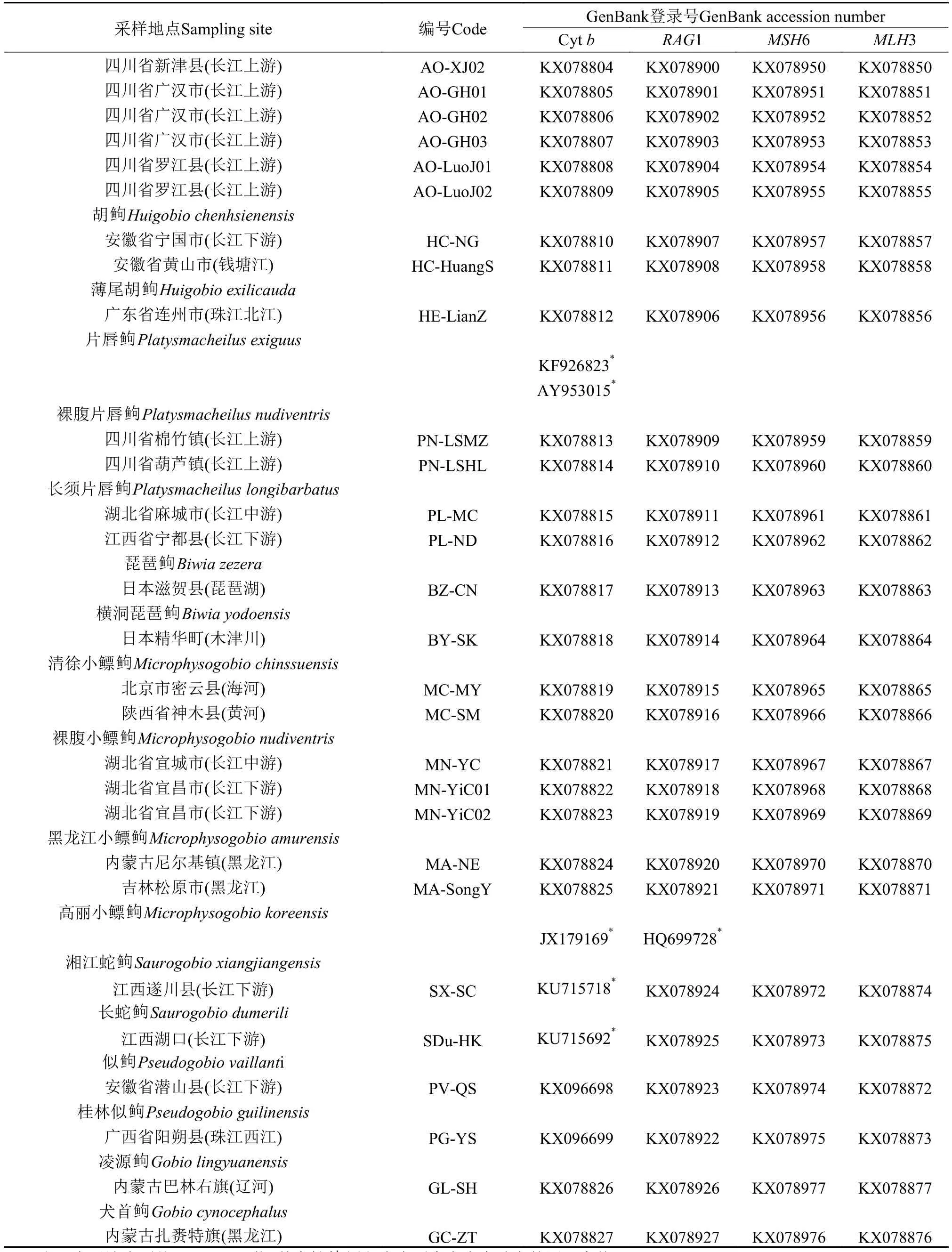
续表1
1.2 DNA提取、PCR扩增与测序
采用高盐法[14]从95%酒精保存的肌肉组织中提取全基因组DNA。PCR扩增引物[15,16]见表 2, 反应条件如下: 94℃预变性5min; 94℃变性50s, 52℃退火60s或者50s, 72℃延伸70s或者60s; 循环40或者35次; 72℃最后延伸8min。PCR产物纯化后在ABI 3730 DNA测序仪中使用扩增引物进行测序。
1.3 序列对位与系统发育关系重建
基因的序列对位在DAMBE ver.5.6.7软件[17]中完成, 包括3个步骤: (1)将核苷酸序列翻译成氨基酸序列; (2)使用Clustal W算法对位氨基酸序列[18]; (3)将对位好的氨基酸序列转换为核苷酸序列。序列对位后排除插入或缺失区域。基因的分子特征计算在MEGA ver.6.06软件[19]中完成。采用线粒体基因数据集、核基因多位点数据集、线粒体与核基因联合数据集进行系统发育关系的重建。按基因进行分区, 基于赤池信息准则(AIC)选择最适碱基替换模型, 在jModeltest ver.2.1.7软件[20]中完成。使用Mrbayes ver.3.2.2软件[21]进行贝叶斯分析。基于马尔可夫链蒙特卡罗(Markov chain Monte Carlo, MCMC)进行2000万代运算, 取样频率为1000, 保证分离频率平均标准偏差小于0.01。运行完成后舍弃前1/4的取样树, 根据剩余的3/4取样树构建50%的多数原则一致树, 并计算每个节点的后验概率值。使用RAxML ver.7.2.6软件[22]进行最大似然法分析。在GTRGAMMA模型下进行100次重复推算以搜索分值最高的最大似然树并进行1000次自展分析估计节点的支持度。
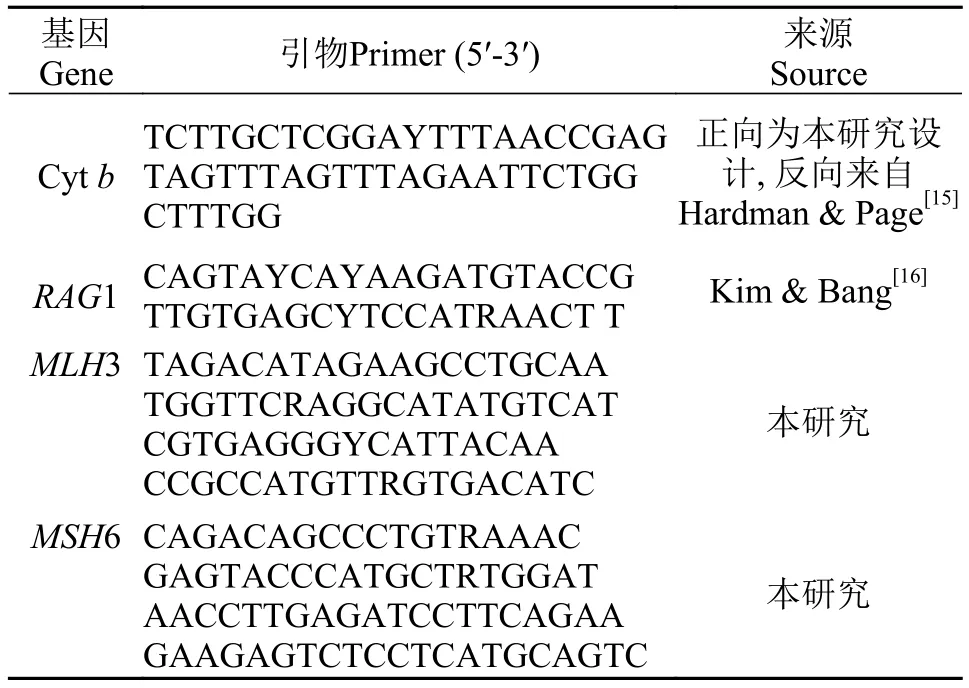
表 2 分子标记扩增使用的引物Tab.2 Primers for the amplification of molecular markers
使用*BEAST ver.2.3.0软件[23]对线粒体与核基因联合数据进行分析, 重建物种树。选择对数正态分布的松散钟模型作为分子钟模型, 树的先验模型设定为Yule过程。运行4个独立的重复, MCMC代数设置为15000万代, 取样频率为5000, 舍弃前1/5的样本后独立重复的log文档和trees文档在Log-Combiner ver.2.3.0软件[23]中合并。利用Tracer ver.1.5软件[24]估算合并后的log文档参数, 保证每个参数有效取样大小超过200。最后, 在TreeAnnotator ver.2.3.0软件[23]中生成最大谱系置信树。
1.4 形态特征的计数与测量
可数性状的计数依据陈宜瑜[25], 可量性状的测量依据Kottelat和Freyhof[26](图 1), 统一采用对鱼体背部俯视方向的左侧进行计数与测量。可量性状数据的主成分分析在Past ver.1.90软件[27]中完成。为消除异速生长的影响, 可量性状数据进行对数转换(Log-transformed)并使用Burnaby方法进行校正[28,29]。
2 结果
2.1 序列特征
本研究使用的Cyt b序列长度为1140 bp, 变异位点470个、简约信息位点451个; RAG1序列长度为1446 bp, 变异位点230个、简约信息位点175个; MLH3序列长度2139 bp, 变异位点558个、简约信息位点419个; MSH6序列长度2118 bp, 变异位点398个、简约信息位点305个。GenBank登录号信息见表 1。

图 1 可量性状示意图(参考Kottelat & Jorg[26])Fig.1 The diagram of morphometric characters (referring to Kottelat & Jorg[26])
2.2 棒花鱼属鱼类的系统发育关系
基于线粒体细胞色素b基因重建的系统发育树(图 2)和基于核基因多位点联合序列重建的系统发育树(图 3)结果均显示棒花鱼与钝吻棒花鱼是单系种。拉林棒花鱼与辽宁棒花鱼是并系种, 它们一起形成一单系群。基于线粒体细胞色素b基因与核基因多位点联合序列重建的系统发育树(图 4)与*BEAST物种树(图 5)树形一致。一般认为*BEAST物种树比基因多位点联合序列重建的系统发育树更能准确反映鱼类的系统发育关系[30—32]。因此, 以下仅描述*BEAST物种树揭示的结果。
*BEAST物种树(图 5)揭示的广义棒花鱼属鱼类系统发育关系为: 拉林棒花鱼和辽宁棒花鱼嵌套在小鳔逗属鱼类内部, 它们一起形成一单系群(贝叶斯后验概率100%); 钝吻棒花鱼嵌套在片唇逗属鱼类内部, 它们一起形成一单系群(贝叶斯后验概率93%), 钝吻棒花鱼与裸腹片唇逗形成姊妹群关系(贝叶斯后验概率100%); 拉林棒花鱼+辽宁棒花鱼+钝吻棒花鱼+片唇逗属+胡逗属+小鳔逗属+琵琶逗属鱼类一起形成一单系群(贝叶斯后验概率100%), 它们一起和棒花鱼形成姊妹群关系(贝叶斯后验概率100%)。
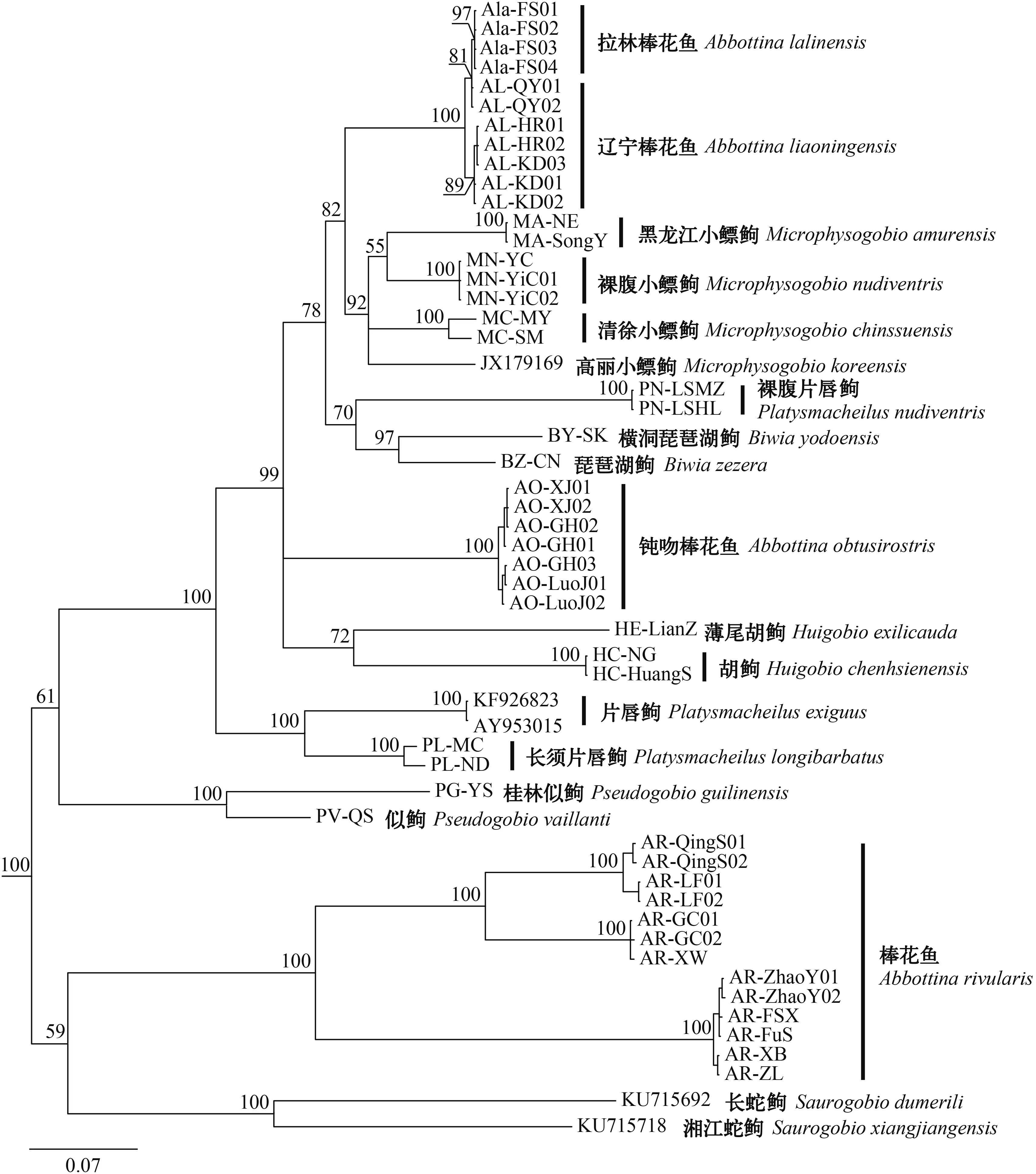
图 2 基于Cyt b序列重建的棒花鱼属鱼类贝叶斯系统发育树Fig.2 Bayesian tree of Abbottina fishes based on Cyt b sequence

图 3 基于核基因多位点联合序列重建的棒花鱼属鱼类贝叶斯系统发育树Fig.3 Bayesian tree of Abbottina fishes based on concatenated sequences of nuclear loci
2.3 辽宁棒花鱼与拉林棒花鱼的形态特征比较
辽宁棒花鱼与拉林棒花鱼的可数性状与可量性状见表 3。结果表明两种鱼类可数性状与可量性状的每一个形态特征均相互重叠, 没有不连续的分离。基于可量性状的主成分分析结果亦表明不支持把辽宁棒花鱼与拉林棒花鱼划分为两个形态群(图 6)。
3 讨论
物种的进化谱系概念认为种是系统发育树中单系群所代表的集合种群[33,34]。最近涉及物种界定的分类与系统发育关系研究广泛地采纳物种的进化谱系概念[35—37]。本研究基于线粒体与核基因多位点重建的系统发育树(图 2—4)显示拉林棒花鱼与辽宁棒花鱼一起形成单系群, 两者相互之间是并系关系。采纳物种的进化谱系概念, 本研究结果表明拉林棒花鱼与辽宁棒花鱼为同一物种, 进一步的形态特征分析支持它们是同种(表 3、图 6)。由于拉林棒花鱼新种发表于1987年4月[38], 辽宁棒花鱼新种发表于1987年8月[39]。基于动物命名法则的优先原则, 辽宁棒花鱼是拉林棒花鱼的同物异名。*Beast物种树(图 5)揭示拉林棒花鱼嵌套在小鳔逗属鱼类内部, 它们一起形成单系群。拉林棒花鱼的分类位置有待进一步全面的小鳔逗属鱼类系统发育关系研究后再确定。
Bănărescu和Nalbant[6,7]与Bănărescu[8]将钝吻棒花鱼放在小鳔逗属, 而罗云林等[9]和乐佩琦[4]将钝吻棒花鱼放在棒花鱼属。本研究重建的系统发育树(图 2—5)不支持他们各自的观点, 揭示了钝吻棒花鱼与裸腹片唇逗的姊妹群关系, 支持它与其他片唇逗属鱼类形成单系群。基于本研究的结果, 为了保持片唇逗属鱼类作为单系群, 钝吻棒花鱼应校订为钝吻片唇逗, 这将导致需要进一步的研究校订片唇逗属的属征。
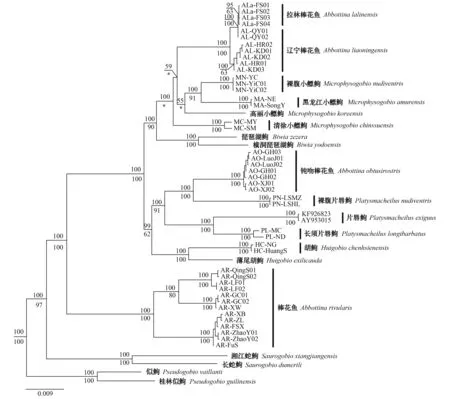
图 4 基于Cyt b和核基因多位点联合序列重建的棒花鱼属鱼类贝叶斯系统发育树Fig.4 Bayesian tree of Abbottina fishes based on concatenated sequences of Cyt b and nuclear loci
本研究重建的系统发育关系(图 2—5)表明片唇逗属+胡逗属+琵琶逗属+小鳔逗属鱼类一起是棒花鱼的姊妹群。这与先前使用形态证据[11]或者多位点分子证据[1]的研究结果一致。由于棒花鱼属的模式种是棒花鱼, 因此我们认为严格意义棒花鱼属鱼类包括棒花鱼与平江棒花鱼(未取样)。先前研究描述的3个外部形态特征能很好地区分严格意义棒花鱼属鱼类与它的姊妹群[4,11,40—42]: (1)“下唇侧叶在中叶前方是否相连”这个形态特征能区分严格意义棒花鱼属鱼类(下唇侧叶在中叶前方相连)与片唇逗属+胡逗属+琵琶逗属+小鳔逗属鱼类(下唇侧叶在中叶前方不相连); (2)“口角须是否存在”这个形态特征能区分严格意义棒花鱼属鱼类(无口角须)与琵琶逗属鱼类(有口角须); (3)“上下颌是否具角质边缘”这个形态特征能区分严格意义的棒花鱼属鱼类(上下颌不具角质边缘)与片唇逗属+胡逗属+小鳔逗属鱼类(上下颌具角质边缘)。拉林棒花鱼与钝吻棒花鱼具有“下唇侧叶在中叶前方不相连”以及“上下颌具角质边缘”两个形态特征, 表明它们不属于严格意义棒花鱼属鱼类。

表 3 辽宁棒花鱼与拉林棒花鱼的可数性状与可量性状Tab.3 Meristic and morphometric characters between Abbottina liaoningensis and A.lalinensis
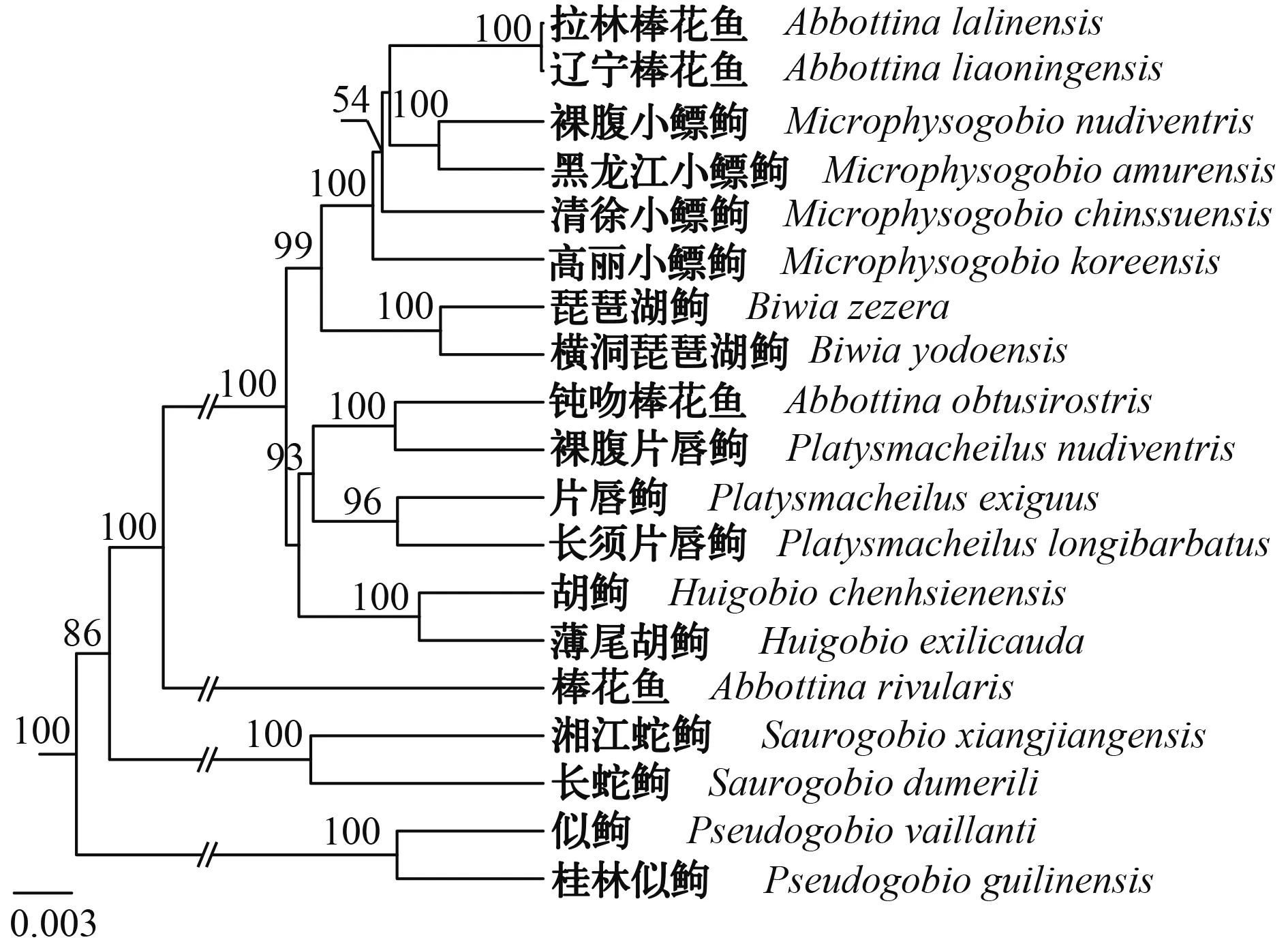
图 5 基于Cyt b和核基因多位点重建的棒花鱼属鱼类*BEAST物种树Fig.5 *BEAST species tree of Abbottina fishes based on Cyt b and nuclear loci

图 6 拉林棒花鱼和辽宁棒花鱼的第一与第二主成分得分散点图Fig.6 Scatter plots of scores on the first and second principal components of Abbottina lalinensis and A.liaoningensis
综上所述, 棒花鱼属鱼类包括棒花鱼和平江棒花鱼; 确认辽宁棒花鱼是拉林棒花鱼的同物异名,后者的分类位置有待进一步校订; 揭示钝吻棒花鱼应该归入片唇逗属, 并校订为钝吻片唇逗。本研究厘定了棒花鱼属鱼类的分类, 为后续进一步解析逗亚科逗族鱼类的系统发育关系与生物地理学研究奠定了基础。
[1]Tang K L, Agnew M K, Chen W J, et al.Phylogeny of the gudgeons (Teleostei: Cyprinidae: Gobioninae) [J].Molecular Phylogenetics and Evolution, 2011, 61(1): 103—124
[2]Jiang Z G, Zhang E.Molecular evidence for taxonomic status of the gudgeon genus Huigobio Fang, 1938 (Teleostei: Cypriniformes), with a description of a new species from Guangdong Province, South China [J].Zootaxa,2013, 3731(1): 171—182
[3]Eschmeyer W N, Fricke R, Van der Laan R.Catalog of Fishes: Genera, Species, References, http://researcharchive.calacademy.org/research/ichthyology/catalog/fishcatmain.asp, accessed 2016.04.15
[4]Yue P Q.Abbottina Jordan et Fowler, 1903 [A].In: Chen Y Y (Eds.), Fauna Sinica, Osteichthyes, Cypriniformes (II) [C].Beijing: Science Press.1998, 347—353 [乐佩琦.棒花鱼属.见: 陈宜瑜, 中国动物志硬骨鱼纲鲤形目(中卷).北京: 科学出版社.1998, 347—353]
[5]Xie Y H.Abbottina Jordan et Fowler, 1903 [A].In: Xie Y H (Eds.), Freshwater Fishes in Northeast Region of China [C].Shenyang: Liaoning Science and Technology Press.2007, 194—198 [解玉浩.棒花鱼属.见:解玉浩, 东北地区淡水鱼类.沈阳: 辽宁科学技术出版社.2007, 194—198]
[6]Bǎnǎrescu P, Nalbant T T.Some New Chinese Minnows (Pisces, Cypriniformes) [J].Proceedings of the Biological Society of Washington, 1968, 81: 335—342
[7]Bǎnǎrescu P, Nalbant T T.Pisces, Teleostei, Cyprinidae (Gobioninae) [M].Das Tierreich, Lieferung 93, Berlin: Walter de Guryter.1973, 1—304
[8]Bǎnǎrescu P.A critical updated checklist of Gobioninae (Pisces, Cyprinidae) [J].Travaux du Muséum National d’Histoire Naturelle “Grigore Antipa”, 1992, 32: 303—330
[9]Luo Y L, Yue P Q, Chen Y Y.Abbottina Jordan et Fowler, 1903 [A].In: Wu X W (Eds.), Fauna of Cyprinidae in China (II) [C].Shanghai: Shanghai People’s Publishing House.1977, 516—529 [罗云林, 乐佩琦, 陈宜瑜.棒花鱼属.见: 伍献文, 中国鲤科鱼类志(下卷).上海: 上海人民出版社.1977, 516—529]
[10]Hosoya K.Interrelationships of the Gobioninae (Cyprinidae) [A].In: Uyeno T, Arai T, Taniuchi T, et al.(Eds.), Indo-Pacific Fish Biology: Proceeding of the Second International Conference on Indo-Pacific Fishes [C].Tokyo: The Ichthyological Society of Japan.1986, 484—501
[11]Yu L R, Yue P Q.Studies on phylogeny of the Pseudogobiini fishes (Cypriniformes: Cyprinidae) [J].Acta Zootaxonomica Sinica, 1996, 21(2): 244—255 [俞利荣, 乐佩琦.似逗类鱼类的系统发育研究(鲤形目: 鲤科).动物分类学报, 1996, 21(2): 244—255]
[12]Yang J Q, He S P, Freyhof J, et al.The phylogenetic relationships of the Gobioninae (Teleostei: Cyprinidae) inferred from mitochondrial cytochrome b gene sequences [J].Hydrobiologia, 2006, 553(1): 255—266
[13]Liu H Z, Yang J Q, Tang Q Y.Estimated evolutionary tempo of East Asian gobionid fishes (Teleostei: Cyprinidae) from mitochondrial DNA sequence data [J].Hydrobiology, 2010, 55(15): 1502—1510
[14]Miller S A, Dykes D D, Polesky H F.A simple salting out procedure for extracting DNA from human nucleated cells [J].Nucleic Acids Research, 1988, 16(3): 12—15
[15]Hardman M, Page L M.Phylogenetic relationships among bullhead catfishes of the Genus Ameiurus (Siluriformes: Ictaluridae) [J].Copeia, 2003, 2003(1): 20—33
[16]Kim K Y, Bang I C.Molecular phylogenetic position of Abbottina springeri (Cypriniformes, Cyprinidae) based on nucleotide sequences of RAG1 gene [J].Korean Journal of Ichthyology, 2010, 22(4): 273—278
[17]Xia X H.DAMBE5: A comprehensive software package for data analysis in molecular biology and evolution [J].Molecular Biology and Evolution, 2013, 30(7): 1720—1728
[18]Thompson J D, Gibson T J, Plewniak F, et al.The Clustal_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J].Nucleic Acids Research, 1997, 25(25): 4876—4882
[19]Tamura K, Stecher G, Peterson D, et al.MEGA6: Molecular evolutionary genetics analysis version 6.0 [J].Molecular Biology and Evolution, 2013, 30(12): 2725—2729
[20]Darriba D, Taboada G L, Doallo R, et al.jModelTest 2: more models, new heuristics and parallel computing [J].Nature Methods, 2012, 9(9): 772
[21]Ronquist F, Teslenko M, Van der Mark P, et al.MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space [J].Systematic Biology, 2012, 61(3): 539—542
[22]Stamatakis A.RAXML-VI-HPC: maximum likelihoodbased phylogenetic analyses with thousands of taxa and mixed models [J].Bioinformatics, 2006, 22(21): 2688—2690
[23]Bouckaert R, Heled J, Kuhnert D, et al.BEAST 2: A software platform for bayesian evolutionary analysis [J].PLoS Computational Biology, 2014, 10(4): 1—6
[24]Rambaut A, Drummond A.Tracer v1.5.2007.Available at: http://tree.bio.ed.ac.uk/software/tracer
[25]Chen Y Y.Fauna Sinica, Osteichthyes, Cypriniformes (II) [M].Beijing: Science Press.1998, 16—18 [陈宜瑜.中国动物志硬骨鱼纲鲤形目(中卷).北京: 科学出版社.1998, 16—18]
[26]Kottelat M, Freyhof J.Handbook of European freshwater fishes [M].Delémont: Imprimerie du Démocrate SA.2007, 15—16
[27]Hammer Ø, Harper D A T, Ryan P D.Past: Paleontological statistics software package for education and data analysis [J].Palaeontologia Electronica, 2001, 4(1): 9
[28]Burnaby T P.Growth-invariant discriminant functions and generalized distances [J].Biometrics, 1966, 22(1): 96—110
[29]Rolf F J, Bookstein F L.A comment on shearing as a method for ‘size correlation’ [J].Systematic Zoology, 1987, 36(4): 356—367
[30]Waters J M, Rowe D L, Burridge C P.Gene trees versus species trees: reassessing life-history evolution in a freshwater fish radiation [J].Systematic Biology, 2010, 59(5):504—517
[31]Fu C Z, Guo L, Xia R.A multilocus phylogeny of Asian noodlefishes Salangidae (Teleostei: Osmeriformes) with a revised classification of the family [J].Molecular Phylogenetics and Evolution, 2012, 62(3): 848—855
[32]Xia R, Durand J D, Fu C Z.Multilocus resolution of Mugilidae phylogeny (Teleostei: Mugiliformes): Implications for the family’s taxonomy [J].Molecular Phylogenetics and Evolution, 2016, 96: 161—177
[33]De Queiroz K.Species concepts and species delimitation [J].Systematic Biology, 2007, 56(6): 879—886
[34]Wiens J J.Species delimitation: new approaches for discovering diversity [J].Systematic Biology, 2007, 56(6): 875—878
[35]Chen H, Polgar G, Fu C Z, et al.Cryptic species and evolutionary history of Boleophthalmus pectinirostris complex along the northwestern Pacific coast [J].Acta Hydrobiologica Sinica, 2014, 38(1): 75—86 [陈卉, Polgar G, 傅萃长, 等.西北太平洋海岸带大弹涂鱼复合体的隐存种与进化历史.水生生物学报, 2014, 38(1): 75—86]
[36]Hedin M.High-stakes species delimitation in eyeless cave spiders (Cicurina, Dictynidae, Araneae) from central Texas [J].Molecular Ecology, 2015, 24(2): 346—361
[37]Karanovic T, Djurakic M, Eberhard S M.Cryptic Species or Inadequate Taxonomy? Implementation of 2D Geometric Morphometrics Based on Integumental Organs as Landmarks for Delimitation and Description of Copepod Taxa [J].Systematic Biology, 2016, 65(2): 304—327
[38]Huang Z H, Li Z P.A new species of Abbottina: Abbottina lalinensis (Cyprinidae: Gobioninae) [J].Heilongjiang Fisheries, 1987, 1: 7—10 [黄智弘, 李再培.棒花鱼属一新种: 拉林棒花鱼(鲤科: 逗亚科).黑龙江水产, 1987, 1: 7—10]
[39]Qin K J.Abbottina Jordan et Fowler, 1903 [A].In: Liu C X, Qin K J (Eds.), Fauna Sinica (Pisces) [C].Shenyang: Liaoning Science and Technology Press.1987, 167—171 [秦克静.棒花鱼属.见: 刘蝉馨, 秦克静, 辽宁动物志(鱼类).沈阳: 辽宁科学技术出版社.1987, 167—171]
[40]Kim I, Yang H.A Revision of the Genus Microphysogobio in Korea with Description of a New Species (Cypriniformes, Cyprinidae) [J].Korean Journal of Ichthyology, 1999, 11(1): 1—11
[41]Nguyen V H, Ngo S V.Freshwater fishes of Viet Nam, Volume I, Family Cyprinidae [M].Vietnam: Nha Xuat Ban Nong Nghiep.2001, 1—622
[42]Kawase S, Hosoya K.Biwia yodoensis, a new species from the Lake Biwa / Yodo River Basin, Japan (Teleostei: Cyprinidae) [J].Ichthyological Exploration of Freshwaters, 2010, 21(1): 1—7
TAXONOMY AND PHYLOGENETIC RELATIONSHIPS OF THE GENUS ABBOTTINA FISHES IN THE SUBFAMILY GOBIONINAE
HE Huan, LI Yu-Huo, CAO Kai, LI Ming-Yue and FU Cui-Zhang
(Ministry of Education Key Laboratory for Biodiversity Science and Ecological Engineering, Institute of Biodiversity Science and School of Life science, Fudan University, Shanghai 200438, China)
Taxonomy and phylogeny of the genus Abbottina fishes in the subfamily Gobioninae (Cypriniformes: Cyprinidae) remain unresolved.The current study utilized mitochondrial gene (cytochrome b) and nuclear loci (RAG1, MLH3 and MSH6) as molecular markers to reconstruct phylogenetic relationships of Abbottina fishes, and revised their classification by merging morphological evidence.The results showed that Abbottina rivularis and A.obtusirostris were monophyletic species.Abbottina lalinensis and A.liaoningensis were paraphyletic species, which formed a monophyletic group.Abbottina lalinensis+A.liaoningensis were nested within Microphysogobio fishes, and Abbottina obtusirostris was nested within Platysmacheilus fishes.Abbottina lalinensis+A.liaoningensis+A.obtusirostris+Platysmacheilus+Huigobio+Microphysogobio+Biwia fishes formed a monophyletic group, which were a sister taxon of Abbottina rivularis.Meristic and morphometric characters did not separate Abbottina lalinensis from A.liaoningensis.The molecular evidence and morphological evidence demonstrated that the genus Abbottina fishes include Abbottina rivulari and A.binhi (un-sampling in this study), and that Abbottina liaoningensis was a junior synonym of A.lalinensis, which needs further revision; and Abbottina obtusirostris was revised as Platysmacheilus obtusirostris.
Cyprinidae; Gobioninae; Abbottina; Platysmacheilus; Microphysogobio; Phylogeny
Q959.4
A
1000-3207(2017)04-0843-10
10.7541/2017.105
2016-04-28;
2016-08-25
国家自然科学基金(31471982)资助 [Supported by the National Natural Science Foundation of China Science (31471982)]
何欢(1989—), 男, 四川德阳人; 硕士研究生; 主要从事鱼类生态与进化研究。E-mail: 13210700005@fudan.edu.cn
傅萃长, 教授, 博士生导师; E-mail: czfu@fudan.edu.cn
