生物信息学分析CysC特异性肽段及质谱验证
2017-07-07沈蕾王惠民季伙燕沈培王建新
沈蕾,王惠民,季伙燕,沈培,王建新
(南通大学附属医院检验科,江苏南通 226001)
·临床实验研究·
生物信息学分析CysC特异性肽段及质谱验证
沈蕾,王惠民,季伙燕,沈培,王建新
(南通大学附属医院检验科,江苏南通 226001)
目的 用生物信息学技术分析半胱氨酸蛋白酶抑制剂C(CysC)的特异性肽段及其性质,并用质谱技术验证预测结果。方法 用在线软件工具分析胰蛋白酶水解CysC产生的肽段和同源性,预测特异性肽段质谱扫描离子化碎片信息;对人工合成的特异性多肽样品进行母离子扫描和子离子扫描,人重组CysC和血清样品经胰蛋白酶水解后分析质谱,比较分析结果与预测结果。结果 选择T3(ALDFAVGEYNK)肽段作为CysC的特异性肽段,用串联质谱对T3样品[M+2H]2+进行子离子扫描,几乎可发现Skyline软件预测的所有b离子和y离子。人重组CysC和血清样品经胰蛋白酶水解出的肽段与T3*在同一时间出峰。结论 用生物信息学技术快速准确地筛选出了CysC特异性肽段并准确预测该肽段质谱扫描离子化碎片。
半胱氨酸蛋白酶抑制剂C;生物信息学技术;特异性肽段;质谱
血清半胱氨酸蛋白酶抑制剂C(Cystatin C,CysC)是半胱氨酸蛋白酶抑制剂家族Ⅱ的成员,由120个氨基酸组成,相对分子质量(Mr)为13 300,是一种低分子量的碱性非糖基化蛋白质,是反映肾小球滤过率变化的理想标志物[1],也与慢性肾损伤[2]、糖尿病性肾病[3-4]、恶性胃肠道肿瘤[5]等疾病的发生发展有关。临床常用检测方法为免疫比浊法,测量结果尚无法溯源,不同测量系统间结果没有可比性。同位素稀释液相色谱串联质谱技术(ID-LC-MS/MS)为蛋白质定量检测提供了新思路。用同位素标记肽段定量蛋白酶水解蛋白质产生的一个或多个特征肽段,其中如何获得专一的特异性肽段及其离子化信息是蛋白质质谱法准确定量的关键[6]。本研究用在线软件及数据库等生物信息学技术分析胰蛋白酶水解CysC产生的肽段及同源性,预测特异性肽段质谱扫描离子化碎片信息,并用质谱技术验证。
1 材料与方法
1.1 仪器与试剂 Triple Quad 5500三重四级杆质谱仪(美国AB Sciex公司);Nexera X2超高效液相色谱系统(日本株式会社岛津制作所);Allegra 64R高速冷冻离心机(美国Beckman Coulter公司);Milli-Q Direct-q 3UV超纯水一体机(德国Millipore公司);微量移液器(德国Eppendorf公司);人重组CysC(英国BBI Solutions公司);T3(ALDFAVGEYNK)和T3*(AL*DFAVGEYNK)肽段(上海强耀公司);甲醇、乙腈(美国J.T. Baker公司);甲酸、三氟乙酸(美国Fluka公司);碳酸氢铵、碘乙酰胺、二硫苏糖醇、ProteoPreBlue清蛋白及IgG去除试剂盒(美国Sigma公司);序列级胰蛋白酶(美国Promega公司)。
1.2 CysC特异性肽段生物信息学分析 通过ExPASy软件中PeptideMass和ProtParam工具预测CysC经胰蛋白酶水解后产生的肽段并分析肽段Mr、等电点、氨基酸、原子、摩尔吸光系数、半衰期、不稳定性系数、脂肪族氨基酸系数、平均亲水系数等理化性质,选择溶解性、稳定性较好,且满足本实验室质谱仪可检测质荷比小于1 250要求的肽段,用Pubmed 数据库中BlastP在线软件分析肽段同源性,用Skyline软件预测特异性肽段质谱扫描离子化碎片信息。
1.3 质谱验证
1.3.1 肽段水平验证 将人工合成的肽段溶于50%甲醇水溶液中,针泵恒流进样,Q1扫描可得到[M+H]+、[M+2H]2+、[M+3H]3+等准分子离子,再选择[M+2H]2+作为母离子二级扫描得到各个[M+H]+子离子。将质谱扫描结果与软件预测结果比对,观察二者一致性,验证多肽的序列。
1.3.2 蛋白质水平验证 前处理过程如下:(1)高丰度蛋白质去除及待测蛋白质变性:按照试剂盒说明书去除血清中高丰度蛋白质,加入70 μL碳酸氢铵溶液(25 mmol/L)、96.1 mg尿素使待测蛋白质变性,再加入250 μL碳酸氢铵溶液(25 mmol/L);(2)蛋白质的还原和烷基化:加入50 μL二硫苏糖醇(100 mmol/L)60 ℃水浴30 min;加入50 μL碘乙酰胺(550 mmol/L)室温避光30 min;再加入185 μL二硫苏糖醇(100 mmol/L)室温30 min以终止烷基化,加入250 μL碳酸氢铵溶液(25 mmol/L)以稀释;(3)胰蛋白酶水解:加入同位素标记肽段T3*,胰蛋白酶37 ℃水浴4 h,再加入5% TFA终止酶解反应;(4)萃取及上样:酶解产物经固相萃取柱萃取后氮气吹干,复溶于400 μL 0.1%甲酸-水溶液中,上样检测。对人重组CysC和血清前处理,用Waters Symmetry Shield RP18(2.1×150 mm,3.5 μm)色谱柱分离肽段。以0.01%甲酸水溶液作为流动相A,0.01%甲酸-乙腈作为流动相B,梯度洗脱,流速0.200 mL/min。选择[M+2H]2+二级扫描得到的[M+H]+中信号较高的子离子进行多反应监测(MRM),观察水解后肽段的分离及离子化效果,且是否与同位素标记T3*同时出峰。
2 结果
2.1 肽段生物信息学分析 预测胰蛋白酶水解CysC可获得10条肽段,按蛋白质氨基酸序列先后顺序分别命名为T1~T10,各肽段理化性质见表1。对符合要求的T3、T10肽段与Pubmed数据库中已知蛋白质比对分析,未发现人源性其他蛋白质中存在相同肽段,从而选择T3作为研究CysC的特异性肽段。
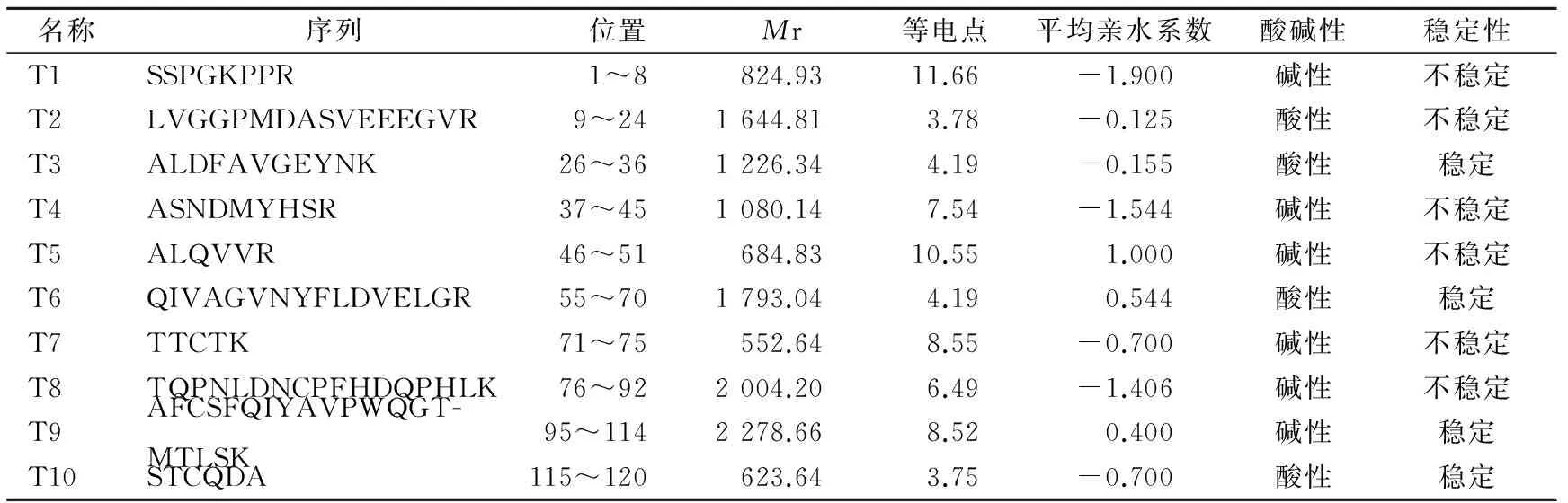
表1 10条胰蛋白酶水解CysC肽段的性质
用Skyline软件预测T3肽段的质谱扫描离子化碎片信息,[M+H]+、[M+2H]2+、[M+3H]3+的质荷比(m/z)分别为1 226.605 2、613.806 2、409.539 9。以[M+2H]2+(613.806 2)为准分子离子预测MS/MS, y+1~y+10结果分别为147.112 8、261.155 7、424.219 1、553.261 7、610.283 1、709.351 5、780.388 6、927.457 1、1 042.484 0、1 155.568 1,b+1~b+10结果为72.0444、185.128 5、300.155 4、447.223 8、518.260 9、617.329 3、674.350 8、803.393 4、966.456 7、1 080.499 7。
2.2 T3肽段样品质谱扫描结果 对T3肽段样品溶液一级扫描,可见[M+H]+、[M+2H]2+、[M+3H]3+准分子离子峰,质荷比分别为1 226.6、613.4、409.5,见图1。以[M+2H]2+为母离子,去簇电压(DP)设置为80 V,调节碰撞能量(CE)为25 V,通过MS/MS扫描得到碎片离子,y+1~y+9结果分别为147.1、261.4、424.3、553.3、610.4、709.4、780.4、927.6、1 042.5,b+2~b+10结果分别为185.1、300.1、447.2、518.3、617.4、674.3、803.4、966.4、1 080.6。CysC特异性肽段生物信息学预测结果与质谱扫描结果一致。除b+1和y+10外,二级扫描可发现所有b离子和y离子,见图2。
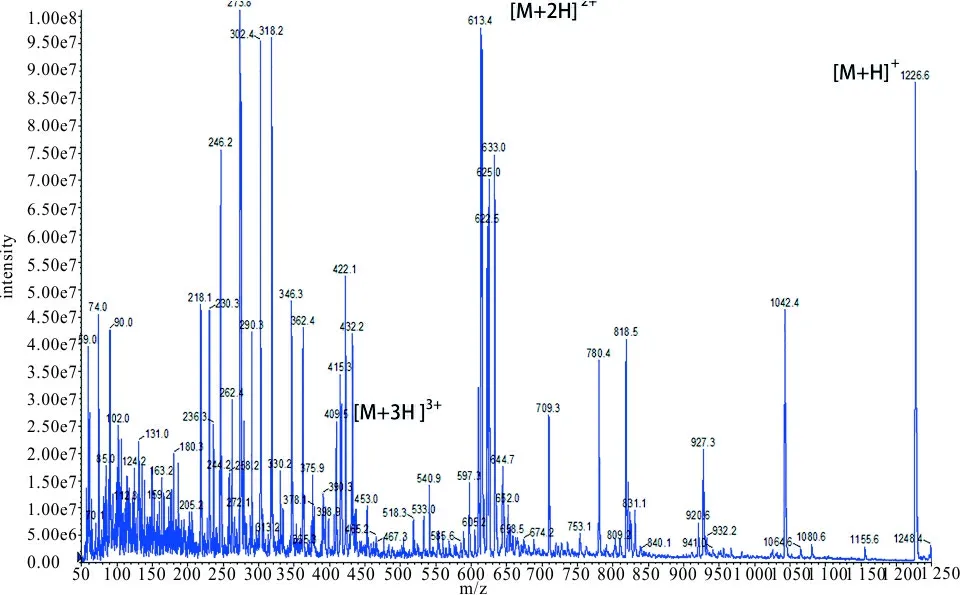
图1 T3质谱一级扫描图
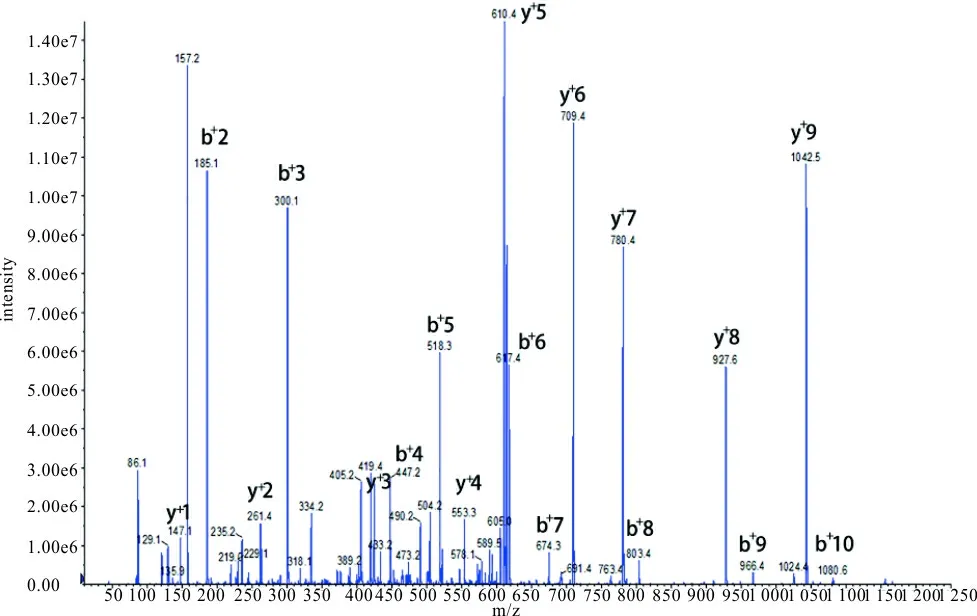
图2 T3质谱二级扫描图
2.3 蛋白质水平验证 选择613.8/709.4、613.8/1 042.5两对离子对T3验证蛋白质水平,对应的同位素标记肽段T3*离子对为617.6/709.4、617.6/1 042.5。胰蛋白酶水解人重组CysC的MRM图见图3,酶解后肽段与T3*的出峰时间均为10.05 min;胰蛋白酶水解血清的MRM图见图4,酶解后肽段与T3*的出峰时间均为10.09 min;验证了胰蛋白酶水解复杂基质中的CysC也可生成T3肽段。
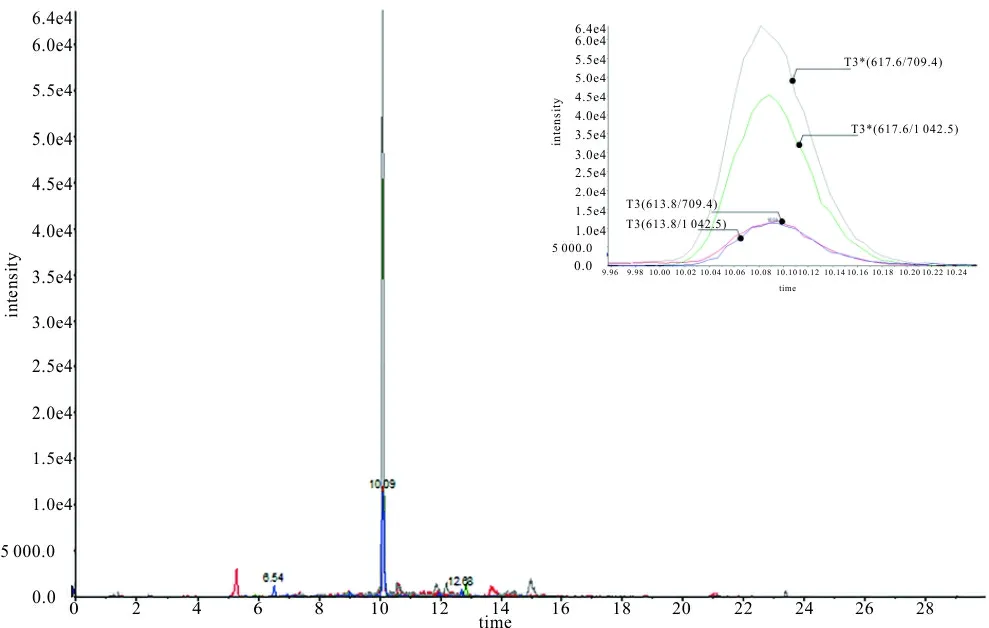
图4 血清样品酶解后MRM质谱图
3 讨论
临床实验室常用的蛋白质定量分析法主要为免疫法,由于非特异性抗体干扰、交叉反应、钩状效应等影响[7],不同检测平台检测同一项目结果差异较大。ID-LC-MS/MS蛋白质定量方法能直接检测目标分析物或其特异性肽段,虽然基质效应仍对测量结果产生某些影响,但相较于免疫法,假阳性率可显著降低。蛋白质分子量较大,蛋白质内标合成困难且价格昂贵,一般不用质谱法定量检测完整蛋白质。常用方法是用蛋白酶水解蛋白质产生一个或多个能够代表待测蛋白质的特征肽段,这些肽段在物质的量上可代表待测蛋白质的含量,从而定量检测蛋白质[8]。胰蛋白酶可特异性地切割赖氨酸(K)和精氨酸(R)的C端,水解方式较简单,因而常用胰蛋白酶作为蛋白质水解酶。传统方法是根据不同酶的水解特点直接估计肽键断裂部位,人为选择特异性肽段,估算肽段离子碎片分子量等信息。由于多肽在质谱中碎裂反应的复杂性,有时所选择的肽段在质谱中的实际响应值并不高,而同位素标记肽段较昂贵且合成周期长,重新合成新的肽段不仅多花费上万元,更浪费时间。生物信息学技术的应用可很大程度地减少试错概率,通过肽段理化性质分析,可很清晰地了解各个肽段的溶解性、稳定性、酸碱性等理化性质,为后续实验过程中对肽段合理有效的处理提供依据;通过同源性分析,非特异性肽段被排除,保证了定量检测时所选肽段的物质的量能够真正代表完整蛋白质的量;通过离子碎片信号分析,可筛选出离子信号较高的离子对,大大缩短预实验的范围和时间。用生物信息学分析技术省时省力,甚至可根据需求筛选肽段,肽段的选择原则如下:(1)肽段应具有代表性、专一性,不能在其他蛋白质的序列中出现;(2)肽段长度7~20个氨基酸为宜,在质谱中应有良好的离子化能力,响应强;(3)避免选择疏水性或亲水性太强的肽段;(4)避免选择易氧化、易环化、含二硫键、易脱酰胺基或者双碱基氨基酸的肽段,如蛋氨酸、半胱氨酸、KK、KR等。这些筛选原则在Skyline预测前均可设定。本实验中,排除T10是因其肽段长度太短,仅包含6个氨基酸,且不包含热稳定性的缬氨酸、亮氨酸、异亮氨酸、苯丙氨酸,后期蛋白质溯源工作无法开展;T3肽段稳定性好,可溶于水,在现有仪器设备上具有可检测性,且包含缬氨酸、亮氨酸和苯丙氨酸3种热稳定性氨基酸,各方面均符合要求,故选择T3肽段分析同源性和用质谱验证。此外,条件允许时还应用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)分析胰蛋白酶水解CysC纯蛋白质产生的所有肽段及相应的信号。González-Antua等[9]做了这方面的工作,显示T3肽段在质谱中的响应值最高,进一步证明选择T3肽段是正确的。本实验中,人重组CysC与血清经过一系列前处理及酶解过程后,MRM质谱图均显示酶解后肽段与T3*在同一时间出峰,也从另一个角度说明了人重组CysC与血清这样的复杂生物基质酶解后能够产生T3肽段,进一步验证生物信息学分析结果准确、可靠。综上所述,用生物信息学技术快速准确地筛选出CysC特异性肽段,显著地节约了成本,提高了工作效率,为蛋白质定量方法的研究奠定了良好的基础。
[1]Otsuka T, Kato K, Kachi Y,etal. Serum cystatin C, creatinine-based estimated glomerular filtration rate, and the risk of incident hypertension in middle-aged men[J]. Am J Hypertens, 2014,27(4): 596-602.
[2]Delanaye P, Cavalier E. Staging chronic kidney disease and estimating glomerular filtration rate: an opinion paper about the new international recommendations[J]. Clin Chem Lab Med, 2013, 51(10): 1911-1917.
[3]谭婷婷,程莉,魏红霞,等. HbA1c、尿AAG 和血清CysC 对早期糖尿病肾病的诊断价值[J].临床检验杂志,2015,33(11):827-829.
[4]王亚平,姜宇海,余伟,等.胱抑素C在糖尿病肾病早期诊断中的作用[J]. 临床检验杂志,2006,24(3):240.
[5]Zhang J, He P, Zhong Q,etal. Increasing Cystatin C and Cathepsin B in serum of colorectal cancer patients[J]. Clin Lab, 2017, 63(2): 365-371.
[6]Brun V, Masselon C, Garin J,etal. Isotope dilution strategies for absolute quantitative proteomics[J]. J Proteomics, 2009, 72(5):740-749.
[7]Hoofnagle AN, Wener MH. The fundamental flaws of immunoassays and potential solutions using tandem mass spectrometry[J]. J Immunol Methods, 2009, 347(1-2): 3-11.
[8]Yu Y, Xu JH, Liu Y,etal. Quantification of human serum transferrin using liquid chromatography-tandem mass spectrometry based targeted proteomics[J]. J Chromatogr B, 2012(902):10-15.
[9]González-Antua A, Rodríguez-González P, Ohlendorf R,etal. Determination of Cystatin C in human serum by isotope dilution mass spectrometry using mass overlapping peptides[J]. J Proteomics, 2015, 112:141-155.
(本文编辑:王海燕)
CysC specific peptides: bioinformatics analysis and mass spectrometry verification
SHENLei,WANGHui-min,JIHuo-yan,SHENPei,WANGJian-xin
(DepartmentofLaboratoryMedicine,AffiliatedHospitalofNantongUniversity,Nantong226001,Jiangsu,China)
Objective To analyze the specific peptide of cystatin C (CysC) and its characteristics by bioinformatics technology, and verify the predicted results by mass spectrometry. Methods Online software was applied to analyze the physicochemical properties and homology of CysC peptides hydrolyzed by trypsin and predict the associated parameters of ionized fragmentation of specific peptide by mass spectrometry. Precursor ion scan and product ion scan were conducted on the samples of synthetic specific peptide. The recombinant human CysC and serum samples were analyzed by mass spectrometry after trypsin digestion. The results of analysis were compared with the outcomes predicted by bioinformatics. Results T3 (ALDFAVGEYNK) was considered as the specific peptide of CysC by software analysis. When selecting[M+2H]2+for product ion scan, almost all the y and b ions of fragmentation were observed using tandem mass spectrometry (MS/MS), showing consistency with Skyline predictions. Moreover, both the peptides from the human recombinant CysC and serum sample following the trypsin digestion were eluted at the same time with the isotope-labeled T3* under the fixed conditions. Conclusion Bioinformatics technology could be available for picking out the specific peptides of target protein quickly and efficiently and predicting the ionized fragmentation precisely by mass spectrometry scanning.
Cystatin C;bioinformatics technology;specific peptide;mass spectrometry
10.13602/j.cnki.jcls.2017.06.13
沈蕾,1987年生,女,技师,硕士,主要从事临床检验工作。
王建新,副主任技师,E-mail:tdfyiyk@126.com。
R446.1
A
2017-01-18)
