基于转录组数据的印度谷螟微卫星位点分析
2017-06-05唐培安陶冶心袁明龙
唐培安, 陶冶心, 薛 昊, 袁明龙
(1. 南京财经大学食品科学与工程学院/江苏省现代粮食流通与安全协同创新中心, 南京 210023; 2. 草地农业生态系统国家重点实验室/兰州大学草地农业科技学院, 兰州 730020)
研究报告
ResearchReports
基于转录组数据的印度谷螟微卫星位点分析
唐培安1, 陶冶心1, 薛 昊1, 袁明龙2
(1. 南京财经大学食品科学与工程学院/江苏省现代粮食流通与安全协同创新中心, 南京 210023; 2. 草地农业生态系统国家重点实验室/兰州大学草地农业科技学院, 兰州 730020)
基于印度谷螟转录组测序数据筛选其功能微卫星(EST-SSR)分子标记,设计该虫的微卫星(SSR)引物并验证其适用性。用MicroSAtellite微卫星搜索软件查找印度谷螟转录组中微卫星的数量、重复次数以及所有微卫星的位置信息,采用Primer Premier 5软件设计SSR引物,并进行PCR验证。结果表明含EST-SSR的序列3 173个,占总搜索序列的8.52%,平均每13 kb中就出现1个EST-SSR。共发现192种微卫星类型,其中重复单元为单碱基、二碱基和三碱基的比例较高,依次是28.21%、39.84%、25.43%。除单碱基重复单元外,出现频率最高的是AT/AT,有593次。在66对印度谷螟EST-SSR引物中,有28对PCR扩增成功。这说明基于印度谷螟转录组数据开发微卫星标记是可行的,本研究获得的印度谷螟EST-SSR位点为今后开展该虫的种群遗传学研究提供了基础数据。
转录组; 高通量测序; 螟蛾科; 微卫星; 分子标记
微卫星(microsatellite)又称SSR(simple sequence repeat, 简单重复序列),是指以1~6个核苷酸为重复单元串联组成的重复序列[1],1981年,微卫星首次被发现,它们大都以2~3个碱基重复类型为主,主要分布于原核生物和真核生物的基因组中[2-3]。微卫星具有高多态性、多等位性、共显性、高可重复性、数量丰富和对基因组有很好覆盖性等特征[4-5]。因此,该标记已被广泛应用于生物遗传研究中[6]。
印度谷螟Plodiainterpunctella属鳞翅目Lepidoptera螟蛾科Pyralidae,是世界性分布的重大储粮害虫。该虫在我国南北方广泛分布[7],依据2004-2005年第6次全国储粮昆虫调查,印度谷螟被列为危害严重且分布广泛的主要鳞翅目储粮害虫之一[8]。目前,关于印度谷螟SSR的研究还未见报道,在GenBank中也没有发现登录的印度谷螟SSR序列,不能满足其种群遗传结构研究的需要。因此,开发印度谷螟的微卫星位点,是有效开展其种群遗传结构和基因交流、系统发育与分子进化等方面研究的重要前提和首要工作,同时对于揭示其危害规律和综合防治等都具有重要意义。传统开发SSR标记的方法需要构建文库、筛选SSR克隆、测序等复杂且工作量大的步骤[9-11]。随着新一代高通量测序技术的成长,尤其是转录组测序的飞速发展,降低了微卫星分子标记的开发成本,提高了开发效率,为非模式物种SSR位点的大批量开发提供了一种经济、高效的途径[12-13]。从转录组数据中获得的与功能基因相关SSR位点又叫表达序列标签SSR(expressed sequence tag SSR,EST-SSR)。目前,从昆虫转录组数据库中筛选功能EST-SSR标记已有许多成功的报道[14-17]。本研究基于已测印度谷螟转录组数据,挖掘其EST-SSR位点,分析这些微卫星位点的类型和分布特点,探讨鉴定到的EST-SSR位点的适用性,以期获得可供该虫种群遗传学研究所需的核分子标记,也为印度谷螟近缘物种微卫星位点的发掘提供可借鉴的方法。
1 材料与方法
1.1 材料及转录组数据来源
印度谷螟采自江苏吴江,以大米粉为饲料,在温度为(30±1)℃、湿度为75%±5%、24 h无光照条件的恒温培养箱中培养10代以上,选取4龄幼虫作为试验材料。采用德国Qiagen公司的RNeasy Plus Universal Mini Kit试剂盒提取RNA,由华大科技进行高通量转录组测序(转录组数据的NCBI登录号:SRP060836)。通过Illumina Hiseq2000平台测序,总计产出5.7 GB数据,共组装Unigene 37 246个,总长49 682 542 bp,平均长度1 334 bp,N50达到2 307 bp,过滤后质量不低于20的碱基比例(Q20,测序错误率<1%)为98.31%,表明测序结果良好。
1.2 微卫星序列筛选
以组装出来的Unigene作为参考序列,使用Perl操作平台下的SSR分析软件MicroSAtellite (MISA) (http:∥pgrc.ipkgatersleben.de/misa/)在Unigene中搜索SSR位点。筛选条件为:单碱基重复次数≥12,二碱基重复次数≥6,三碱基重复次数≥5,四、五、六碱基重复次数≥4,并且两个相邻微卫星重复单元之间的最小长度设为100 bp。
1.3 印度谷螟SSR引物设计及PCR扩增
基于SSR重复单元两翼序列设计引物,采用Primer 5.0对印度谷螟EST-SSR设计了66对引物。引物筛选条件如下:①引物不含有重复序列;②将获得的引物比对到Unigene序列,引物5′端允许有3个碱基的错配,而3′端允许有1个碱基的错配;③去除比对到非目标Unigene的引物,保证引物与Unigene唯一匹配。④使用ssr_finder校验SSR,使用产物序列来寻找SSR,检验结果是否与MISA结果相同,并筛选出相同的SSR产物。设计好的引物由金斯瑞生物技术公司(江苏南京)合成。
采用TIANamp Genomic DNA Kit(北京天根)试剂盒提取单头印度谷螟4龄幼虫的基因组DNA。PCR扩增体系:基因组DNA 1 μL,上游引物2 μL,下游引物2 μL,灭菌水7.5 μL,ExTaqMix 12.5 μL。PCR反应条件:94℃预变性3 min,再进行30个循环,每个循环包括94℃变性30 s,Tm复性30 s, 72 ℃延伸1 min;最后72 ℃延伸10 min。PCR产物用2%的琼脂糖凝胶电泳检测。
2 结果与分析
从构建的印度谷螟转录组数据库中,获得EST-SSR序列3 173个,占总搜索基因的8.52%,平均每13 380 bp中就出现1个SSR位点。
2.1 微卫星数量及分布特征
单碱基、二碱基、三碱基、四碱基、五碱基和六碱基重复的EST-SSR数分别为895、1 264、807、100、28、79,依次占总SSR个数的28.21%、39.84%、25.43%、3.15%、0.88%、2.49%(表1)。可见,在印度谷螟基因数据库中二碱基重复序列占据优势,其次是单碱基和三碱基重复序列。此外,复合型SSR有72个,占SSR总数的2.27%。在单碱基、二碱基、三碱基、四碱基、五碱基、六碱基SSR中出现频率最多的分别是A/T、AT/AT、CCG/CGG、AAAT/ATTT、ACG-CGC/GCGCGT(图1),在各自重复单元类型中所占比例依次为98.55%、46.91%、36.31%、33.00%、17.86%、12.66%。在五碱基和六碱基中各类重复基元分布较为平均,这是由于此类重复较少的原因。
单碱基重复次数集中在12~15次,二碱基重复次数集中在6~8次,三碱基重复次数集中在5~8次,四碱基重复次数集中于5~6次,五碱基和六碱基则以重复4次为主。且随着碱基重复单元的重复次数增加,其数量均呈下降趋势。

表1 印度谷螟转录组中不同微卫星类型分布
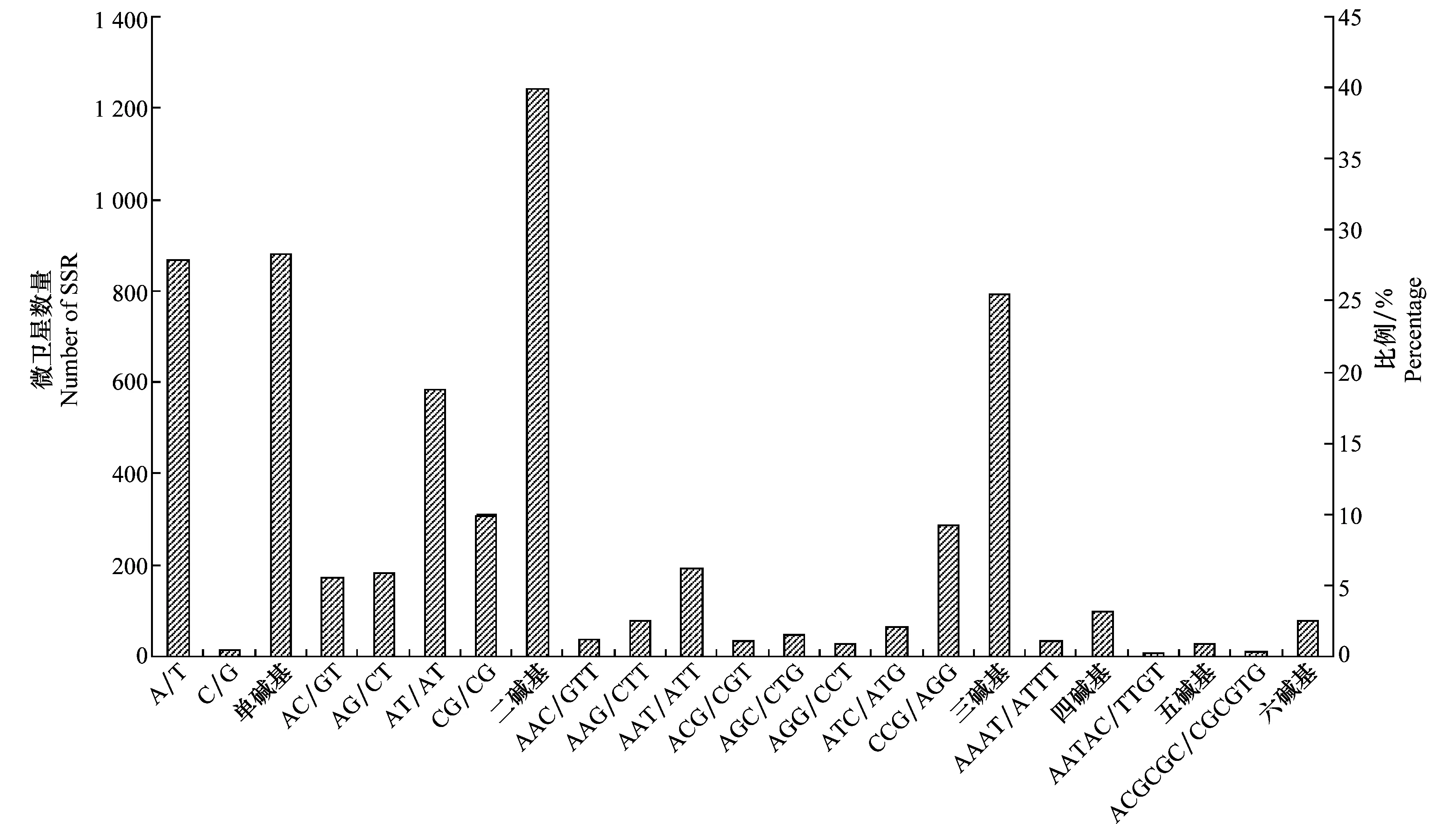
图1 印度谷螟转录组不同碱基类型微卫星位点分布Fig.1 Distribution of the microsatellites with different repeat motifs in the Plodia interpunctella transcriptome
2.2 微卫星长度分布特征
印度谷螟转录组中发现的3 173个EST-SSR长度从12~192个碱基不等,平均长度为16.24个碱基。序列长度12~15个碱基的有2 175个,占总SSR个数的68.5%,其中单碱基、二碱基和三碱基重复单元所占比例分别为35.54%、39.72%、24.74%,分布较为平均。序列长度16~20个碱基有560个,所占比例为17.6%,其中二碱基重复单元比例最高(48.04%),其次是三碱基重复单元(27.50%)。序列长度21~25个碱基的有297个,占总EST-SSR的9.3%,其中三碱基重复单元占29.97%,其次是二碱基重复单元(27.27%),再次是六碱基重复单元(24.58%),其他重复单元数量较少。序列长度大于25的数量较少,仅占总EST-SSR的4.4%,且全部是复合型(图2)。

图2 印度谷螟转录组中EST-SSR的长度分布Fig.2 Length distribution of EST-SSRs in the Plodia interpunctella transcriptome
2.3 微卫星在转录组编码区分布特征
印度谷螟转录组数据中,有23 310个Unigene序列功能注释成功。在23 310个注释成功的Unigene序列中发现EST-SSR位点2 226个,总长度13 182 617 bp,其中编码区858个EST-SSR位点。编码区EST-SSR中二碱基重复319个,三碱基重复378个,在非编码区二碱基重复517个,三碱基重复291个。说明在编码区以三碱基重复为主,非编码区以二碱基重复为主。
2.4 微卫星引物设计与PCR扩增
根据印度谷螟可用于引物设计的EST-SSR序列共随机设计了66对引物,部分引物信息见表2。以单头印度谷螟4龄幼虫DNA为模板,进行PCR扩增,结果表明66对引物中的31对引物可以扩增得到条带(图3)。其中两对引物扩增产物与预期相差较大,一对引物产生非特异性条带,其余28对引物与预期相符。
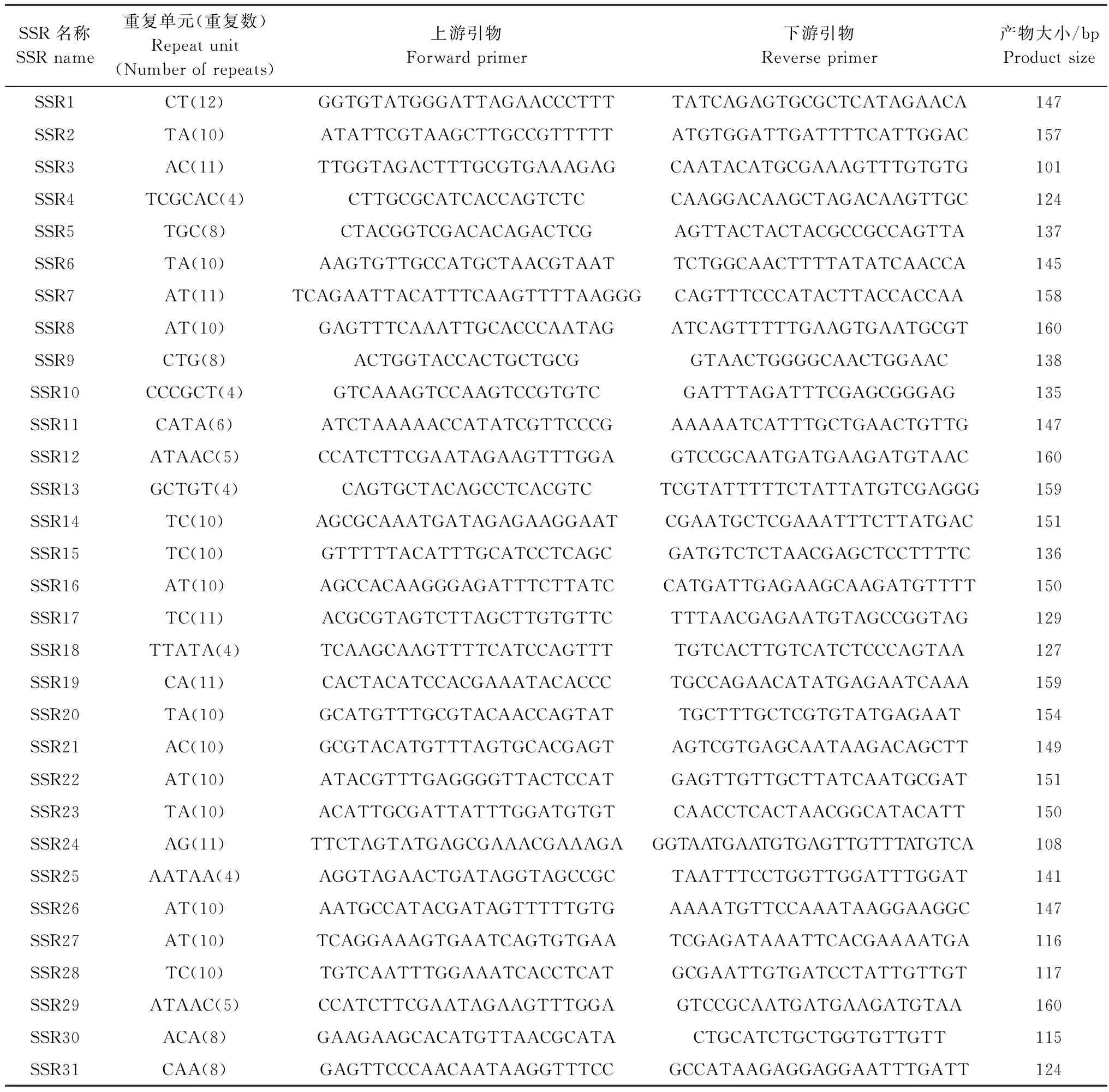
表2 印度谷螟31对EST-SSR引物信息

图3 印度谷螟31个EST-SSR位点的扩增电泳图Fig.3 Agarose gel showing the amplification of 31 EST-SSR loci in the Plodia interpunctella transcriptome
3 讨论
本研究通过生物信息学方法从印度谷螟转录组数据库37 246条Unigenes中共获得EST-SSR位点3 173个,其出现频率(8.52%)高于大多数的昆虫,如扶桑绵粉蚧Phenacoccussolenopsis(6.33%)[18],烟粉虱Bemisiatabaci(5.07%)[19],橘小实蝇Bactroceradorsalis(4.23%)[11],黄粉虫Tenebriomolitor(1.67%)[20],云南切梢小蠹Tomicusyunnanensis(1.29%)[21]和葱地种蝇Deliaantiqua(1.12%)[22]等;而目前仅有少数昆虫的EST-SSR位点出现频率高于印度谷螟,如黑翅土白蚁Odontotermesformosanus(9.98%)[23]。导致这种差异的原因,可能与EST-SSR筛选的标准不同以及用于测序的RNA质量相关。当然,最本质的原因应是物种本身的差异所致。
本研究表明,印度谷螟转录组中微卫星的种类较为丰富,1~6 核苷酸重复类型都有出现,主要重复类型为1~3核苷酸重复,占SSR总数的93.48%。在单碱基重复基元中, A/T是占优势的重复基元,这与已研究的大多数昆虫类似,如褐飞虱Nilaparvatalugens[24]、黏虫Mythimnaseparata[15]和扶桑绵粉蚧P.solenopsis[18]等。研究发现,基于转录组数据库发掘的昆虫微卫星以三碱基重复SSR出现的频率最高,如二点委夜蛾Athetislepigone[25]、灰飞虱Laodelphaxstriatellus[26]、橘小实蝇B.dorsalis[11]、黄粉虫T.molitor[20]和温带臭虫Cimexlectularius[16]等。普遍的观点认为,三碱基重复序列占据主导地位,是由于三联体密码子的选择作用所致[27]。因为密码子的基本单位就是三碱基,其他类型的重复单元会引起阅读框的改变,增加错配或者移码突变的机会。本研究发现,印度谷螟编码区中三碱基重复也为优势重复单元,支持该论点。然而,在印度谷螟的所有EST-SSR中最优势重复单元为二碱基重复,这与丽蝇蛹集金小蜂Nasoniavitripennis、N.giraulti和N.longicornis[28]、黑腹果蝇Drosophilamelanogaster[29]和细梢小卷蛾Rhyacionialeptotubula[17]等类似。以往对昆虫EST-SSR的研究中,以GC/GC为核心基元的两碱基重复SSR的存在非常稀少,几乎为零。但是在印度谷螟二碱基重复单元中GC/GC含量占了1/3左右,仅仅低于重复单元AT/AT,这与同属于鳞翅目的黏虫[15]、二点委夜蛾[25]和细梢小卷蛾[17]相似。因此,我们推断GC/GC重复基元可能与鳞翅目的某些特殊功能有关系。此外,研究表明,SSR的多态性与其长度以及核心基元的重复次数的多少具有相关性,即长度越长、核心基元的重复次数越多,其多态性越高[30]。印度谷螟的EST-SSR长度集中于12~15个碱基占据了70%,而且还有12%(多为二、三碱基重复单元)长度大于20个碱基。因此,印度谷螟可能具有较高的遗传多态性,这也是该虫高度适应环境而成为世界性害虫的遗传基础。
众所周知,鳞翅目昆虫SSR开发难度较大,由于其SSR的多态性较低、存在微卫星家族现象并有大量的无效等位基因[31]。为得到质量较高且具有潜在多态性的引物,在筛选引物时应尽量选择侧翼长度大于30 bp的位点,且相似度低于70%,核心重复单元重复次数二碱基在10次以上,三碱基应在8次以上,可降低家族现象的影响并且避免非特异性扩增[11]。本文利用转录组数据,设计得到的66对EST-SSR引物,28对扩增得到预期大小的条带,占合成引物的42.42%。尽管这些位点的多态性还有待进一步检验,但其数量足够开展印度谷螟种群遗传学等相关研究。导致EST-SSR位点扩增失败有诸多原因:扩增得到条带远大于预期,可能由于基因组DNA中含有内含子而在转录后未出现,干扰了扩增过程;而其中一对引物产生非特异性条带可能是由于EST-SSR位点位于同源基因序列上;扩增失败无条带,可能是因为EST-SSR在基因组中的表达丰度较低而导致产物浓度很低,未能检测到。
目前,SSR研究主要应用于遗传学分析。对于非模式昆虫的研究其难点在于SSR引物的筛选,因为在昆虫等节肢动物基因组内SSR的含量较少,且存在家族现象,这加大了研究难度。传统微卫星筛选方法费时费力,且费用高昂。转录组测序技术的发展解决了这一难题,从转录组测序结果中可以直接筛选出质量合格的SSR,经过验证其在相近物种间具有较高的通用性。随着转录组测序技术的进一步发展,测序通量加大,成本降低,这为昆虫SSR的大规模开发提供了可能,且SSR所在基因的表达水平也可以监测到,为深入研究SSR在基因调控和修饰中所产生的作用提供了技术保障[32]。但基于转录组测序获得的EST-SSR位点,其核心重复次数相较于基因组的SSR少,这是因为在组装过程中重复次数多的SSR会发生断裂或者丢失,这影响了EST-SSR位点的多态性。另外,cDNA中缺少内含子或者内含子位于SSR引物之间,这就导致部分EST-SSR位点的扩增失败。另一方面,基于转录组测序数据发掘EST-SSR位点具有快速、方便、低廉以及数据量大等优点,已成为当前发掘昆虫微卫星位点的重要策略。
综上所述,本研究基于印度谷螟转录组数据筛选获得了大量EST-SSR位点,并对其数量分布、相关特性及适用性进行了分析。结果表明,基于转录组数据发掘印度谷螟微卫星位点是高效可行的,也是发掘非模式生物EST-SSR位点的一种经济、快速、有效的方法。本研究获得的印度谷螟EST-SSR位点,为今后开展该虫的遗传多样性及种群遗传结构研究,阐明其种群遗传分化及抗药性基因流动规律,以及制定有效防控策略等提供了基础数据。
[1] 孙荆涛,杨现明,葛成,等. 微卫星分子标记在昆虫分子生态学研究上的应用[J]. 南京农业大学学报, 2012, 35(5): 103-112.
[2] Spritz R A.Duplication/deletion polymorphism 5′-to the human β globin gene [J]. Nucleic Acids Research,1981,9(19): 5037-5048.
[3] Selkoe K A, Toonen R J. Microsatellites for ecologists: a practical guide to using and evaluating microsatellite markers[J]. Ecology Letters, 2006,9(5): 615-629.
[4] Sandhu K S, Karaoglu H, Zhang P, et al. Simple sequence repeat markers support the presence of a single genotype ofPucciniapsidiiin Australia[J]. Plant Pathology, 2016,65(7): 1084-1094.
[5] Madesis P, Ganopoulos I, Tsaftaris A.Microsatellites: evolution and contribution [M]∥Walker J M.Methods in molecular biology, USA: Humana Press, 2013: 1-13.
[6] Hadonou A M, Sargent D J, Wilson F, et al. Development of microsatellite markers inFragaria, their use in genetic diversity analysis, and their potential for genetic linkage mapping [J]. Genome, 2004, 47(3): 429-438.
[7] 简富明. 印度谷螟生物学特性初步研究[J]. 西南农业学报, 1993, 6(3): 80-84.
[8] 严晓平,宋永成,沈兆鹏,等.中国储粮昆虫2005年最新名录[J]. 粮食储藏, 2006,35(2):3-9.
[9] Andrés J A, Bogdanowicz S M. Isolating microsatellite loci: looking back, looking ahead [J]. Molecular Methods for Evolutionary Genetics, 2011, 772: 211-232.
[10]Zane L, Bargelloni L, Patarnello T.Strategies for microsatellite isolation: a review [J]. Molecular Ecology, 2002, 11(1): 1-16.
[11]魏丹丹,石俊霞,张夏瑄,等.基于转录组数据的桔小实蝇微卫星位点信息分析[J].应用生态学报,2014,25(6):1799-1805.
[12]Schoebel C N, Brodbeck S, Buehler D, et al. Lessons learned from microsatellite development for nonmodel organisms using 454 pyrosequencing[J]. Journal of Evolutionary Biology, 2013, 26 (3): 600-611.
[13]Fernandez-Silva I, Whitney J, Wainwright B, et al. Microsatellites for next-generation ecologists: a post-sequencing bioinformatics pipeline [J]. PLoS ONE, 2013, 8 (2): e55990.
[14]Comeault A A, Sommers M, Schwander T, et al.Denovocharacterization of theTimemacristinaetranscriptome facilitates marker discovery and inference of genetic divergence [J]. Molecular Ecology Resources, 2012, 12 (3): 549-561.
[15]胡艳华, 李敏, 张虎芳, 等. 粘虫转录组中SSR位点的信息分析[J]. 山西农业大学学报: 自然科学版, 2015, 35 (5): 484-489.
[16]Bai X, Mamidala P, Rajarapu S P, et al. Transcriptomics of the bed bug (Cimexlectularius)[J]. PLoS ONE, 2011, 6 (1): e16336.
[17]Zhu J Y, Li Y H, Yang S, et al.Denovoassembly and characterization of the global transcriptome forRhyacionialeptotubulausing illumina paired-end sequencing[J]. PLoS ONE, 2013, 8 (11): e81096.
[18]罗梅, 张鹤, 宾淑英, 等. 基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物[J]. 昆虫学报, 2014, 57 (4): 395-400.
[19]Xie W, Meng Q S, Wu Q J, et al. Pyrosequencing theBemisiatabacitranscriptome reveals a highly diverse bacterial community and a robust system for insecticide resistance[J]. PLoS ONE, 2012, 7 (4): e35181.
[20]朱家颖, 吴国星, 杨斌. 基于转录组数据高通量发掘黄粉甲微卫星引物[J]. 昆虫学报, 2013, 56 (7): 724-728.
[21]袁远, 张丽芳, 吴国星,等. 云南切梢小蠹微卫星的高通量发掘[J]. 环境昆虫学报, 2014, 36(2): 166-170.
[22]Zhang Yujuan, Hao Youjin, Si Fengling, et al. Thedenovotranscriptome and its analysis in the worldwide vegetable pest,Deliaantiqua(Diptera: Anthomyiidae)[J]. G3: Genes| Genomes| Genetics, 2014, 4 (5): 851-859.
[23]Huang Q Y, Sun P D, Zhou X G, et al. Characterization of head transcriptome and analysis of gene expression involved in caste differentiation and aggression inOdontotermesformosanus(Shiraki)[J]. PLoS ONE, 2012, 7 (11): e50383.
[24]刘玉娣, 侯茂林. 褐飞虱EST资源的微卫星信息分析[J]. 昆虫学报, 2010, 53(3): 239-247.
[25]Li L T, Zhu Y B, Ma J F, et al. An analysis of theAthetislepigonetranscriptome from four developmental stages[J]. PLoS ONE, 2013, 8 (9): e73911.
[26]Zhang F G, Guo H Y, Zheng H J, et al. Massively parallel pyrosequencing-based transcriptome analyses of small brown planthopper (Laodelphaxstriatellus), a vector insect transmitting rice stripe virus (RSV)[J]. BMC Genomics, 2010, 11 (1): 303.
[27]潘海涛, 汪俊君, 王盈盈, 等. 小麦EST-SSR标记的开发和遗传作图[J]. 中国农业科学, 2010, 43 (3): 452-461.
[28]Pannebakker B A, Niehuis O, Hedley A, et al. The distribution of microsatellites in theNasoniaparasitoid wasp genome[J]. Insect Molecular Biology, 2010, 19(S1): 91-98.
[29]Schug M D, Wetterstrand K A, Gaudette M S, et al. The distribution and frequency of microsatellite loci inDrosophilamelanogaster[J].Molecular Ecology, 1998, 7(1): 57-70.
[30]Meglécz E, Nève G, Biffin E, et al. Breakdown of phylogenetic signal: a survey of microsatellite densities in 454 shotgun sequences from 154 non model eukaryote species[J]. PLoS ONE, 2012, 7 (7): e40861.
[31]Zhang D X. Lepidopteran microsatellite DNA: redundant but promising [J]. Trends in Ecology & Evolution, 2004, 19 (10): 507-509.
[32]Jhanwar S, Priya P, Garg R, et al. Transcriptome sequencing of wild chickpea as a rich resource for marker development [J]. Plant Biotechnology Journal, 2012, 10 (6): 690-702.
(责任编辑:田 喆)
Analysis of microsatellite loci inPlodiainterpunctellabased ontranscriptome dataset
Tang Peian1, Tao Yexin1, Xue Hao1, Yuan Minglong2
(1.College of Food Science and Engineering/Collaborative Innovation Center for Modern Grain Circulation and Safety,Nanjing University of Finance and Economics, Nanjing 210023, China; 2.State Key Laboratory of Grassland Agro-Ecosystems/College of Pastoral Agricultural Science and Technology, Lanzhou University, Lanzhou 730020, China)
This study is aimed to identify gene microsatellite (EST-SSR) loci from the transcriptome database ofPlodiainterpunctella. Based on these SSR sequences, the selected SSR primer pairs were validated. The number, repetition time and location information of all microsatellites obtained with the microsatellite search tool MicroSAtellite were analyzed.Primer Premier 5 was used to designP.interpunctellaSSR primers, and then these primer pairs were verified by PCR. A total of 3 173 SSRs were identified in 37 246 Unigenes, with one SSR per 13 kb. The majority of microsatellite loci consisted of mono-, di- and tri-nucleotide motifs (28.21%, 39.84% and 25.43%, respectively). However, different types of repeat SSRs had considerably different distribution. Except mono-nucleotide, AT/AT was the most frequent repeat motif (593 repeats) among all SSR motifs. Among the 66 designed primer pairs, 28 pairs were amplified successfully. Our study shows that it is feasible to develop microsatellite markers based onP.interpunctellatranscriptome. The EST-SSRs ofP.interpunctellaobtained in this study are helpful for population genetics studies of this important stored-product pest.
transcriptome; high-throughput sequencing; Pyralidae; microsatellite; molecular marker
2016-06-22
2016-08-02
粮食公益性行业科研专项(201413007-2,201513002-5-3);国家重点研发计划专项(2016YFD0401004-04);江苏省高校优势学科建设工程
Q 968.1
A
10.3969/j.issn.0529-1542.2017.03.007
联系方式 E-mail:tangpeian@163.com
