酿酒酵母高效自主复制区的筛选与鉴定
2017-04-26沈方琳黄双成侯朋晨耿安丽阮文权
沈方琳, 黄双成,侯朋晨,耿安丽, 阮文权*
1(江南大学 环境与土木工程学院,江苏 无锡,214122) 2(义安理工学院,生命科学与化学技术实验室,新加坡) 3(江苏省厌氧生物技术重点实验室,江苏 无锡,214122)
酿酒酵母高效自主复制区的筛选与鉴定
沈方琳1,3, 黄双成2,侯朋晨2,耿安丽2, 阮文权1,3*
1(江南大学 环境与土木工程学院,江苏 无锡,214122) 2(义安理工学院,生命科学与化学技术实验室,新加坡) 3(江苏省厌氧生物技术重点实验室,江苏 无锡,214122)
在酵母表达过程中,传统的表达载体存在着拷贝数低以及稳定性差的缺点。因此,找到一个可用于构建复制整合型载体的高效酵母自动复制区(Autonomously Replicating Sequence,ARS)是解决问题的关键。研究中,从酿酒酵母基因组中扩增得到4种不同的ARS(ARS304、ARS315、ARS735、ARS1512), 然后连接到整合载体pNTS2K中,得到4种复制整合型载体(pNTS2K-ARS304、pNTS2K-ARS315、 pNTS2K-ARS735、pNTS2K-ARS1512)。经电转化后,4种转化子(36a(pNTS2K-ARS304)、36a(pNTS2K-ARS315)、36a(pNTS2K-ARS735)、36a(pNTS2K-ARS1512))都能在含有300 μg/mL G418的YPD平板上生长。然而,只有转化子36a(pNTS2K-ARS315)能在含有500 μg/mL G418的YPD液体培养基里较快生长。经过G418耐受性检测后,36a(pNTS2K-ARS315)仍能在含有1 mg/mL G418的YPD培养基中很好地生长。此外,通过质粒pNTS2K-ARS315表达荧光蛋白,其表达效果高于普通商业质粒。实验结果证明,4种复制区都能在酿酒酵母中独立自主复制,其中ARS315复制能力最强。因此,ARS315可用于构建高拷贝且稳定的重组质粒,为构建高表达且稳定的工程酵母菌株奠定了基础。
酿酒酵母;自动复制区;高拷贝复制整合型质粒;荧光蛋白
在酵母表达蛋白以及其他有机产物的过程中,通过增强转化过程中的酶活性来加快产物生成是研究者公认的观点,而高表达特异性酶是解决问题的主要途径。寻找1个新颖匹配的启动子或构建1个高效表达系统是非常困难且需要大量的时间。向宿主细胞中引入大量目的基因是最先广泛采用的方法。在酵母表达过程中,传统的表达载体YEP,YCP和YIP常常被用来引入外源基因。但是,YCP和YIP存在低拷贝的缺点,而YEP表达不稳定[1]。高拷贝数地将目的基因整合入酵母基因组是高表达且稳定的必要途径。酵母自动复制区作为酵母染色体复制起点,在染色体复制过程中起着至关重要的作用[2-3]。如果这些位点发生突变会导致ARS失去活性,进而影响到复制起始的功能[4]。其活性还可能与其他诸如ARS侧翼序列特征,染色体定位,染色体修饰等因素有关。每条染色体复制都是在间隔40~100 kb的多个位点起始的,每个起点在1个细胞周期的中期S期相继被激活,起始1次染色体复制[5-7]。在酵母基因组中,存在着300多个自动复制区,而找到高效的自动复制区是构建高拷贝质粒的前提。本实验在酿酒酵母整合载体pNTS2K中的NdeI和EcoRI之间(见图1)分别引入4种ARS,筛选出高效的复制区ARS315,从而构建了一种高效的复制整合型载体pNTS2K-ARS315。通过质粒pNTS2K-ARS315表达荧光蛋白,其表达效果高于普通商业质粒。因此,这种质粒可以在G418抗性基因KanMX两侧,2个rDNA片段之间(见图1)引入多个表达框。随着G418抗性的选择压力,尽可能多的将外源基因表达框整合在酵母基因组多拷贝rDNA区域,为以后外源基因的高效稳定表达奠定了基础。

图1 pNTS2K质粒图谱Fig.1 pNTS2K plasmid map
1 材料与方法
1.1 材料
1.1.1 质粒与菌种
酿酒酵母整合载体pNTS2K由本实验室构建,以pUC19为骨架,引入rDNA 整合区片段及KanMX表达框,如图1所示。大肠杆菌(E.coli)DH5α,酿酒酵母(S.cerevisiae)Juk36a (URA3缺陷型)由本实验室保存。
1.1.2 酶和试剂
酵母基因组提取试剂盒、DNA聚合酶以及Phusion高保真酶购买于Thermo Scientific 公司;限制性内切酶和T4连接酶购买于NEB公司;LB培养基、YPD培养基、氨苄青霉素(Amp)以及G418购买于Sigma公司,引物也由Sigma 公司合成。
1.2 方法
1.2.1 质粒的构建
用酵母基因组提取试剂盒提取Juk36a基因组DNA。由于ARS活性可能与诸如ARS侧翼序列特征、染色体定位和染色体修饰等因素有关[8],所以根据NCBI上S288c基因组序列,针对各ARS序列上下游200 bp处设计4对特异性引物(见表1)分别扩增ARS304、ARS315、ARS735和ARS1512片段,并在上下游引物5’末端引入NdeI和EcoRI酶切位点。PCR扩增条件为98 ℃ 30 s;98 ℃ 10 s, 60 ℃ 20 s, 72 ℃ 1 min, 循环30次;72 ℃ 2 mins;4 ℃保存。用1%琼脂糖凝胶电泳鉴定PCR产物,用胶回收试剂盒回收特异性条带。用限制性内切酶NdeI和EcoRI双酶切胶回收产物和pNTS2K质粒。纯化酶切反应液后,用T4连接酶连接,转化DH5α感受态细胞。经菌落PCR鉴定后,挑取阳性转化子37 ℃过夜培养。然后提取质粒,并用NdeI和EcoRI酶切鉴定。

表1 PCR引物序列
1.2.2 酵母转化
将所得到的正确质粒pNTS2K-ARS304、pNTS2K-ARS315、pNTS2K-ARS 735和 pNTS2K-ARS1512(如表2所示)用Nanodrop检测浓度后,分别稀释到100 ng/μL。按照Gene Pulser XcellTMElectroporation System 标准方法,分别将5 μL质粒加入到40 μL酵母感受态细胞中进行电转[9],然后分别涂布于含有300 μg/mL G418的YPD平板上,培养3天后挑取转化子。转化率=转化子数/质粒DNA质量。

表2 质粒
1.2.3 转化子的复筛及比较
最终OD600 nm的测量是衡量细菌产量的标准,也是细菌生长率的体现,是外源基因高表达的必要前提[10]。因此,从生长出的不同转化子中(如表3所示)分别挑出5个最大的单克隆于2 mL YPD培养基中培养过夜,以OD600nm为0.05转接到5 mL含有500 μg/mL G418的YPD培养基中培养48 h,测OD600nm吸光值。
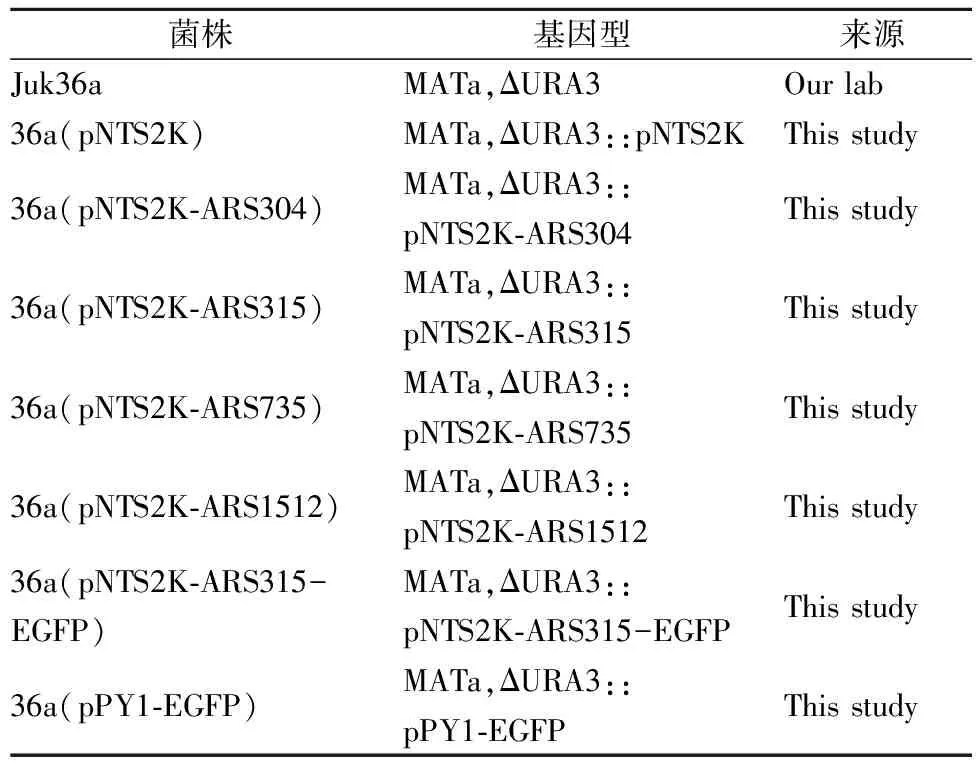
表3 菌株
1.2.4 复制能力的比较及检测
将生长能力最强的Juk36a(pNTS2K-ARS315)菌株分别接种于含有不同梯度的G418的YPD培养基的24孔培养板中,每孔培养体积为0.6 mL,放入InfiniteF200PRO (TECAN)中监测40 h得到生长曲线。
1.2.5 质粒的应用及检测
将得到的复制能力最强的质粒pNTS2K-ARS315为骨架,以本实验室普通商业酵母表达载体pPY1(以2micron 为复制区,由本实验室保存)作为对照,分别引入荧光蛋白表达框。经过酶切、连接,转化到DH5α感受态细胞中。经菌落PCR鉴定后,挑取阳性转化子37℃过夜培养,提取重组质粒pNTS2K-ARS315-EGFP和pPY1-EGFP(如表2所示)。然后将得到的质粒转化到酿酒酵母Juk36a(URA3缺陷型)中,检测转化子36a(pNTS2K-ARS315-EGFP)和36a(pPY1-EGFP)(如表3所示)的荧光强度。
2 结果与分析
2.1 ARS片段的获得及质粒的构建
以酿酒酵母Juk36a基因组为模板扩增ARS304(930 bp),ARS315(584 bp),ARS735(844 bp)和ARS1512(1042 bp)4条序列,采用1%的琼脂糖凝胶电泳检测PCR产物结果如图2所示。从图2中可以看出,目的条带单一,大小与预期结果一致。

M-DNA标记;1-ARS304 PCR片段;2-ARS315 PCR片段;3-ARS735 PCR片段;4-ARS1512 PCR片段图2 自动复制区序列 PCR扩增产物琼脂糖凝胶电泳图Fig.2 Agarose gel electrophoresis of ARS PCR production
将PCR产物回收后,分别用NdeI和EcoRI双酶切,然后连接到整合载体pNTS2K上,转化到大肠杆菌DH5α中,进行大肠杆菌菌落PCR鉴定,挑取鉴定后的阳性克隆培养,提取质粒,NdeI和EcoRI双酶切鉴定结果如图3所示。
从图3中可以看出,各重组质粒经双酶切得到2条带,对照质粒pNTS2K得到1条带,且大小都与预期相符.同时,将所得阳性质粒送往公司测后,NCBI比对与S288C酿酒酵母相关ARS序列一致。

M-DNA标记;C-对照质粒pNTS2K;1-质粒pNTS2K-ARS304;2-质粒pNTS2K-ARS315;3-质粒pNTS2K-ARS735;4-质粒pNTS2K-ARS1512图3 重组质粒酶切鉴定琼脂糖凝胶电泳图Fig.3 Digest verification of recombinant plasmid
2.2 酵母转化子的获得
在相同条件下,由于单细胞内质粒拷贝数的多少直接影响细胞抗性能力的强弱,所以酵母转化效率以及转化子单克隆大小是鉴定ARS自主复制活性强弱的直接指标。本实验以整合质粒pNTS2K为阳性对照,将构建好的含ARS复制区的复制整合型质粒pNTS2K-304、pNTS2K-315、pNTS2K-735和pNTS2K-1512电转化酿酒酵母Juk36a后,在YPD平板上筛选耐G418的转化子。根据酵母转化率以及克隆大小初步评估4种复制区自主复制能力,结果见图4。从图4中可以看出,4种重组质粒转化酵母后都能在含有300 μg/mL G418的YPD平板上产生大量的转化子,且克隆明显多且大于对照组。还可以看出,转化子36a(pNTS2K-ARS315)和36a(pNTS2K-ARS1512)克隆大小较其他2组要大。上述结果说明这4条ARS都具有独立自主复制能力,且ARS315和ARS1512复制能力可能较强。经统计分析,各种重组质粒转化率如图5所示。从图5中可以看出,4种重组质粒的转化率(转化子数/ng DNA)分别为63,57,41和32,明显高于对照组。对照组pNTS2K的复制依赖于其整合到染色体DNA上,转化率低(5个转化子/ng DNA)。这也初步说明了4条ARS都具有独立自主复制能力。转化率只是代表质粒是否存在于细胞中,可初步地判断质粒在细胞中的稳定性,而单克隆的大小及抗性的强弱则代表拷贝数的多少及稳定性。所以,虽然质粒pNTS2K-315 的转化率略低于pNTS2K-304,但转化子单克隆明显较大,这说明质粒pNTS2K-315以更多的拷贝存在于单细胞中,表达特异性蛋白更多,对G418耐受性更强。这进一步说明了ARS315复制能力可能会更强。

质粒pNTS2K的酵母转化子;36a(pNTS2K-ARS304):质粒pNTS2K-ARS304的酵母转化子;36a(pNTS2K-ARS315):质粒pNTS2K-ARS315的酵母转化子;36a(pNTS2K-ARS735):质粒pNTS2K-ARS735的酵母转化子;36a(pNTS2K-ARS1512):质粒pNTS2K-ARS1512的酵母转化子图4 转化子在含300 μg/mL G418的YPD平板上生长情况Fig.4 Transformants growth on the YPD plate with 300 μg/mL

C-对照质粒pNTS2K转化率;ARS304-重组质粒pNTS2K-ARS304转化率;ARS315-重组质粒pNTS2K-ARS315转化率;ARS735-重组质粒pNTS2K-ARS735转化率;ARS1512-重组质粒pNTS2K-ARS1512转化率图5 重组质粒转化率(转化率=转化子数/质粒DNA的质量)Fig.5 Transformation rate of recombinant plasmids (Transformation rate=Number of transformants/quality of plasmid DNA)
2.3 四种转化子抗性生长比较
为了进一步筛选出复制能力最强的ARS,从所得到的转化子中各挑取5个最大的单克隆于2 mL YPD 培养基中培养过夜,以OD600nm为0.05转接到含有500 μg/mLG418的5 mL YPD培养基中培养48 h后,分别测OD600nm处的吸光值,结果如图6所示。含pNTS2K-ARS315重组质粒的转化子生长OD最高,平均为8.55;对照组pNTS2K质粒的转化子生长OD最低,平均为0.57。这也进一步说明了4条ARS都具有独立自主复制能力,其中ARS315复制能力最强。

C-对照转化子36a(PNTS2K)吸光值;ARS304-转化子36a(pNTS2K-ARS304)吸光值;ARS315-转化子36a(pNTS2K-ARS315)吸光值;ARS735-转化子36a(pNTS2K-ARS735)吸光值;ARS1512-转化子36a(pNTS2K-ARS1512)吸光值图6 四种转化子抗性生长比较(该数值为5组独立实验的平均值和标准差)Fig.6 The resistance comparison of the four transformants (Values are given as the average and standard deviation of five independent experiments)
2.4 ARS315复制能力的检测
为了检测ARS315的独立自主复制能力,将转化子36a(pNTS2K-ARS315)接种于含不同梯度的G418的YPD培养基中,在InfiniteF200PRO中监测生长曲线,结果如图7所示。从图7可以看出,36a(pNTS2K-ARS315)在含1 mg/mL G418以下浓度的YPD中均能很好地生长,在含1 mg/mL G418以上浓度的YPD中生长明显受到抑制。这说明pNTS2K-ARS315能够在酿酒酵母中高表达且能抵御1 mg/mL 高浓度G418的毒性,进一步说明了pNTS2K-ARS315有很强的复制能力且以高拷贝的形式存在于酵母细胞中。

图7 转化子36a (pNTS2K-ARS315)不同浓度梯度G418耐受性检测(该数值为5组独立实验的平均值和标准差)Fig.7 The tolerance of transformant 36a (pNTS2K-ARS315) to different concentrations of G418(Values are given as the average and standard deviation of five independent experiments)
2.5 复制整合型载体pNTS2K-ARS315表达能力检测
为了检测质粒pNTS2K-ARS315是否能正常表达其它蛋白及其表达能力。将得到的含荧光蛋白的质粒pNTS2K-ARS315-EGFP和pPY1-EGFP转化酵母后,都能得到发荧光的转化子36a(pNTS2K-ARS315-EGFP)和36a(pPY1-EGFP),如图8所示。

图8 转化子在荧光显微镜下,放大1 000倍的发光状态Fig.8 The fluorescence performance of transformants under fluorescence microscope magnified 1 000 times
在荧光显微镜下,以ARS315为复制区的表达载体表达的荧光蛋白的转化子36a(pNTS2K-ARS315-EGFP)产生的荧光强度明显较强。此外,使用流式细胞仪进一步检测两种转化子,结果如图9所示,从图9可以看出,转化子36a(pNTS2K-ARS315-EGFP)所产生的带荧光的细胞较36a(pPY1-EGFP) 明显多且强[11]。这说明以高效复制能力的ARS315为复制区的表达载体表达水平更高。

图9 流式细胞仪检测转化子荧光强度Fig.9 The fluorescence idensity of transformants measured by flow cytometry
为了进一步检查表达水平的稳定性,将带有荧光的转化子分别挑取10个单克隆,接种于YPD培养基中培养48 h,在InfiniteF200PRO中检测比荧光强度(FIC)和OD595nm,结果如图10所示。从图10可以看出,转化子36a(pNTS2K-ARS315-EGFP)在YPD中培养48h后,产生的比荧光强度较36a(pPY1-EGFP)强3倍。这也进一步证明了pNTS2K-ARS315有很强的复制能力且以高拷贝的形式稳定存在于酵母细胞中。

图10 转化子比荧光强度的检测(该数值为10个单克隆菌株独立实验的平均值和标准差)Fig.10 The measurement of transformants fluorescence density rate (Values are given as the average and standard deviation of ten single colonies independent experiments)
3 讨论
在酿酒酵母的每个复制周期中,实际被激活的复制区较少。虽然大多数ARS都能被复制起始识别复合体(Origin recognition complex, ORC)结合[12],但其中有些复制区被激活的频率比其他要高,ARS315能在约90%的细胞循环中启动复制[13]。当这些复制区克隆到载体上时,这些序列也能行使独立自主复制的功能[14]。而来自转座子Tn903的G418抗性基因KanMX应用较为广泛,并能通过提高G418的浓度筛选出高拷贝的转化子[15]。构建一种既能独立自主复制,又能将外源基因整合到染色体的载体,优点是可以自由调节外源基因在宿主中的拷贝数[16],从而达到代谢路径通畅且代谢平衡。本实验证实四种复制区都能在酿酒酵母中独立自主复制,其中ARS315复制能力最强,其转化子36a(pNTS2K-ARS315)能在含高达1 mg/mL G418的YPD培养基中正常生长, 而且该质粒能高效表达荧光蛋白。因此,ARS315可用于构建高拷贝且稳定的重组质粒,为酿酒酵母进行基因工程改造建立了技术平台。
[1] YAMADA R,TANAKA T,OGINO C,et al.Gene copy number and polyploidy on products formation in yeast[J].Applied Microbiology and Biotechnology, 2010,88(4):849-857.
[2] DESHPANDE A M,NEWLON C S.The ARS consensus sequence is required for chromosomal origin function inSaccharomycescerevisiae[J]. Molecular and Cellular Biology,1992,12(10):4 305-4 313.
[3] YANG C,THEIS J F,NEWLON C S.Conservation of ARS elements and chromosomal DNA replication origins on chromosomes III ofSaccharomycescerevisiaeandS.carlsbergensis[J].Genetics,1999,152(3):933-941.
[4] THEIS J F,NEWLON C S.Two compound replication origins inSaccharomycescerevisiaecontain redundant origin recognition complex binding sites[J].Molecular and Cellular Biology, 2001,21(8):2 790-2 801.
[5] FENG W, COLLINGWOOD D, BOECK M E,et al.Genomic mapping of single-stranded DNA in hydroxyurea-challenged yeasts identifies origins of replication[J].Nature Cell Biology, 2006,8(2):148-155.
[6] YABUKI N,TERASHIMA H,KITADA K.Mapping of early firing origins on a replication profile of budding yeast[J].Genes to Cells,2002,7(8):781-789.
[7] WYRICK J J,APARICIO J G,CHEN T, et al.Genome-wide distribution of ORC and MCM proteins inS.cerevisiae: high-resolution mapping of replication origins[J].Science,2001,294(5 550):2 357-2 360.
[8] EATON M L,GALANI K,KANG S,et al.Conserved nucleosome positioning defines replication origins[J].Genes & Development,2010,24(8):748-753.
[9] SUGA M,HATAKEYAMA T.High-efficiency electroporation by freezing intact yeast cells with addition of calcium[J]. Current Genetics,2003,43(3):206-211.
[10] KIM B, DU J, ERIKSEN D T, et al. Combinatorial design of a highly efficient xylose-utilizing pathway inSaccharomycescerevisiaefor the production of cellulosic biofuels[J]. Applied and Environmental Microbiology, 2013, 79(3): 931-941.
[11] SHI Shuo-bo,LIANG You-yun,ZHANG Ming-zi M,et al.A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways inSaccharomycescerevisiae[J].Metabolic Engineering,2016,33: 19-27.
[12] SANTOCANALE C,DIFFLEY J F.ORC-and Cdc6-dependent complexes at active and inactive chromosomal replication origins inSaccharomycescerevisiae[J].The EMBO Journal,1996,15(23):6 671.
[13] NEWLON C S, COLLINS I, DERSHOWITZ A, et al. Analysis of replication origin function on chromosome III ofSaccharomycescerevisiae[C].Cold Spring Harbor Symposia on Quantitative Biology. Cold Spring Harbor Laboratory Press, 1993, 58: 415-423.
[14] VUJCIC M,MILLER C A,KOWALSKI D.Activation of silent replication origins at autonomously replicating sequence elements near the HML locus in budding yeast[J].Molecular and Cellular Biology,1999,19(9):6 098-6 109.
[15] SCORER C A,CLARE J J,MCCOMBIE W R,et al.Rapid selection using G418 of high copy number transformants ofPichiapastorisfor high-level foreign gene expression[J].Nature Biotechnology,1994,12(2):181-184.
[16] SHEN Y, MURAMATSU S I, IKEGUCHI K,et al.Triple transduction with adeno-associated virus vectors expressing tyrosine hydroxylase, aromatic-L-amino-acid decarboxylase, and GTP cyclohydrolase I for gene therapy of Parkinson's disease[J].Human Gene Therapy,2000,11(11):1 509-1 519.
A high effective autonomous replicative sequence inSaccharomycescerevisiae
SHEN Fang-lin1,3, HUANG Shuang-cheng2, HOU Peng-chen2, GENG An-li2, RUAN Wen-quan1,3*
1(School of Environment and Civil Engineering, Jiangnan University, Wuxi 214122, China) 2(School of Life Sciences and Chemical Technology, Ngee Ann Polytechnic, Singapore) 3(Jiangsu Key Laboratory of Anaerobic Biotechnology, Jiangnan University, Wuxi 214122, China)
In the case of yeast expression, low-copy number of transformed genes and low stability are disadvantages of the conventional types of expression vectors. Therefore, finding a high effectiveSaccharomycescerevisiaeautonomously replicative sequence (ARS) that can be used for construction of replicative/ integrated plasmid is significantly important. In this study, four different ARSs (ARS304, ARS315, ARS735, and ARS1512) were amplified fromSaccharomycescerevisiaeand inserted into integrated yeast vector pNTS2K and four plasmids pNTS2K-ARS304, pNTS2K-ARS315, pNTS2K-ARS735, pNTS2K-ARS1512 were obtained. All the four plasmids were successfully transformed intoS.cerevisiaeand all transformants were able to grow on YPD plate containing 300 μg/mL of G418. However, only transformant containing plasmid pNTS2K-ARS315 was able to grow well in the YPD medium containing 500 μg/mL of G418. After G418 tolerance detection, 36a (pNTS2K-ARS315) still could grow well in YPD medium containing 1 mg/mL of G418. Furthermore, the expression of the fluorescent protein by the plasmid pNTS2K-ARS 315 was higher than that by the ordinary commercial plasmid. It can be concluded that all of the four ARSs functioned and the ARS315 had the highest replicate ability inS.cerevisiae. Consequently, ARS315 can be potentially used for high expression and stable recombinant strain construction and metabolic engineering ofS.cerevisiae.
Saccharomycescerevisiae; autonomously replicative sequence; high copy number replicative/ integrated plasmid;fluorescent protein
硕士研究生(阮文权教授为通讯作者,E-mail: wqruan@jiangnan.edu.cn)。
Science and Engineering Research Council of the Agency for Science, Technology and Research(A*STAR)(029 139 0035)
2016-09-23,改回日期:2016-12-07
