冷鲜鸡污染微生物磺胺类和喹诺酮类耐药基因分析
2017-04-26何祥祥戴宝玲夏效东肖英平
何祥祥,杨 华,戴宝玲,夏效东,肖英平,*
(1.西北农林科技大学 食品科学与工程学院,陕西 杨凌 712100; 2.浙江省农业科学院 农产品质量标准研究所,浙江 杭州 310021; 3.浙江省植物有害生物防控省部共建国家重点实验室培育基地,浙江 杭州 310021)
冷鲜鸡污染微生物磺胺类和喹诺酮类耐药基因分析
何祥祥1,2,3,杨 华2,3,戴宝玲2,3,夏效东1,肖英平2,3,*
(1.西北农林科技大学 食品科学与工程学院,陕西 杨凌 712100; 2.浙江省农业科学院 农产品质量标准研究所,浙江 杭州 310021; 3.浙江省植物有害生物防控省部共建国家重点实验室培育基地,浙江 杭州 310021)
动物养殖业的发展过程中抗生素的长期使用,导致耐药菌的快速发展。这些耐药菌在动物养殖过程中逐渐被抗生素筛选,在动物性食品生产链中容易被携带传播,对人类食品安全产生很大的威胁。试验选取浙江省绍兴市8个大型超市和11个农贸市场销售的冷鲜鸡为检测对象,采用PCR和测序验证的技术手段,对冷鲜鸡表面微生物中的2种磺胺类耐药基因sulⅠ、sulⅡ和8种喹诺酮类基因qnrA、qnrB、qnrC、qnrD、qnrS、qepA、oqxA与oqxB进行检测。在所检测的样品中,检测出sulⅠ、sulⅡ、qnrD、qnrS、qepA和oqxB基因,检出率分别为100%、100%、100%、89.5%、100%和89.5%。并对所检出的耐药基因进行测序和NCBI收录的耐药基因序列进行Blast比对验证,比对结果同源性分别为100%、99.44%、100%、92.74%、99.82%和100%。试验结果将为冷鲜鸡中耐药基因的风险评估提供有力支持。
冷鲜鸡;细菌;磺胺类;喹诺酮类;耐药基因
目前国内多数城市如上海、北京和杭州已经全面禁止活禽买卖交易[1],冷鲜鸡因其肉质纯熟,适合烹饪和口味鲜美的特点代替了“活杀鸡”走向百姓的餐桌[2]。然而,在禽类养殖中抗生素的普遍使用引起了人们较大的关注[3-6]。抗菌药物在欧洲[7-8]和北美[9]已经被广泛使用了近半个世纪,在提高肉制品产量的同时也导致了细菌耐药性的产生[10-11]。例如Yildirim等[12]在200份零售的冷鲜鸡中分离出68株沙门氏菌,通过对耐药基因的检测,这些沙门氏菌对青霉素、苯唑西林、克林霉素、万古霉素、红霉素和氨苄西林等抗生素具有不同程度的耐药性,同时也有半数以上的沙门氏菌对四环素、链霉素、新霉素和头孢霉素产生耐药性。动物性食品中耐药性基因的出现引起了人们的极大关注,因为耐药性基因是通过可移动的介质比如质粒、转座子或染色体[13-14]来传播,另外抗生素耐药基因也可以在细菌之间相互传播[15]。冷鲜鸡在从养殖到宰杀、运输储藏再到消费者餐桌上[16-17]的过程的产业链中也会携带大量的耐药细菌,对食品安全和人们的健康将产生很大的威胁[18]。
本试验从浙江绍兴市19个不同的农贸市场和超市分别采冷鲜鸡,然后通过直接提取表面微生物的DNA,通过PCR扩增耐药基因的方法,检测总细菌的耐药性。试验结果将为冷鲜鸡表面微生物的耐药监测提供有力依据。
1 材料与方法
1.1 材料与试剂
1.1.1 样品来源
19只冷鲜鸡样品于2016年8月6日采自浙江省绍兴市的8个大型超市和11个农贸市场,编号为1~19。
1.1.2 仪器与试剂
PCR扩增仪(美国bio-rad公司),凝胶成像系统(美国bio-rad公司),微量核酸蛋白分析仪(Thermo Fisher公司)。BPW培养基(杭州微生物试剂有限公司),微生物基因组DNA提取试剂盒(Zymo Research Fungal/Bacterial DNA MiniPrepTM)。
1.2 实验方法
1.2.1 样品处理
将从市场上采集来的冷鲜鸡放置于无菌袋中,按照每500 g冷鲜鸡加入500 mLBPW冲洗液,密封后于摇床上振荡(200 r·min-1)15~20 min,然后取50 mL冲洗下来的BPW溶液于37 ℃烘箱,培养12 h。
1.2.2 基因组DNA提取
将增菌后的BPW冲洗液按照微生物基因组DNA提取试剂盒(Zymo Research Fungal/Bacterial DNA MiniPrepTM)说明书,提取总细菌DNA。然后用微量核酸蛋白质分析仪(Nanodrop)对所提取的DNA进行含量和纯度(D260/D280)的测定。
1.2.3 耐药基因的PCR扩增
本试验从磺胺类和喹诺酮类耐药基因中共选取了10种耐药基因,其中包括2种磺胺类基因(sulⅠ、sulⅡ)和8种喹诺酮类基因(qnrA、qnrB、qnrC、qnrD、qnrS、qepA、oqxA和oqxB)。所用引物参照文献[19],见表1。PCR扩增反应程序为95 ℃,5 min预变性,然后94 ℃,30 s,58 ℃,30 s;72 ℃,1 min,共32个循环;最后16 ℃保存。扩增反应后进行琼脂糖凝胶电泳并成像。
表1 PCR反应引物
Table 1 PCR primers used in this study

目的基因Targetgene引物序列(5’-3’)Primersequence产物长度Length/bpsulⅠF:CGCACCGGAAACATCGCTGCACR:TGAAGTTCCGCCGCAAGGCTCG163sulⅡF:TCCGGTGGAGGCCGGTATCTGGR:CGGGAATGCCATCTGCCTTGAG191qnrAF:AGAGGATTTCTCACGCCAGGR:TGCCAGGCACAGATCTTGAC580qnrBF:GGATGAAATTCGCCACTGR:TTTGCGCGCCAGTCGAA264qnrCF:GGGTTGTACATTTATT-GAATCGR:CACCTACCCATTTATTTTCA307qnrDF:CGAGATCAATTTACGGGGAA-TAR:AACAAGCTGAAGCGCCTG465qnrSF:GCAAGTTCATTGAACAGGGTR:TCTAAACCGTCGAGTTCGGCG428qepAF:CCAGCTCGGCAACTTGATACR:ATGCTCGCCTTCCAGAAAA570oqxAF:CTCGGCGCGATGATGCTR:CCACTCTTCACGGGAGACGA392oqxBF:TCCTGATCTCCATTAACGCCCAR:ACCGGAACCCATCTCGATGC131
1.2.4 检出耐药基因的测序和序列比对
将目的条带割胶后送至杭州浩基生物公司,对PCR产物切胶回收,加尾后和pGM-Simple-T Fast Vector进行连接,转化到感受态细胞DH5α,筛选阳性克隆进行测序。将返回的测序结果通过NCBI网站(https://www.ncbi.nlm.nih.gov/blast)比对。
2 结果与分析
2.1 基因组DNA提取和耐药基因检测结果
通过对浙江省某市超市和农贸市场上共19只冷鲜鸡的表面微生物提取总DNA如图1,然后以总DNA为模板,对10种耐药基因的检测,共检测出6种耐药基因如表2和图2。其中包括2种磺胺类耐药基因(sulⅠ、sulⅡ)和4种喹诺酮类耐药基因(qnrD、qnrS、qepA、oqxB)。
2.2 被检出的耐药基因测序比对结果
将测序结果使用NCBI网站(https://www.ncbi.nlm.nih.gov/blast)的BLAST进行序列同源性比对,发现检测到的耐药基因PCR产物与以往文献报道的耐药基因有较高的同源性。在BLAST中进行的序列比对同源性结果如表3。

M,DNA Marker(DL5000); 1~8分别为来自大型超市的样品M, DNA ladder; 1-8, Samples from supermarkets图1 基因组DNA电泳图Fig.1 Electrophoretic results of genome DNA
表2 耐药基因的PCR检测结果
Table 2 The PCR results of antibiotic genes

耐药基因Drugresistancegene检出率Detectionrate/%超市样品Samplesfromsupermarket农贸市场样品Samplesfromfarmersmarket磺胺类sulⅠ100100SulfonamidessulⅡ100100喹诺酮类qnrA00QuinolonesqnrB00qnrC00qnrD100100qnrS10081.8qepA100100oqxA00oqxB87.590.9
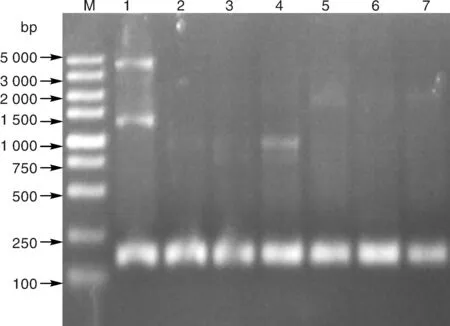
M: DNA marker(DL5000); 1~8:来自大型超市的样品M: DNA ladder; 1-8: Samples from supermarkets图2 耐药基因电泳结果Fig.2 Electrophoretic results of antibiotic resistance gene
表3 耐药基因序列同源性比对结果
Table 3 The results of antibiotic resistance gene sequences comparing to ones who have been deposited into the GenBank

耐药基因DrugresistancegeneNCBI中基因序列号AccessionnumberinGenBank相似度Similarity/%sulⅠJN003421100sulⅡHQ44116999.44qnrDHM056769100qnrSHQ64066192.74qepAFJ74412199.82oqxBHQ674771100
3 结论与讨论
由于抗生素的大量使用,导致家禽养殖中细菌耐药性的提高。岳磊等[20]对广东地区分离得到的84株鸡源肠杆菌进行药敏检测,结果表明,这些鸡源肠杆菌对氟罗沙星(98.8%),恩诺沙星(84.5%)等喹诺酮类抗生素产生不同程度的抗性。磺胺类药物在家禽养殖中,主要被用来治疗和预防鸡白痢。然而近些年的广泛使用,逐渐造成鸡白痢沙门氏菌对磺胺类药物的耐药性[21]。董洪燕等[22]对江苏东台地区分离得到的33株鸡白痢沙门氏菌检测,94.4%的菌株含有耐药基因sulⅠ。宁家宝等[23]对珠江三角洲地区分离到的69株鸡白痢沙门氏菌进行检测,其中sulⅠ和sulⅡ基因的检出率分别为33.3%和79.7%。
本试验所选取的2个磺胺类耐药基因和8个喹诺酮类耐药基因,具有一定的代表性。这些耐药基因在家禽类的养殖中较为常见,对于细菌耐药基因在环境中的传播、扩散具有一定的指示作用。本试验没有采用细菌的分离培养的方法,而是直接对冷鲜鸡表面总细菌进行DNA的提取,对具有代表性的磺胺类和喹诺酮类耐药基因进行PCR扩增和电泳,共有6种基因被检测出来,经过序列比对后,所检出的基因相似度均在92.74%以上,说明检测结果可靠。其中包括2种磺胺类耐药基因(sulⅠ、sulⅡ)和3种喹诺酮类耐药基因(qnrS、qnrD、qepA、oqxB)。从电泳图中亮度可以看出,sulⅠ、sulⅡ、qnrD、qnrS和oqxB基因在样本间的差异不大,qepA在样品间的差异较大。这可能与样品采自不同市场有关。而其他5个喹诺酮类耐药基因(qnrA、qnrB、qnrC、oqxA、oqxB)均未检出。
细菌的耐药性与耐药基因产生的关系。微生物的耐药基因的产生一方面来源于微生物固有的耐药基因,另一方面来源于自身基因突变或通过微生物之间的的接合、转化、转导等相互作用而获得[24-25]。细菌的抗生素耐药基因主要有三种作用机制,第一种是通过产生抗生素的水解酶,使抗生素失去活性或分解。比如aac(6’)-Ib-cr可以编码灭活酶,使喹诺酮类抗生素灭活而失去活性[26]。第二种是通过改变抗生素作用的靶点,降低抗生素与细菌的结合能力,这种方式比较普遍。比如四环素耐药基因tetM、tetW、tetO、tetQ介导的耐药机制就是通过产生的核糖体保护蛋白,抑制核糖体与四环素的相互作用[27]。第三种机制是通过改变细菌膜的通透性而形成药物外排系统,将药物外排,使得菌体内抗生素达不到有效作用浓度。比如tetC等基因介导的耐药机制[27]。
综合本试验对磺胺类和喹诺酮类耐药基因的检测结果,可以得出家禽养殖场的耐药基因可能会通过微生物携带到冷鲜鸡的表面,对消费者的健康会产生一定的影响。另一方面,养殖场抗生素残留和耐药基因产生的相关性有待进一步研究,这对于分析耐药基因的产生是由于自然因素还是人为因素导致的,意义重大。
[1] 胡卫中, 周自明. 消费者对冷鲜鸡支付意愿的影响因素研究[J]. 中国畜牧杂志, 2016, 52(4):48-51. HU W Z, ZHOU Z M. Factors influencing consumer’s willingness to pay for chilled chicken[J].ChineseJournalofAnimalScience, 2016, 52(4):48-51. (in Chinese with English abstract)
[2] 范京辉, 张永华. 冷鲜鸡的质量要求及其影响因素[J]. 杭州农业与科技, 2014(3):23-25. FAN J H, ZHANG Y H. Chilled chicken quality and its influencing factors[J].HangzhouAgriculturalScienceandTechnology, 2014(3):23-25. (in Chinese)
[3] TEUBER M. Spread of antibiotic resistance with food-borne pathogens [J].Cellular&MolecularLifeSciences, 1999, 56(9/10): 755-763.
[4] SCHWARZ S, KEHRENBERG C, WALSH T R. Use of antimicrobial agents in veterinary medicine and food animal production [J].InternationalJournalofAntimicrobialAgents, 2001, 17(6): 431-437.
[5] NORMARK B H, NORMARK S. Evolution and spread of antibiotic resistance [J].JournalofInternalMedicine, 2002, 252(2): 91-106.
[6] TURNIDGE J. Antibiotic use in animals—prejudices, perceptions and realities [J].JournalofAntimicrobialChemotherapy, 2004, 53(1): 26-27.
[7] WEGENER H C, AARESTRUP F M, JENSEN L B, et al. Use of antimicrobial growth promoters in food animals andEnterococcusfaeciumresistance to therapeutic antimicrobial drugs in Europe[J].EmergingInfectiousDiseases, 1999, 5(3): 329-335.
[8] DRITZ S S, TOKACH M D, GOODBAND R D, et al. Effects of administration of antimicrobials in feed on growth rate and feed efficiency of pigs in multisite production systems[J].JournaloftheAmericanVeterinaryMedicalAssociation, 2002, 220(11): 1690-1695.
[9] National Research Council. The use of drugs in food animals, benefits and risks[M]. Washington DC: National Academy Press, 1999.
[10] PHILLIPS I, CASEWELL M, COX T, et al. Does the use of antibiotics in food animals pose a risk to human health? A critical review of published data[J].JournalofAntimicrobialChemotherapy, 2004, 53(1): 28-52.
[11] LU K, ASANO R, DAVIES J. Antimicrobial resistance gene delivery in animal feeds[J].EmergingInfectiousDiseases, 2004, 10(4): 679-683.
[12] YILDIRIM Y, GONULALAN Z, PAMUK S, et al. Incidence and antibiotic resistance ofSalmonellaspp. on raw chicken carcasses[J].FoodResearchInternational, 2011, 44(3):725-728.
[13] SALYERS A A, SHOEMAKER N B, STEVENS A M, et al. Conjugative transposons: an unusual and diverse set of integrated gene transfer elements[J].MicrobiologicalReviews, 1995, 59(4): 579-590.
[14] ROWE-MAGNUS D A, GUEROUT A M, PLONCARD P, et al. The evolutionary history of chromosomal super-integrons provide sanancestry for multiresistant integrons[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2001, 98(2): 652-657.
[15] TEALE C J. Antimicrobial resistance and the food chain[J].JournalofAppliedMicrobiology, 2002, 92(S1): 85-89.
[16] HAYES J R, ENGLISH L L, CARTER P J, et al. Prevalence and antimicrobial resistance ofEnterococcusspecies isolated from retail meats[J].Applied&EnvironmentalMicrobiology, 2003, 69(12): 7153-7160.
[17] FRANZ C M, STILES M E, SCHLEIFER K H, et al. Enterococci in food-a conundrum for food safety[J].InternationalJournalofFoodMicrobiology, 2003, 88(2/3): 105-122.
[18] DONABEDIAN S M, THAL L A, HERSHBERGER E, et al. Molecular characterization of gentamicin-resistant enterococci in the United States: evidence of spread from animals to humans through food[J].JournalofClinicalMicrobiology, 2003, 41(3): 1109-1113.
[19] 李娟.养猪场废水、周边土壤中抗生素及耐药基因污染状况的研究[D]. 北京:中国农业大学,2012. LI J. The levels of pollution about antibiotic residues and antibiotic resistance genes(ARGs) in swine feedlot wastewater and surrounding lands[D]. Beijing: China Agricultural University, 2012.
[20] 岳磊, 蒋红霞, 刘健华,等. 鸡源肠杆菌质粒介导喹诺酮类耐药基因检测[J]. 中国农业科学, 2009, 42(8):2966-2971. YUE L, JIANG H X, LIU J H, et al. Detection of plasmid-mediated quinolone resistance in clinical isolates of enterobacteriaceae from avian[J].ScientiaAgriculturaSinica, 2009, 42(8):2966-2971. (in Chinese with English abstract)
[21] 潘志明, 焦新安, 刘学贤,等. 鸡白痢沙门氏菌耐药性的变化趋势[J]. 中国预防兽医学报, 1999(4):305-307. PAN Z M, JIAO X A, LIU X X, et al. Changes of drug resistance of salmonella pullorum[J].ChineseJournalofPreventiveVeterinaryMedicine, 1999(4):305-307. (in Chinese with English abstract)
[22] 董洪燕, 缪晓斌, 李鑫,等. 鸡白痢沙门菌分离株的耐药性及磺胺类耐药机制研究[J]. 中国家禽, 2010, 32(9):29-33. DONG H Y, MIU X B, LI X, et al. Drug resistance pattern ofSalmonellapullorumisolates and mechanism of sulfonamides resistance[J].ChinaPoultry, 2010, 32(9):29-33. (in Chinese with English abstract)
[23] 宁家宝, 陈建红, 张济培,等. 鸡白痢沙门菌的耐药性及磺胺类耐药基因的研究[J]. 中国家禽, 2014, 36(3):18-20. NING J B, CHEN J H, ZHANG J P, et al. Drug resistance pattern and resistance genes for sulfonamides ofSalmonellapullorum[J].ChinaPoultry, 2014, 36(3):18-20. (in Chinese with English abstract)
[24] CHOPRA I, ROBERTS M. Tetracycline antibiotics: mode of action, applications, molecular biology, and epidemiology of bacterial resistance[J].Microbiology&MolecularBiologyReviews, 2001,65(2):232-260.
[25] ZHANG X X, ZHANG T, FANG H H P. Antibiotic resistance genes in water environment[J].AppliedMicrobiologyandBiotechnology, 2009, 82(3):397-414.
[26] ROBICSEK A, STRAHILEVITZ J, JACOBY G A, et al. Fluoroquinolone-modifying enzyme: a new adaptation of a common aminoglycoside acetyltransferase[J].NatureMedicine, 2006, 12(1):83-88.
[27] DE WASCH K, OKERMAN L, CROUBELS S, et al. Detection of residues of tetracycline antibiotics in pork and chicken meat: correlation between results of screening and confirmatory tests [J].Analyst, 1998, 123(12):2737-2741.
(责任编辑 张 韵)
Detection of the antibiotic genes of sulfonamides and quinolones of bacteria in refrigerated chicken
HE Xiangxiang1,2,3, YANG Hua2,3, DAI Baoling2,3, XIA Xiaodong1, XIAO Yingping2,3,*
(1.CollegeofFoodScienceandEngineering,NorthwestA&FUniversity,Yangling712100,China;2.InstituteofQualityandStandardforAgro-products,ZhejiangAcademyofAgriculturalSciences,Hangzhou310021,China;3.StateKeyLaboratoryBreedingBaseforZhejiangSustainablePestandDiseaseControl,ZhejiangAcademyofAgriculturalSciences,Hangzhou310021,China)
The long-term use of antibiotics in the animal farming industry has caused rapid expansion of drug resistant bacteria. These drug resistant bacteria are screened during the process of animal farming, and are easily transmitted in the animal production chain, which is a huge threat to human’s food safety. In this study, we aimed at testing the safety of refrigerated chicken collected from eight supermarkets and eleven agricultural markets in Shaoxing city, Zhejiang Province. We detected the antibiotic genes of sulfonamides and quinolones of surface microorganisms of the samples by PCR reaction and sequencing. We detectedsulⅠ,sulⅡ,qnrD,qnrS,qepAandoqxBin the samples and the detection rate was 100%, 100%, 100%, 89.5%, 100% and 89.5%, respectively. We also sequenced the antibiotic genes with the sequences in NCBI and the coincidence rate was 100%, 99.44%, 100%, 92.74%, 99.82% and 100%, respectively. This study will provide effective support for risk assessment of antibiotic genes in the refrigerated chicken.
refrigerated chicken; bacteria; sulfonamides; quinolones; drug-resistance genes
http://www.zjnyxb.cn何祥祥,杨华,戴宝玲,等. 冷鲜鸡污染微生物磺胺类和喹诺酮类耐药基因分析[J].浙江农业学报,2017,29(4): 555-559.
10.3969/j.issn.1004-1524.2017.04.06
2016-11-07
浙江省公益技术运用研究项目(2016C32073);浙江省植物有害生物防控重点实验室——省部共建国家重点实验室培育基地(2010DS700124-ZM1608)
何祥祥(1991—),男,河南洛阳人,硕士研究生,研究方向为食品科学。E-mail:xianghe2009@126.com
*通信作者,肖英平,E-mail: ypxiaozju@126.com
S879.2
A
1004-1524(2017)04-0555-05
浙江农业学报ActaAgriculturaeZhejiangensis, 2017,29(4): 555-559
