CD1蛋白家族及CD1蛋白和TCR相互作用的研究进展
2017-02-27富霄鹏
富霄鹏
(英国牛津大学医学院,英国牛津OX3 9DS)
CD1蛋白家族及CD1蛋白和TCR相互作用的研究进展
富霄鹏
(英国牛津大学医学院,英国牛津OX3 9DS)
从20世纪发现主要组织相容复合体(MHC)分子以来,对MHC、多肽、T细胞抗原受体(TCR)之间相互识别和相互作用的机制的研究进行了几十年,是免疫学最重要的研究领域之一。多肽可以作为抗原被MHC分子识别并结合,呈递给T细胞,形成TCR-抗原-MHC复合物,进而激活下游信号通路,开启免疫反应:这一过程是由T细胞介导的细胞免疫的核心[1-3]。围绕抗原呈递细胞(APC)、多肽抗原及特异性T细胞的广泛研究不断拓宽了人类对免疫系统的认识,在此基础上研发的疫苗和药物产品在疾病预防、诊断和治疗领域也有大范围的应用。例如,利用HIV病毒毒株的蛋白片段筛选并研究HIV广谱中和性抗体的作用机制,其中部分广谱中和性抗体经实验验证可能对艾滋病的治疗有帮助[4]。
然而最近的研究表明,多肽-MHC复合物并不是唯一可以被T细胞识别的分子。越来越多的实验证据表明,部分αβ T细胞能够识别由非多态性抗原呈递分子呈递的抗原,例如由HLA-E呈递的多肽[5-7]、CD1蛋白呈递的脂质[8,9]、MR1蛋白呈递的核黄素衍生物等[10,11]。在这一过程中,分布在抗原呈递细胞表面的非多态性抗原呈递分子扮演了类似MHC分子的角色。相比较于MHC分子,我们对于非多态性抗原呈递分子了解甚少。研究表明,非多态性抗原呈递分子在识别自身抗原及来自致病菌(如分枝杆菌[12,13])的外源抗原的过程中发挥作用,对于它们的研究将有助于进一步了解过敏、自身免疫疾病、细菌和真菌感染等疾病的致病机理,并为临床治疗提供思路。
本文将介绍非多态性抗原呈递蛋白家族CD1,并着重论述CD1蛋白和TCR相互作用的研究进展。该蛋白能够将来自自身和病原体的脂质及小分子物质作为抗原呈递给T细胞,并激活相应的免疫反应。除αβ T细胞外[14],γδ T细胞[15,16]和δ/αβ T细胞[17]也能识别CD1蛋白,暗示这一蛋白家族成员可能具有广泛的免疫调节作用。
1 CD1蛋白家族及其分类
CD1蛋白是一类非多态性抗原呈递分子,其编码基因位于MHC基因位点之外[13,18-21]。该种蛋白的分布十分广泛,在所有的胎盘哺乳动物、鸟类和有袋类动物中均有表达[22]。人类能够表达5种CD1基因[23],部分物种(如豚鼠[24]和狗[25])能够表达10种以上。鼠类则是一个特例,仅表达两种CD1基因CD1d1和CD1d2[26],从而为研究其余CD1蛋白亚型带来难度。
人类CD1基因位于1号染色体上[9],表达五种同源异构体,分别命名为CD1a、CD1b、CD1c、CD1d和CD1e[23]。五种CD1蛋白亚型的分布和运输都不相同,暗示它们具有不同的生理功能。根据序列同源性,CD1a、CD1b、CD1c和CD1e又称Ⅰ型CD1蛋白,CD1b称为Ⅱ型CD1蛋白[23]。
Ⅰ型和Ⅱ型CD1蛋白的划分主要根据序列同源性,同时,他们在生物体内的表达和调节也有所不同。
Ⅰ型CD1蛋白表达于胸腺细胞和成熟的造血细胞,例如B淋巴细胞、骨髓细胞、单核细胞等;Ⅱ型CD1蛋白则在造血和非造血细胞中均能表达,例如CD1d可以在肠道上皮细胞和角质细胞中表达[26,27]。Ⅱ型CD1蛋白在生物体内组成性表达[28],而Ⅰ型CD1蛋白诱导性表达并且能够被骨髓细胞调节[29,30]。例如在细菌刺激的条件下,Ⅰ型CD1蛋白的表达可以被选择性上调[30,31]。此外,两种类型的CD1基因转录对于免疫刺激信号分子(如GM-CSF、TNF-α、IL-3、IL-4、IL-6、IL-1β、视黄酸或TLR配体)有不同响应,从而产生携带不同CD1亚型的抗原呈递细胞[32]。
2 CD1蛋白亚型的分布
CD1蛋白五种亚型具有不同的转运和器官分布,暗示它们具有不同的生理功能[33]。
CD1a早期作为人类表皮朗格汉斯细胞的特异性标记蛋白而被熟知。骨髓树突细胞也同样能够表达这种蛋白[28]。近期研究表明,在几乎所有人类外周血样品中都检测出CD1a自身免疫T细胞的存在,侧面证明这类T细胞是人类免疫系统中的一个组成部分[18,19,34]。CD1a自身免疫T细胞占血液中T细胞总数的0.1%~10%,表达皮肤特异性归巢受体,大量在皮肤归巢[18]。皮肤脂质是一类已知的典型CD1a配体,在皮肤中大量存在,作为CD1a识别的自身抗原之一[34]。以上信息提示,CD1a蛋白可能是一种特异性的CD1蛋白亚型,在皮肤中大量表达并发挥免疫作用。
CD1b主要在淋巴器官中的骨髓树突细胞中表达[28]。由于CD1b具有CD1蛋白家族中容积最大的抗原呈递区,它可能主要负责呈递体积较大的外源脂质[35]。
CD1c表达于B细胞和树突细胞中[28],已有证据显示它与αβ T细胞和γδ T细胞均能相互作用[36,37]。CD1c能够调节T细胞对结核杆菌的免疫[38,39]。
CD1d是唯一一种在鼠类实验动物模型中表达的CD1蛋白,围绕它的研究最为广泛[40]。CD1d是唯一一种Ⅱ型CD1蛋白,主要表达于上皮细胞、B细胞和骨髓树突状细胞中[28]。由于CD1d限制性T细胞即是自然杀伤细胞(NTK cell)[40],因此对CD1d的研究大多紧密围绕着CD1d-抗原复合物与NKT细胞之间的相互作用。
CD1e是一种可溶性蛋白,分布在内吞小体(Endocytic compartment)中,主要功能是辅助脂质的修饰过程,以便脂质与其他CD1分子结合形成复合物[41]。由于CD1e的功能与其他CD1蛋白差异较大,在某些分类方法中将CD1e单独命名为Ⅰ、Ⅱ型CD1蛋白,而将CD1a、CD1b和CD1c合称为Ⅰ型CD1蛋白[42]。
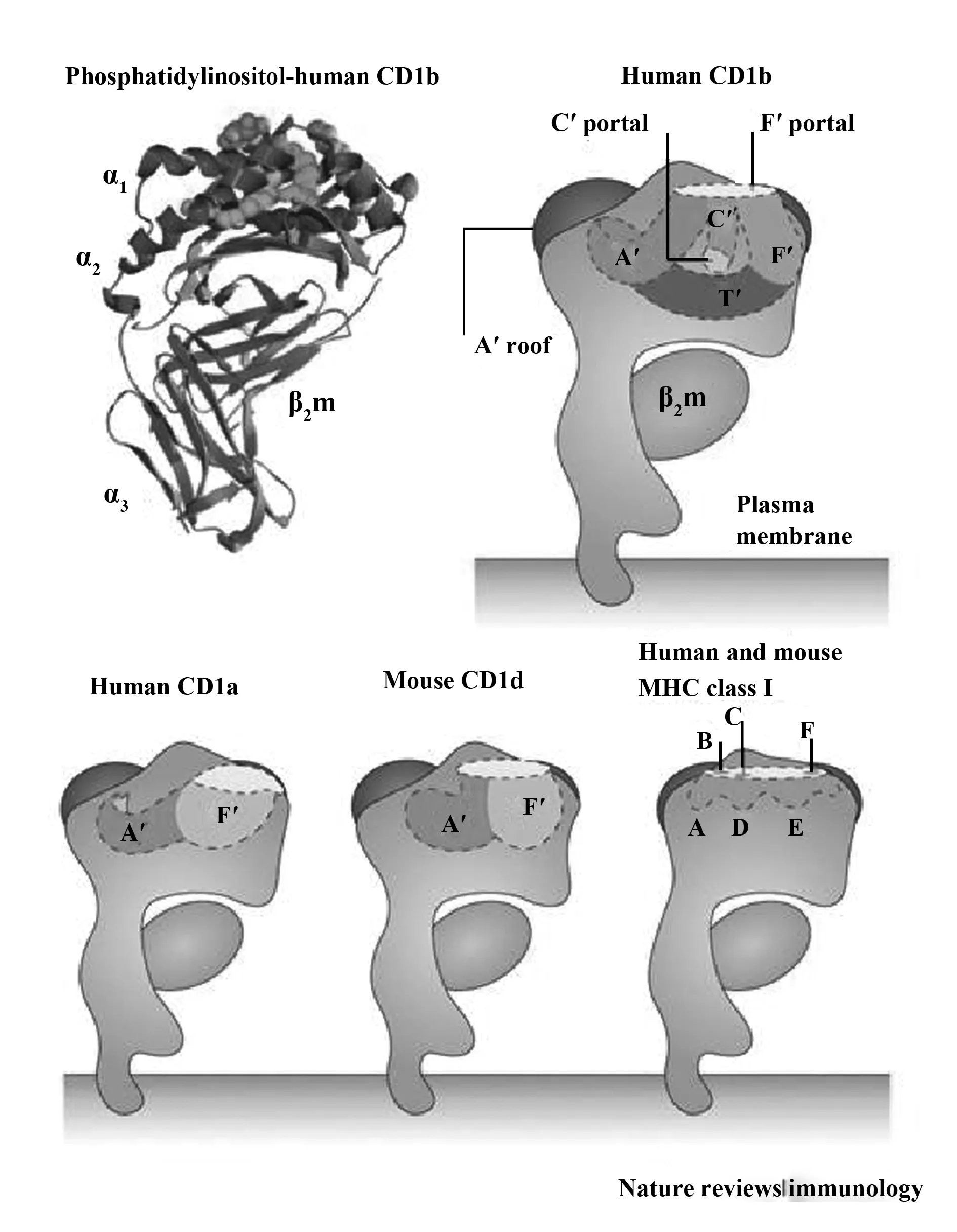
图1 CD1蛋白分子结构示意及与MHCⅠ分子的对比[43]Fig.1 Comparison of CD1 and MHCⅠ molecular structure[43]
3 CD1蛋白的结构
CD1蛋白是跨膜糖蛋白,是由三条重链α1、α2、α3和β2微球蛋白以非共价键的方式结合形成的异二聚体,其分子结构类似MHC Ⅰ型分子(见图1)[43]。
CD1糖蛋白在粗面内质网中合成,与β2微球蛋白结合,随后加载内源脂质。成熟的CD1蛋白被运输到细胞膜表面,将所加载的内源脂质呈递给T细胞[44,45]。
一般来说,CD1蛋白抗原结合区域在α1和α2重链之间,由非极性氨基酸组成,CD1蛋白可能利用这些疏水区域来捕获脂质。CD1蛋白结合域与外部环境连接的开口非常狭窄,但内部空间较大,形成类似口袋状结构。这种口袋状结构允许脂质的烷基链深入插进蛋白内部,使这些分子链长、易弯曲、高度疏水的结构与蛋白结合得更紧密。CD1蛋白内的口袋状结构一般有多个,分别被命名为A′、C′、F′和T′袋;每一种CD1蛋白亚型都有独特的抗原结合域结构[46-50]。
4 CD1蛋白与抗原的结合
每一种CD1蛋白的抗原结合区域容积都相对固定,然而与之结合的配体却多种多样,体积也不尽相同[51]。为了与不同分子大小的脂质结合,CD1蛋白使用支架脂质(Scaffold lipids)、空间脂质(Spacer lipids)和辅助出口(Accessory portals)来修饰抗原结合域的容积。
CD1b蛋白结合域的容积比其他CD1亚型大50%,然而其配体的体积却并不比其他CD1亚型的配体大。研究表明,CD1b配体只占据抗原结合域的上半部分空间;支架脂质占据下半部分空间,结合在抗原的下方,将抗原推向TCR。两类已知的CD1b支架脂质是甘油酯(Glycerides)和Deoxydihydroceramides[52,53]。空间脂质工作原理与支架脂质类似,不过分布更为广泛。空间脂质与抗原一起占据CD1蛋白结合域,使体积较小的脂质也能与CD1蛋白结合并被呈递[54,55]。
对于体积较大的脂质,CD1蛋白上的辅助出口允许配体将多余部分伸出蛋白结合域,远离抗原呈递面。这些辅助出口被命名为C′和D′/E′出口,分别位于CD1b和CD1c蛋白。CD1a和CD1c在F′袋也有类似的缺口,允许所结合的脂质向外伸出[47,48,56]。
5 CD1-抗原复合物与TCR相互作用
CD1分子与MHC分子的抗原呈递区不同(见图1)。MHCⅠ分子的抗原呈递区与外界直接相连,抗原位于两个α螺旋形成的夹缝中间,TCR同时与α重链和抗原相接触,接触面左右对称;而CD1蛋白的抗原呈递区在α1和α2重链之间,TCR与A′袋的顶部以及抗原的亲水端相接触,形成左右不对称的接触面[35]。
一般来说,CD1蛋白的配体是脂质,具有较长且易折叠的烷基链和一个亲水端[45,57]。烷基链深入CD1蛋白结合域,而亲水端留在蛋白外与TCR相接触。脂质与抗原结合域的相互作用一般来说是非特异性的;亲水端的朝向以及其与TCR的相互作用则相对精确[31,46]。与MHC分子不同的是,CD1蛋白并没有水平的抗原呈递面;它与TCR相接触的部分是不对称的,是由位于左侧的CD1蛋白外表面和位于右侧的脂质配体共同组成的呈递面[35]。插入蛋白内部的烷基链能够调整CD1蛋白外表面的结构,从而使TCR能够识别化学结构不同的抗原[58]。
6 CD1a蛋白的“缺乏接触”特性
CD1家族蛋白识别的绝大多数抗原都是具有极性基团或者携带电荷的脂质,尽管也有文献报道CD1d可以呈递一种人工合成的缺乏脂肪族链和亲水端的非脂质小分子PPBT并激活T细胞[59-64]。然而,CD1a是个特例,它呈递抗原的方式与其他CD1亚型略有不同。一般认为,TCR识别CD1-抗原复合物时需要脂质亲水端参与相互作用;然而,CD1a所呈递的一类典型抗原是皮肤脂质,包括蜡酯(Wax esters)、鲨烯(Squalene)和三酰甘油(Triacylgly-cerides),均是高度疏水、缺乏亲水基团的分子[34]。这说明CD1a呈递抗原时或许并不需要TCR和脂质之间的直接相互作用。这一特性被命名为“缺乏接触(Absence of interference)”。CD1a抗原插入蛋白内部的长烷基链被认为能够调整CD1a外表面的结构,从而影响CD1a-配体复合物与TCR之间的相互作用[58]。
7 CD1a蛋白存在自身抑制类配体
与其他CD1同源异构体不同,CD1a的胞内端非常短,缺少酪氨酸内化基序(Tyrosine-based internalization motifs)。因此,CD1a仅参与早期胞内体循环,随后便回到细胞膜表面;这一点与MHCⅠ分子类似[61,62],同时也解释了为什么CD1a主要分布于细胞表面。相反,CD1b和CD1c则会参与晚期胞内体循环,在细胞内的时间也更长[63]。
在胞内体循环过程中,CD1a蛋白一般会加载一个内源配体,这将影响其与TCR的相互作用[34]。研究发现CD1a蛋白需要始终与抗原结合以维持稳定。当CD1a蛋白在细胞表面聚集时,蛋白自身携带不具有激活作用的配体以抑制CD1a诱导的自身免疫反应。这类不具有激活作用的配体被称为抑制类配体(Non-permissive ligand)。这些配体具有较大的亲水端,阻碍CD1a与TCR发生自体免疫[34]。当皮肤有伤口时,具有激活作用的配体聚集,替换起抑制作用的内源配体,从而激活下游免疫反应。
比如,蜡酯多分布在角化上皮,鲨烯分布在皮脂层,这两类分子在上皮中含量不高,因此很少与抗原呈递细胞接触。当皮肤被破坏,蜡酯和鲨烯能够大量进入上皮,替换抑制类配体,与CD1a结合,刺激T细胞,使之产生免疫反应,以应对皮肤的伤口。由于CD1a能够经过早期胞内体循环快速回到细胞膜表面,它能够及时识别并呈递外源抗体,从而感知上皮的环境变化[35]。
8 总结与展望
与MHC分子类似,CD1蛋白家族能够作为一类抗原呈递分子激活和调节由T细胞介导的免疫反应[8,9]。该类蛋白在生物界分布广泛,其不同亚型具有不同的转运和器官分布,暗示它们具有不同的生理功能。CD1蛋白的分子结构与MHCⅠ分子类似,由三条α重链与β2微球蛋白形成异二聚体[43],抗原结合域是由非极性氨基酸构成的疏水区域,与外部环境连接有限,允许脂质的烷基链深入插进蛋白内部,将蛋白外部的抗原亲水端呈递给TCR[44,45]。TCR同时识别CD1蛋白表面和抗原的亲水端[35]。CD1a能够向TCR呈递缺乏亲水端的皮肤脂质,暗示其与其他CD1蛋白在抗原呈递方式上存在差异[34]。
CD1蛋白等非多态性抗原呈递分子与MHC分子具有不同的生理功能,极大地拓宽了TCR能够识别的分子范围,提示我们T细胞所具有的更丰富的免疫作用。在理论方面,了解CD1蛋白及非多态性抗原分子有助于我们深入理解αβ T细胞的激活和调节机制,理解不同亚群的T细胞在不同器官中的功能,让我们能更好地了解皮肤和胃肠道等局部器官免疫系统是如何发挥作用的。同时,由于CD1蛋白能够与γδ T细胞相互作用[17],通过对CD1蛋白的研究或许能帮助我们理解这一类目前尚未完全了解清楚的T细胞的功能和调节方式。已知γδ T细胞在肿瘤免疫中发挥重要作用,对其的深入研究非常具有吸引力和临床应用价值。
由于CD1蛋白的抗原多为来自自身或来自致病菌的脂质,对它的研究将有助于进一步了解过敏、自身免疫疾病、细菌和真菌感染等疾病的致病机理。例如,在研究蜜蜂和黄蜂毒素中潜在的CD1a抗原时,我们发现蜂毒的脂质成分中并不存在能够被CD1a直接呈递的脂质抗原;蜂毒中的磷脂酶A2(PLA2)能够将细胞膜脂质作为底物,分解产生具有CD1a活性的脂质小分子作为新抗原,从而通过CD1a蛋白的呈递间接激活相应T细胞[65]。这一结果便为我们在筛查病原体、研究致病机理时提供了新思路,使我们的考虑范围不仅仅局限在可能存在的多肽抗原内。
CD1蛋白使我们意识到多肽并不是唯一具有抗原性的分子,这为临床预防、诊断和治疗提供新思路。例如,可以基于脂质或酯酶研制新型疫苗,通过检测特定脂质来作为疾病诊断的依据之一,或者利用非多态性抗原呈递分子的作用机制在皮肤、胃肠道等特定器官设计药物,以治疗过敏、自身免疫疾病、细菌和真菌感染等疾病。
考虑到基于多肽抗原的诸多临床和科研应用,相信以脂质作为抗原的相关免疫学研究,其理论及实际应用前景十分广泛。
[1] Garboczi DN,Ghosh P,Utz U,etal.Structure of the complex between human T-cell receptor,viral peptide and HLA-A2 [J].Nature,1996,384(6605):134.
[2] Garcia KC,Degano M,Stanfield RL,etal.An αβ T cell receptor structure at 2.5 Å and its orientation in the TCR-MHC complex[J].Science,1996,274(5285):209-219.
[3] Zinkernagel RM.Cellular immune recognition and the biological role of major transplantation antigens[J].Biosci Rep,1997,17(2):91-111.
[4] Stephenson KE,Barouch DH.Broadly Neutralizing Antibodies for HIV Eradication [J].Current HIV/AIDS Reports,2016,13(1):31-37.
[5] Salerno-Gonçalves R,Fernandez-Via M,Lewinsohn DM,etal.Identification of a human HLA-E-restricted CD8+T cell subset in volunteers immunized with Salmonella enterica serovar Typhi strain Ty21a typhoid vaccine [J].J Immunol,2004,173(9):5852-5862.
[6] Heinzel AS,Grotzke JE,Lines RA,etal.HLA-E-dependent presentation of Mtb-derived antigen to human CD8+T cells [J].J Exp Med,2002,196(11):1473-1481.
[7] Adams EJ,Luoma AM.The adaptable major histocompatibility complex (MHC) fold:structure and function of nonclassical and MHC class I-like molecules [J].Annual Rev Immunol,2013,31:529-561.
[8] McMichael AJ,Pilch JR,Fabre JW,etal.A human thymocyte antigen defined by a hybrid myeloma monoclonal antibody[J].Eur J Immunol,1979,9(3):205-210.
[9] Calabi F,Milstein C.A novel family of human major histocompatibility complex-related genes not mapping to chromosome 6 [J].Nature,1986,323(6088):540-543.
[10] Gold MC,Cerri S,Smyk-Pearson S,etal.Human mucosal associated invariant T cells detect bacterially infected cells [J].PLoS Biol,2010,8(6):e1000407.
[11] Le Bourhis L,Martin E,Péguillet I,etal.Antimicrobial activity of mucosal-associated invariant T cells [J].Nat Immunol,2010,11(8):701-708.
[12] Young DC,Kasmar A,Moraski G,etal.Synthesis of dideoxymycobactin antigens presented by CD1a reveals T cell fine specificity for natural lipopeptide structures [J].J Biol Chem,2009,284(37):25087-25096.
[13] Kasmar AG,van Rhijn I,Cheng TY,etal.CD1b tetramers bind αβ T cell receptors to identify a mycobacterial glycolipid-reactive T cell repertoire in humans [J].J Exp Med,2011,208(9):1741-1747.
[14] Borg NA,Wun KS,Kjer-Nielsen L,etal.CD1d-lipid-antigen recognition by the semi-invariant NKT T-cell receptor [J].Nature,2007,448(7149):44-49.
[15] Luoma AM,Castro CD,Mayassi T,etal.Crystal structure of Vδ1 T cell receptor in complex with CD1d-sulfatide shows MHC-like recognition of a self-lipid by human γδ T cells [J].Immunity,2013,39(6):1032-1042.
[16] Uldrich AP,Le Nours J,Pellicci DG,etal.CD1d-lipid antigen recognition by the [gamma][delta] TCR [J].Nat Immunol,2013,14(11):1137-1145.
[17] Pellicci DG,Uldrich AP,Le Nours J,etal.The molecular bases of δ/αβ T cell-mediated antigen recognition [J].J Exp Med,2014,211(13):2599-2615.
[18] De Jong A,Pea-Cruz V,Cheng TY,etal.CD1a-autoreactive T cells are a normal component of the human αβ T cell repertoire [J].Nat Immunol,2010,11(12):1102-1109.
[19] De Lalla C,Lepore M,Piccolo FM,etal.High-frequency and adaptive-like dynamics of human CD1 self-reactive T cells [J].Eur J Immunol,2011,41(3):602-610.
[20] Ly D,Kasmar AG,Cheng TY,etal.CD1c tetramers detect ex vivo T cell responses to processed phosphomycoketide antigens [J].J Exp Med,2013,210(4):729-741.
[21] Dellabona P,Casorati G,Friedli B,etal.In vivo persistence of expanded clones specific for bacterial antigens within the human T cell receptor alpha/beta CD4-8-subset [J].J Exp Med,1993,177(6):1763-1771.
[22] Baker ML,Miller RD.Evolution of mammalian CD1:marsupial CD1 is not orthologous to the eutherian isoforms and is a pseudogene in the opossum Monodelphis domestica [J].Immunology,2007,121(1):113-121.
[23] Calabi F,Jarvis JM,Martin L,etal.Two classes of CD1 genes [J].Eur J Immunol,1989,19(2):285-292.
[24] Dascher CC,Hiromatsu K,Naylor JW,etal.Conservation of a CD1 multigene family in the guinea pig [J].J Immunol,1999,163(10):5478-5488.
[25] Van Beeck FAL,Zajonc DM,Moore PF,etal.Two canine CD1a proteins are differentially expressed in skin [J].Immunogenetics,2008,60(6):315-324.
[26] Brigl M,Brenner MB.CD1:antigen presentation and T cell function [J].Annu Rev Immunol,2004,22:817-890.
[27] Im JS,Tapinos N,Chae GT,etal.Expression of CD1d molecules by human schwann cells and potential interactions with immunoregulatory invariant NK T cells [J].J Immunol,2006,177(8):5226-5235.
[28] Dougan SK,Kaser A,Blumberg RS.CD1 expression on antigen-presenting cells[M]//T cell activation by CD1 and Lipid Antigens.Springer Berlin Heidelberg,2007:113-141.
[29] Porcelli S,Brenner MB,Greenstein JL,etal.Recognition of cluster of differentiation 1 antigens by human CD4-CD8>-cytolytic T lymphocyte [J].Nature,1989,341(6241):447-450.
[30] Roura-Mir C,Wang L,Cheng TY,etal.Mycobacterium tuberculosis regulates CD1 antigen presentation pathways through TLR-2 [J].J Immunol,2005,175(3):1758-1766.
[31] Yakimchuk K,Roura-Mir C,Magalhaes KG,etal.Borrelia burgdorferi infection regulates CD1 expression in human cells and tissues via IL1-β [J].Eur J Immunol,2011,41(3):694-705.
[32] Moody DB.TLR gateways to CD1 function [J].Nat Immunol,2006,7(8):811-817.
[33] Kasmar A,Van Rhijn I,Moody DB.The evolved functions of CD1 during infection [J].Curr Opin Immunol,2009,21(4):397-403.
[34] De Jong A,Cheng TY,Huang S,etal.CD1a-autoreactive T cells recognize natural skin oils that function as headless antigens [J].Nat Immunol,2014,15(2):177-185.
[35] Van Rhijn I,Godfrey D I,Rossjohn J,etal.Lipid and small-molecule display by CD1 and MR1 [J].Nat Rev Immunol,2015,15(10):643-654.
[36] Vincent MS,Leslie DS,Gumperz JE,etal.CD1-dependent dendritic cell instruction [J].Nat Immunol,2002,3(12):1163-1168.
[37] Spada FM,Grant EP,Peters PJ,etal.Self-Recognition of Cd1 by γ/δ T cells implications for innate immunity [J].J Exp Med,2000,191(6):937-948.
[38] Beckman EM,Melian A,Behar SM,etal.CD1c restricts responses of mycobacteria-specific T cells.Evidence for antigen presentation by a second member of the human CD1 family [J].J Immunol,1996,157(7):2795-2803.
[39] Moody DB,Ulrichs T,Mühlecker W,etal.CD1c-mediated T-cell recognition of isoprenoid glycolipids in Mycobacterium tuberculosis infection [J].Nature,2000,404(6780):884-888.
[40] Godfrey DI,Rossjohn J.New ways to turn on NKT cells [J].J Exp Med,2011,208(6):1121-1125.
[41] De la Salle H,Mariotti S,Angenieux C,etal.Assistance of microbial glycolipid antigen processing by CD1e [J].Science,2005,310(5752):1321-1324.
[42] Porcelli SA,Modlin RL.The CD1 system:antigen-presenting molecules for T cell recognition of lipids and glycolipids [J].Annual Rev Immunol,1999,17(1):297-329.
[43] Moody DB,Zajonc DM,Wilson IA.Anatomy of CD1-lipid antigen complexes [J].Nat Rev Immunol,2005,5(5):387-399.
[44] Kang SJ,Cresswell P.Calnexin,calreticulin,and ERp57 cooperate in disulfide bond formation in human CD1d heavy chain [J].J Biol Chem,2002,277(47):44838-44844.
[45] Barral DC,Brenner MB.CD1 antigen presentation:how it works [J].Nat Rev Immunol,2007,7(12):929-941.
[46] Gadola SD,Zaccai NR,Harlos K,etal.Structure of human CD1b with bound ligands at 2.3 Å,a maze for alkyl chains [J].Nat Immunol,2002,3(8):721-726.
[47] Scharf L,Li NS,Hawk AJ,etal.The 2.5 Å structure of CD1c in complex with a mycobacterial lipid reveals an open groove ideally suited for diverse antigen presentation [J].Immunity,2010,33(6):853-862.
[48] Zajonc DM,Elsliger MA,Teyton L,etal.Crystal structure of CD1a in complex with a sulfatide self antigen at a resolution of 2.15Å [J].Nat Immunol,2003,4(8):808-815.
[49] Zeng ZH,Castano AR,Segelke BW,etal.Crystal structure of mouse CD1:an MHC-like fold with a large hydrophobic binding groove[J].Science,1997,277(5324):339-345.
[50] Adams EJ.Lipid presentation by human CD1 molecules and the diverse T cell populations that respond to them [J].Curr Opin Immunol,2014,26:1-6.
[51] Cheng TY,Relloso M,Van Rhijn I,etal.Role of lipid trimming and CD1 groove size in cellular antigen presentation [J].EMBO J,2006,25(13):2989-2999.
[52] Huang S,Cheng TY,Young DC,etal.Discovery of deoxyceramides and diacylglycerols as CD1b scaffold lipids among diverse groove-blocking lipids of the human CD1 system [J].Proc Natl Acad Sci,2011,108(48):19335-19340.
[53] Garcia-Alles LF,Collmann A,Versluis C,etal.Structural reorganization of the antigen-binding groove of human CD1b for presentation of mycobacterial sulfoglycolipids [J].Proc Natl Acad Sci,2011,108(43):17755-17760.
[54] Wun KS,Cameron G,Patel O,etal.A molecular basis for the exquisite CD1d-restricted antigen specificity and functional responses of natural killer T cells [J].Immunity,2011,34(3):327-339.
[55] Zajonc DM,Crispin MDM,Bowden TA,etal.Molecular mechanism of lipopeptide presentation by CD1a[J].Immunity,2005,22(2):209-219.
[56] Beckman EM,Porcelli SA,Morita CT,etal.Recognition of a lipid antigen by GDI-restricted αβ+T cells [J].Nature,1994,372(6507):691.
[57] De Libero G,Mori L.Recognition of lipid antigens by T cells [J].Nat Rev Immunol,2005,5(6):485-496.
[58] Birkinshaw RW,Pellicci DG,Cheng TY,etal.[alpha][beta] T cell antigen receptor recognition of CD1a presenting self lipid ligands [J].Nat Immunol,2015,16(3):258-266.
[59] Manolova V,Kistowska M,Paoletti S,etal.Functional CD1a is stabilized by exogenous lipids [J].Eur J Immunol,2006,36(5):1083-1092.
[60] Moody DB,Young DC,Cheng TY,etal.T cell activation by lipopeptide antigens [J].Science,2004,303(5657):527-531.
[61] Sugita M,Grant EP,van Donselaar E,etal.Separate pathways for antigen presentation by CD1 molecules [J].Immunity,1999,11(6):743-752.
[62] Barral DC,Cavallari M,McCormick PJ,etal.CD1a and MHC class I follow a similar endocytic recycling pathway [J].Traffic,2008,9(9):1446-1457.
[63] Van Rhijn I,Ly D,Moody DB.CD1a,CD1b,and CD1c in immunity against mycobacteria [M]//The new paradigm of immunity to tuberculosis.Springer New York,2013:181-197.
[64] Van Rhijn I,Young DC,Im JS,etal.CD1d-restricted T cell activation by nonlipidic small molecules [J].Proc Natl Acad Sci U S A,2004,101(37):13578-13583.
[65] Bourgeois EA,Subramaniam S,Cheng TY,etal.Bee venom processes human skin lipids for presentation by CD1a[J].J Exp Med,2015,212(2):149-163.
[收稿2016-02-25 修回2016-05-09]
(编辑 倪 鹏)
·启事·
《中国免疫学杂志》关于彩图处理的有关说明
《中国免疫学杂志》的全部来稿一经采用,来稿所附图片根据具体需要,酌情制作彩图,彩图制作费包含在该稿件的版面制作费中,一并开具发票,望周知!
《中国免疫学杂志》编辑部
doi:10.3969/j.issn.1000-484X.2017.02.033
10.3969/j.issn.1000-484X.2017.02.032
富霄鹏(1991年-),女,在读博士,主要从事免疫学方面研究,E-mail:xiaopeng.fu@lincoln.ox.ac.uk。
R392.3
A
1000-484X(2017)02-0308-06
