沉默生物钟基因PER1对口腔鳞状细胞癌细胞中生物钟基因网络的影响
2017-02-23赵钦敖奕然杨凯苏小丽吕晓强
赵钦 敖奕然 杨凯 苏小丽 吕晓强
重庆医科大学附属第一医院口腔颌面外科,重庆 400016
沉默生物钟基因PER1对口腔鳞状细胞癌细胞中生物钟基因网络的影响
赵钦 敖奕然 杨凯 苏小丽 吕晓强
重庆医科大学附属第一医院口腔颌面外科,重庆 400016
目的 探讨口腔鳞状细胞癌细胞中生物钟基因PER1对生物钟基因网络中其他生物钟基因表达的影响。方法采用RNA干扰技术沉默人口腔鳞状细胞癌SCC15细胞中PER1基因,应用流式细胞仪检测沉默后细胞的增殖和凋亡水平,实时荧光定量聚合酶链式反应检测生物钟基因CLOCK、BMAL1、PER1、PER2、PER3、DEC1、DEC2、CRY1、CRY2、TIM、CKIE、RORA、NPAS2、REV-ERBA mRNA的表达情况。结果 PER1基因沉默后,SCC15细胞增殖指数上升,凋亡指数下降(P<0.05);PER1、PER2、DEC1、DEC2、CRY1、CRY2和NPAS2 mRNA的表达水平降低(P<0.05),PER3、TIM、RORA和REV-ERBA mRNA的表达水平升高(P<0.05),CLOCK、BMAL1和CKIE mRNA的表达水平无统计学改变(P>0.05)。结论 生物钟基因PER1能够调控生物钟基因网络中众多其他生物钟基因PER2、DEC1、DEC2、CRY1、CRY2、NPAS2、PER3、TIM、RORA和REV-ERBA的表达,PER1在生物钟基因网络中具有重要调控作用。
生物钟; 鳞状细胞癌; 口腔; 基因
生物钟对调控体内各种复杂生命活动协同有序进行和维持正常生命活动具有重要作用[1-2]。生物钟基因是构成生物钟的核心,迄今为止已经发现14个生物钟基因,即:CLOCK、BMAL1、PER1、PER2、PER3、DEC1、DEC2、CRY1、CRY2、TIM、CKIE、RORA、NPAS2和REV-ERBA[2-8]。这些生物钟基因存在于人体内几乎所有的细胞[9-10],其通过相互作用,在转录和翻译水平形成多条正负反馈环路的网络结构[3-6]。哺乳类动物基因组中有2%~10%的基因受到这些生物钟基因的调控,被称为钟控基因[3],不同生物钟基因可通过对下游不同钟控基因的调控影响细胞的生命活动[3]。生物钟基因的异常表达和昼夜节律紊乱是导致癌症、内分泌疾病、心血管疾病和抑郁症等诸多疾病发生发展的重要原因[3,7]。
PER1是重要的生物钟基因之一[4-5]。PER1在头颈部癌、前列腺癌和乳腺癌等多种实体癌中表达降低[11-12],PER1表达降低可导致细胞增殖和凋亡平衡失调,促进细胞恶性转变[7-8,13]。本课题组前期研究也证明:PER1 mRNA和蛋白在口腔鳞状细胞癌(以下简称鳞癌)中的表达显著低于癌旁组织,沉默口腔鳞癌SCC15细胞中PER1后导致下游众多细胞周期基因表达改变,癌细胞增殖能力增强[2,11]。PER1在癌症中表达降低后通过调控下游众多细胞周期基因的改变,从而与癌症的发生发展密切相关[10,13]。PER1在癌细胞中表达降低对其本身生物钟基因网络中的其他生物钟基因表达的影响目前还不清楚。本研究通过沉默人口腔鳞癌SCC15细胞中PER1后,全面检测生物钟基因网络中PER2、PER3、DEC1、DEC2、CRY1、CRY2、TIM、RORA、NPAS2和REV-ERBA mRNA的表达改变。
1 材料和方法
1.1 主要仪器和试剂
梯度聚合酶链式反应(polymerase chain reaction,PCR)仪、荧光定量PCR仪(S1000TM Thermal Cycler)(Bio-Rad公司,美国),RNAiso Plus试剂盒和实时荧光定量PCR试剂盒(TaKaRa公司,日本),QIAGEN质粒抽提试剂盒和QIAGEN质粒包装试剂盒(Qiagen公司,德国),BCA蛋白浓度测定试剂盒(Sigma公司,美国),人口腔鳞癌SCC15细胞和293T细胞(重庆医科大学生命科学院)。
1.2 短发夹RNA(short hairpin RNA,shRNA)慢病毒载体质粒的构建和鉴定
根据人PER1 mRNA序列(Genbank Accession:NM_002616.2),用Reynolds A对照筛选原则[14],筛选获得PER1基因的3个不同干扰靶点,即PER-Ⅰ:CAGCACCACTAAGCGTAAATG;PER1-Ⅱ:CCAGCACCACTAAGCGTAAAT;PER1-Ⅲ:CCATGGACATGTCCACCTATA。根据RNA干扰序列设计原则[14],分别设计合成3条PER1干扰序列,即PER1-shRNA-Ⅰ、PER1-shRNA-Ⅱ和PER1-shRNA-Ⅲ(表1)。然后使用Age Ⅰ/EcoR Ⅰ双酶切,将载体质粒PLKO.1酶切后行凝胶电泳并回收,用T4 DNA连接酶分别与PER1-shRNA-Ⅰ~Ⅲ退火引物链接,构建PER1-shRNA-Ⅰ~Ⅲ慢病毒质粒。以不含任何干扰基因片段的scramble质粒为阴性对照(Control-shRNA)。将以上慢病毒质粒分别转入感受态大肠杆菌DH5A,接种于含氨基核苷类抗生素的LB固体培养基上,然后按照Qiagen质粒抽提说明书提取质粒,经PCR反应后将扩增产物行DNA测序,测序结果用软件Chromas V2.4(Technelysium公司,美国)进行分析鉴定。
1.3 shRNA的包装
根据QIAGEN质粒包装试剂盒说明书进行操作,简述如下:将1 mL DMEM、10 μL质粒、10 μL慢病毒包装混合质粒(Lenti-HG Mix)和60 μL核酸转染剂(HG transgene reagent)在室温下混合20 min后,转移至细胞密度为70%~80%的293T细胞培养皿中,12 h后滴加120 μL 100×Enhancing buffer。培养48 h后用孔径为0.45 μm的过滤器过滤293T细胞,获上清液,即为质粒病毒液,分装后于-80 ℃保存备用。
1.4 PER1-shRNA质粒病毒感染SCC15细胞及沉默PER1基因
将对数生长期的SCC15细胞均匀接种于六孔板中(编号1~6),加入1.5 mL含10%胎牛血清的DMEM/ F12培养液,第1~4孔先加2.5 μL聚凝胺(polybrene),然后分别加入500 μL的PER1-shRNA-Ⅰ、PER1-shRNA-Ⅱ、PER1-shRNA-Ⅲ、Control-shRNA质粒病毒液,第5和第6孔只加入无血清DMEM/F12培养液500 μL,培养24 h(37 ℃,5%CO2)后更换培养液(第1~5孔为含浓度为2 μg·mL-1的嘌呤霉素DMEM/ F12培养液,第6孔为DMEM/F12培养液),每日更换培养液,连续7 d后获得PER1稳定干扰的SCC15细胞。实验分为5组:PER1-shRNA-Ⅰ组、PER1-shRNA-Ⅱ组、PER1-shRNA-Ⅲ组、Control-shRNA组和SCC15组,分别转染PER1-shRNA-Ⅰ、PER1-shRNA-Ⅱ、PER1-shRNA-Ⅲ和Control-shRNA质粒病毒的SCC15细胞以及未做任何处理的SCC15 细胞。
1.5 蛋白质印迹法(Western blot)检测
将对数生长期的SCC15细胞用RIPA裂解液裂解30 min,然后用细胞刮刀收集后离心(12 000 r·min-1,4 ℃)5 min,收集上清液。用BCA蛋白测定试剂盒测定蛋白质浓度。取50 μg蛋白的样品溶液行十二烷基硫酸钠聚丙烯酰胺凝胶电泳后凝胶湿转PVDF膜,用5%脱脂奶粉室温封闭 1 h,然后加入一抗为兔抗人PER1多克隆抗体和鼠抗人GAPDH单克隆抗体,在室温下摇床孵育2 h,TBS洗膜3次后加入二抗为辣根酶标记山羊抗兔IgG抗体,孵育2 h后洗膜3遍。暗室下将ECL发光液滴加在PVDF膜上,化学发光仪曝光后用凝胶成像系统拍照并测量条带灰度值。计算PER1和内参GAPDH条带灰度值比值。实验重复3次。
1.6 流式细胞仪检测
将对数生长期的SCC15细胞用0.25%胰蛋白酶消化后吹打成单细胞悬液,用PBS调整细胞浓度为1× 106个·mL-1。1)细胞增殖检测:将1 mL细胞悬液离心(4 ℃,800 r·min-1)5 min后弃上清液,然后加入-20 ℃、70%的乙醇1 mL,反复吹打混匀后于4 ℃过夜固定。固定后的细胞用PBS洗涤2遍后用1 mL PBS重悬,然后加入1 mL碘化丙啶染液,4 ℃避光30 min。用流式细胞仪检测,细胞增殖指数=(S+ G2/M)/(G0/G1+S+G2/M)×100%。2)细胞凋亡检测:取1 mL单细胞悬液用70%的乙醇固定2 h,换用0.5 mL结合缓冲液(Binding Buffer)重悬细胞,然后加入200 μL Annexin V-FITC混匀,室温避光15 min后加入1 mL碘化丙啶染液混匀,室温避光2 min。用流式细胞仪检测,细胞凋亡指数=(凋亡细胞数/所测细胞总数)×100%。实验重复3次。
1.7 实时荧光定量PCR(quantitative real time polymerase chain reaction,qRT-PCR)检测
根据RNAiso Plus试剂盒说明书提取细胞总RNA,用核酸蛋白分析仪检测波长在260 nm和280 nm处的光密度值,计算RNA浓度和纯度。将RNA反转录为cDNA,反应体系条件为37 ℃、15 min,85 ℃、5 s。Oligo7.0软件(DBA Oligo公司,美国)设计目的基因引物序列,合成目的基因CLOCK、BMAL1、PER1、PER2、PER3、DEC1、DEC2、CRY1、CRY2、TIM、CKIE、RORA、NPAS2、REV-ERBA和内参β-actin的引物(表2)。反应体系为:2×SYBR Premix Ex TaqTMⅡ 12.5 μL,浓度均为0.4 μmol·L-1的上游引物和下游引物各1 μL,浓度为50 ng·μL-1的DNA 模板2 μL,灭菌灭酶蒸馏水8.5 μL。反应条件均为:95 ℃预变性90 s,95 ℃变性10 s,60 ℃退火延伸30 s,扩增40个循环,60 ℃延伸时采集荧光信号,2-ΔΔCt法计算各基因mRNA表达。实验重复3次。
1.8 统计学分析
采用SPSS 19.0软件进行分析,组间比较进行单因素方差分析,两两比较采用LSD检验。

表 2 各生物钟基因qRT-PCR引物序列Tab 2 Primer sequences for clock genes used for qRT-PCR
2 结果
2.1 shRNA慢病毒载体质粒的构建和测序
PER1-shRNA-Ⅰ~Ⅲ慢病毒载体质粒DNA的测序结果见图1。

图 1 PER1-shRNA慢病毒载体质粒DNA测序结果Fig 1 The sequencing atlas of PER1-shRNA recombinant plasmids
PER1-shRNA-Ⅰ~Ⅲ慢病毒载体质粒DNA与设计的寡核苷酸干扰靶点组合序列的PER1-shRNA-Ⅰ~Ⅲ正义链一致,证明3条PER1-shRNA质粒构建成功。
2.2 SCC15细胞感染shRNA质粒病毒后PER1蛋白和mRNA的表达
PER1-shRNA-Ⅰ~Ⅲ、Control-shRNA和SCC15组中,PER1蛋白表达分别为0.18±0.07、0.61±0.06、0.56±0.06、1.14±0.05和1.18±0.13(图2);PER1 mRNA表达分别为0.22±0.05、0.56±0.07、0.63±0.11、0.94±0.20和1.12±0.10。PER1-shRNA-Ⅰ组细胞中PER1 mRNA和蛋白的表达均低于其他组(P<0.05)。表明PER1-shRNA-Ⅰ组对PER1的干扰效果最明显。
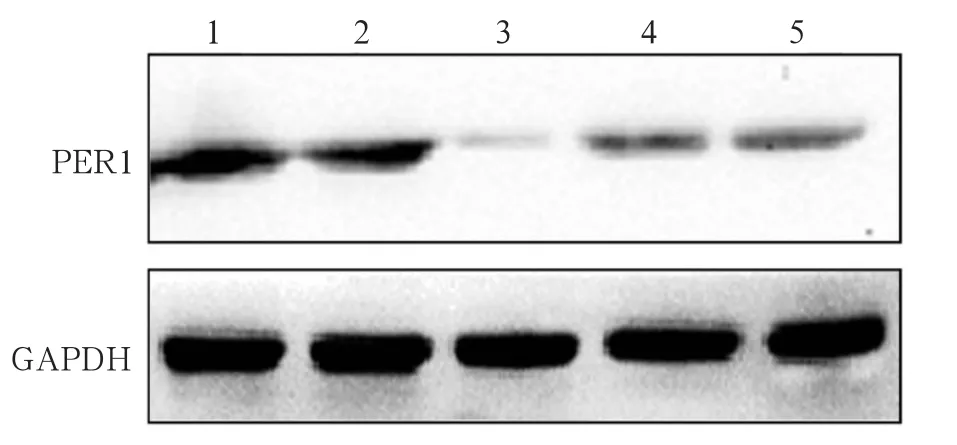
图 2 Western blot 检测5组细胞PER1蛋白的表达Fig 2 The protein expression of PER1 in fi ve groups by Western blot
2.3 PER1基因沉默后SCC15细胞的增殖和凋亡
PER1-shRNA-Ⅰ组细胞的增殖指数高于ControlshRNA组和SCC15组(P<0.05),凋亡指数低于Control-shRNA组和SCC15组(P<0.05);ControlshRNA组与SCC15组细胞的增殖指数和凋亡指数均无统计学差异(P>0.05)(图3)。
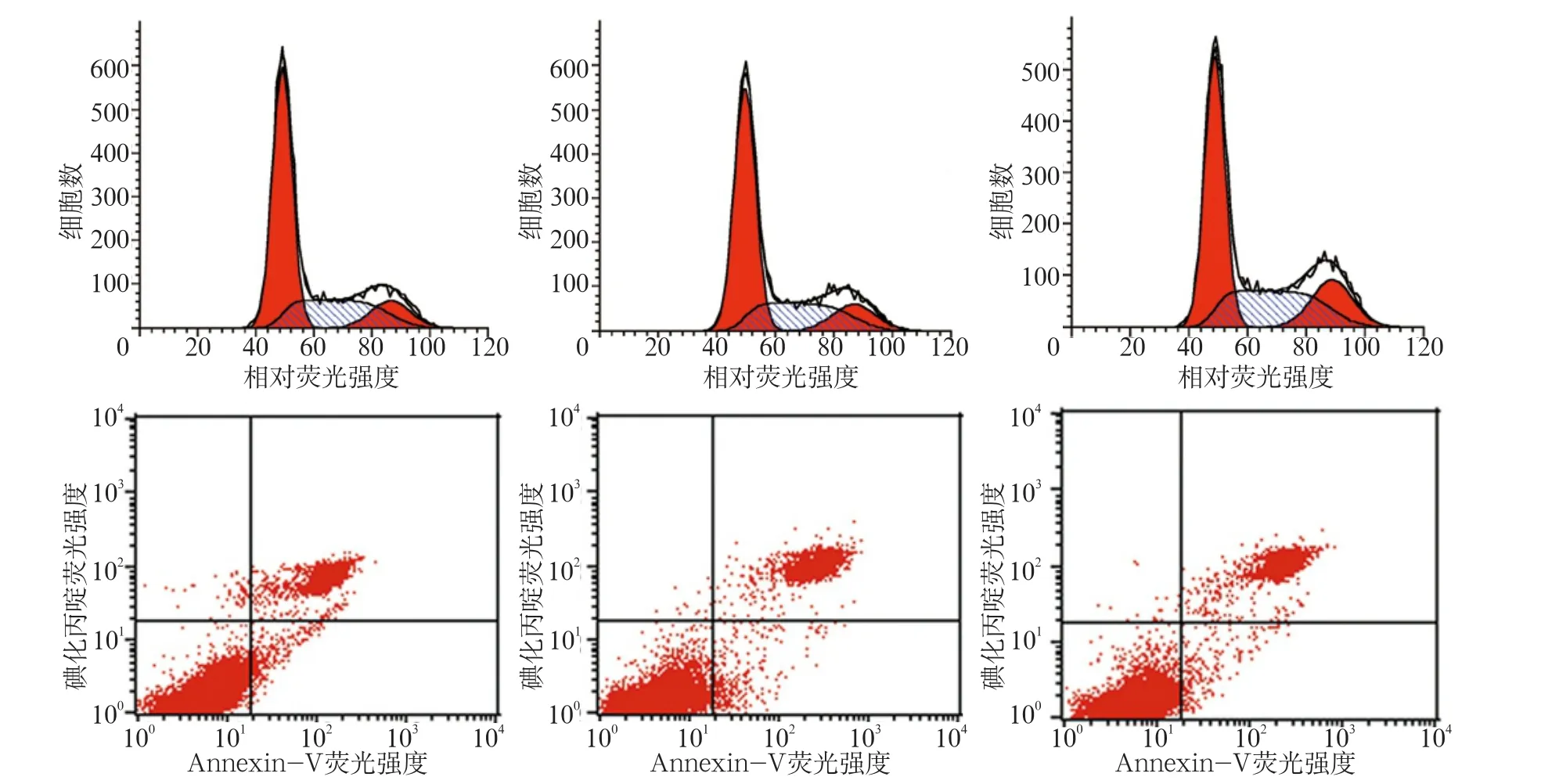
图 3 PER1沉默后SCC15细胞的流式细胞仪检测结果Fig 3 Flow cytometry results after PER1 knockdown in SCC15 cells
2.4 PER1沉默后SCC15细胞内生物钟基因的表达
qRT-PCR检测显示:PER1-shRNA-Ⅰ组细胞中PER3、TIM、RORA和REV-ERBA mRNA的表达水平高于Control-shRNA组和SCC15组(P<0.05),而PER1、PER2、DEC1、DEC2、CRY1、CRY2和 NPAS2 mRNA的表达水平降低(P<0.05);ControlshRNA组和SCC15组细胞中各基因mRNA的表达水平无统计学差异(P>0.05);CLOCK、BMAL1和CKIE mRNA的表达水平在3组中均无统计学差异(P>0.05)(图4)。
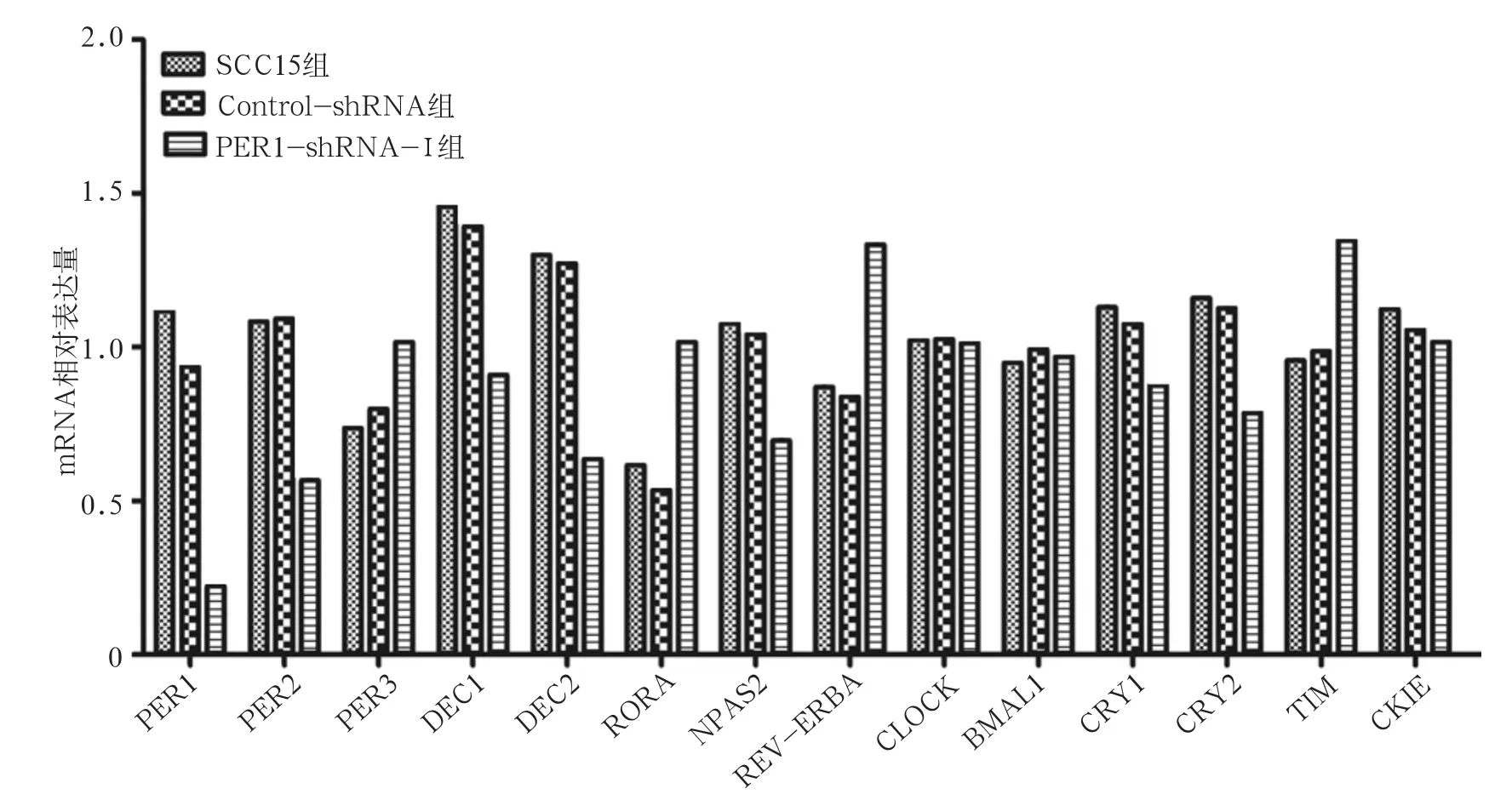
图 4 沉默PER1后SCC15细胞中生物钟基因mRNA的表达Fig 4 Levels of mRNA expression of clock genes in SCC15 cells after PER1 knockdown
3 讨论
目前已证明生物钟基因PER1在多种癌症中表达降低,并通过对下游众多细胞周期基因的调控,从而与癌症的发生发展密切相关[10-13]。PER1作为重要生物钟基因之一[4-5],PER1在癌症中表达降低是否导致生物钟基因网络中其他生物钟基因表达的改变?目前对这个问题还不清楚。
研究[4-6]表明:正常细胞内生物钟基因之间在转录和翻译水平主要包括3条正负反馈环路的网络结构。3条正负反馈环路中的正反馈因子均为CLOCK和BMAL1结合形成的CLOCK/BMAL1异二聚体,作为转录因子激活PERs、CRYs、DECs、REV-ERBA和RORA基因的转录,启动这些基因的表达,形成正反馈通路;而形成的PERs和CRYs结合为异二聚体发生核转移与CLOCK/BMAL1结合抑制CLCOK/ BMAL1活性,反馈性降低其转录激活作用,形成第一条负反馈通路,这条通路也是主要的通路。其次,DEC1和DEC2结合形成二聚体或异二聚体,作为转录因子转运至核内与CLCOK/BMAL1具有竞争性结合作用,从而降低CLOCK/BMAL1的转录激活作用,形成第二条负反馈通路;REV-ERBA和RORA转运至核内,分别抑制和促进BMAL1的表达,形成第三条负反馈通路。本研究中,沉默SCC15癌细胞中PER1后,正反馈因子CLOCK和BMAL1 mRNA表达水平没有显著改变,提示在转录水平上CLOCK和BMAL1不受PER1的调控。Lee等[15]报道:在哺乳动物正常肝细胞,生物钟反馈环路中CLOCK/BMAL1具有能保持持续稳定水平的特点,CLOCK/BMAL1的正向调控功能是通过发生核转移作为转录因子而发挥作用,因此,笔者推测在癌细胞中PER1的表达改变也是通过调控CLOCK/BMAL1的活性或在细胞内的分布来实现的,而不是表达水平的改变。
从以上正常细胞内生物钟基因的反馈环路来看,当PER1表达减低时,由于降低了对正向调控转录因子CLOCK/BMAL1的抑制作用,导致CLOCK/BMAL1转录激活作用增强,因而负向调控基因PER2、PER3、CRY1、CRY2、DEC1、DEC2、REV-ERBA和RORA的表达应该升高。Zheng等[9]报道在PER1缺陷的小鼠体内,PER2蛋白表达升高。Richards等[16]发现在小鼠正常肝细胞和内皮细胞中敲除PER1能导致CRY2 mRNA和蛋白表达上调。本研究证明:在SCC15细胞中沉默PER1后,负向调控基因中只有PER3、REVERBA和RORA mRNA的表达升高,符合已知的正常细胞内生物钟基因正负反馈调节特点,而其他负向调控基因PER2、CRY1、CRY2、DEC1、DEC2 mRNA的表达反而显著降低,这与正常细胞内生物钟基因正负反馈调节特点不符。笔者推测原因是:首先,先前的研究均是正常细胞,而本研究是癌细胞,可能在癌细胞中生物钟基因正负反馈通路网络的调控作用发生变化,也可能在癌细胞中出现了其他补充调控途径。另外,在癌细胞中,可能还存在其他调控生物钟基因表达的因素,导致生物钟基因的反馈环路出现异常。
本研究还发现癌细胞中沉默PER1后,生物钟基因TIM和NPAS2 mRNA分别显著升高和降低。目前已知的正常细胞内生物钟基因反馈环路中未包括TIM[4-6]。本研究发现在癌细胞中沉默PER1后TIM mRNA表达升高,这表明TIM也可能是生物钟基因反馈环路中的因子之一。NPAS2是CLOCK的同源类似物,能与BMAL1结合形成NPAS2/BMAL1异二聚体,NPAS2/BMAL1的功能与CLOCK/BMAL1相似,主要对CLOCK/BMAL1具有代偿作用,本研究沉默PER1后NPAS2 mRNA表达显著降低,提示在癌细胞中PER1沉默后,NPAS2/BMAL1对CLOCK/ BMAL1的代偿作用减弱。
目前认为PER1的表达变化导致下游众多的细胞周期基因改变,从而导致肿瘤的发生[10,13]。本研究进一步发现在癌细胞中PER1对生物钟基因网络中PER2、PER3、CRY1、CRY2、DEC1、DEC2、TIM、RORA、REV-ERBA和NPAS2基因也具有重要的调控作用,证明了PER1对癌症发生发展的影响并非通过单一对下游钟控基因的调控,而同时对生物钟基因网络本身也具有调控作用。
[1] 叶华, 杨凯, 谭雪梅, 等. 钟基因Per2和肿瘤相关钟控基因在金黄地鼠口腔颊黏膜癌变不同阶段的昼夜节律改变[J]. 华西口腔医学杂志, 2015, 33(5):513-518.
Ye H, Yang K, Tan XM, et al. Circadian variation of clock gene Per2 and cancer-related clock-controlled genes in buccal mucosa carcinoma of golden hamster at different cancer stages[J]. West Chin J Stomatol, 2015, 33(5):513-518.
[2] 谭雪梅, 叶华, 杨凯, 等. 鼠口腔黏膜癌变过程中生物钟基因Per1与细胞周期基因昼夜节律的表达[J]. 中华口腔医学杂志, 2015, 50(7):392-398.
Tan XM, Ye H, Yang K, et al. Circadian rhythm variation of the clock genes Per1 and cell cycle related genes in different stages of carcinogenesis of buccal mucosa in animal model [J]. Chin J Stomatol, 2015, 50(7):392-398.
[3] Storch KF, Lipan O, Leykin I, et al. Extensive and divergent circadian gene expression in liver and heart[J]. Nature, 2002, 417(6884):78-83.
[4] Siepka SM, Yoo SH, Park J, et al. Circadian mutant Overtime reveals F-box protein FBXL3 regulation of cryptochrome and period gene expression[J]. Cell, 2007, 129(5): 1011-1023.
[5] Shearman LP, Sriram S, Weaver DR, et al. Interacting molecular loops in the mammalian circadian clock[J]. Science, 2000, 288(5468):1013-1019.
[6] Honma S, Kawamoto T, Takagi Y, et al. Dec1 and Dec2 are regulators of the mammalian molecular clock[J]. Nature, 2002, 419(6909):841-844.
[7] Cao Q, Gery S, Dashti A, et al. A role for the clock gene per1 in prostate cancer[J]. Cancer Res, 2009, 69(19):7619-7625.
[8] Zhao N, Yang K, Yang G, et al. Aberrant expression of clock gene period1 and its correlations with the growth, proliferation and metastasis of buccal squamous cell carcinoma[J]. PLoS ONE, 2013, 8(2):e55894.
[9] Zheng B, Albrecht U, Kaasik K, et al. Nonredundant roles of the mPer1 and mPer2 genes in the mammalian circadian clock[J]. Cell, 2001, 105(5):683-694.
[10] Yang X, Wood PA, Ansell CM, et al. The circadian clock gene Per1 suppresses cancer cell proliferation and tumor growth at specifi c times of day[J]. Chronobiol Int, 2009, 26(7):1323-1339.
[11] Chen R, Yang K, Zhao NB, et al. Abnormal expression of PER1 circadian-clock gene in oral squamous cell carcinoma [J]. Onco Targets Ther, 2012, 5:403-407.
[12] Krugluger W, Brandstaetter A, Kállay E, et al. Regulation of genes of the circadian clock in human colon cancer: reduced period-1 and dihydropyrimidine dehydrogenase transcription correlates in high-grade tumors[J]. Cancer Res, 2007, 67(16):7917-7922.
[13] Gery S, Komatsu N, Baldjyan L, et al. The circadian gene per1 plays an important role in cell growth and DNA damage control in human cancer cells[J]. Mol Cell, 2006, 22(3): 375-382.
[14] Reynolds A, Leake D, Boese Q, et al. Rational siRNA design for RNA interference[J]. Nat Biotechnol, 2004, 22(3):326-330.
[15] Lee C, Etchegaray JP, Cagampang FR, et al. Posttranslational mechanisms regulate the mammalian circadian clock[J]. Cell, 2001, 107(7):855-867.
[16] Richards J, All S, Skopis G, et al. Opposing actions of Per1 and Cry2 in the regulation of Per1 target gene expression in the liver and kidney[J]. Am J Physiol Regul Integr Comp Physiol, 2013, 305(7):R735-R747.
(本文编辑 李彩)
Effect of clock gene PER1 knockdown on clock gene networks in human oral squamous cell carcinoma
Zhao Qin, Ao Yiran, Yang Kai, Su Xiaoli, Lü Xiaoqiang. (Dept. of Oral and Maxillofacial Surgery, The First Affi liated Hospital of Chongqing Medical University, Chongqing 400016, China)
Objective This study investigated the effect of clock gene PER1 on the expression levels of other clock genes in clock gene networks in oral squamous cell carcinoma cells. Methods We used RNA interference mediated by short hairpin RNAs (shRNAs) to effectively knock down PER1 in SCC15 human oral squamous cell carcinoma cells. Flow cytometry was used to detect the degree of proliferation and apoptosis of the cells after PER1 knockdown, and quantitative real-time PCR was used to detect the mRNA expression levels of the clock genes CLOCK, BMAL1, PER1, PER2, PER3, DEC1, DEC2, CRY1, CRY2, TIM, CKIE, RORA, NPAS2, and REV-ERBA. Results The proliferation index of SCC15 cells increased signifi cantly while the apoptotic index decreased signifi cantly after PER1 knockdown (P<0.05). The mRNA expression levels of PER1, PER2, DEC1, DEC2, CRY1, CRY2, and NPAS2 markedly decreased (P<0.05) while those of PER3, TIM, RORA, and REV-ERBA markedly increased (P<0.05). By contrast, no obvious changes were observed in the mRNA expression levels of CLOCK, BMAL1, and CKIE (P>0.05). Conclusion The clock gene PER1 can regulate the expression levels of other clock genes in the clock gene networks; these genes include PER2, DEC1, DEC2, CRY1, CRY2, NPAS2, PER3, TIM, RORA, and REV-ERBA. PER1 gene thus plays an important role in the regulation of clock gene networks.
circadian clock; squamous cell carcinoma; oral cavity; gene
R 739.8
A
10.7518/hxkq.2017.01.008
2016-07-10;
2016-09-10
赵钦,硕士,E-mail:zhaoqinzxc123@163.com
杨凯,教授,博士,E-mail:cqfyyk@aliyun.com
Correspondence: Yang Kai, E-mail: cqfyyk@aliyun.com.
