基因芯片技术检测结核分枝杆菌利福平和异烟肼耐药性临床应用评价
2017-02-17许榕青林银霞黄明翔陈新朝
许榕青,李 丹,林银霞,黄明翔,陈新朝
基因芯片技术检测结核分枝杆菌利福平和异烟肼耐药性临床应用评价
许榕青,李 丹,林银霞,黄明翔,陈新朝
目的 评价基因芯片技术检测结核分枝杆菌利福平和异烟肼耐药性的临床应用效果。方法 利用基因芯片技术对涂阳肺结核患者的痰标本和临床分离株进行利福平和异烟肼耐药基因位点检测,比较痰标本与菌株检测结果的差异;并以BACTEC MGIT 960药敏试验结果为对照评价基因芯片的检测性能。另外,通过对999株鉴定为结核分枝杆菌的临床分离菌株进行rpoB、katG和inhA基因耐药相关区域扩增产物测序,以验证基因芯片对核酸序列检测的准确性。结果 在涂阳的1 108例标本中有100例经基因芯片菌种鉴定为非结核分枝杆菌。其余的1 008例痰标本基因芯片结果与BACTEC MGIT 960培养阳性的菌株基因芯片结果比较仅有9例结果不一致,符合率为99.1%。以BACTEC MGIT960培养法药敏结果为对照,999株菌株基因芯片法检测利福平耐药性的符合率为98.1%,异烟肼的耐药性的符合率为94.5%。与DNA测序法相比,基因芯片法检测利福平和异烟肼耐药性的准确率分别为99.6%和99.8%。结论 基因芯片技术能够快速、准确地进行结核分枝杆菌利福平和异烟肼的耐药性检测,并能直接用于痰标本检测。
结核分枝杆菌;利福平;异烟肼;耐药性;基因芯片
Supported by the Young Research Program of Department of Public Health of Fujian Province (No. 2013-2-77) Corresponding author: Huang Ming-xiang, Email: hmg119@163.com
结核病是全世界范围内严重威胁公共健康的传染病。近年来,由于不规范治疗导致的耐多药结核病(至少对利福平和异烟肼同时耐药)是结核病疫情上升的最主要原因之一,因此,结核分枝杆菌及其耐药性的快速鉴定成为结核病防治的关键。目前,我国结核分枝杆菌的药物敏感性试验以改良L-J法和BACTEC MGIT 960液体培养法为主,但传统的药敏试验方法费时、烦琐,在得到分离株后3~4周后才能获得结果[1];即使应用BACTEC MGIT 960液体快培法在培养阳性后得到药敏结果也需要9~13 d[2],不能满足临床快速诊断的需求。临床亟需一种快速鉴定利福平和异烟肼耐药的实验室诊断方法,以利于耐多药结核病 (multidrug resistant TB, MDR-TB) 的早期诊断和治疗。
基因芯片技术具有高通量、高特异性的特点,可快速检测标本中的分枝杆菌耐利福平基因rpoB,及耐异烟肼katG、inhA基因常见突变位点[3]。本研究拟通过临床大样本检测以评估该技术直接应用于痰标本检测的效果。同时以BACTEC MGIT 960药敏试验结果为对照评价基因芯片技术对于结核分枝杆菌利福平和异烟肼耐药检测的敏感性、特异性、Kappa值及符合率,并探讨该方法在临床诊断耐多药结核病中的应用。
1 材料与方法
1.1 标本来源 2014年9月至2015年6月福州肺科医院的涂阳肺结核患者的痰标本,最终纳入的样本为分枝杆菌培养阳性者。
1.2 主要仪器与试剂 晶芯○R Extractor TM 36核酸快速提取仪、BioMixer TM II芯片杂交仪、SlideWasher芯片洗干仪、LuxScan 10K-B微阵列芯片扫描仪和晶芯结核分枝杆菌耐药基因检测试剂盒,均购自博奥生物有限公司。BACTEC MGIT960分枝杆菌培养仪购自美国BD公司。
1.3 BACTEC MGIT960分枝杆菌培养、菌种鉴定和药敏试验 BACTEC MGIT960分枝杆菌培养试验方法参照《结核病诊断实验室检验规程》[4]。培养阳性菌株用对硝基苯甲酸(PNB)生长试验进行菌种鉴定,PNB培养基不生长鉴定为结核分枝杆菌复合群。BACTEC MGIT960药敏试验具体操作按照BACTEC MGIT960仪器操作说明进行。
1.4 基因芯片检测利福平和异烟肼耐药基因位点
基因芯片技术检测一线药物利福平及异烟肼的3个耐药相关基因rpoB基因、katG基因及inhA基因启动子的野生型及不同突变型[3]。
1.4.1 样本制备及核酸提取 痰标本加入等量10%NaOH,液化15~20 min,12 000 r/min,离心后弃上清。再加入1 mL的生理盐水,充分震荡后12 000 r/min离心5 min,弃上清。向核酸提取管中加入80 μL核酸提取液,加入上述样本后,使用Extractor36核酸快速提取仪振荡5 min,95 ℃水浴5 min,10 000 r/min离心1 min。
1.4.2 PCR扩增 采用不对称扩增法对相关基因位点进行扩增[5]。每个样品进行3管 PCR扩增反应,扩增的分别是PCR扩增试剂1、2、3。分别向3管中加入模板DNA 2 μL和18 μL PCR扩增试剂。每管PCR反应体系总体积为20 μL,置于PCR扩增仪中进行扩增。PCR反应条件为:37 ℃激活10 min;94 ℃变性600 s; 94 ℃ 30 s、60 ℃ 30 s、72 ℃ 40 s,35个循环; 94 ℃30 s,72 ℃ 60 s,10个循环;72 ℃ 420 s。
1.4.3 芯片杂交与结果判读 扩增反应完成后,在利福平芯片杂交管加入3 μL扩增产物1、2和9 μL杂交缓冲液,在异烟肼芯片杂交管加入3 μL扩增产物1 μL、3 μL和9 μL杂交缓冲液。15 μL杂交反应混合物95 ℃变性5 min,立即冰水浴3 min。吸出13.5 μL杂交反应混合物加入芯片阵列,再放在50 ℃杂交仪中杂交120 min。杂交后经洗涤甩干后使用LuxScan10K-B微阵列芯片扫描仪和晶芯软件进行信号的读取及结果的判读。
1.5 测序验证 对999株经BACTEC MGIT 960分枝杆菌培养阳性的菌株rpoB、katG和inhA基因耐药相关区域的扩增产物全部进行测序,由博奥生物有限公司完成。一代测序,Sanger终止法,仪器为ABI 3730XL。
1.6 统计学分析 两种方法检测结果的一致性用Kappa检验判别。Kappa≥0.75,两者一致性较好。采用PASW Statistics 18软件统计Kappa值。用配对计数资料的χ2检验分析两种检测方法的检测结果差异,P<0.05为差异有统计学意义。
2 结 果
2.1 在所有涂阳的1 108例标本中,有100例经基因芯片初筛鉴定为分枝杆菌属,再进一步进行基因芯片菌种鉴定为非结核分枝杆菌,与MGIT960培养的菌株鉴定结果一致。其余的1 008例痰标本基因芯片结果与BACTEC MGIT 960培养的菌株基因芯片结果比较仅有9例结果不一致,符合率达99.1%。这9例痰标本经BACTEC MGIT960培养的菌株不纳入最后结果分析。
2.2 基因芯片检测相关突变位点的结果 基因芯片检测999株BACTEC MGIT960培养阳性的菌株利福平和异烟肼耐药相关突变位点。基因芯片检测107株rpoB突变型的菌株中,106株存在单一位点突变,1株存在双位点突变。rpoB突变频率最高的位点是531,突变频率是56%,其次是526位点,突变频率是22.4%。基因芯片检测出159株katG/inhA突变株,111株是katG315位点的单一突变,突变率是70.7%。基因芯片检出异烟肼katG位点缺失2株。katG的 315位点和inhA的启动子区域存在双突变的菌株有3株,占全部突变株的1.9%,见表1。
表1 基因芯片法检测rpoB、katG和inhA耐药相关突变位点的分布
Tab.1 Distribution of mutations loci in therpoB,katGandinhAgenes detected by gene chip

基因Genes突变位点Loci突变类型Specificmutation菌株数No.ofstrains突变率(%)MutationraterpoB511T→C87.5513A→C43.7516G→T或A→T或A→G76.5526C→T或C→G或A→T或A→G2422.4531C→T或C→G6056533T→C32.8511,516T511C,G516T或T511C,A516T或T511C,A516G10.9katG315G→A或G→C11170.7inhA-15C→T4327.3katG,inhA315,-15G315A,C(-15)T或G315C,C(-15)T31.9
注:数字为突变位点编号,数字前字母为野生型碱基,数字后字母为突变后碱基。
Note: the numbers indicate mutation loci, the letters before the numbers indicate wild-type bases and those after the numbers indicate mutation-type bases.
2.3 基因芯片技术与BACTEC MGIT960培养法检测菌株利福平、异烟肼耐药结果比较分析 利福平基因芯片与BATECT MGIT 960分枝杆菌药敏试验结果符合率为98.1%,敏感度和特异度分别是94%和98.5%,χ2值为1.89,P>0.05,Kappa值为0.898,两种方法一致性较好;异烟肼基因芯片与BACTEC MGIT 960分枝杆菌药敏试验结果符合率为94.5%,敏感度和特异度分别是74.5%和99.9%,Kappa值为0.819。基因芯片技术与BACTEC MGIT 960培养法检测菌株异烟肼耐药结果的χ2值为51.07,P<0.05,提示两种方法检测异烟肼耐药的结果存在差异,见表2、表3。
表2 基因芯片技术与BACTEC MGIT 960培养法检测菌株利福平、异烟肼耐药结果比较分析
Tab. 2 Results of gene chip and BACTEC MGIT 960 culture method for detection of drug resistance to rifampin and isoniazid in MTB isolates

基因芯片法GenechipmethodBACTECMGIT960培养法BACTECMGIT960culturemethod耐药Resistance敏感Sensitivityχ2P利福平Rifampin突变型Mutationtype94131.89>0.05野生型Wildtype6886异烟肼Isoniazid突变型Mutationtype158151.07<0.05野生型Wildtype54786
表3 基因芯片技术检测菌株利福平、异烟肼耐药效果分析
Tab.3 Effect of gene chip for detection of rifampin and isoniazid resistance in clinic strains
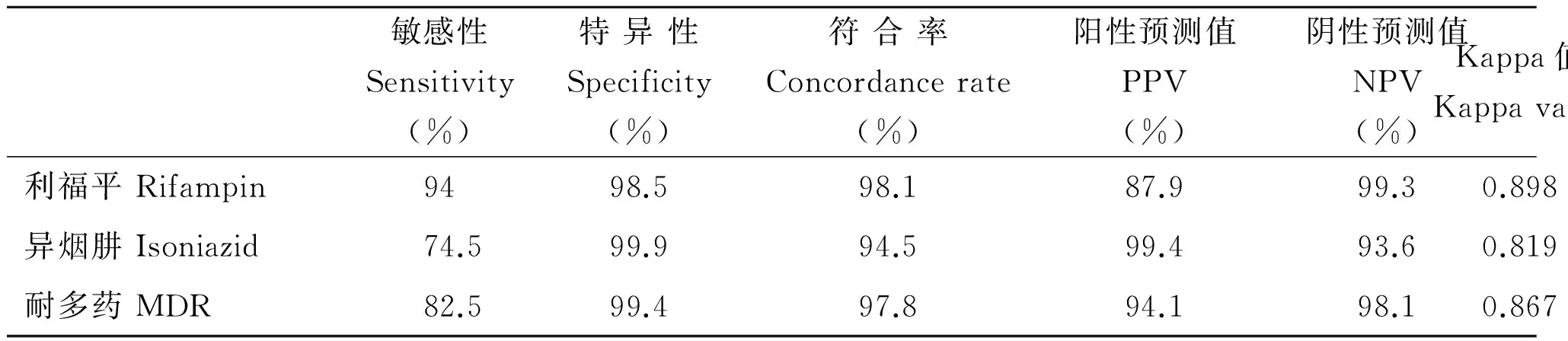
敏感性Sensitivity(%)特异性Specificity(%)符合率Concordancerate(%)阳性预测值PPV(%)阴性预测值NPV(%)Kappa值Kappavalue利福平Rifampin9498.598.187.999.30.898异烟肼Isoniazid74.599.994.599.493.60.819耐多药MDR82.599.497.894.198.10.867
Note:MDR=Multidrug resistant; PPV=Positive predictive value; NPV=Negative predictive value
2.4 基因芯片技术与BACTEC MGIT 960 培养法检测菌株耐多药结果比较分析 基因芯片技术检测耐多药(MDR)结核结果与BACTEC MGIT 960分枝杆菌药敏试验结果符合率为97.8%,敏感度和特异度分别是82.5%和99.4%,Kappa值为0.867,两种方法一致性较好,见表3、表4。
表4 基因芯片技术与BACTEC MGIT 960法检测菌株耐多药结果比较分析
Tab. 4 Comparison of the results of gene chip and BACTEC MGIT 960 culture method for detection of MDR

基因芯片法GenechipmethodBACTECMGIT960培养法BACTECMGIT960culturemethod耐多药MDR非耐多药Non-MDR符合率Concordancerate(%)耐多药MDR80597.8非耐多药Non-MDR17897
Note: MDR=Multidrug resistant
2.5 基因芯片技术与基因测序结果比较分析 对999株经BACTEC MGIT 960分枝杆菌培养阳性的菌株rpoB、katG和inhA基因耐药相关区域的扩增产物全部进行测序以验证基因芯片的准确性。基因芯片法检测利福平耐药结果有4株与DNA测序结果不同,检测的准确率为99.6%,基因芯片法检测异烟肼耐药结果有2株与DNA测序结果不同,检测的准确率为99.8%,见表5。
表5 基因芯片技术与基因测序结果比较分析
Tab.5 Comparison on the results of gene chip assay and DNA sequencing
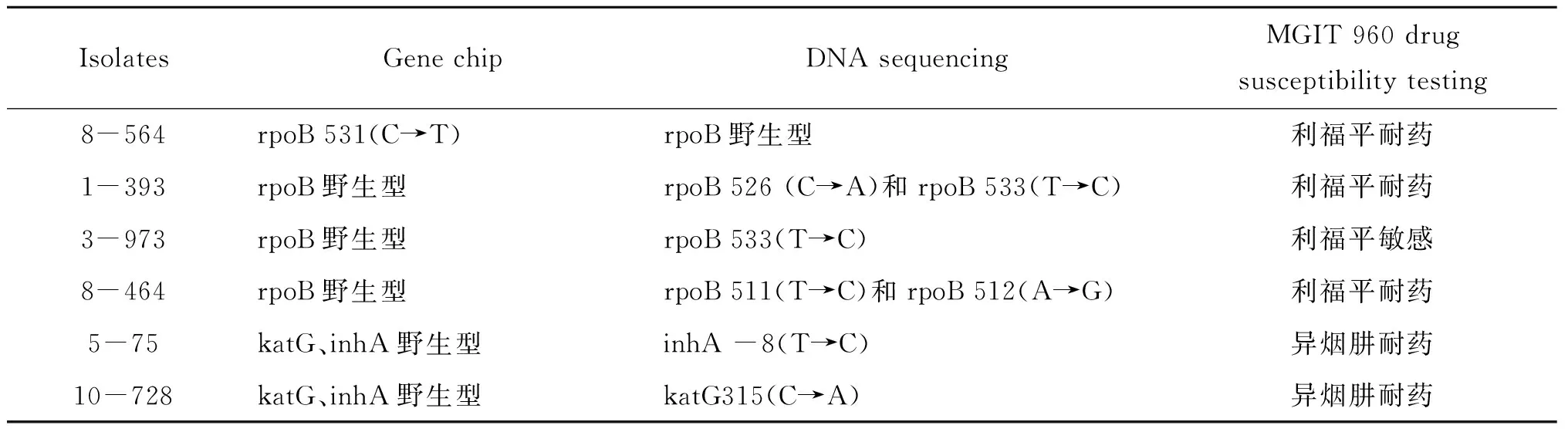
IsolatesGenechipDNAsequencingMGIT960drugsusceptibilitytesting8-564rpoB531(C→T)rpoB野生型利福平耐药1-393rpoB野生型rpoB526(C→A)和rpoB533(T→C)利福平耐药3-973rpoB野生型rpoB533(T→C)利福平敏感8-464rpoB野生型rpoB511(T→C)和rpoB512(A→G)利福平耐药5-75katG、inhA野生型inhA-8(T→C)异烟肼耐药10-728katG、inhA野生型katG315(C→A)异烟肼耐药
注:括号内为检测位点的碱基信息,箭头前为野生型的碱基,箭头后为突变后碱基。
Note: the brackets contain information on bases of detection loci., with wild-type bases before the arrows and mutation-types behind .
3 讨 论
耐多药结核菌(MDR-TB)感染问题已严重威胁社会公共卫生安全。对耐药结核患者早期诊断,及时给予充分有效治疗,对于切断耐药结核传播、减少药物不良反应十分必要。以细菌学为基础的药敏检测方法,无论是传统方法还是液体快速培养法都需要较长时间,无法满足临床快速诊断的需求。
基因芯片技术是20世纪90年代发展起来的分子生物学技术,结合了分子生物学高灵敏度和生物芯片的高通量性,具有快速、灵敏、准确等特点,该技术在在结核菌耐药性检测方面应用越来越广泛。本研究将基因芯片技术应用于痰标本直接检测,在1 008例痰标本基因芯片检测结果中仅有9例与BACTEC MGIT 960阳性菌株的基因芯片结果不一致,符合率高达99.1%。研究结果表明,从病人痰标本开始直接进行基因芯片检测,结果与菌株的结果符合率高,节省了等待培养阳性菌株时间,大大缩短了检测时间,仅需6 h就能得到药敏结果,满足了临床快速检测需求。
本研究中对于异烟肼耐药结果基因芯片法与BACTEC MGIT 960药敏试验结果符合率为94.5%,敏感度和特异度分别为74.5%和99.9%,与赵雁林报道的77.4%的敏感度及96.9%的特异度相近[6]。结核分枝杆菌异烟肼耐药株主要有katG基因和inhA基因碱基突变[7-8], 有文献报道两者总突变率达90.7%[9]。基因芯片法对于异烟肼耐药性检测敏感度较低,推测可能与这些菌株突变位置发生在基因芯片检测的位点范围之外;另外可能与INH其他耐药机制有关,诸如ahpC、oxyR、kasA、药物外排泵基因突变等。其中,inhA是活化的异烟肼作用靶位,inhA与KasA参与分枝菌酸的生物合成,ahpC参与氧化一应激应答oxyR是调节分枝杆菌氧化一应激的基因,参与调节katG和ahpC基因的表达[10-13]。有研究表明10%~15%的异烟肼耐药菌株中可见ahpC-oxyR区突变[14]。该区可作为INH耐药筛查的重要指标[15]。我们认为扩大基因芯片检测的位点范围,可能有助于提高方法的敏感度。
本研究利福平耐药结果基因芯片法与BACTEC MGIT 960分枝杆菌药敏试验结果符合率为98.1%,敏感度和特异度分别是94%和98.5%,Kappa值为0.898,两种方法一致性较好。文献报道利福平耐药的病例中有95%~98%的利福平耐药临床分离株在结核分枝杆菌的80 bp的rpoB基因的利福平耐药决定区(RRDR)有突变[16-17]。
基因芯片对耐多药结核病的诊断符合率为97.8%,其灵敏度和特异度分别为82.5%和99.4%,提示基因芯片技术对耐多药结核病有一定的筛查与较高的排除诊断价值。
基因芯片法检测利福平耐药结果有4株与DNA测序结果不同,检测的准确率为99.6%。本研究在利福平耐药菌株中,通过测序发现了基因芯片法未包含的突变类型rpoB526(C→A)及新的突变位点rpoB512(A→G),而其与利福平耐药的相关性仍有待进一步的研究证明。基因芯片法检测异烟肼耐药结果有2株与DNA测序结果不同,检测的准确率为99.8%。本研究中异烟肼的基因芯片法结果与测序法结果不符的菌株通过测序发现了基因芯片未包含的突变类型katG315(C→A)及突变位点inhA-8 (T→C)。其中有文献报道inhA的启动子区域inhA-8 (T→C)与异烟肼的耐药相关[18]。
基因芯片检测为rpoB突变型的107株菌株中,106株存在单一位点突变,1株存在双位点突变。突变频率最高的位点是531,突变频率是56%,其次是526位点,突变频率是22.4%。基因芯片检测出159株katG/inhA突变株,111株是katG315位点的单一突变,突变率是70.7%。福州地区结核耐药最主要是rpoB531,526位点突变,katG315位点突变。文献报道,异烟肼耐药主要与katG基因突变密切相关[19]。在异烟肼基因芯片检测为突变的标本中突变频率最高的位点是315,突变频率是70.7%,与浙江地区(69.3%)315突变位点突变率相当[20],但与武汉地区(60.9%)[21]、深圳地区(84.6%)[22]等的报道区别较大。这说明katG315突变存在一定的地理差异。
综上所述,基因芯片技术能够直接利用痰标本进行检测。对于利福平的耐药性检测结果与BACTEC MGIT 960药敏试验结果具有很高的符合率而检测异烟肼的敏感性则有待提高。基因芯片技术是一种快速检测结核分枝杆菌耐药性的分子生物学方法,弥补了以细菌学为基础的药敏方法检测时间长的缺点,为临床提供快速诊断。
[1] Zhu L, Jiang G, Wang S, et al. Biochip system for rapid and accurate identification of mycobacterial species from isolates and sputum[J]. J Clin Microbiol, 2010, 48(10): 3654-3660. DOI:10.1128/JCM.00158-10
[2] Sun ML, Chen X, Hua YC. Application of BACTEC MGIT 960 system in preliminary identification of mycobacterium and antimicrobial susceptibility test[J]. Chin J Nosocomiol, 2011, 21(11): 2372-2374. (in Chinese)
孙美兰,陈晓,华永川. BACTEC MGIT 960 系统在分枝杆菌属菌种初步鉴定及药敏中的应用[J]. 中华医院感染学杂志, 2011, 21(11): 2372-2374.
[3] Guo Y, Zhou Y, Wang C, et al. Rapid, accurate determination of multidrug resistance inM.tuberculosisisolates and sputum using a biochip system[J]. Int J Tuberc Lung Dis, 2009, 13(7): 914-920.
[4] Basic Specialized Committee of Chinese Antituberculosis Association. Test procedure in tuberculosis Diagnosis Laboratory[M]. Beijing: China Education and Culture Press, 2006: 68-72. (in Chinese)
中国防痨协会基础专业委员会. 结核病诊断实验室检验规程[M]. 北京: 中国教育文化出版社, 2006: 68-72
[5] Zhu LX, Zhang ZW, Liang D, et al. Multiplex asymmetric PCR-based oligonucleotide microarray for detection of drug resistance genes containing single mutations inEnterobacteriaceae[J].Antimicrobial Agents Chemother, 2007, 51: 3707-3713. DOI: 10.1128/AAC.01461-06
[6] Zhou Y, Ou XC, Le J, et al. A multicenter evaluation of a biochip system for detection of rifampin and isoniozid resistance in clinic ofMycobacteriumtuberculosis[J]. Chin J Lab Med, 2011, 34(9): 76-82. (in Chinese)
周杨, 欧喜超, 乐军,等. 基因芯片诊断耐多药结核病的临床多中心研究[J]. 中华检验医学杂志, 2011, 34(9): 76-82.
[7] Cade CE, Dlouhy AC, Medzihradszky KF, et al. Isoniazid-resistance conferring mutations inMycobacteriumtuberculosisKatG: catalase, peroxidase, and INH-NADH adduct formation activities[J]. Protein Sci, 2010, 19(3): 458-474. DOI: 10.1002/pro.324
[8] Lucknert SR, Liu N, am Ende CW, et al. A slow, tight binding inhibitor of InhA, the enoyl-acyl carrier protein reductase fromMycobacteriumtuberculosis[J]. J Biol Chem, 2010, 285(19): 14330-14337. DOI: 10.1074/jbc.M109.090373
[9] Lin SF, Liang QF, Chen QY, et al. Mutations of drug-resistance genes related to INH inMycobacteriumtuberculosisisolates in Fujian province, China[J]. Chin J Zoonoses, 2014, 30(6): 588-593. (in Chinese)
林淑芳,梁庆福, 陈求扬,等. 福建省结核分枝杆菌异烟肼耐药相关基因突变特征初步分析[J]. 中国人兽共患病学报, 2014, 30(6):588-593.
[10] Silva MS, Senna SG, Ribeiro MO, et al. Mutations in katG, inhA, and ahpC genes of Brazilian isoniazid-resistant isolates of Mycobacterium tuberculosis[J]. J Clin Microbiol, 2003, 41(9): 4471-4474. DOI: 10.1128/JCM.41.9.4471-4474.2003
[11] Sajduda A, Brzostek A, Poplawska M, et al. Molecular characterization of rifampin- and isoniazid-resistantMycobacteriumtuberculosisstrains isolated in Poland[J]. J Clin Microbiol, 2004, 42(6): 2425-2431. DOI: 10.1128/JCM.42.6.2425-2431.2004
[12] Jiao WW, Mokrousov I, Sun GZ, et al. Molecular characteristics of rifampin and isoniazid resistantMycobacteriumtuberculosisstrains from Beijing, China[J].Chin Med J (Engl), 2007, 120(9): 814-819.
[13] Almeida Da Silva PE, Palomino JC. Molecular basis and mechanisms of drug resistance inMycobacteriumtuberculosis: classical and new drugs[J]. J Antimicrob Chemother, 2011, 66(7): 1417-1430. DOI: 10.1093/jac/dkr173
[14] Vijdea R, Stegger M, Sosnovskaja A, et al. Multidrug-resistant tuberculosis: rapid detection of resistance to rifampin and high or low levels of isoniazid in clinical specimens and isolates[J]. Eur J Clin Microbiol Infect Dis, 2008, 27(11): 1079-1086. DOI: 10.1007/s10096-008-0548-9
[15] Laurenzo D, Mousa SA. Mechanisms of drug resistance inMycobacteriumtuberculosisand current status of rapid molecular diagnostic testing[J]. Acta Trop, 2011, 119(1): 5-10. DOI: 10.1016/j.actatropica.2011.04.008
[16] Chakravorty S, Kothari H, Aladegbami B, et al. Rapid, high-throughput detection of rifampin resistance and heteroresistance inMycobacteriumtuberculosisby use of sloppy molecular beacon melting temperature coding[J]. J Clin Microbiol, 2012, 50(7): 2194-2202. DOI: 10.1128/JCM.00143-12
[17] Garcia-Sierra N, Lacoma A, Prat C, et al. Pyrosequencing for rapid molecular detection of rifampin and isoniazid resistance inMycobacteriumtuberculosisstrains and clinical specimens[J]. J Clin Microbiol, 2011, 49(10): 3683-3686. DOI: 10.1128/JCM.01239-11
[18] Hillemann D, Rush-Gerdes S, Richter E. Evaluation of the genotype MTBDRplus assay for rifamin and isoniazid susceptibility testing ofMycobacteriumtuberculosisstrains and clinical specimens[J]. J Clin Microbiol, 2007,45: 2635-2640. DOI: 10.1128/JCM.00521-07
[19] Hoshide M, Qian L, Rodrigues C, et al. Geographical differences associated with single-nucleotide polymorphisms (SNPs) in nine gene targets among resistant clinical isolates ofMycobacteriumtuberculosis[J]. J Clin Microbiol, 2014, 52(5): 1322-1329. DOI: 10.1128/JCM.00857-13
[20] Rong QJ, Lu HX, Sun AH. Rapid detection on katG gene mutation ofMycobacteriumtuberculosisusing genetic microarray and the correlation between mutation and isoniazid resistance[J]. Chin J Zoonoses, 2011, 27(3): 233-237. (in Chinese)
戎奇吉, 吕火祥, 孙爱华.基因芯片快速检测结核分枝杆菌katG基因突变及其与异烟肼耐药相关性[J].中国人兽共患病学报, 2011, 27(3): 233-237. DOI:10.3969/j.issn.1002-2694.2011.03.014
[21] Ma J, Chen GZ, Dong JL, et al. Molecular characterization of katG gene mutations in isoniazid-resistantMycobacteriumtuberculosisstrains in wu han of China[J]. Chin J Antituberc, 2014, 37(1): 95-97. (in Chinese)
马峻, 陈高瞻, 董洁莉,等. 武汉地区耐异烟肼结核分枝杆菌临床分离株katG基因突变的分子特征分析[J]. 中国防痨杂志, 2014,37(1): 95-97. DOI:10.3969/j.issn.1000-6621.2015.01.018.
[22] Gui J, Wang F. Genetic and phenotypic characterization of drug-resistantMycobacteriumtuberculosisisolates in Shenzhen of China[J]. Chin J Microbiol Immunol, 2010, 30(5): 466-471. (in Chinese)
桂静, 王峰. 深圳地区结核分枝杆菌耐药分离株分子特征与表型特征相关性研究[J].中华微生物学和免疫学杂志, 2010, 30(5): 466-471
Evaluation for clinical application effect of gene chip for detection of rifampin and isoniazid resistance inMycobacteriumtuberculosis
XU Rong-qing, LI Dan, LIN Yin-xia, HUANG Ming-xiang, CHEN Xin-chao
(FuzhouPulmonaryHospital/ClinicalTeachingHospitalofFujianMedicalUniversity,Fuzhou350008,China)
We evaluated clinical application effect of gene chip for detection of rifampin (RFP) and isoniazid (INH) resistance inMycobacteriumtuberculosis(MTB). Rifampin and isoniazid drug-resistance gene loci were detected by gene chip with sputum specimens from smear-positive tuberculosis patients and clinical strains, comparing the results of detection. BACTEC MGIT 960 drug susceptibility test results were used as control to evaluate the detection performance of gene chip. The sequences of the polymerase chain reaction products of therpoB,katGandinhAgenes from 999 strains identified asMycobacteriumtuberculosiswere determined to confirm the mutations by DNA sequencing. Results showed that 100 cases were identified as nontuberculous mycobacteria by gene chip in the 1 108 cases of smear-positive samples. Among the rest 1 008 samples, there were only 9 cases of microarray results different from BACTEC MGIT960 culture-positive strains, achieving the coincidences of 99.1%. Compared with BACTEC MGIT 960 drug susceptibility test results, the gene chip method displayed a concordance of 98.1% and 94.5% for RFP and INH respectively in the 999 strains. Compared with the DNA sequencing method, the accuracy of gene chip method was 99.6% for rifampin resistance and 99.8% for isoniazid resistance. It's concluded that the gene chip technology can quickly and accurately detect rifampin and isoniazid resistance in MTB and can be used directly for the detection of sputum samples.
Mycobacteriumtuberculosis; rifampin; isoniazid; drug resistance; gene chip
10.3969/j.issn.1002-2694.2017.01.008
福建省卫生厅青年科研课题项目(No. 2013-2-77)
黄明翔,Email: hmg119@163.com
福建省福州肺科医院检验科,福建医科大学临床教学医院,福州 350008
R378.91
A
1002-2694(2017)01-0043-06
2015-12-26 编辑:梁小洁
