中国地方黄牛PPARα exon7基因多态性与生长性状的关联研究
2016-12-27李荣荣张琼琼李暑燕卢建雄
李 芬,李荣荣,张琼琼,李暑燕,马 云,卢建雄
(1.西北民族大学生命科学与工程学院,甘肃 兰州 730030;2.山东省滕州市至善中学,山东 滕州 277599;3.信阳师范学院生命科学学院,河南 信阳 464000)
科学试验
中国地方黄牛PPARα exon7基因多态性与生长性状的关联研究
李 芬1,3,李荣荣2,张琼琼3,李暑燕3,马 云3,卢建雄1*
(1.西北民族大学生命科学与工程学院,甘肃 兰州 730030;2.山东省滕州市至善中学,山东 滕州 277599;3.信阳师范学院生命科学学院,河南 信阳 464000)
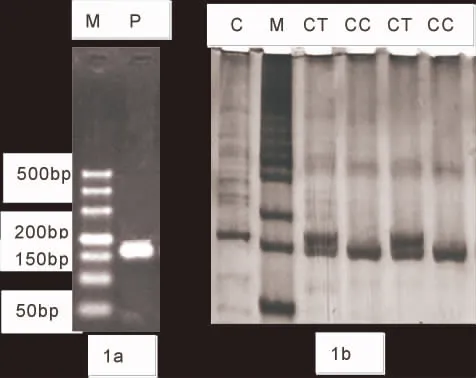
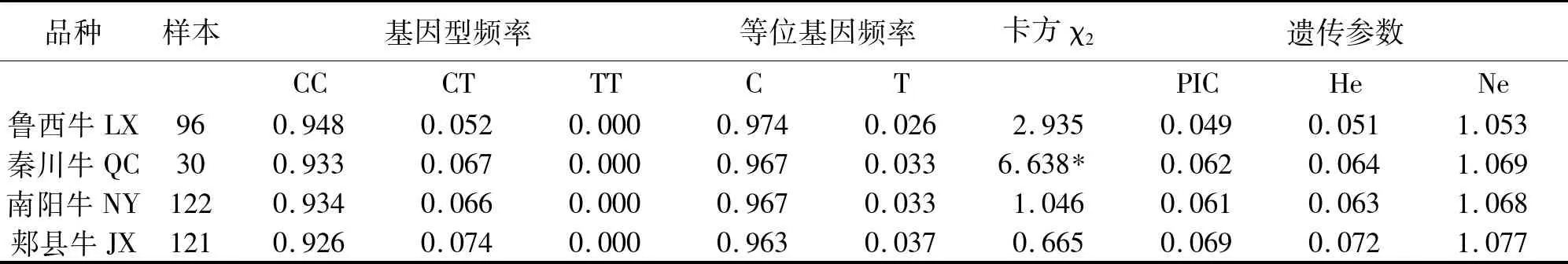
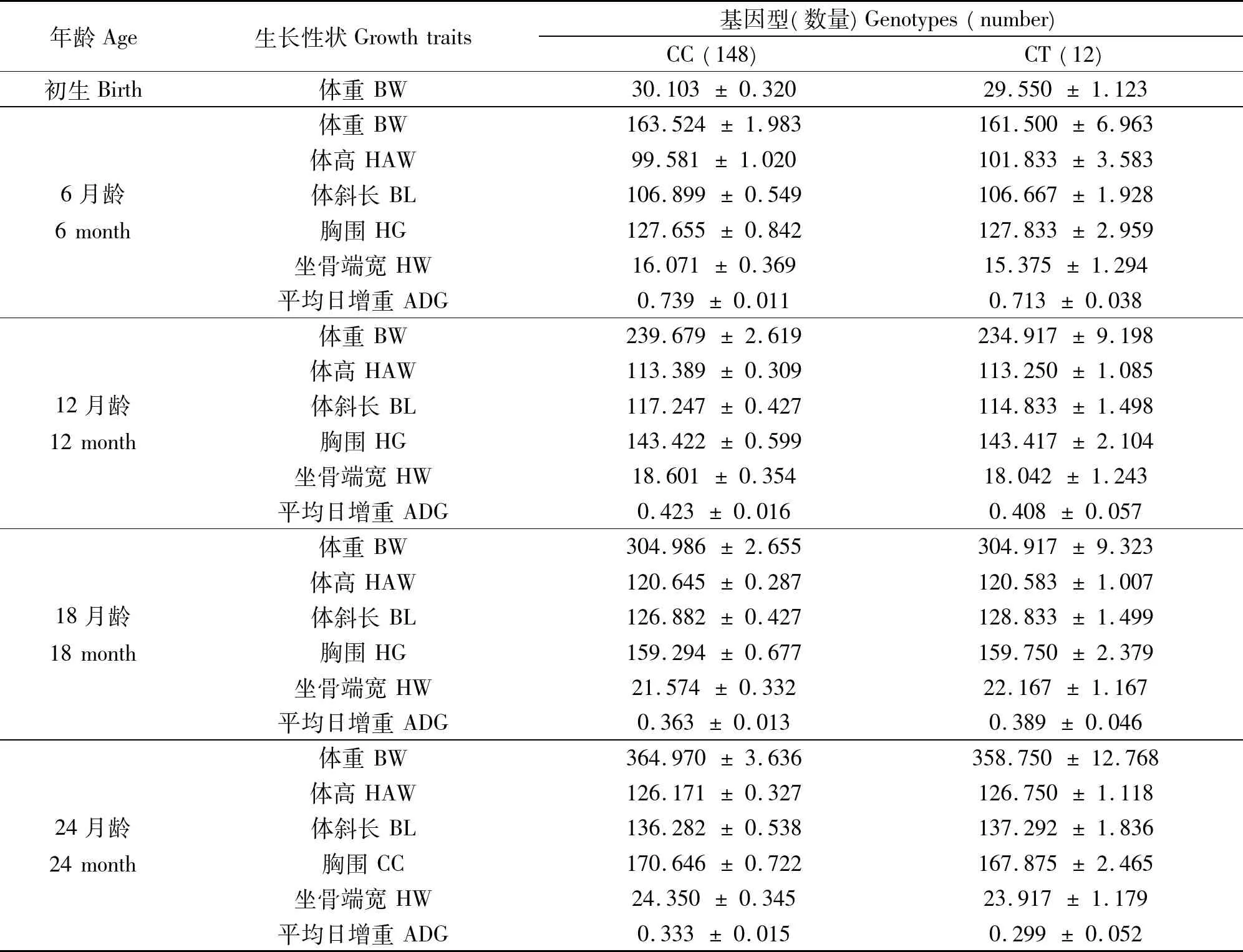
[目的]以期为我国地方黄牛生长性状分子育种提供理论依据。[方法]本研究以南阳牛、郏县红牛、鲁西牛和秦川牛4个品种共计424头中国地方黄牛为材料,以与牛脂肪代谢相关的基因PPARα为目标基因,利用生物信息学、PCR-RFLP结合DNA池测序等技术,对其第7外显子(PPARα_exon7)进行遗传特征分析,检测SNPs,并分析该序列的多态性与试验黄牛生长性状指标的相关性。[结果]发现在PPARα基因(AC_000162.1)exon7的164bp处发现了一处SNP(C/T),检测出CC和CT两种基因型。群体遗传分析显示,该位点在试验牛群体中均属于中度多态(0.25 中国地方黄牛;PPARα基因;SNPs;关联分析 过氧化物酶体增殖物激活型受体(peroxisome proliferator-activated receptors, PPARs )是一组调节脂肪酸代谢的配体激活核受体转录因子,作为激素激活核受体转录因子,PPARs在调节脂肪代谢、胰岛素敏感、炎症反应、脂肪细胞生长和分化等方面发挥着重要作用[1-5]。 PPARs家族已发现含有α、β和γ三个成员,其中PPARα基因主要参与脂肪酸氧化代谢,通过调节控制肝脏内脂肪酸氧化相关基因的表达,从而对肝脏中脂肪细胞的分化、脂质转运及贮存、脂肪酸氧化等脂肪和能量代谢过程产生影响[6]。因此,近年来备受关注。在家养动物PPARα基因的研究方面,Shimada[7]、Sebastian[8]、樊月园[9]、晋大鹏[10]、邵根宝[11]和孟和[12]等分别在牛、猪和鸡上开展了一些研究,但总体上关于PPARα基因与牛肉用性状相关研究的报道较少。 我国地方黄牛品种遗传资源丰富,而且种间差异较大,适应性和繁殖能力强、耐粗饲、有害性状的遗传频率低、肉脂品质好等优势特征明显,因而成为我国乃至全世界牛繁殖育种的重要遗传资源[13]。但是,地方黄牛在牛肉产量和肉品质提高方面还存在许多问题,与国外专门化肉牛品种还存在不小的差距。本研究选择4个地方黄牛品种南阳牛、郏县红牛、秦川牛和鲁西牛,以其作为试验材料,以PPARα基因作为目标基因进行遗传特征分析,检测其SNPs,并分析该基因的多态性与我国地方代表性黄牛生长性状指标的相关性,以期进一步阐明和确证PPARα基因多态与中国地方黄牛肉用性状之间是否存在相关性,研究结果可为我国地方黄牛肉用性状遗传改良提供理论依据。 1.1 材料 本实验所使用的血液样品分别来源于4个中国地方黄牛品种(包括南阳牛、郏县红牛、鲁西牛和秦川牛),试验动物总计424头,全部为母牛,其中139份南阳牛血样来自南阳市黄牛良种繁育场,141份郏县红牛血样采自河南省平顶山市郏县红牛良种繁育场,114份鲁西黄牛血样采自山东鄄城鲁西黄牛原种场,30份秦川牛样品由陕西省秦川牛良种繁育中心。成年牛生产性能数据主要由本课题组相关人员现场测定,测定指标包括体高(BH)、体斜长(BL)、胸围(CC)、腹围(AC)、腰角宽(HBW)、坐骨端宽(HW)、十字部高(HHC)和体重(BW)等。其中73头郏县红牛和100头南阳牛不同生长阶段体尺数据分别由郏县红牛国家级保种场和南阳牛良种繁育中心提供,具体测定指标包括初生重以及6、12、18和24月龄的体高、体斜长、胸围、腰角宽、体重和日增重。 1.2 实验方法 1.2.1 基因组DNA提取 本实验血液基因组DNA提取均采用购自天根生化科技(北京)有限公司的TIANamp Blood全血基因组DNA提取试剂盒(离心柱型,产品编号:DP318-02)提取。 1.2.2 PCR引物设计 根据GenBank中公布的牛PPARα基因DNA序列(AC_000162.1),mRNA 序列(NM_001034036.1)以及dbSNP数据库(http://www.ncbi.nlm.nih.gov/SNP/)提供的SNP参考信息,运用引入错配碱基创造限制性核酸内切酶酶切位点法设计1对引物,用Forced-PCR-RFLP 技术对牛PPARα基因第7外显子区域突变rs110745628(C/T)进行基因型分型,然后再对不同基因型进行DNA 测序验证。上游引物引物序列为:TCCGTGGAGACCGGCAC;下游引物序列为:TAGGCTACCAACATCCCATCTTTAT;目的片段164bp,设计好的引物序列交由南京金斯瑞生物科技有限公司合成。 1.2.3 PCR扩增 本实验中PCR反应体系分别是dNTP mix 7.5μL,上下游引物(10 μM)各0.1μL,Taq DNA聚合酶0.1μL(250U),DNA 模板0.5μL,ddH2O 6.7μL,总体积15.0μL。 PCR扩增程序:95 ℃预变性5 min;94 ℃变性30 s;57.7 ℃退火30 s;72 ℃延伸30~50 s;72 ℃终延伸10 min;4 ℃保存。其中变性、退火、延伸为30~40个循环。72 ℃延伸时间根据扩增片段大小确定。 1.2.4 PCR-RFLP分析及测序验证 根据碱基突变位点选择合适的限制性核酸内切酶,根据优化的PCR反应条件进行扩增,并对产物进行检测,目的条带清晰且无非特异性扩增时即可进行酶切。酶切反应结束后将酶切产物用浓度为10%~14%的聚丙烯酰胺凝胶进行电泳。 根据聚丙烯酰胺凝胶电泳结果选择不同基因型个体分别进行PCR扩增,然后将PCR产物寄送至南京金斯瑞生物科技有限公司测序验证。 1.2.5 数据统计 对不同的牛群体的基因型,计算基因频率和基因型频率,并进行 2检验。用Popgene计算各牛品种在该位点的多态信息含量(PIC)、有效等位基因数(Ne)和基因杂合度(He)。 运用SPSS17.0软件的一般线性模型(GLM)分析个体基因型对牛生长性状的影响。相关分析模型如下:Yijk= u + Bi+ Gj+ eijk。基中:Yijk为个体表型记录;u为群体平均值;Bi为品种效应;Gj为标记基因型效应;eijk为随机误差。4个黄牛群体作为一个整体,由于牛群为成年母牛,故没有考虑性别效应和年龄效应。 2.1 牛PPARα exon7的PCR扩增及SNP分析 以试验牛DNA样品为模板,对牛PPARα基因的第7外显子利用酶切引物进行扩增, 产物经2.5%的琼脂糖凝胶检测,获得与目的片段大小一致的产物164 bp,产物带型清晰且无杂带, 稳定性和特异性好(图1-1a)。根据PCR扩增产物混合测序结果,利用分析软件将其与候选基因参照序列进行对比,筛查得到第7外显子164 bp处C/T突变位点。根据碱基突变位点选择限制性核酸内切酶HgiCI进行酶切分型,酶切反应结束后将酶切产物用浓度为10%~14%的聚丙烯酰胺凝胶进行电泳,结果呈现出CC和CT两种基因型(图1-1b)。 图1 牛PPARα exon7 多态位点的电泳图谱 2.3 牛PPARα exon7遗传平衡检测和多态性指标分析 本研究对4个品种牛个体PPARα exon7基因(C/T)位点多态信息含量(PIC)、杂合度(He)、有效等位基因数(Ne)进行了统计分析,结果见表1。 表1 牛 PPARα exon7基因(C/T)位点遗传参数分析 由表1可知,牛PPARα exon7基因(C/T)位点,四个地方黄牛群体均属于低度多态(0 2.4 牛PPARα exon7多态性与生长发育性状的关联分析 应用所列出的一般线性模型对4个黄牛群体PPARα exon7的多态性与所对应的体高、体重等指标进行关联分析,结果见表2。 表2 牛 PPARα exon7基因(C/T)位点不同基因型对体尺性状的影响 注: A,B代表数值的显著水平为P < 0.01; a,b代表数值的显著水平为P < 0.05。 表3 PPARα exon7基因(C/T)位点多态性与黄牛不同月龄生长性状的关联分析 由表2可知,(C/T)位点仅有两种不同基因型(CC和CT),但在体斜长指标上,这两种不同基因型的个体之间有显著的差异(\%P\%<0.05)。 2.5 牛PPARα exon7多态性与黄牛不同月龄生长性状的关联分析 应用所列出的一般线性模型对4个黄牛群体PPARα exon7的多态性与黄牛不同月龄生长性状的关联分析结果见表3。 由表3可知,(C/T)位点两种不同基因型(CC和CT)的个体之间没有显著的差异(\%P\%>0.05)。 3.1 关于牛肉用性能改良与生长发育性状指标的选择 我国是世界第三大牛肉生产大国,但是我国用以生产牛肉的主体是过去曾经长期役用的地方黄牛,虽然建国后尤其是近年来先后选育出了5个肉牛品种(夏南牛、延黄牛、辽育白牛、蜀宣花牛、云岭牛)及4个兼用品种(新疆褐牛、三河牛、草原红牛、中国西门塔尔牛),但总体上这些培育的肉牛(兼用)品种和国外专门化肉牛品种相比较,肉用性能仍然不尽如人意,在生长速度、优质肉比率、高档肉产率及肉品质等指标方面都亟待进一步选育提高。当前,随着常规育种技术选择增益的日渐减缓,分子标记辅助选择技术在牛的育种方面发挥越来越重要的作用。利用分子标记辅助选择技术结合常规育种技术制订综合选择方案,对牛的经济性状进行改良已成为牛育种的主流趋势,而生长发育指标是衡量肉用动物的产肉性能的非常重要的评价指标,因此制订包含有影响生长发育性状指标分子标记的选择方案,对于改良我国黄牛肉用性能具有重要理论和现实意义。 3.2 牛PPARα exon7基因的遗传变异分析 牛肉品质与肉中脂肪沉积密切相关。而脂肪沉积来源于脂肪细胞的分化与增殖,过氧化物酶体增殖物激活型受体(peroxisome proliferator-activated receptors,PPARs )是一组调节脂肪酸代谢的配体激活核受体转录因子。研究显示,PPARs家族在脂质代谢和能量代谢,脂肪细胞分化方面发挥重要作用。 本研究以PPARs家族成员PPARα基因为目标基因,利用生物信息学技术、质谱SNP分型技术、PCR-RFLP技术结合DNA池测序对牛PPARα exon7基因进行筛查。四个地方黄牛群体均属于低度多态(0 < PIC < 0.25),鲁西牛、南阳牛和郏县红牛在该位点处于Hardy-Weinberg平衡状态,说明采集的样本来自于一个大的孟德尔群体中;而秦川牛处于不平衡状态,此结果可能与秦川牛样本群数量少有关。 3.3 牛PPARα exon7基因多态性与地方黄牛生长性状相关性分析 本文对牛PPARα exon7的多态性与4个黄牛品种的生长性状进行了相关性分析, 发现除体斜长指标上CC和CT两种基因型存在显著差异(\%P\%<0. 05)外,其他指标无差异或差异较小,且CC和CT基因型在黄牛不同月龄生长指标上不存在显著差异。 PPARα基因定位于牛5号染色体(BTA5),大约位于117.15到117.23 Mb处。笔者利用辐射杂种细胞系定位图和QTL数据库(http://www.genome.iastate.edu/cgi-bin/QTLdb/BT/index),寻找与牛PPARα基因相关的数量性状位点,通过对比发现与牛平均日增重相关的QTLs (124 cM)[8]。PPARα的功能改变与肥胖和胰岛素抵抗的发病机制有密切关系,大量研究结果表明人的PPARα基因第5 外显子 L162V 多态性与低体重糖尿病或糖尿病脂质代谢异常有关[10]。樊月圆等在牛 PPARα基因第 5 外显子的 138 bp 处发生 T→C 突变,发现该突变对于秦川牛肉质有显著影响,这些结果提示该位点突变可能是影响牛肉背膘厚、眼肌面积和系水力的主效 QTN 或与之紧密连锁,可作为肉牛产肉性状的辅助选择标[9]。我们的研究结果表明PPARα基因的杂合基因型CT与黄牛体斜长显著相关。牛体重与体斜长相关,这说明牛PPARα exon7遗传多态性与体重指标直接关联,与之前所报道的牛PPARα基因影响脂质代谢和能量代谢的报道一致。然而本研究未涉及肉品质指标, 因此牛PPARα基因是否会影响肉牛肉品质有待进一步研究。所以,PPARα基因可能在家畜生长发育过程中发挥一定作用。 [1] Yuan G, Chen X, Li D. Modulation of peroxisome proliferator-activated receptor gamma (PPARγ) by conjugated fatty acid in obesity and inflammatory bowel disease[J]. Journal of agricultural and food chemistry, 2015, 63(7): 1883-1895. [2] Penas F, Mirkin G A, Vera M, et al. Treatment in vitro with PPARα and PPARγ ligands drives M1-to-M2 polarization of macrophages from T. cruzi-infected mice[J]. Biochimica et biophysica acta-molecular basis of disease, 2015, 1852(5) :893-904. [3] Falcone R, Florio T M, Di G E, et al. PPARβ/δ and γ in a rat model of parkinson's disease: possible involvement in PD symptoms[J]. Journal of cellular biochemistry, 2015, 116(5): 844-855. [4] Peng Z, Ban K, Wawrose R A, et al. Protection by enteral glutamine is mediated by intestinal epithelial cell peroxisome proliferator activated receptor gamma during intestinal ischemia/reperfusion[J]. Shock, 2015, 43(4): 327-333. [5] Gu S J, Chen D H, Guo Z R, et al. Effect of obesity on the association between common variations in the PPAR gene and C-reactive protein level in Chinese Han population[J]. Endocrine, 2015, 48(1): 195-202. [6] Van Diepen J A, Jansen P A, Ballak D B, et al. PPAR-alpha dependent regulation of vanin-1 mediates hepatic lipid metabolism[J]. Journal of Hepatology, 2014, 61(2): 366-372. [7] Montiel P G, Antaramian A, Miyasaka A S, et al. Role of coactivator PGC-1α in energy balance: Importance in the production of beef cattle[J]. Archivos Latinoamericanos de Produccion Animal, 2008, 16(2):63-77. [8] Nkrumah J D, Sherman E L, Li C, et al. Primary genome scan to identify putative quantitative trait loci for feedlot growth rate, feed intake, and feed efficiency of beef cattle[J].Journal of Animal Science, 2007, 85 (12): 3170-3181. [9] Sebastian L, Beatrice G, Holger K, et al. Clofibrate causes an upregulation of PPAR-αtarget genes but does not alter expression of SREBP target genes in liver and adipose tissue of pigs[J]. American Journal of Physiology, 2007 , 293(1): 70-77. [10] 樊月圆,林森,王洪宝,等. 牛过氧化物酶体增殖激活受体 (PPAR)基因第5外显子多态性与胴体,肉质性状的相关性[J].农业生物技术学报,2010,18(40):713-714. [11] 晋大鹏. 猪PPARGC1A和CAPNS1基因多态性及其与肉质性状的关联性分析[J].中国实验动物学杂志,2002,12(4):45-48. [12] 邵根宝,贾超,赵如茜,等. 猪13号染色体部分微卫星标记与肉质性状关系的研究[J].遗传学报,2005,32(5):466-467. [13] 孟和,王桂华,王启贵,等. 鸡PPAR基因单核苷酸多态与脂肪性状相关的研究[J].遗传学报, 2002,29(2):119-123. [14] 张英汉. 对中国黄牛资源认知及持续利用问题的冷思考[J]. 中国牛业科学, 2013, 39(6):1-6. Polymorphism of the Exon 7 of PPARα Gene and Correlation with Growth Traits in Chinese Indigenous Cattle LI Fen1,3, LI Rong-rong2, ZHANG Qiong-qiong3, MA Yun3, LU Jian-xiong1 (1.Collegeoflifescienceandengineering,NorthwestUniversityforNationalities,Lanzhou,Gansu730030;2.ZhishanSchoolofTengzhou,Zaozhuang,Shandong277599;3.CollegeofLifeSciences,XinYangNormalUniversity,Xinyang,Henan464000) 【Objective】 The aim of this study was to provide theoretical basis for the molecular breeding of local cattle growth traits in China. 【Method】In order to better understand the effect of PPARα gene which related with bovine fat metabolism in 424 Chinese indigenous cattle samples, including Nanyang cattle (NY), Jiaxian red cattle (JX), Luxi cattle (LX) and Qinchuan cattle (QC). A novel SNP of exon 7 locus of the bovine PPARα gene was detected with bioinformatics, PCR-RFLP, and DNA sequencing to investigate the genetic variations of PPARα. In addition, an association study was carried out to determine the effect of the SNPs of PPARα gene in exon 7 on the growth and meat quality traits. 【Result】 As a result, the SNP (C/T) was identified at 164 bp of PPARα gene in exon 7 (AC_000162.1), which contained two genotypes (CC and CT). The genetic analysis showed that SNP (C/T) was midrange polymorphism (0.25 Chinese indigenous cattle; PPARα gene; SNPs; association analysis 2015-07-20修改日期:2015-07-25 国家自然科学基金(No. 31172193), 河南省科技创新人才项目 (No. 134100510012), 河南省高校科技创新团队项目 (No. 14IRTSTHN012) 及信阳师范学院青年科学基金(2015037)资助。 李芬(1976-),女,陕西安康人,实验师,在读硕士,研究方向:动物遗传育种与繁殖。E-mail:lifen2843@126.com 卢建雄,男,博士,教授,研究方向:动物遗传育种与繁殖。 E-mail:lu2003jx@163.com S823.2 A 1001-9111(2016)01-0001-051 材料与方法
2 结果与分析

3 讨论
